Los hongos patógenos han desarrollado estrategias que involucran la expresión de genes que les ayudan a permanecer y multiplicarse en el huésped. La ausencia de las moléculas codificadas por estos genes podría interferir en el crecimiento y muerte de estos hongos. En el pasado, se reportó un gen (Hcp100) que codifica para una proteína coactivadora del hongo Histoplasma capsulatum, el cual se sobreexpresa después de 1h de contacto entre las levaduras del hongo y los macrófagos murinos. El producto de este gen, una proteína de 100kDa (Hcp100) de H. capsulatum, es probablemente una proteína regulatoria involucrada en los procesos necesarios de adaptación del hongo para la supervivencia en las condiciones hostiles intracelulares de los macrófagos. Un fragmento de 210pb del marcador Hcp100ha revelado ser una excelente herramienta para la detección molecular de H. capsulatum en muestras clínicas. El potencial uso de este gen como blanco terapéutico en Plasmodium falciparum ha sido explorado a través de la inhibición del gen y de la proteína p100 del parásito, logrando bloquear su crecimiento.
MétodosTomando como base los antecedentes mencionados, la Hcp100 podría tener un papel clave en el desarrollo y mantenimiento de las levaduras de H. capsulatum dentro de los macrófagos.
Resultados y conclusionesEstudiar la probable función de la Hcp100 en la fase de levadura de este patógeno fúngico es relevante para entender su actividad y poder proponerla como un blanco terapéutico para el tratamiento de la histoplasmosis.
Fungal pathogens have developed strategies, involving genes expression that favors their persistence and multiplication in the host. The absence of molecules encoded by these genes could interfere with the growth and death of these fungi. In the past, a coactivator protein coding gene (Hcp100) of the fungus Histoplasma capsulatum was reported, which is overexpressed after 1h of contact between fungal yeast-cells and murine macrophages. The product of this gene, a protein of 100kDa (Hcp100) of H. capsulatum, is probably a regulatory protein involved in the processes required for fungal adaptation and its survival in the intracellular hostile conditions of the macrophages. A 210-bp fragment of the Hcp100 marker has proved to be an excellent tool for H. capsulatum molecular detection in clinical samples. The potential use of this gene as a therapeutic target in Plasmodium falciparum has been explored through the inhibition of both, the gene and the protein p100 of the parasite, by blocking its growth.
MethodsBased on the above mentioned antecedents, we believe that the Hcp100 has an important role in the development and maintenance of the H. capsulatum yeast cells within macrophages.
Results and conclusionsTo study the probable function of Hcp100 in the yeast-phase of this fungal pathogen is relevant to understand its activity and to propose it as a therapeutic target for histoplasmosis treatment.
Histoplasma capsulatum es un hongo dimórfico que tiene una fase micelial saprobia geofílica (forma infectiva) que se asocia con guano de murciélagos y aves, mientras que la fase de levadura (forma virulenta-parasitaria) es frecuentemente encontrada dentro de fagocitos de mamíferos. Este hongo es el agente etiológico de la histoplasmosis, micosis sistémica más común del aparato respiratorio que afecta a humanos y otros mamíferos. La enfermedad es endémica en varias regiones de América y es relevante en áreas geográficas como los valles de Mississippi y Ohio en los Estados Unidos. Por otro lado, la forma epidémica de la histoplasmosis tiene gran impacto en regiones del mundo que exponen poblaciones humanas a alto riesgo de infección, tales como la mayoría de los países latinoamericanos, en particular México debido a que muchos trabajadores rurales pueden desarrollar la forma epidémica por su tipo de actividad ocupacional. En la actualidad, la relevancia de la histoplasmosis se destaca por su asociación con individuos inmunocomprometidos.
Interacciones huésped-patógenoPatógenos fúngicos como H. capsulatum, Paracoccidioides brasiliensis o Blastomyces dermatitidis, que crecen como saprobios en nichos ecológicos definidos, pueden infectar a un huésped susceptible por inhalación de esporas o fragmentos hifales aerosolizados que se convierten en formas parasitarias (fase levaduriforme) preferentemente dentro del ambiente intracelular del huésped. Las levaduras, potencialmente virulentas, pueden ser destruidas por fagocitos; sin embargo, ciertas especies de patógenos fúngicos han desarrollado estrategias para evitar la muerte por los mecanismos de defensa del huésped21.
Las interacciones que se llevan a cabo entre el huésped y el parásito fúngico conducen al disparo de una serie de eventos tanto en uno como en otro. En los primeros, con frecuencia, se expresan moléculas de diferentes tipos, algunas de las cuales participan en la activación de mecanismos para combatir y eliminar al agresor. Por su parte, el parásito también expresa moléculas, algunas de las cuales son morfotipo-específicas, que le ayudarán a evadir los mecanismos de defensa del huésped y a sobrevivir dentro de este. Entre las estrategias que los patógenos fúngicos han desarrollado, destaca la expresión o sobreexpresión de genes cuyos productos actúan en distintos aspectos del binomio huésped-parásito. Entre las moléculas codificadas por estos genes sobresalen los factores de virulencia y moléculas coactivadoras de la transcripción. La ausencia de algunas de estas moléculas puede conllevar la inhibición del crecimiento y la muerte del patógeno.
En H. capsulatum se han descrito algunos factores de virulencia que cumplen con los postulados moleculares de Koch y que son característicos de la fase levaduriforme del hongo, a saber: la proteína Yps3 localizada en la pared celular de la levadura, la proteína de unión a calcio CBP que se expresa fundamentalmente a bajas concentraciones de calcio durante la estancia intracelular y el carbohidrato de pared celular α-(1,3)-glucano18,30. Sin embargo, otras moléculas de H. capsulatum han sido consideradas como factores asociados a la virulencia, como el producto del gen URA5, que participa en la vía biosintética de las pirimidinas siendo importante para la supervivencia del hongo en el huésped19,30, y la proteína de choque térmico Hsp60, que actúa como ligando para el receptor CR3 (CD11b/CD18) de macrófagos, promoviendo el parasitismo intracelular15. Es importante destacar que en ensayos de inmunización con la proteína recombinante Hsp60 de H. capsulatum se generó una respuesta inmune protectora en el modelo murino5, lo que la hace candidata para ser utilizada como vacuna.
Sobreexpresión del gen Hcp100 de Histoplasma capsulatumLa interacción entre los fagocitos del huésped y H. capsulatum es un determinante primario en el curso de la infección y define el destino de H. capsulatum en el huésped. En estas interacciones se encuentran involucrados factores de virulencia fúngicos así como componentes de las inmunidades innata y adaptativa del huésped, particularmente de la inmunidad celular. Los hongos patógenos, una vez fagocitados, exhiben una importante reprogramación transcripcional y traduccional que se refleja en las estrategias de supervivencia y adaptación a las condiciones hostiles del fagocito. Las células fúngicas tienen la capacidad para explorar el ambiente circundante y modificar apropiadamente su perfil transcripcional21.
Colonna-Romano et al.4 realizaron un estudio comparativo, por despliegue diferencial/RT-PCR, del patrón de expresión de genes de H. capsulatum antes y después de la exposición de levaduras del hongo a macrófagos murinos y, asimismo, durante su internalización. Los resultados mostraron varias copias de cADN a partir de los transcritos obtenidos. Seis de los transcritos fueron expresados diferencialmente, y 5 manifestaron sobreexpresión en los momentos iniciales de la interacción macrófago-hongo (15min) y hasta después de 1h de internalización. Uno de estos transcritos presentó los niveles más altos de sobreexpresión después de 1h de infección, cuando el 95% de las levaduras se encontraban intracelularmente. Porta et al.17 caracterizaron la proteína codificada por una de las copias de cADN (pHc12) obtenida por Colonna-Romano et al.4 y, mediante un ensayo de Northern blot, confirmaron que el transcrito se expresaba 5 veces más después de 1h de contacto entre los macrófagos murinos y las levaduras de H. capsulatum, considerando la transcripción basal antes de la infección.
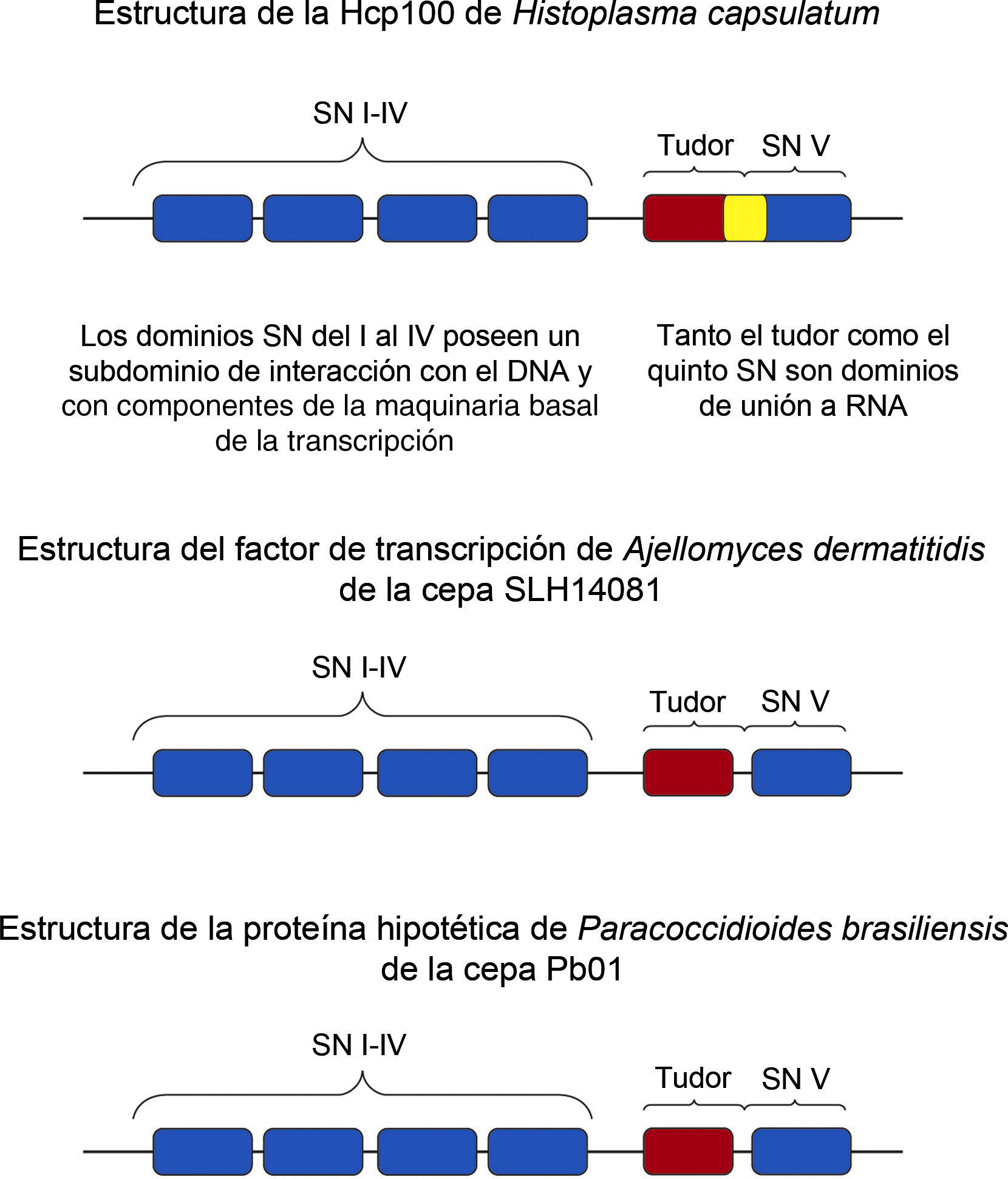
Estructura de la proteína de 100kDa de Histoplasma capsulatum (Hcp100)El análisis de la secuencia del cADN del transcrito de la secuencia pHc12 reveló un marco de lectura de 890 residuos de aminoácidos. El análisis por BLAST de la secuencia de aminoácidos presentó una identidad entre el 30-33% con las secuencias de las proteínas coactivadoras de 100kDa de humano y Caenorhabditis elegans, así como con la proteína de 105kDa de rata. La identidad entre las secuencias de aminoácidos de la Hcp100 de H. capsulatum y las proteínas de la familia p100 fue analizada por hydrophobic cluster analysis y los valores obtenidos reflejaron una relación putativa entre las proteínas analizadas. Con base en estos análisis, se estableció que la proteína obtenida de la secuencia pHc12 de H. capsulatum es un miembro de la familia de las proteínas de 100kDa (p100).
El marco de lectura encontrado por Porta et al.17, en la secuencia de la Hcp100 al igual que en las secuencias de las proteínas de 100kDa de sus homólogos de mamíferos y C. elegans, sugiere una estructura que contiene 4 dominios (i al iv) staphylococcal nuclease (SN) de aproximadamente 150 aminoácidos cada uno, los cuales se disponen en tándem. Los dominios SN fueron en un inicio reportados para eubacterias y arqueobacterias, siendo posteriormente identificados en eucariotas9. En el ser humano, los dominios SN del i al iv de la proteína p100 interactúan con componentes de la maquinaria basal de la transcripción que incluye la ARN helicasa dependiente de ATP (RHA), la subunidad grande de la polimerasa ii (PolR2A) y la proteína CREB (CBP/p300)22. Los dominios SN constan de 2 subdominios A y B. El subdominio A está relacionado con una estructura OB-plegada (oligonucleotide-oligosaccharide binding fold), se presenta en diversas proteínas y tiene la capacidad de unirse a oligonucleótidos y oligosacáridos14. No está claro si la Hcp100 a través de sus dominios SN se une directamente al ADN o si lo hace a través de interacciones proteína-proteína.
El subdominio B consiste en dos α-hélices plegadas independientemente17. La repetición de los dominios SN puede ser esencial para alcanzar una completa actividad biológica de la proteína16. Porta et al.17 reportaron que la Hcp100 contiene un quinto dominio SN modificado, más largo que los otros 4 (200 aminoácidos) y que posee las dos α-hélices del subdominio B y, adicionalmente, un subdominio A que no corresponde al SN OB-plegada.
Estas observaciones permitieron sugerir que la primera parte del quinto dominio de la Hcp100, designado como subdominio A’, es diferente de los subdominios A de los primeros 4 dominios SN. Este subdominio A’ fue asociado al dominio tudor, nombre derivado de la proteína tudor de Drosophyla melanogaster, donde fue primeramente descrito17.
Los dominios tudor se encuentran presentes en proteínas de unión a ARN7,24. Se ha propuesto que los dominios tudor en algunas ocasiones están en contacto directo con los ácidos nucleicos y, en otras ocasiones, este contacto está mediado por interacciones proteína-proteína2,29. Los dominios tudor han sido encontrados en proteínas importantes para el desarrollo de la célula, siendo que muchas de estas proteínas han mostrado estar involucradas en la unión, metabolismo y transporte del ARN3,16. El dominio tudor y el quinto dominio SN interactúan con componentes importantes del ARN splicing entre los cuales se incluyen componentes proteicos de las ribonucleoproteínas nucleares pequeñas (snRNP) y la proteína nuclear SKIP (SKIIP)22.
Los dominios tudor muestran un alto grado de divergencia entre organismos de distintas especies (comparten menos del 20% de identidad) y se caracterizan por tener un segmento más conservado de aproximadamente 80 residuos de aminoácidos y, dentro de este segmento, se encuentran aminoácidos que son invariables. El dominio tudor de la Hcp100 comparte 32,4, 32,4 y 25,1% de identidad con los dominios tudor del ser humano, la rata y C. elegans, respectivamente, manteniendo sus características. En este dominio hay 3 motivos conservados, designados como x, y, z, que incluyen los aminoácidos invariables D722, Y726, R727 (motivo y), D746, G748 (motivo z), así como el altamente conservado Q676 (motivo x) y los aminoácidos aromáticos en la posición 725 (motivo x) y 747 (motivo z)17. Los aminoácidos conservados en esta familia de dominios tudor tienen un importante papel en la función de las proteínas p100 que Porta et al.17 designaron como Hcp100 para H. capsulatum. Porta et al.,17 sugieren en su estudio que la composición multidominio de esta proteína se asocia a funciones esenciales, entre las que se incluyen la unión a ADN y ARN, debido a la presencia de los dominios SN y tudor. Porta et al.17 proponen además que la Hcp100 inducida en etapas primarias de la infección puede actuar como una proteína regulatoria involucrada en los procesos de adaptación del hongo dentro de macrófagos murinos, ayudando al patógeno a manifestar su virulencia.
Utilización del marcador Hcp100 para diagnóstico molecularLa infección por H. capsulatum ha sido exitosamente monitoreada en tejidos del huésped a través de una PCR-anidada para un fragmento de 210pb del gen Hcp100. Este marcador molecular es altamente sensible y considerado único para H. capsulatum. En los últimos años, ha sido utilizado para determinar la presencia del hongo en muestras clínicas de humanos como herramienta diagnóstica1,12,13,26.
La PCR-anidada realizada con este marcador también ha permitido la detección exitosa de la carga fúngica en tejidos de animales silvestres y cautivos que cursaban histoplasmosis diseminada, aportando datos para la epidemiología molecular de la histoplasmosis6,20. La detección del gen Hcp100 de H. capsulatum en muestras de pulmón de murciélagos infectados y su posterior amplificación rindió el fragmento esperado de 210pb de este marcador. El análisis de las secuencias obtenidas se realizó sobre un fragmento de 156 nucleótidos (nt) que se encuentra incluido en el fragmento de 210pb, ya que 54nt fueron eliminados del análisis por presentar inconsistencias en 71 secuencias estudiadas. El alineamiento de este fragmento de 156nt y subsecuente análisis comparativo por BLASTn con las secuencias de Ajellomyces dermatitidis cepa SLH14081 y P. brasiliensis cepa Pb01 depositadas en la base de datos del GenBank, reveló un 83 y 75% de similitud con estas secuencias, respectivamente. Estos porcentajes de similitud pueden ser explicados debido a las cercanas relaciones filogenéticas de H. capsulatum con A. dermatitidis y P. brasiliensis y, asimismo, porque los fragmentos amplificados forman parte de posibles genes ortólogos (datos no publicados).
Además, los 156nt fueron analizados por el algoritmo BLASTx para una búsqueda en el banco de secuencias de proteínas del GenBank, obteniéndose un fragmento de 51 aminoácidos que corresponden a la proteína de 100kDa de H. capsulatum (Hcp100). Cuando este fragmento de 51 aminoácidos fue comparado con secuencias de proteínas de otros hongos, los valores más altos de identidad se obtuvieron con la secuencia de un factor de transcripción de A. dermatitidis (91%) y con una proteína hipotética de P. brasiliensis (87%). Estos valores tan altos de identidad nos indican que probablemente se trate de la misma proteína en organismos distintos. La figura 1 muestra las identidades y diferencias en la estructura de las proteínas de 100kDa de H. capsulatum, A. dermatitidis y P. brasiliensis.
Estructura de las proteínas de la familia p100 de hongos dimórficos. La figura muestra un esquema de las proteínas de 100kDa de Histoplasma capsulatum, Ajellomyces dermatitidis y Paracoccidioides brasiliensis. Se destaca la estructura multidominios de las proteínas y la diferencia del dominio tudor y del quinto dominio SN de la Hcp100, considerando que en H. capsulatum el extremo terminal del dominio tudor se superpone con el inicio del quinto dominio SN (resaltado en color amarillo).
Como ha sido señalado, la familia de proteínas de 100kDa es multifuncional y esto se debe principalmente a su compleja estructura de dominios. En células de humano, la p100 fue caracterizada como un coactivador de la transcripción, interactuando con varios factores de transcripción para aumentar su actividad, incluyendo el EBNA-28,25, STAT5, STAT611,27 y el c-Myb10.
Evidencias recientes sugieren que la p100 de humano actúa como puente para la unión de los factores de transcripción con los componentes de la maquinaria de transcripción; por ejemplo, forma parte de la unión entre c-Myb y Pim-110, así como de la unión entre STAT6 y CBP, STAT6 y ARN-helicasa A27,28. Igualmente, se ha demostrado que la p100 de humano interactúa con snRNP facilitando el ensamblaje del spliceosome31.
Las proteínas de la familia p100 realizan múltiples funciones en la célula de diferentes organismos, lo que sugiere su importancia en procesos vitales de esta y la hacen meritoria de estudios exhaustivos.
Propuesta para considerar el gen Hcp100 y su proteína codificante como posible blanco terapéutico para la histoplasmosisDebido a que las proteínas que se unen a ARN tienen un importante papel en la regulación postranscripcional de la expresión de genes y, por lo tanto, en las funciones de la célula, se estudiaron 2 proteínas de Plasmodium falciparum (agente etiológico de la malaria) que tienen la capacidad de unirse a ARN, para probarlas como posibles blancos terapéuticos9. La búsqueda mediante BLASTn, en la base de datos PlasmoDB, reveló que la proteína p100 de P. falciparum posee un dominio tudor que se une a ARN. Mediante el uso del inhibidor de nucleasas 3′,5′-desoxitimidina bifosfato se reprimió la función de la proteína, lo que ocasionó la inhibición del crecimiento y desarrollo del parásito, lo que sugiere un papel importante de esta proteína en el ciclo de vida del parásito9.
En un estudio muy interesante, realizado por Sunil et al.23, la actividad del gen de la p100 de P. falciparum fue silenciada por ARN de interferencia con el objetivo de detener el crecimiento y desarrollo del parásito in vitro y proponer este gen y su producto como posibles blancos terapéuticos para el agente etiológico de la malaria. Además, utilizando estadios sanguíneos asexuales del parásito, el silenciamiento del gen provocó el cese tanto del crecimiento del parásito como de su desarrollo en el estado de trofozoito. Estos resultados, junto con los obtenidos por Hossain et al.9, demostraron que la p100 es esencial para el ciclo de vida del parásito.
Tomando como base los ensayos de Hossain et al.9 y Sunil et al.23 sobre el gen y la proteína p100 de P. falciparum utilizados como posible blanco terapéutico, en nuestro grupo de investigación se considera la posibilidad de aplicar estas estrategias para H. capsulatum, ya que ha sido demostrado que el gen, y principalmente la proteína codificada, cumplen con importantes funciones en la célula fúngica. Aunque el Hcp100 es un gen que posee ortólogos en diversos organismos, la diversidad tanto del gen como de la proteína los presentan como posibles candidatos para blancos terapéuticos, ya que existen regiones específicas únicas de estos marcadores para H. capsulatum.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Este trabajo se realizó con el apoyo de SEP-CONACYT-ANUIES-ECOS-M05-A03 (México-Francia) y DGAPA-PAPIIT-IN203407-3, UNAM. A. E. González-González agradece al Posgrado en Ciencias Biológicas de la UNAM por el apoyo para realizar sus estudios de doctorado y al Consejo Nacional de Ciencia y Tecnología por la beca otorgada (CONACYT No. 23492) para llevar a cabo dichos estudios.