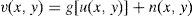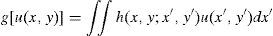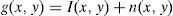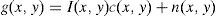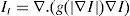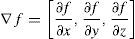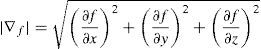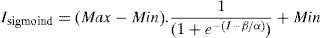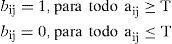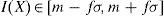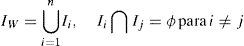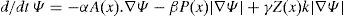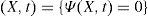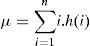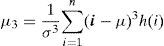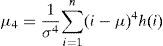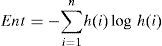Los tejidos humanos son estructuras anatómicas que se caracterizan por tener una morfología compleja y solapada entre sí, razones por las cuales la generación de modelos geométricos precisos no resulta una tarea fácil. En la actualidad, esta tarea se ha beneficiado por el desarrollo de técnicas de diagnóstico por imágenes médicas, las cuales permiten visualizar el cuerpo humano en una forma confiable y no invasiva, generando cortes transversales que representan una sección de los tejidos bajo evaluación. En este trabajo, se propone el uso de un conjunto de técnicas numéricas de procesamiento digital de imágenes implementadas en una herramienta de software para la obtención de modelos geométricos a través de cinco etapas: (1) lectura y reconstrucción tridimensional (3D) inicial de los cortes originales de imágenes de tomografía computacional y resonancia magnética; (2) corrección de la baja calidad de las imágenes utilizando algoritmos de preprocesamiento para suavizar el ruido y realzar los bordes de los tejidos; (3) segmentación híbrida para obtener la geometría 3D de los tejidos de interés; (4) posprocesamiento para corregir errores de segmentación, y (5) exportación de los volúmenes en formatos legibles por otras herramientas de visualización médica y de Diseño Asistido por Computador (CAD) para verificar su utilidad para la generación de modelos discretos a través del análisis con los métodos numéricos. Las técnicas utilizadas fueron validadas calculando descriptores estadísticos en los modelos generados y los modelos proporcionados por otros medios como base de datos libres disponibles en sitios web. Los resultados demostraron que las técnicas implementadas generan modelos precisos y útiles para el análisis numérico, de manera versátil y en corto tiempo de procesamiento.
The generation of anatomical models is one the most important concern to biomedical researchers as well as to medical doctors, due to needed to understand the human tissues. Is know that the soft tissues like heart, brain, prostate and hard tissues like jaw, bones, skull, etc are structures of complex morphologies, so, the anatomical models generation is not an easy and trivial task. Currently, this task has benefited of advances of imaging diagnostic, which permit obtain cross and longitudinal sections of human body. In this research, we describe a method to obtain 3D discrete models of human body given by a dataset of medical images. Five main modules were implemented in prototype software: (1) Reading and 3D reconstruction of Computerized Axial Tomography and Magnetic Resonance Images. (2) Preprocessing techniques for improve the low medical images quality by using enhancement algorithms to reduce image noise and to increase structures contrast. (3) Combined segmentation techniques for tissue identification, which were applied through a multi-stage approach. (4) Post processing techniques to improve segmented volumes and (5) Exportation task of volumes to readable formats by Computer Aided Design (CAD) tools to be later analyzed by numerical methods. The performance of our method is shown on several medical examples and the techniques were validated using statistical descriptors to compare our models with models from free databases. Results showed that the implemented techniques generate precise and useful models for numerical analysis and medical survey, planning and surgery in a short processing time.
La obtención de modelos geométricos de las estructuras del cuerpo humano y sus enfermedades para fines de estudio, planificación e intervención quirúrgica se resuelve con la utilización de los métodos numéricos que aproximan soluciones a través del modelado anatómico de estos tejidos. Esta aproximación se ve afectada por la complejidad de las estructuras anatómicas, generalmente asimétricas y complejas, siendo difícil realizar simplificaciones en la resolución de las ecuaciones diferenciales del problema y la presencia de errores ocasionados por una inadecuada imposición de las condiciones de contorno y las cargas externas utilizadas en el modelado. Considerando que los tejidos blandos como el corazón, el cerebro y duros como el hueso mandibular, fémur, cráneo, etc. se caracterizan por tener una morfología variada, compleja y muchas veces superpuesta, la obtención de modelos precisos no resulta una tarea fácil y trivial. En la actualidad, esta tarea se ha visto beneficiada por diversas técnicas de diagnóstico por imagen como la Imagen por Resonancia Magnética (IRM), la cual es una técnica no invasiva que utiliza el fenómeno de la resonancia magnética para obtener información sobre la estructura y composición del cuerpo; Tomografía Axial Computarizada (TAC o TC), que utilizando la exploración de rayos X produce imágenes detalladas de cortes axiales del cuerpo; y la Tomografía por Emisión de Positrones (TEP), con la cual se utilizan tomógrafos capaces de detectar los fotones gamma emitidos por los pacientes para generar imágenes que muestran la actividad metabólica del cuerpo humano, entre otras. A estas imágenes se aplican técnicas de procesamiento digital en un conjunto de etapas que van desde la lectura y reconstrucción tridimensional de cada corte transversal inicial hasta la obtención de volúmenes geométricos finales de las estructuras anatómicas de interés. Existe variada bibliografía [1–3] así como software que estudian los procesos para extraer y analizar estructuras anatómicas humanas a partir de estas técnicas de radiología. Aunque algunos autores consideran procedimientos adicionales para estas tareas, la mayoría coincide en establecer tres etapas principales aplicadas después de que la data médica ha sido adquirida y digitalizada: (a) preprocesamiento o mejora de las imágenes para reducir ruido, eliminar artefactos, mejorar contraste, etc.; (b) segmentación o extracción de regiones de interés para su posterior análisis, y (c) visualización de las regiones segmentadas en vistas adecuadas (volumen, superficie, malla) para su posterior manipulación. La amplia gama de estudios basada en el procesamiento y visualización de estas imágenes demuestran que los métodos clínicos de diagnosticar y tratar las enfermedades han sido revolucionados. No solo permiten a los médicos y científicos obtener información vital observando el interior del cuerpo humano de una manera no invasiva, sino que además, constituyen una herramienta para la obtención de modelos geométricos más precisos. Algunos de los trabajos enmarcados en esta línea para la reconstrucción de tejidos duros son: Müller-Karger et al. [4], quienes obtuvieron un modelo tridimensional del hueso a partir de la lectura de TC. Posteriormente emplearon diferentes métodos numéricos para analizar mecánicamente el hueso y obtuvieron modelos discretos a través de programas CAD. Accardo et al. [5] reconstruyeron hueso trabecular a partir de IRM y TC y realizaron un análisis a los modelos obtenidos empleando el método de los elementos finitos. Pattijn et al. [6] propusieron una metodología para diseños especializados de prótesis de titanio a partir de las estructuras de hueso de fémur en TC. Los autores aplicaron técnicas de procesamiento de imágenes y modelado con elementos finitos. En el trabajo de Isaza et al. [7], se reconstruyeron estructuras cráneo-faciales a partir de TC, aplicando técnicas de procesamiento de imágenes para segmentar las estructuras de interés y obtener una nube de puntos. Finalmente emplearon los elementos finitos para simular la acción de un dispositivo utilizado en ortodoncia, tanto para el manejo dental como esquelético por medio de la tracción cervical. En la reconstrucción de tejidos blandos tenemos el trabajo de Peitgen et al. [8], donde se implementaron técnicas de procesamiento para el análisis y la visualización de tejidos del sistema vascular y aneurismas en IRM, TC, angiografías. Los volúmenes obtenidos fueron mejorados y visualizados en diferentes vistas. Goldenberg et al. [9] realizaron una revisión de las técnicas de procesamiento más utilizadas para la reconstrucción 3D de la superficie cortical del cerebro en IRM, los autores emplearon técnicas de segmentación basada en los modelos deformables para segmentar la materia gris del cerebro, sus resultados fueron comparados base de datos de phantoms de IRM. Otro trabajos enmarcados en la reconstrucción 3D de tejidos del cerebro a partir de IRM fueron presentados por Liew et al. [10] y Park et al. [11], los últimos también presentaron diferentes técnicas de visualización 3D.
En este trabajo se define una metodología eficiente para la obtención de modelos geométricos de tejidos del cuerpo humano a partir de las imágenes médicas, los cuales resulten útiles para su mallado y el análisis de su comportamiento con métodos numéricos como los Elementos Finitos (FEM). Esta metodología propone el uso de un conjunto de rutinas de procesamiento digital de imágenes y su adecuada combinación para cada etapa implicada en la obtención de modelos geométricos del cuerpo humano, las cuales fueron implementadas en una herramienta computacional desarrollada en MATLAB [12] con una interfaz de usuario que permitió configurar los parámetros y procesos dependiendo del tejido bajo estudio. Las etapas de procesamiento consideradas son: la etapa de lectura y reconstrucción de imágenes 3D, preproceso, segmentación, remuestreo, y exportación de modelos en formatos legibles por herramientas de visualización médica o CAD. Para la validación de la funcionalidad de las rutinas se consideraron dos criterios: los tiempos de ejecución y la precisión de los volúmenes obtenidos, para lo cual se aplicó el análisis de texturas a través de descriptores estadísticos calculados en la matriz 3D de los volúmenes generados.
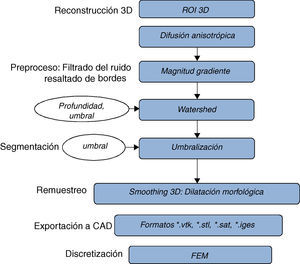
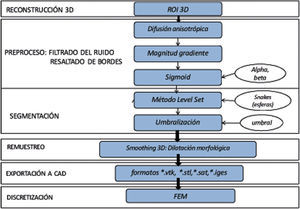
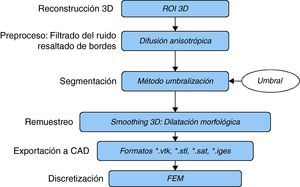
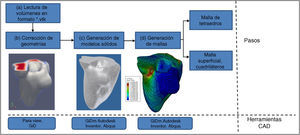
2Conceptualización y metodologíaEl problema de obtener modelos geométricos a partir de imágenes médicas implica la utilización de un conjunto de rutinas de procesamiento aplicadas a la matriz 3D de las imágenes médicas a lo largo de varias etapas de procesamiento. En la figura 1 se observan las cinco etapas propuestas con las herramientas de software utilizadas en cada una de ellas, las cuales son: (1) etapa de lectura y reconstrucción, en la cual se implementó una rutina para obtener una imagen 3D de dimensiones m×n×o obtenida por el apilamiento paralelo de o cortes ortogonales (axial, coronal o sagital) del mismo tamaño de m × n pixels, donde cada elemento de la matriz representa un valor de intensidad de gris calculado por la interacción de la radiación en el tejido. (2) Preproceso, en la cual se aplicaron rutinas de suavizado de ruido y realzado de bordes, de este modo se mejoró la calidad de las imágenes, preparándolas para la siguiente etapa. (3) Segmentación, en la cual se utilizaron rutinas de extracción del volumen de los tejidos u órganos de interés. (4) Remuestreo, donde se emplearon rutinas de posprocesamiento para suavizar las superficies y eliminar elementos no conectados presentes en los volúmenes segmentados. (5) Exportación de modelos, para lo cual se implementaron rutinas para almacenar los volúmenes obtenidos en formatos legibles por herramientas de visualización médica y CAD, en las cuales se visualice en sólidos, superficies, mallas, etc.
Esquema de procesos y rutinas implementados en una herramienta de procesamiento de imágenes médicas desarrollada en MATLAB [13].
La visualización 3D de modelos geométricos en bioingeniería depende de ambientes computacionales, hardware gráfico y herramientas de software que faciliten la interacción humano-máquina-data para la exploración y análisis de estos tejidos. Otro aspecto crítico es garantizar el realismo en la perspectiva tridimensional para la representación espacial de los datos, la representación de la información temporal y otras formas de señales visuales como texturas y tonos, así como, resolver el paradigma de interacción entre los usuarios y la información a través de los sistemas de visualización.
Para la obtención de los modelos geométricos y la interacción con los algoritmos de procesamiento considerados en este trabajo, se desarrolló una herramienta computacional [13] bajo la plataforma de MATLAB [12], en la cual se integraron las rutinas por etapas (fig. 1).
A continuación se describen los algoritmos de procesamiento considerados por cada una de estas etapas, y como fueron implementados en este trabajo.
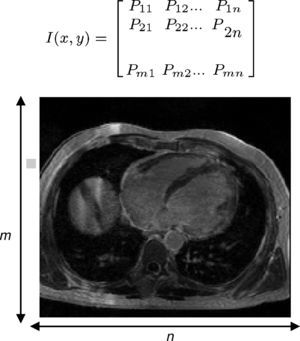
2.1Lectura y reconstrucción 3DCada corte adquirido en una sesión de diagnóstico por imagen es representado por una matriz bidimensional de tamaño m ×n (fig. 2), donde cada elemento Px,y de la matriz es conocido como pixel (picture element), n representa el número de pixels a lo ancho de la imagen y m es el número de pixels a lo largo. En la figura, cada elemento Px,y representa un valor en escala de gris, el cual refleja el grado de atenuación del haz radiológico sobre el tejido humano.
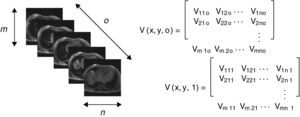
La reconstrucción 3D de estos cortes iniciales es obtenida por el apilamiento paralelo de o cortes de la misma resolución (tamaño m × n pixels), lo cual es representado por una matriz 3D de dimensiones m × n × o, donde cada elemento Vx,y,z de esta matriz es denominado voxel, el cual es el elemento básico de un volumen (fig. 3). En la figura, se presenta la representación matricial del primer y último corte de la imagen 3D.
Para mantener la relación del tamaño del volumen reconstruido con el tamaño real del tejido, se tiene en cuenta el espaciado de cada voxel (voxel spacing) que conforma el volumen, el cual es obtenido de la información incluida en la imagen médica. El procesamiento de las imágenes se lleva a cabo procesando los valores de niveles de gris contenido en la matriz que representa la imagen.
Un paso posterior fue la selección de un Region of Interest (ROI) en la imagen 3D inicial para obtener sub-volúmenes que contengan las zonas de interés. En cada uno de estos sub-volúmenes se aplicaron flujogramas conformados por algoritmos desde el preprocesamiento hasta la obtención de los volúmenes de los tejidos de interés.
2.2Preprocesamiento de imágenes médicasLas imágenes médicas son usualmente afectadas por ruido generado por interferencia u otros fenómenos que afectan los procesos de medición en los sistemas de adquisición de imágenes. La presencia de ruido es inherente a los sistemas de medición y usualmente es un factor limitante en el desempeño de los instrumentos médicos [3]. Asimismo, la naturaleza de los sistemas fisiológicos bajo investigación y los procedimientos usados en radiología también afectan el contraste y la visibilidad de detalles [2], siendo necesario modificar el rango de intensidades de gris de las imágenes para mejorar la visualización de aquellas zonas más brillantes que por la característica del ojo humano son difíciles de diferenciar en comparación a las zonas más oscuras que se aprecian con mejor detalle.
El éxito en la obtención de los modelos geométricos de tejidos dependerá de las técnicas empleadas en esta primera etapa. Dos de las técnicas más empleadas son: (a) técnicas de reducción del ruido, y (b) técnicas de realzado de bordes, las cuales se implementaron en este trabajo y son descritas a continuación.
2.2.1Reducción de ruidoEl ruido es caracterizado por un cierto valor de amplitud y distribución y su nivel depende del tejido tratado y de la resolución espacial de cada imagen, por ejemplo, imágenes de gran resolución como TC con 0,5mm de grosor de corte (slice trickness), presentan mayor nivel de ruido que aquellas imágenes de 0,1mm de grosor. En las imágenes médicas, el ruido se debe a procesos estocásticos de la adquisición de la imagen o durante su reconstrucción, lo que disminuye su calidad. Para resolver este problema, se han desarrollado filtros de reducción, la mayoría de ellos han sido diseñados empleando la distribución de Gauss. Matemáticamente, una imagen corrupta con ruido puede ser representada por la ecuación (1).
donde u(x,y) es la imagen original, y v(x,y) es la imagen observada (corrupta con ruido) y n(x,y) representa el ruido aditivo. El proceso de formación de la imagen puede ser modelado por el sistema lineal descrito en la ecuación (2), donde h(x,y;x’,y’) representa la respuesta de impulso del proceso de adquisición de la imagen.El ruido presente en una imagen se manifiesta de diferente manera y su interpretación dependerá de la imagen en sí y de su percepción visual. La estimación de las características estadísticas de ruido presente en una imagen es necesaria para ayudar a separar el ruido de la imagen. Acharya y Ray [14] describen cuatro clases de ruido: el ruido aditivo, ruido multiplicativo, ruido impulsivo y ruido de cuantificación, siendo los dos primeros los tipos de ruido más comunes en imágenes médicas, los cuales han sido considerados en este trabajo.
2.2.1.1Ruido aditivoEs el ruido generado por los sensores del tipo Gaussiano blanco y es definido en la ecuación (3), donde g(x,y) es la imagen con ruido observada resultante de la imagen I(x,y) corrupta con el ruido aditivo n(x,y).
En la figura 4, se muestra un ejemplo de ruido aditivo al agregar ruido aditivo del tipo Gaussiano a una imagen de phantom de BrainWeb[15] que simula una IRM del cerebro. El comportamiento del ruido agregado puede ser observado en el histograma de la figura (4.d).
2.2.1.2Ruido multiplicativoEn esta clase encontramos el tipo de ruido speckle muy común en las imágenes médicas, principalmente en las imágenes de ultrasonido. Este tipo de ruido puede ser representado por la ecuación (4), donde g(x,y) es la imagen observada con ruido, I(x,y) es la imagen en formación, c(x,y) es el componente de ruido multiplicativo y n(x,y) es el componente de ruido adicionado.
En la figura 5 es presentado un ejemplo de ruido multiplicativo agregado a la imagen de phantom que simula una resonancia magnética del cerebro. El comportamiento del ruido agregado puede ser observado en el histograma de la figura (2.d).
Existen diversos tipos de filtros dedicados a reducir la presencia de ruido en las imágenes, la mayoría de ellos han sido diseñados empleando la distribución de Gauss. Dentro de los filtros más usados tenemos: el filtro Gaussiano, filtro media, filtro mediana. Existen otros filtros más sofisticados como los filtros basados en ondículas (wavelets) y los filtros de difusión anisotrópica. Este último es el empleado en este trabajo.
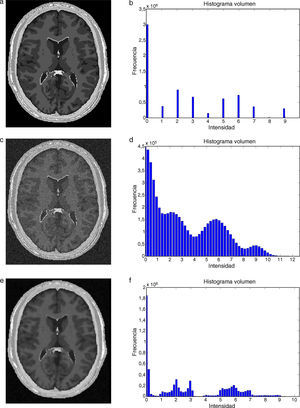
2.2.1.3Filtro de difusión anisotrópicaEs un filtro del tipo no lineal propuesto por Perona and Malik [16], quienes asumieron que el comportamiento del ruido en la imagen es similar a la «propagación del calor en un cuarto vacío», por lo que modificaron la conocida ecuación de conducción del calor, obteniendo la ecuación (5). Este filtro utiliza gradientes locales para controlar la anisotropía del filtro.
En la ecuación se utiliza un detector de bordes |∇I|, responsable de suavizar el ruido, cuyo valor tiende al infinito al acercarnos a un borde perfecto. La función g(|∇I|) controla la fuerza de difusión, reduciendo la conductancia en áreas de valores de |∇I| grandes. Se le asigna un valor 0 donde el valor del gradiente es grande y disminuye completamente cuando el gradiente es bajo, es decir: g(x)→0, si x→∞ (valor alcanzando en un borde); y g(x)→1, si x→0 (valor alcanzando dentro de una región). De esto deducimos que la operación de suavizado del ruido es un proceso de difusión que se suprime o detiene en las fronteras mediante la selección adecuada de valores de difusión espacial. Dependiendo de los valores asumidos por la fuerza de difusión, el filtro es capaz de suavizar entre regiones sin afectar los bordes.
La difusión anisotrópica ha tenido bastante aceptación en el filtrado del ruido debido a su velocidad y simplicidad de cálculo algorítmico. En Gerig et al. [17] fue aplicado en la reducción de ruido y suavizado de bordes en IRM de secuencia de onda eco spin y eco gradiente en 2D y 3D y en Santareli et al. [18] se demostró su máximo rendimiento en el filtrado local para segmentación cardiaca. Asimismo, en el trabajo de Landini et al. [19] se aplicó el filtro de difusión anisotrópica en un phantom de IRM del ventrículo izquierdo y en una IRM original.
En este trabajo, se implementó una rutina de suavizado de ruido con preservación de bordes empleando la librería de difusión anisotrópica: itk::GradientAnisotropicDiffusionImageFilter [20]. En la figura 6 se presentan los resultados obtenidos al aplicar los filtros en un phantom de IRM [15] que simulan IRM del cerebro a través de volúmenes «fuzzy». Esta imagen de phantom tiene dimensiones de 181 × 217 × 181 (X × Y × Z), con voxels isotrópicos de 1,0mm3. Por visualización se presenta el corte axial 98, sin embargo, los filtros e histogramas mostrados fueron aplicados sobre el volumen completo. En las figuras 6.a y b se presenta el corte axial 98 del phantom y el histograma del phantom completo, respectivamente. En las figuras 6.c y 6.d se presenta el corte axial 98 con ruido gaussiano aditivo y el histograma de este nuevo volumen con ruido, respectivamente. En la figura 6.e se presenta la imagen resultante luego de aplicar al volumen de la figura 6.e el filtro de difusión anisotrópica itk::GradientAnisotropicDiffusionImageFilter. En la figura 6.f es mostrado el histograma de esta imagen filtrada.
Aplicación de filtros en imagen phantom de IRM corrompida con ruido gaussiano. (a). Imagen de phantom original, vista del corte 98. (b) Histograma de (a). (c) Imagen (a) corrompido con ruido gaussiano. (d) Histograma de (c). (e) Imagen (a) suavizada con filtro de difusion anisotrópica. (f) Histograma de (e).
El borde de una imagen se define como un salto brusco en los valores de intensidad e indica las fronteras o líneas de separación entre los distintos objetos presentes en ella. Los bordes de los objetos se ven en la imagen como discontinuidades de ciertas propiedades: intensidad, color, textura. En las imágenes médicas, los tejidos están separados por bordes y regiones discontinuas que pueden ser detectables a través de diversas técnicas, pero sin un mayor procesamiento no necesariamente se extraerá una región de interés.
2.2.2.1Generación de imágenes módulo del gradienteSon diversas las técnicas aplicadas para realzar los bordes en imágenes médicas, la mayoría son basadas en el cálculo del gradiente y su módulo. Estos filtros, al ser aplicados sobre una imagen en escala de grises, calculan el gradiente de la intensidad de brillo de cada punto (pixel o voxel) proporcionando la dirección del mayor incremento posible (de negro a blanco), además calculan el valor de cambio en esa dirección, es decir, devuelven un vector. En la ecuación (6) se presenta la función para el cálculo del gradiente de un punto en las direcciones X, Y, Z. El resultado final muestra que tan brusca o suavemente cambia una imagen en cada punto analizado, y a su vez que tanto un punto determinado representa un borde en la imagen y también la orientación a la que tiende ese borde. En la práctica, el cálculo de la magnitud de la gradiente brinda nociones de un borde y ayuda a la separación de regiones homogéneas, lo que resulta más sencillo que la interpretación de la dirección. En la ecuación (7) se presenta la función empleada para el cálculo de la magnitud del gradiente en las tres direcciones.
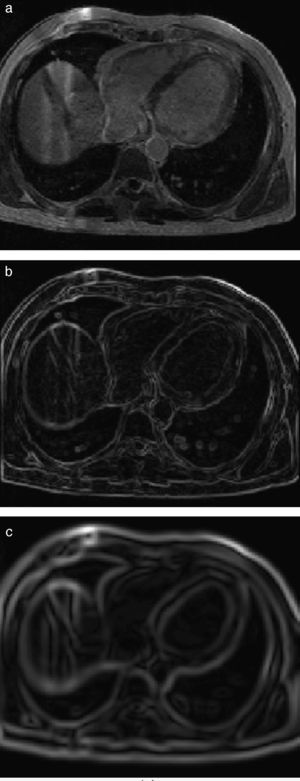
En la figura 7 se presenta la aplicación de dos rutinas del cálculo del gradiente en IRM cardiovascular. Obsérvese como los contornos son mejorados y se puede distinguir mejor el músculo miocardio y el ventrículo izquierdo. En la figura 7.b se aplicó el operador Sobel en las direcciones x,y,z. En la figura 7.c se presenta el resultado de aplicar el filtro itk::GradientMagnitudeRecursiveGaussianImageFilter[20]. Este filtro calcula la magnitud de la imagen gradiente por cada pixel o voxel. El proceso computacional consiste en primero suavizar la imagen a través de la convolución con una máscara Gaussiana y luego aplicar el operador diferencial.
Aplicación de las rutinas de módulo de una imagen gradiente. (a) IRM cardiovascular original, solo se visualiza el corte axial número 33. (b) Aplicación del operador Sobel en x,y,z en la imagen (a). (c) Imagen módulo del gradiente de (a) empleando itk::GradientMagnitudeRecursiveGaussianImageFilter.
El uso del gradiente puede ser muy sensible al ruido si no se aplica ningún suavizado previo, por ello, las imágenes de entrada a este filtro fueron obtenidas al aplicar los filtros de suavizado de ruido mencionados en la sección anterior.
2.2.2.2Reforzamiento adicional de bordesEn algunos casos, después de mejorar los bordes con el cálculo del gradiente, se aplicó un filtro adicional con la finalidad de reforzar los bordes y garantizar una adecuada segmentación. Se integró el filtro sigmoid proporcionado por ITK [20] en itk::SigmoidImageFilter, el cual transforma la intensidad de los valores de gris de la imagen, generando una imagen Isigmoid con los voxels de los bordes reforzados y los demás voxels de las regiones atenuados progresivamente. Este filtro fue configurado por cuatro parámetros y se define según la ecuación (8).
donde I contiene las intensidades de los voxels de entrada. La imagen Isigmoid contiene las intensidades de los voxels de salida, Min y Max son los valores mínimo y máximo de la imagen de salida, α define el ancho del rango de intensidades de entrada, y β define la intensidad alrededor de la cual el rango es centrado.En la figura 8 se presenta la aplicación de la rutina del filtro sigmoid a partir de la imagen módulo gradiente mostrada en la figura 7.c.
2.3SegmentaciónDespués de mejorar los niveles de intensidad y corregir artefactos en las imágenes médicas, la siguiente etapa es la segmentación que consiste en dividir las imágenes en regiones contiguas (subregiones o sub-volúmenes) cuyos elementos miembros (pixels o voxels) tienen propiedades de cohesión comunes. Es un paso previo para extraer parámetros cuantitativos, cualitativos y evaluar la morfología y funcionamiento del objeto segmentado, su éxito depende de la realización de un óptimo preprocesado.
Los algoritmos de segmentación de imágenes médicas tienen un papel importante en el diagnóstico y tratamiento médico, son usados para el análisis de estructuras anatómicas y los tipos de tejidos, la distribución espacial de la función y la actividad de los órganos, y las regiones patológicas. Su aplicación incluye diversas aplicaciones médicas como la detección de tumores cerebrales [21], extracción de la zona afectada por tuberculosis extra pulmonar [22], visualización de patologías del corazón [23], la detección de contornos coronarios en angiogramas, cuantificación de lesiones de esclerosis múltiple, simulación y planificación de cirugías, medición del volumen de tumores y su respuesta a terapias, clasificación automatizada de células sanguíneas, estudio del desarrollo del cerebro, detección de micro calcificaciones en mamografías, entre otras aplicaciones.
Los métodos para llevar a cabo el proceso de segmentación varían ampliamente dependiendo de la necesidad específica, tipo de la imagen, y otros factores. Por ejemplo, la segmentación del tejido del cerebro tiene diferentes requerimientos que la segmentación del corazón o la segmentación de estructuras óseas como la mandíbula o el fémur. Se ha comprobado que los métodos especializados para aplicaciones particulares pueden obtener mejores resultados tomando en cuenta conocimiento a priori. Sin embargo, la selección de un método apropiado para un problema de segmentación resulta una tarea muy difícil en algunos casos. Debido a que actualmente no existen métodos de segmentación generales que puedan ser aplicados a cualquier variedad de datos y que alcancen resultados aceptables para todo tipo de imagen médica, en este trabajo se implementaron y combinaron varios métodos de segmentación, los cuales son introducidos en las siguientes secciones.
2.3.1Segmentación basada en umbralesLa umbralización es una técnica de segmentación efectiva en imágenes cuyas estructuras tienen intensidades u otras características diferenciables. Algunas de las técnicas de umbralización están basadas en el histograma de la imagen y otras están basadas en propiedades locales como el valor promedio local, la desviación estándar o el gradiente local. En su forma más simple, esta técnica es llamada umbralización global [2,24] o umbralización bimodal, en la cual, a partir de un histograma bimodal para una imagen f(x,y,z), el objeto puede ser extraído del fondo con una simple operación que compara los valores de f(x,y,z) con un umbral T que puede separar las dos modas del histograma generando como resultado una imagen binaria. En otros casos se pueden definir varios umbrales para segmentar la imagen, en este caso se está hablando de una umbralización multinivel. En la ecuación (9) se representa la umbralización bimodal.
donde T es un valor umbral, entonces bij=1 para todos los pixels del objeto de interés, y bij=0 para todos los pixels del fondo (background). La ecuación (9) puede ser extendida a una umbralización multinivel al definir varios valores de umbral.Las técnicas de umbralización fueron utilizadas en imágenes, cuyo histograma presentaba picos bien definidos que permitían segmentar con facilidad diferentes tipos de tejidos. Estos tipos de histograma son característicos de las imágenes de TC de estructuras óseas, en vista de que los niveles de gris del hueso son mayores a las intensidades de otros tejidos como piel, grasa, músculo, etc., según la escala de Hounsfield[1].
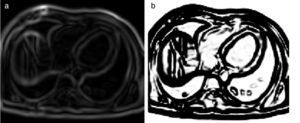
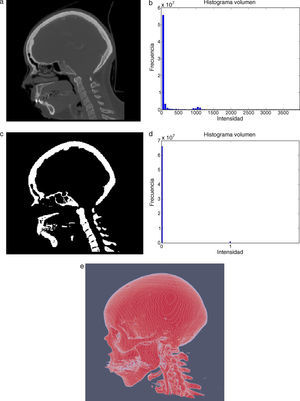
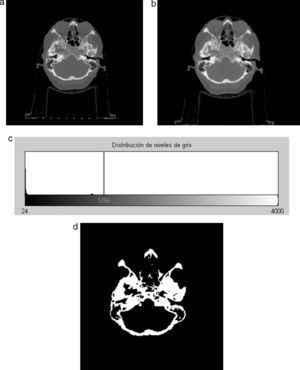
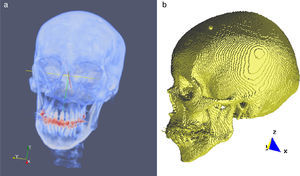
En la figura 9 se presenta el resultado de aplicar la rutina de umbralización global a una imagen tridimensional reconstruida a partir de imágenes de TC cráneo-facial, para segmentar el hueso del cráneo del background y los demás tejidos. En la figura 9.b se presenta el histograma del volumen total de la figura 9.a, en el cual se observan varios picos. La aplicación de esta rutina consistió en seleccionar el valor del umbral T igual a 1.235, asignándose el valor de 0 (negro) a aquellos voxels menores a T un umbral de valor 1.235. A los voxels con valores mayores o igual a T se les asignaron el valor 1 (blanco). De esta manera se obtuvo una nueva imagen binaria donde se ha segmentado el hueso cráneo-facial y parte de las vértebras (fig. 9.c). El histograma de esta nueva imagen binaria es presentado en la figura 9.d y la vista 3D del hueso segmentado es presentada en la figura 9.e.
Técnica de umbralización aplicada a imágenes de TC cráneo-facial. (a) Vista original de un corte sagital. (b) Histograma de imagen a. (c) Imagen resultante de umbralizar imagen (a) con un umbral T=1235. (d) Nuevo histograma de la imagen binaria (c). (e) Vista 3D del volumen del hueso cráneo-facial y parte de las vértebras segmentado con umbralización.
Comúnmente, esta técnica es empleada para extraer regiones de la imagen que están conectadas según cierto criterio predefinido (los objetos a segmentar son regiones con características similares). En su forma más sencilla, se inicia con el establecimiento de una semilla que puede ser un pixel, voxel o conjunto de ellos, los cuales son seleccionados manualmente por el usuario. En el siguiente paso los elementos vecinos son examinados y adicionados a la región sí son suficientemente similares basados en un test de uniformidad (criterio de homogeneidad como intensidad de gris, promedio, desviación estándar, etc.). El procedimiento continúa hasta que no puedan ser adicionados más voxels. Finalmente, el objeto segmentado es representado por todos los elementos que han sido aceptados durante el procedimiento de búsqueda.
Existen métodos de Region Growing avanzados, los cuales son obtenidos mediante la combinación de diferentes criterios de inclusión, como umbrales, gradientes, media, desviación estándar, etc.
Region Growing es muy utilizada en aplicaciones médicas para extraer estructuras del cuerpo y sus patologías. Liu et al. [25] emplearon este algoritmo para segmentar los ventrículos cerebrales. Avazpour et al. [22] segmentaron la infección por tuberculosis extra-pulmorar, modificando el algoritmo de Region Growing. Mühlenbruch et al. [26] consiguieron extraer el ventrículo izquierdo en TC para posterior análisis numérico. Asimismo, esta técnica es usualmente utilizada para segmentar estructuras vasculares [27,28].
Una técnica avanzada de segmentación Region Growing fue implementada en la herramienta para extraer regiones con texturas de características similares y bordes claramente definidos. Esta técnica fue empleada para extraer regiones conectadas de texturas con características similares. El primer paso del algoritmo consistió en establecer manualmente una o más semillas (volumen esférico) dentro de la región de los tejidos de interés. En el siguiente paso se analizaron los voxels vecinos a la región, calculando la media m y la desviación estándar σ, agregando los pixels de posición X cuyo valores de intensidad de gris cumplían la condición establecida en la ecuación (10).
donde, I: imagen, X: posición del voxel vecino analizado, m: media, σ: desviación estándar, f: factor de multiplicación.El segundo paso fue ejecutado hasta no poder adicionar más voxels. Finalmente, el objeto segmentado fue representado por todos los elementos aceptados durante el procedimiento de búsqueda.
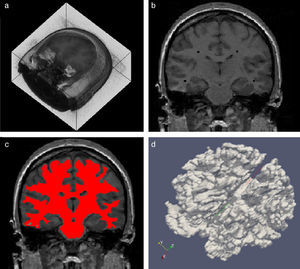
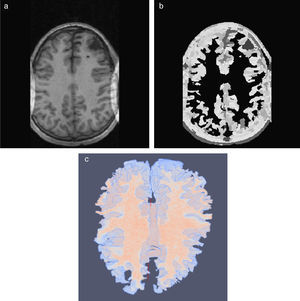
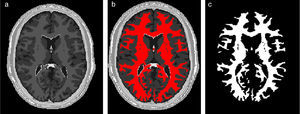
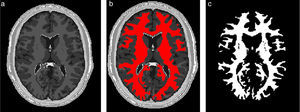
En la figura 10 se presenta el resultado de segmentar la materia blanca del cerebro en IRM empleando la rutina de Region Growing mencionada. En la figura 10.a se observa la vista 3D del volumen inicial. En la figura 10.b se observa uno de los cortes de la figura 10.a con la selección de cuatro semillas iniciales de forma esférica dentro de la zona de la materia blanca. En la figura 10.c se observa en color rojo la zona región de la materia blanca obtenida al finalizar la rutina de segmentación. En la figura 10.d se presenta una vista 3D de la zona de la materia blanca segmentada.
Segmentación de materia blanca empleando Region Growing en IRM del cerebro. (a) Volumen formado por varios cortes de IRM cerebral original. (b) Vista de un corte coronal con la lección de cuatro semillas iniciales. (c) Vista del corte coronal (b) con la materia blanca segmentada con Region Growing. (d). Vista volumétrica de la materia blanca segmentada en (c).
La segmentación Watershed es otro método basado en región que tiene sus orígenes en la morfología matemática. El concepto general fue introducido por Digabel y Lantuéjoul en 1978 [29]. En este algoritmo, la imagen se ve como una superficie topográfica con ríos y valles, donde los valores de elevación del paisaje son definidos por el valor de gris de cada pixel o su magnitud gradiente. Basados en una reconstrucción 3D, el algoritmo divide la imagen en regiones llamadas «cuencas hidrográficas» (catchment basins). Para cada mínimo local, una cuenca hidrográfica comprende todos los puntos cuyo camino más empinado desciende terminado sobre este mínimo. Finalmente, Watershed queda definido por las líneas de borde que separa una cuenca de otra. Las cuencas hidrográficas (segmentos obtenidos) son distinguidas por etiquetas con diferente intensidad de gris. Watershed es utilizado en una variedad de aplicaciones de segmentación. Por mencionar algunas, Hahn y Peitgen [30] segmentaron la zona del cerebro y los ventrículos cerebrales aplicando Watershed sobre IRM. Asimismo, Kuhnigk et al. [31] utilizaron Watershed para delimitar los lóbulos pulmonares en TC.
En este trabajo, Watershed fue empleado para segmentar estructuras grandes como el ventrículo izquierdo y con textura continua como las estructuras óseas. En nuestro algoritmo, la imagen de entrada fue la imagen magnitud gradiente Img obtenida en la etapa de preproceso (sección 2.2.2), la cual es considerada como una función de altura donde los valores altos indican la presencia de bordes. El primer paso fue eliminar las regiones poco profundas que se encuentren por debajo de un mínimo valor de umbral, lo cual ayudó a controlar la sobre segmentación. A partir de esto, el algoritmo creó una segmentación inicial siguiendo el más rápido descenso de cada pixel hasta los mínimos locales. El resultado inicial fue pasado a un segundo filtro que consideró solo aquellas regiones con una profundidad menor a un nivel de profundidad máximo establecido, de esta manera se controló hasta donde descendía la segmentación (llenado de cuencas). Los parámetros umbral y profundidad fueron establecidos dentro del rango [0,0-1,0] y elegidos de manera arbitraria. Como resultado de esta función, se obtuvo una primera segmentación de la imagen Iw conformada por varios segmentados conectados de manera no conexa y etiquetados con un nivel de gris distinto, como se describe a continuación:
Uno de los principales problemas de esta técnica, es la sobre segmentación de regiones ocasionada por el ruido presente en las imágenes, de ahí la importancia de la etapa de preproceso que reduzca el ruido y resalte los bordes.
Para obtener el volumen completo de las zonas de interés fue necesario agrupar algunos de los segmentos adyacentes considerando los niveles de gris de las regiones etiquetadas. Es así, que en algunos casos, los volúmenes finales If se obtuvieron a partir de la técnica de umbralización, estableciendo un umbral inferior t0 y un umbral superior tf, donde: t0=If=tf.
En la Fig. 11 se presenta la aplicación de la rutina Watershed para segmentar la materia blanca del cerebro en la reconstrucción 3D de IRM. En la Fig. 11.a se observa uno de los cortes axiales de la imagen original utilizada, obsérvese que la imagen presenta artefactos (aliasing) ocasionados por la baja resolución utilizada en la reconstrucción inicial de la imagen. En la Fig. 11.b se visualizan los segmentos encontrados al aplicar esta técnica en la imagen original, etiquetados con diferentes niveles de gris, entre ellos la zona de la materia blanca. Es importante destacar que las técnicas de segmentación son aplicadas sobre imágenes previamente pre-procesadas con la finalidad de corregir artefactos presentes en las imágenes originales como el mostrado en la Fig. 11.a, la eficiente combinación y aplicación de estas técnicas son la base de este trabajo y serán presentadas en los siguientes apartados. En la Fig. 11.c se visualiza la vista 3D de la zona de la materia blanca segmentada.
Segmentación 3D de la zona de la materia blanca en IRM del cerebro empleando Watershed. (a) Vista de un corte axial original. (b) Regiones segmentadas con Watershed y etiquetadas con diferentes intensidades de gris. (c) Vista volumétrica de la zona de la materia blanca segmentada.
Uno de los principales problemas de esta técnica, es la sobre-segmentación de regiones ocasionado por el ruido presente en las imágenes. Para resolver este problema, se aplicó previamente filtros de reducción del ruido.
2.3.4Métodos Level SetLos algoritmos de segmentación basados en modelos, implican tareas más especializadas que las técnicas mencionadas anteriormente. Estas técnicas utilizan información del tamaño, la forma de los objetos, las distribuciones de gris, características de simetría, orientación y gradiente, entre otras. Dentro de los algoritmos más conocidos tenemos el modelo de los contornos activos (active contour models) y la técnica Level Set and Fast Marching propuestas por Sethian [32], ambos son una variante generaliza de los modelos deformables para la segmentación de estructuras.
La segmentación a través de la técnica Level Set es ampliamente utilizada para segmentar estructuras anatómicas de forma variable y solapadas con otras, generalmente difíciles de segmentar con las técnicas anteriores. Se basa en la aplicación de métodos numéricos para rastrear la evolución de contornos y superficies denominados snakes que son colocados inicialmente sobre la imagen y van modificándose hasta encontrar bordes y adquirir la forma de las zonas de interés. Un snake puede ser una curva o superficie que se deforma en dirección de características de interés en la imagen como líneas, bordes, y es controlado a través de una ecuación diferencial, ver ecuación (12), que establece en valor de la función Level Set Ψ basada en tres velocidades: velocidad de propagación que es la responsable de la extensión del snake hacia dentro o hacia fuera; velocidad de curvatura: responsable de controlar la forma del snake; velocidad de advección: es la más crítica y es responsable de que el snake frene ante la presencia de bordes en la regiones.
donde A: velocidad de advección; P: velocidad de propagación; Z: modificador de la curvatura k; α,β,γ: modifican cada velocidad en cada movimiento del snake.La técnica Level Set fue implementada en la herramienta para segmentar tejidos de estructuras más complejas y poco definidas. Se implementó una rutina que integró la librería itk::FastMarchingImageFilter[20] que es una versión más simplificada de los métodos Level Set. En el presente trabajo, el algoritmo se inicia con la implantación de snakes de forma esférica en las zonas de interés. En un instante de tiempo, la forma del snake pudo ser obtenida extrayendo la función Γ (zero-Level Set) de la imagen salida, como se presenta en la ecuación (14). Esta imagen extraída representa un mapa de distancias, siendo necesario aplicar umbralización, eligiendo los valores cercanos a cero para extraer la zona de la cicatriz.
En la figura 12, se presenta la aplicación de esta rutina para segmentar la zona de la aorta descendente en la reconstrucción 3D de IRM cardiovascular. En la figura 12.a se presenta uno de los cortes de la imagen original, en la cual se observa la implantación de un snake inicial en la zona de la aorta. En la figura 12.b se observa la imagen mapa de distancias generada luego de aplicar el algoritmo Level Set, obsérvese que los voxels dentro de la región de la aorta tienen valores de intensidad más oscuros comparados con aquéllos que se van alejando de esta zona, cuyos valores de intensidad son más. En la figura 12.c se observa en color rojo la zona de la aorta segmentada, la cual fue obtenida con umbralización al seleccionar los voxels de la figura 12.b con niveles de gris cercanos de cero.
Segmentación de aorta descendente empleando Level Set. (a) Vista de un corte axial de IRM cardiovascular con la implantación de un snake en la zona de la aorta. (b) Imagen mapa de distancias obtenida luego de aplicar la técnica Level Set. (c) Selección de la zona de aorta extrayendo la función zero level (d) Vista 3D de la aorta descendente segmentada.
Tras aplicar las técnicas de segmentación fue necesario someter los volúmenes obtenidos a un proceso de remuestreo para corregir posibles agujeros dentro de las regiones y suavizar superficies superpuestas. Para esta tarea, se implementaron las siguientes rutinas de posprocesamiento:
2.4.1Corrección de zonas no conectadasSe implementó una rutina interactiva, utilizando la librería itk::VotingBinaryIterativeHoleFillingImageFilter[20], la cual a través de un análisis de cada uno de lo voxels del volumen, rellena aquellos agujeros encontrados, eliminando de esta manera las zonas no conectadas.
2.4.2Suavizado de superficiesLas superficies rugosas o superpuestas presentes en las segmentaciones iniciales fueron corregidas con una rutina de suavizado combinando las técnicas de morfología matemática: dilatación y erosión con el filtro de Gauss. En Gonzalez y Woods [24], se explica con mayor detalle estas técnicas. En la figura 13 se presenta un ejemplo de la aplicación de estas rutinas de remuestreo. En la figura 13.a se muestra el volumen original segmentado con el método Level Set, en el cual se observa con superficies rugosas. En la figura 13.b se observa el remuestreo aplicado al volumen de la figura 13.a, lo cual generó un volumen más liso.
2.5Exportación de modelos geométricosPara procesar los modelos geométricos en otras herramientas de visualización médica y CAD, se implementó una rutina de exportación de la data de los volúmenes en formatos *.vtk[33] que permite guardar las coordenadas de los voxels y el tamaño de ellos en las direcciones x, y, z y en formato *.stl[34] que almacena una malla de triángulos sobre las superficies para definir la forma del objeto. Este es un formato de salida estándar para la mayor parte de los programas CAD. Utilizamos GiD [35] y ParaView [36] para visualizar los modelos en superficie y generar el mallado. Se empleó Autodesk Inventor [37] para convertir los modelos en sólidos y finalmente, se utilizó Abaqus [38] para discretizar los modelos y hacer análisis con elementos finitos.
Se emplearon las herramientas Abaqus y Autodesk Inventor para leer los archivos generados y verificar si los modelos geométricos obtenidos con la metodología eran útiles para discretizar, para ello se asignaron valores de contorno de prueba en diferentes zonas del volumen sólido de los modelos.
2.6Análisis estadístico de modelos geométricosPara analizar la precisión y confiabilidad de las técnicas implementadas y los resultados obtenidos, se implementaron rutinas de análisis estadístico de texturas, empleando descriptores descritos para comparar los volúmenes obtenidos con nuestras rutinas de procesamiento con los volúmenes obtenidos por otros medios como segmentación manual o proporcionados por sitios web.
El sistema de visión humano percibe escenas con variaciones de intensidad y color, los cuales forman ciertos patrones que se repiten, llamada texturas. Acharya y Ray [14] definen las siguientes afirmaciones sobre las texturas: (a) una textura es un conjunto de patrones repetitivos, los cuales caracterizan a las superficies de varias clases de objetos. La clasificación de estos patrones resultará fácil si las texturas presentes en las imágenes se pueden identificar y diferenciar entre sí; (b) las texturas proporcionan información importante de la disposición de los elementos importantes de una imagen, y (c) los atributos de una textura se pueden describir en términos cualitativos como la homogeneidad, la orientación de las estructuras y la relación espacial entre las intensidades de la imagen.
El análisis de texturas aplicada al mundo de las imágenes está relacionada con la distribución espacial de los niveles digitales presentes en la imagen. Dependiendo de la selección de las características y la filosofía de la clasificación, el análisis de texturas en una imagen es clasificado en tres grandes métodos: métodos espaciales, métodos estructuras y métodos estadísticos [14]. El análisis de texturas estadístico se basa en la cuantificación y caracterización de las propiedades estocásticas de la distribución espacial de los niveles de gris en una imagen a través del cálculo de descriptores estadísticos.
En este trabajo se implementaron seis descriptores estadísticos, los cuales estudian la relación de un pixel con su entorno, y caracterizan la suavidad, rugosidad, etc. Para calcular este tipo de descriptores se utilizó el histograma de probabilidades hp obtenido al dividir cada valor del histograma original de la imagen entre el número total de pixels. Los descriptores se presentan a continuación.
2.6.1MediaEmpleado para calcular el promedio de los niveles de gris de la imagen. Este descriptor queda definido como:
donde μ es la media, h(i) es el histograma de probabilidades para los niveles de gris i de la imagen que van desde 1 a n.2.6.2Momento de segundo orden (desviación estándar)Mide la dispersión o contraste entre los niveles digitales. Se identifica con la homogeneidad que se percibe en la imagen. En una imagen oscura la desviación estándar σ es alta si hay pixels de alto nivel de gris en un fondo de bajo nivel de gris. Si σ=0, entonces la intensidad de la imagen es constante, si σ=1, la intensidad de la imagen posee valores altos de varianza.
2.6.3Momento de 3.er orden (asimetría)Mide la asimetría del histograma. Un histograma simétrico tendrá un momento de tercer orden con valor cercano a cero. Toma valor positivo (negativo) si el histograma está más alargado a la derecha (izquierda). En términos matemáticos, la asimetría es una medida de la asimetría de los datos alrededor de la media muestral. Si el valor de asimetría es negativo, los datos son distribuidos de manera más a la izquierda de la media que a la derecha. Si la asimetría es positiva, los datos se extienden más a la derecha. La asimetría de la distribución normal (o cualquier distribución perfectamente simétrica) es cero. Este descriptor es definido por la siguiente ecuación:
donde μ es la media de la imagen, σ es la desviación estándar, h(i) es el histograma de probabilidades para los niveles de gris i de la imagen que van desde 1 a n.2.6.4Momento de 4.° orden (homogeneidad)Propiedad conocida como curtosis mide el achatamiento del pico superior del histograma. Mientras más pequeño es su valor el pico es más redondeado. Es un indicador de uniformidad de la imagen que mide la distribución de los valores del histograma. La curtosis de una distribución normal es de 3. Las distribuciones que son más propensas que los valores atípicos de la distribución normal tienen curtosis mayor a 3; distribuciones que son menos propensas a tener valores atípicos tienen curtosis menor a 3. La homogeneidad en una imagen queda definida como:
donde μ es la media de la imagen, σ es la desviación estándar, h(i) es el histograma de probabilidades para los niveles de gris i de la imagen que van desde 1 a n.2.6.5Entropía promedioMide la granularidad de la imagen, es una medida estadística de la aleatoriedad que puede ser utilizada para caracterizar la textura de la imagen, un valor alto indica una textura gruesa, tendrá valor cero si es constante. La entropía queda definida como:
donde h(i) es el histograma de probabilidades para los niveles de gris i de la imagen que van desde 1 a n.3Casos de estudioLas rutinas presentadas en el apartado anterior fueron aplicadas en imágenes médicas, orientados por flujogramas de algoritmos establecidos para las etapas representadas en la figura 1. A continuación presentamos los resultados obtenidos en cuatro casos de estudio.
3.1Modelado del ventrículo izquierdoLas IRM del ventrículo izquierdo se caracterizan porque la fuerza del gradiente en el endocardio es por lo general diferente a la del epicardio. Asimismo, el miocardio es fuertemente influenciado por inhomogeneidades en escala de grises responsables de los cambios locales en la media y la varianza de los tejidos. Considerando estas peculiaridades, es necesario utilizar más de una sola técnica en las etapas de preprocesado y segmentación. El flujograma de algoritmos utilizado es presentado en la figura 14. Las técnicas son detalladas a continuación.
3.1.1PreprocesoPara conseguir reducir el ruido de las imágenes sin atenuar las zonas de interés y resaltar adecuadamente las estructuras que conforman el corazón se necesitaba de una técnica de filtrado con preservación de bordes. El filtro de difusión anisotrópica comentado en el apartado 2.2.1 resultó ser ideal para este tipo de tareas. Como siguiente paso, se aplicó el cálculo del módulo gradiente sobre la imagen filtrada, lo cual permitió diferenciar de mejor manera los contornos de las estructuras del corazón.
3.1.2SegmentaciónSegmentar el volumen del ventrículo izquierdo puede resultar una tarea no tan complicada si se combinan técnicas de segmentación adecuadas para extraer estructuras grandes y bien definidas. En este caso, se obtuvieron mejores resultados al combinar dos técnicas de segmentación muy utilizadas en este tipo de tareas. En primer lugar se aplicó el algoritmo Watershed en la imagen gradiente, obteniéndose una imagen en escala de gris con las regiones uniformes etiquetadas por una intensidad de gris diferente para cada segmento obtenido. Watershed tiene la desventaja de producir sobre-segmentación si es aplicado sobre imágenes ruidosas y niveles de gris poco uniformes, sin embargo, esta dificultad fue superada al aplicar esta técnica sobre la imagen preprocesada. Posteriormente se utilizó la técnica de umbralización para determinar el umbral (o umbrales) que conforman la zona del ventrículo izquierdo, entre el conjunto de segmentados etiquetados.
3.1.3Remuestreo y exportación a CADEn el siguiente paso, se realizó el remuestreo del modelo geométrico segmentado empleando dilatación morfológica con un elemento estructural en forma de esfera de radio 3 × 3 × 3. Esta tarea fue necesaria para suavizar superficies superpuestas y rellenar posibles agujeros generados durante la segmentación. Este modelo fue guardado en formatos legibles por software de visualización y herramientas CAD como GiD, ParaView, Autodesk Inventor y Abaqus.
3.1.4Pruebas de discretizaciónFinalmente, empleando estas herramientas, se aplicaron condiciones de contorno de prueba en zonas aleatorias del modelo, verificándose la utilidad del modelo geométrico para su discretización con el método de elementos finitos.
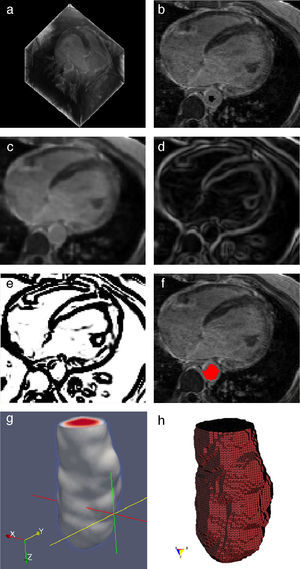
En la figura 15 se presentan los resultados obtenidos por cada etapa de procesamiento en IRM cardiovascular. Las imágenes médicas utilizadas tienen formato DICOM [39], con 59 cortes de tamaño 192 × 192 pixels, voxel spacing: 1,5625 × 1,5625 × 2,5mm. Por efectos de visualización, solamente se presenta uno de los cortes axiales de la imagen 3D.
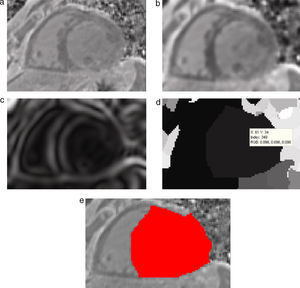
Preproceso y segmentación el volumen del ventrículo izquierdo. (a) Corte axial de la IRM cardiovascular original. (b) Imagen (a) filtrada con difusión anisotrópica. (c) Imagen gradiente obtenida a partir de (b). (d) Imagen Watershed con segmentos etiquetados obtenida a partir de (c). (e) Selección del segmento del ventrículo izquierdo empleando umbralización.
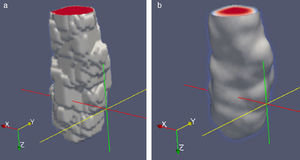
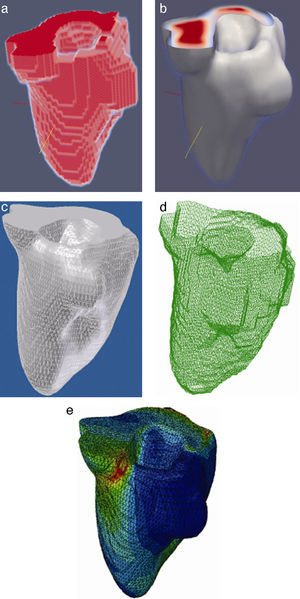
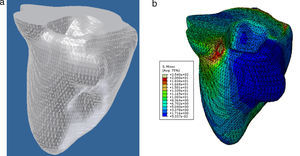
En la figura 16 se presenta el volumen final del ventrículo izquierdo con el inicio de la válvula aórtica (fig. 16.a), el modelo final suavizado visualizado en ParaView (fig. 16.b), el modelo en sólido visualizado en Autodesk Inventor (fig. 16.c), el modelo en malla visualizado en GiD (fig. 16.d) y el modelo discreto con los elementos finitos realizado con Abaqus (fig. 16.e). Estos modelos han sido obtenidos a partir de IRM cardiovascular de un paciente con cardiopatía isquémica. Obsérvese que la protuberancia presente en la zona superior derecha del ventrículo izquierdo constituye una zona de necrosis conocida como cicatriz isquémica, la cual es alojada en el músculo del miocardio.
Vista tridimensional de ventrículo izquierdo. (a) Volumen original visualizado con ParaView. (b) Volumen original suavizado con morfología matemática visualizado con ParaView. (c) Sólido del volumen generado con Autodesk Inventor. (d) Mallado del volumen generado con GiD. (e) Modelo discreto con el método de elementos finitos generado con Abaqus empleando condiciones de contorno de prueba.
Para determinar las técnicas adecuadas en la obtención del modelo de la aorta descendente se consideró una de sus principales características anatómicas: la estructura tubular de la aorta tiene una sección transversal circular. Utilizando este conocimiento a priori nos permitió establecer que la técnica de segmentación adecuada sería la técnica Level Set debido a que el algoritmo utiliza la deformación de semillas esféricas (snakes) que se pueden ir controlando adecuadamente hasta obtener la forma de la estructura que se desea segmentar, obteniéndose buenos resultados en estructuras con bordes redondeados. Asimismo, conocemos que para el eficaz funcionamiento del algoritmo Level Set es necesario realizar un preproceso previo en las imágenes originales que corrija artefactos y ruido presentes en ella y fortalezca los contornos de la aorta con la finalidad de garantizar que el crecimiento de los snakes no se desborde.
En la figura 17 es presentado el flujograma de algoritmos utilizados para obtener el modelo geométrico de la aorta ascendente.
3.2.1PreprocesoEl ruido de las imágenes fue filtrado empleando el algoritmo de difusión anisotrópica, el cual atenúa el ruido preservando los bordes. Con el fin de mejorar la diferenciación de la aorta del resto de tejidos (ventrículos), se aplicó el cálculo del módulo del gradiente sobre la imagen filtrada, asimismo, a la imagen resultante se le aplicó el filtro sigmoid. Ambos filtros reforzaron de manera óptima el contorno de la aorta.
3.2.2SegmentaciónSe aplicó el algoritmo Level Set sobre la imagen sigmoid obtenida en la etapa anterior. Para ello se implantó un snake de forma esférica de 2 pixels de radio. El resultado fue un mapa de imagen en escala de grises con la región de la aorta mapeada a valores de gris oscilando alrededor del valor 0. Para extraer el conjunto Zero Level que constituye la zona de interés, se empleó la técnica de umbralización, definiéndose los umbrales inferior y superior que permitían extraer la zona aórtica.
3.2.3Remuestreo y exportación a CADPara mejorar el modelo geométrico inicial obtenido de la segmentación, se realizó el remuestreo a través de la técnica dilatación morfológica con un elemento estructural en forma de esfera de radio 3 × 3 × 3, lo cual fue realizado con el fin de suavizar superficies superpuestas y rellenar posibles agujeros generados al utilizar la técnica de umbralización. El modelo geométrico fue guardado en formatos legibles por software de visualización y herramientas CAD como GiD, Paraview, Autodesk Inventor y Abaqus.
En la figura 18 se presentan los resultados obtenidos por cada etapa en imágenes médicas de IRM cardiovascular en formato DICOM (38), con 72 cortes de tamaño 192 × 192 pixels, voxel spacing: 1,5625 × 1,5625 × 2,5mm. En las figuras, se presenta uno de los cortes axiales utilizados.
Preproceso y segmentación para el modelado geométrico de la aorta descendente (a) Vista 3D original de imágenes originales de IRM cardiovascular. (b) Vista de un corte axial de (a) con la implantación de un snake en la zona de la aorta. (c) Filtrado del ruido de (b) con la técnica de difusión anisotrópica. (d) Imagen módulo gradiente de (c). (e) Imagen sigmoid de (d). (f) Segmentación de aorta descendente empleando Level Set. (g) Vista tridimensional en ParaView de la aorta descendente segmentada. (h) Vista de la malla en GiD del volumen de la aorta descendente.
El cerebro, como todas las partes del sistema nervioso central contiene una sustancia blanca y una sustancia gris, siendo la última la de menor cantidad. La segmentación de estas estructuras permite realizar un análisis cuantitativo morfométrico necesario para el diagnóstico de distintas patologías y en la evaluación de la respuesta a un determinado tratamiento. Sin embargo, esta tarea se ve afectada por la presencia de distintos tejidos con niveles de gris similares, la ausencia de valores constantes en los niveles de gris. Asimismo, las imágenes médicas del cerebro como las IRM y las PET, entre otras, presentan ruido característico asociado a este tipo de imagenología.
La técnica de segmentación Region Growing puede resultar útil para extraer este tipo de sustancias cerebrales debido a la flexibilidad del método para indicar pequeñas zonas iniciales en los tejidos que se desean segmentar a través de la selección de semillas, asimismo, es posible controlar y restringir las zonas que se van sumando en cada interacción del algoritmo, estableciendo características más sofisticadas como la combinación de la media, la desviación estándar, la entropía, la correlación, entre otros clasificadores estadísticos.
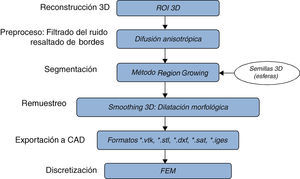
En este caso de estudio seleccionado, el volumen de la zona de la materia blanca fue obtenido aplicando el flujograma de algoritmos presentado en la figura 19.
3.3.1PreprocesoEl ruido de las imágenes fue filtrado empleando el algoritmo de difusión anisotrópica, suavizando así el ruido y preservando los bordes de la imagen. De esta manera se consiguió mantener los contornos que dividen la materia blanca de los demás tejidos y uniformizar sus valores de gris.
3.3.2SegmentaciónSe aplicó el algoritmo Region Growing sobre la imagen filtrada, colocando cuatro esferas (semillas) en la zona de interés, buscando ubicarlas en los extremos más arraigados de este tejido. La condición de inclusión utilizada para hacer crecer la región fue la descrita en la ecuación (10), en base a la media y la desviación estándar de los voxels vecinos. El volumen resultante fue una imagen binaria con la zona de la materia blanca coloreada en valores de 255 (blanco).
3.3.3Remuestreo y exportación a CADPara mejorar el modelo geométrico inicial, se realizó el remuestreo del volumen a través de dilatación morfológica con un elemento estructural en forma de esfera de radio 3 × 3 × 3 a lo largo de todo el tejido segmentado. Este paso es necesario para suavizar superficies superpuestas y rellenar los agujeros generados durante la segmentación debido a la sensibilidad de la condición de segmentación. El modelo geométrico final fue guardado en formatos legibles por software de visualización y herramientas CAD como GiD, ParaView, Autodesk Inventor y Abaqus.
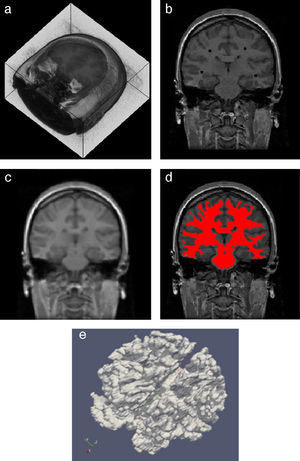
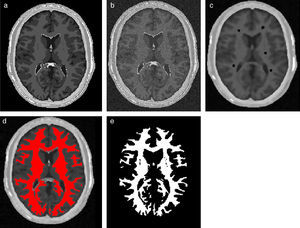
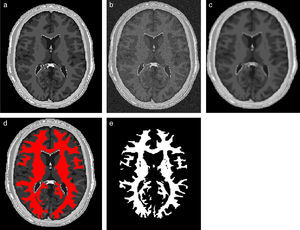
En la figura 20 se presentan los resultados obtenidos por cada etapa en imágenes médicas de IRM del cerebro en formato DICOM, 60 slices, tamaño de 256 × 256 pixels, voxel spacing: 0,86 × 0,86mm × 3,0mm. Por efectos de visualización, solamente se presenta uno de los cortes axiales utilizados. Obsérvese en la figura 20.b la selección de cuatro semillas sobre la zona de interés, las cuales fueron asignadas de manera arbitraria. El éxito de la segmentación dependerá del lugar donde se coloquen estas semillas.
Segmentación de materia blanca empleando Region Growing en IRM del cerebro. (a) Volumen de IRM cerebral original. (b) Vista de un corte axial con la lección de cuatro semillas iniciales. (c) Imagen (b) filtrada con difusión anisotrópica. (d) Vista del corte axial (b) con la materia blanca segmentada a través de Region Growing. (e). Vista volumétrica de la materia blanca segmentada en (d).
En imágenes de TC, las estructuras óseas son representadas con niveles de gris más altos comparado con los otros tipos de tejidos presentes en estas imágenes, debido a esta característica resulta útil aplicar técnicas de umbralización para separar este tipo de tejidos óseos de los demás. Sin embargo, el uso de umbralización puede tener la desventaja de generar pequeñas zonas desconectadas dentro de la misma estructura o fuera de ésta y a la vez generar zonas superpuestas en las superficies de los huesos, por los cuales se hace necesario aplicar técnicas de filtrado y suavizado de ruido antes de segmentar, así como realizar un remuestreo de los modelos segmentados aplicando técnicas de dilatación morfológica para rellenar los agujeros presentes y suavizar capas externas de los modelos óseos.
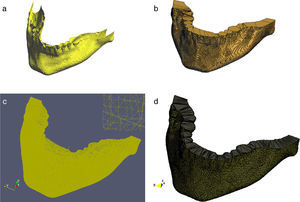
Para verificar la correcta combinación de estas técnicas y su utilidad, éstas fueron aplicadas en imágenes de TC del cerebro para obtener el modelo geométrico del hueso cráneo-facial, el flujograma de algoritmos utilizado es presentado en la figura 21. Cada proceso es descrito a continuación.
3.4.1PreprocesoPara la reducción del ruido de las imágenes de TC se aplicó el filtro de difusión anisotrópica, asimismo, se consiguió uniformizar los niveles de gris de los tejidos en la imagen.
3.4.2SegmentaciónPara segmentar el hueso del cráneo se utilizó la técnica de umbralización. Se observó el histograma global de la imagen y se seleccionó un valor umbral que separara el tejido óseo de los demás tejidos, obteniéndose una imagen binaria. Orientados por la escala de Hounsfield[1], en imágenes de TC correctamente calibradas, los valores del hueso compacto son superiores a 1.000.
3.4.3Remuestreo y exportación a CADPara suavizar las superficies y rellenar los agujeros generados por la técnica de segmentación empleada se aplicó dilatación morfológica con un elemento estructural esférico de radio 3 × 3 × 3. Este modelo fue guardado en formatos legibles por software de visualización y herramientas CAD como GiD, ParaView, Autodesk Inventor y Abaqus.
En la figura 22 se presenta la segmentación del cráneo en imágenes de TC en formato DICOM, 256 slices, tamaño de corte de 512 × 512 pixels, voxel spacing: 0,98 × 0,98 × 1,0mm. Para obtener un valor umbral que distinga el tejido óseo de los demás tejidos, se analizó el histograma global de la imagen, donde claramente se observa que el tejido óseo posee los niveles de gris más altos. En este caso de estudio en espacial, los voxels de la imagen de entrada menores a un umbral de valor 1.266 fueron convertidos a negro, y los voxels con valores mayores al umbral fueron convertidos a blanco. De este modo, se obtuvo un volumen binario del hueso craneal.
El modelo geométrico del cráneo obtenido con la metodología es presentado en la figura 23. Las vistas presentadas en la figura han sido generadas empleando ParaView.
4Validación de técnicas empleando descriptores estadísticosPara validar la funcionalidad y confiabilidad de la metodología propuesta fue necesario comparar nuestros modelos obtenidos con otros los modelos anatómicos proporcionados por otras fuentes a partir de las mismas imágenes médicas. Se utilizó el phantom del cerebro proporcionado por la base de datos libre BrainWeb[15] que simula IRM del cerebro a través de volúmenes «fuzzy», donde se representa de manera discreta cada clase de tejido: Materia Blanca (MB), Materia Gris (MG), Líquido Cefalorraquídeo (CSF), Grasa (G), etc. y volúmenes anatómicos discretos globales con cada clase conformada por voxels etiquetados con valores enteros (0=Fondo, 1=CSF, 2=Materia Gris, 3=Materia Blanca, 4=Grasa, 5=Músculo/Piel, 6=Piel, 7=Cráneo, 8=Materia Glial, 9=Tejido Conectivo).
El proceso de validación consistió en comparar dos volúmenes obtenidos a partir de IRM proporcionadas por esta base de datos: (a) un volumen segmentado de la zona de la materia blanca proporcionado por BrainWeb, y (b) el volumen de la materia blanca obtenido del phantom completo, empleando las rutinas de preprocesamiento y segmentación mencionadas a lo largo de este trabajo. Para ambos volúmenes, se calcularon el número total de pixels y los descriptores estadísticos descritos en la sección 2.6 (la media, desviación estándar, asimetría, homogeneidad y entropía). Calculándose finalmente el porcentaje de error absoluto de cada descriptor, entre los dos volúmenes evaluados.
Se aplicaron tres casos de validación, comparando el volumen de la zona de la materia blanca proporcionada por BrainWeb con: (a) El volumen de la zona de la materia blanca obtenida del phantom empleando la rutina de Region Growing. (b) El volumen obtenido empleando la rutina de Watershed. (c) El volumen obtenido con Region Growing aplicado al phantom corrompido con ruido gaussiano y filtrado con difusión anisotrópica. A continuación se describen los tres casos de validación aplicados.
4.1Caso 1: segmentación Region Growing - BrainWebCon el interés de segmentar la zona de la materia blanca, se utilizó el algoritmo de Region Growing en el phantom discreto completo original. La zona segmentada fue comparada con la zona de la materia blanca proporcionada por BrainWeb. Para este fin, se empleó el análisis de texturas con el cálculo de descriptores estadísticos en ambos volúmenes y los respectivos porcentajes de error entre ambos. En la figura 24 se presenta los resultados obtenidos al segmentar la zona de la materia blanca en el volumen phantom con dimensiones de 181 × 217 × 181 (X × Y × Z), con voxels isotrópicos de 1,0mm3, por visualización se presenta el corte axial 98. En la figura 24.a se presenta la imagen phantom original, mostrando el corte número 98 del phantom. En la figura 24.b se presenta la zona segmentada empleando el algoritmo Region Growing con seis seed points (semillas) elegidos de manera arbitraria sobre el área de la materia blanca, en forma de esferas volumétricas de 2mm de radio, con el centro en las coordenadas X,Y,Z: Seed1=(66,59,98), Seed2=(67,101,98), Seed3=(60,158,98), Seed4=(112,55,98), Seed5=(113,103,98) y Seed6=(127,149,58). En la figura 24.c se presenta la zona de la materia blanca proporcionada por BrainWeb.
En la tabla 1 se presentan los valores estadísticos y los respectivos porcentajes de error, donde se puede observar que el porcentaje de error de las zonas segmentadas por Region Growing y la zona de la materia blanca proporcionada por BrainWeb no supera el 0,2487% para el caso del número global de pixels y el 0,2909% para los descriptores estadísticos.
Validación del volumen de la zona de la materia blanca empleando Region Growing utilizando análisis de texturas estadísticos
| Nro. pixels | Media | Desviación estándar | Asimetría | Homogeneidad | Entropía | |
| Region Growing | 682.820 | 0,0947 | 0,2928 | 2,7687 | 8,6655 | 0,4519 |
| Phantom BrainWeb | 674.777 | 0,0949 | 0,2931 | 2,7641 | 8,6404 | 0,4527 |
| %error RegionGrowing-phantom | 0,2487 | 0,2407 | 0,1078 | 0,1644 | 0,2909 | 0,1643 |
Al igual que el caso anterior, el esquema de segmentación Watershed fue validado empleando el phantom de IRM del cerebro. En la figura 25 se presentan los resultados obtenidos al segmentar la zona de la materia blanca en el volumen phantom con dimensiones de 181 × 217 × 181 (X × Y × Z), con voxels isotrópicos de 1,0mm3. En la figura 25.a se presenta el corte número 98 de la imagen original. En la figura 25.b se presenta la segmentación obtenida empleando el algoritmo de Watershed. En la figura 25.c se presenta la zona de la materia blanca proporcionada por BrainWeb.
Para validar los resultados, se empleó el análisis de texturas, calculando los descriptores estadísticos en los volúmenes obtenidos. En la tabla 2 se presentan los valores estadísticos y los respectivos porcentajes de error para ambos volúmenes. Obsérvese que el porcentaje de error del número global de pixels no supera el 1,2418%, y para los descriptores estadísticos no supera el 1,5201%.
Validación del volumen de materia blanca obtenido con Watershed
| Nro. pixels | Media | Desviación estándar | Asimetría | Homogeneidad | Entropía | |
| Watershed | 683.262 | 0,0961 | 0,2947 | 2,7406 | 8,5110 | 0,4565 |
| Phantom BrainWeb | 674.777 | 0,0949 | 0,2931 | 2,7641 | 8,6404 | 0,4527 |
| %error Watershed-phantom | 1,2418 | 1,2418 | 0,5573 | 0,8576 | 1,5201 | 0,8479 |
En la figura 26 son presentados las vistas tridimensional del volumen de la zona de la materia blanca proporcionado por BrainWeb, el volumen obtenido con Region Growing y volumen obtenido con Watershed.
4.3Caso 3: filtrado de difusión anisotrópica y segmentación Region Growing - BrainWebEn este caso, se corrompió el volumen phantom con ruido aditivo gaussiano y se procedió a aplicar las rutinas de filtrado y segmentación. Para suavizar el ruido de la imagen, se aplicó la rutina de filtrado con difusión anisotrópica (sección 2.2.1) y la segmentación de la zona de la materia blanca fue realizada con la rutina Region growing (sección 2.3.2). El flujograma de técnicas empleadas es similar al presentado en la figura 21.
En la figura 27 se presentan los resultados obtenidos al segmentar la zona de la materia blanca con las rutinas y mencionadas. En la figura 27.a se presenta la imagen phantom original, mostrando el corte axial número 98 del phantom. En la figura 27.b se presenta la imagen phantom con ruido aditivo gaussiano. En la figura 27.c es mostrada la imagen resultante luego de filtrar (b) con el filtro de difusión anisotrópica, además se observan las 5 semillas (seed points) seleccionadas de manera arbitraria sobre el área de la materia blanca. Las semillas empleadas tenían forma esférica de 2 pixels de radio, con el centro en las coordenadas X,Y,Z, las coordenadas de las semillas son: Seed1=(65,59,98), Seed2=(112,55,98), Seed3=(117,104,98), Seed4=(127,137,98), Seed5=(55,128,98). En la figura 27.d se presenta el resultado de la segmentación (en rojo). En la figura 27.e se presenta la zona de la materia blanca proporcionada por BrainWeb.
Materia blanca segmentada en volumen phantom. (a) Corte axial número 98 de imagen de phantom original. (b) Imagen original con ruido gaussiano agregado (c) Imagen con ruido filtrada con filtro de difusión anisotrópica. (d) Materia blanca segmentada con algoritmo Region Growing con 5 semillas esféricas. (e) Zona de la materia blanca segmentada por BrainWeb.
Se calcularon los valores estadísticos y los respectivos porcentajes de error entre el volumen segmentado y el volumen proporcionado por BrainWeb, los resultados son presentados en la tabla 3. Obsérvese que el porcentaje de error del número de pixels global no supera el 3,6374%, y para los descriptores estadísticos no supera el 7,1608%.
Validación del volumen de materia blanca obtenido con Region Growing luego de agregar ruido gaussiano en phantom de IRM del cerebro y aplicar el filtro de difusión anisotrópica
| Nro. pixels | Media | Desviación estándar | Asimetría | Homogeneidad | Entropía | |
| Difusión anisotrópica-Region Growing | 650.232 | 0,0899 | 0,2910 | 2,8739 | 9,2591 | 0,4351 |
| Phantom BrainWeb | 674.777 | 0,0949 | 0,2931 | 2,7641 | 8,6404 | 0,4527 |
| %error Region Growing-phantom | 3,6374 | 5,2687 | 0,2100 | 3,9702 | 7,1608 | 3,8745 |
En este caso, se utilizó el volumen phantom corrompido con ruido aditivo gaussiano, al cual se le aplicó la rutina de filtrado con difusión anisotrópica (sección 2.2.1) y la rutina de segmentación Region Growing (sección 2.3.2). El flujograma de técnicas empleadas es similar al presentado en la figura 14.
En la figura 28 se presentan los resultados obtenidos al aplicar las rutinas mencionadas. En la figura 28.a se presenta el corte 98 de la imagen de phantom original. En la figura 28.b se presenta un corte de la imagen de phantom corrompida con ruido gaussiano. En la figura 28.c se presenta la imagen filtrada con difusión anisotrópica. En la figura 28.d se presenta la segmentación obtenida empleando la rutina Watershed. En la figura 28.e se presenta la zona de la materia blanca proporciona por BrainWeb.
Materia blanca segmentada en volumen phantom. (a) Corte axial 98 de imagen de phantom original. (b) Imagen de phantom corrompida con ruido gaussiano. (c) Imagen (b) filtrada con difusión anisotrópica. (d) Materia blanca segmentada con rutina Watershed. (e) Zona de la materia blanca segmentada por BrainWeb.
En la tabla 4 se presentan los valores estadísticos y los respectivos porcentajes de error entre ambos volúmenes, donde se observa que el porcentaje de error del número global de pixels no supera el 9,5849%, y el porcentaje de error de los descriptores estadísticos no superan el 12,7991%.
Validación del volumen de materia blanca obtenido con Watershed
| Nro. pixels | Media | Desviación estándar | Asimetría | Homogeneidad | Entropía | |
| Watershed | 610.100 | 0,0858 | 0,2801 | 2,9574 | 9,7463 | 0,4224 |
| Phantom BrainWeb | 674.777 | 0,0949 | 0,2931 | 2,7641 | 8,6404 | 0,4527 |
| %error Watershed-phantom | 9,5849 | 9,5890 | 4,4353 | 6,9932 | 12,7991 | 6,6931 |
Los porcentajes de error obtenidos en los cuatro casos de validación aplicados fueron mínimos. En el caso 1, validando el volumen de la zona de la materia blanca obtenido con Region Growing con el volumen proporcionado por BrainWeb, el porcentaje de error del número de pixels no superó el 0,2487%, y los porcentajes de error de los descriptores estadísticos no superaron el 0,2909%. En el caso 2, al aplicar la técnica de segmentación Watershed, el porcentaje de error del número total de pixels no supero el 1,2418% y los porcentajes obtenidos para los descriptores estadísticos no superaron el 1,5201%.
Por otro lado, en los casos 3 y 4, corrompiendo las imágenes con ruido gaussiano, lo valores obtenidos también fueron bajos. En el caso 3, empleando el filtro de difusión anisotrópica y Region Growing, el porcentaje de error del número de pixels fue de 3,6374, y con respecto a los descriptores estadísticos, el máximo porcentaje de error alcanzado fue el de homogeneidad con un valor igual a 7,1608. En el caso 4, empleando el filtro de difusión anisotrópica y Watershed, el porcentaje de error del número de pixels fue de 9,5849%, y para los descriptores estadísticos, el máximo valor obtenido fue para la homogeneidad con un valor de 12,7991%.
El resumen de los porcentajes de error obtenidos en los cuatro casos de validación es presentado en la tabla 5.
Porcentajes de error obtenidos en la validación de los modelos geométricos
| % error Nro. pixels | % error Media | % error Desviación estándar | % error Asimetría | % error Homogen. | % error Entropía | |
| Phantom original | ||||||
| Caso 1: Region Growing-phantom | 0,2487 | 0,2407 | 0,1078 | 0,1644 | 0,2909 | 0,1643 |
| Caso 2: Watershed-phantom | 1,2418 | 1,2418 | 0,5573 | 0,8576 | 1,5201 | 0,8479 |
| Phantom corrompido con ruido | ||||||
| Caso 3: Difusión anisotrópica + Region Growing-phantom | 3,6374 | 5,2687 | 0,2100 | 3,9702 | 7,1608 | 3,8745 |
| Caso 4: Difusión anisotrópica + Watershed-phantom | 9,5849 | 9,5890 | 4,4353 | 6,9932 | 12,7991 | 6,6931 |
La última etapa de la metodología consistió en exportar los modelos de tejidos en formatos legibles por herramientas CAD (fig. 1) para ser analizados de manera cualitativa y cuantitativa. Estos modelos iniciales son exportados en formato *.vtk y guardan información de los niveles de gris, el tamaño y la posición de cada voxel que conforma el volumen, sin embargo, dependiendo de su utilización, será necesario realizar un posprocesamiento.
En la figura 29, se presenta un esquema resumido de los pasos realizados para el procesamiento de los modelos geométricos iniciales hasta obtener los modelos discretos con mallas, todas estas tareas fueron realizadas en los ambientes de herramientas libres y comerciales como ParaView, GiD, Autodesk Inventor y Abaqus.
Los volúmenes en formato *.vtk fueron importados desde las herramientas ParaView y GiD. Estos modelos fueron corregidos y simplificados con técnicas de suavizado, eliminación de elementos desconectados y cierre de superficies abiertas en los modelos. El proceso consistió en extraer los contornos, cuyas poli-líneas son convertidas en curvas suaves (splines), de esta manera se obtiene el modelo wireframe (modelo de alambre). Para que las superficies de los tejidos blandos y duros se suavicen, es necesario reducir el número de puntos de control de los splines.
5.2Generación de modelos sólidosEl siguiente paso consistió en convertir el modelo wireframe a sólidos a fin de poder manejar de mejor manera las estructuras para el siguiente paso, el cual es la generación de mallas. Para este fin, se utilizaron los programas CAD para crear secciones transversales planas a partir de las curvas. Finalmente, las superficies son creadas a partir de estos contornos cerrados y los sólidos son obtenidos aplicando la técnica de extrusión.
5.3Obtención de mallasRealizar el análisis numérico en tejido vivo implica aplicar condiciones de contorno en modelos de mallas y la elección de tipo de malla a utilizar dependerá del método numérico que se desee aplicar. En este trabajo, se utilizó el modelo sólido para generar mallas empleando elementos tetraedros, estas tareas fueron realizadas bajo las plataformas de tres herramientas CAD diferentes: GiD, Autodesk Inventor y Abaqus. Se empleó el método de los elementos finitos (FEM), el cual es una técnica numérica muy utilizada para el análisis del stress/strain en sistemas biológicos [4–7,40,41]. Se consideraron valores de prueba en las propiedades mecánicas de los tejidos para determinar tensiones y deformaciones, de este modo verificamos que los modelos obtenidos son útiles para ser analizados con los métodos numéricos.
Por otro lado, se generaron mallas superficiales, empleando iso-superficies triangulares. Este tipo de mallas son adecuadas cuando se desea hacer el análisis y la simulación con el método de los elementos de contorno (BEM).
A continuación se presentan los modelos de mallas obtenidos para diferentes tipos de tejidos. En la figura 30 se presenta el modelo sólido y el modelo discreto del ventrículo izquierdo. En la figura 30.b se presenta un análisis del modelo con el método de los elementos finitos (FEM), empleando valores de prueba para las condiciones asignadas.
En la figura 31 se presenta el modelo de malla en tetraedros de una sección de la aorta descendente. Este modelo fue generado en GiD, en donde se corrigieron previamente las superficies, cerrando caras abiertas y simplificando el número de nodos.
En la figura 32 se presenta los modelos obtenidos del hueso mandibular, el cual fue extraído a partir del hueso craneal. En la figura 32.a se observa la superficie inicial abierta en la parte superior a la altura de los dientes. En la figura 32.b se presenta el modelo anterior corregido con todas las superficies cerradas, lo cual fue realizado en GiD. En la figura 32.c se observa el modelo wireframe visualizado en el software ParaView. En la figura 32.d se observa el modelo en malla con elementos tetraédricos, generado en GiD.
Generación de malla de una sección del hueso mandibular. (a) Iso-superficie del hueso mandibular extraído del hueso del cráneo presentado en la figura 23. (b) Corrección de (a) en GiD. (c) Modelo wireframe visualizado en Paraview. (d) Malla de tetraedros generado en GiD.
Las rutinas de preproceso, segmentación y visualización empleadas en este trabajo permitieron obtener modelos geométricos precisos, confiables, con tiempos de procesamiento cortos, a partir de imágenes médicas, las cuales fueron integradas en una herramienta de software para su fácil interacción con el usuario. A continuación, se presentan las conclusiones derivadas de los resultados presentados en este trabajo, así como líneas de investigación propuestas en esta línea:
6.1Aporte metodológicoEl principal aporte de este trabajo consistió en proponer e implementar una metodología flexible, eficiente y versátil para la obtención de modelos 3D a partir de imágenes médicas desde la lectura y reconstrucción 3D inicial de las imágenes hasta la generación de mallas de los modelos que puedan ser utilizados en herramientas CAD. El planteamiento de esta metodología consistió en combinar técnicas de procesamiento de imágenes existentes, adecuarlas y determinar el orden de aplicación de las mismas en función del tipo de tejido a reconstruir.
6.2Precisión en los volúmenes obtenidosLa precisión y la confiabilidad de los resultados obtenidos con las rutinas implementadas quedaron garantizadas al validar los modelos obtenidos con otros modelos proporcionados (phantom) por sitios Web, mediante cuatro casos de estudio, en los cuales se obtuvieron porcentajes de error mínimos con respecto al número de pixels global de los volúmenes y el cálculo de descriptores estadísticos. Los resultados fueron analizados en la sección 4.5.
6.3Tiempos de ejecuciónLa ejecución secuencial de las técnicas de preproceso, segmentación y visualización propuestas, siguiendo los flujogramas propuestos, permitieron obtener los modelos geométricos de tejidos blandos y duros en tiempos de ejecución cortos. En la tabla 6, se presentan los tiempos empleados para obtener los cinco modelos geométricos de los casos de estudio. El computador empleado fue un desktop de 64 bits, con 2 procesadores (Core 2 Quad), de velocidades 2,66GHz cada uno fueron y memoria RAM de 8 GB.
Tiempos de ejecución obtenidos en las etapas de preproceso y segmentación para la obtención de los modelos geométricos de los casos de estudios analizados
| Ventrículo izquierdo | Aorta descendente | Materia blanca | Hueso del cráneo | Hueso mandibular | |
| T1 (segundos) | 4,90 | 7,38 | 2,68 | 0,03 | 40,42 |
| T2 (segundos) | 5,09 | 5,59 | 2,66 | 0,05 | 39,35 |
| T3 (segundos) | 5,33 | 5,52 | 2,65 | 0,05 | 40,65 |
| T4 (segundos) | 5,24 | 5,54 | 2,69 | 0,05 | 40,92 |
| T5 (segundos) | 5,08 | 5,59 | 2,66 | 0,03 | 40,98 |
Asimismo, una de las principales ventajas de la metodología propuesta es que incluye un conjunto de algoritmos de preproceso y segmentación que pueden ser combinados para formar técnicas híbridas que se adecuen a las estructuras anatómicas bajo estudio y se formulen nuevos flujogramas. Los parámetros de entrada de los algoritmos implementados pueden ser fácilmente calibrados, según la opinión de los expertos.
6.5Utilidad de los modelos en otras herramientas de visualización y CADPor último, se comprobó que las técnicas implementadas permiten generar y exportar volúmenes en formatos *.vtk y *.stl, fácilmente legibles desde otros programas y herramientas CAD, verificándose su utilidad para generar diferentes vistas como mallado, superficies y sólidos. Asimismo, en el entorno de las herramientas CAD se aplicaron valores de prueba en las condiciones de contorno y se consiguieron discretizar los modelos con el método de los elementos finitos, quedando demostrado que los volúmenes generados son útiles para su análisis numérico.
El desarrollo de este trabajo y su publicación ha sido posible gracias al financiamiento y al apoyo institucional del Centro Internacional de Métodos Numéricos en Ingeniería (CIMNE, España), el Instituto Nacional de Bioingeniería (INABIO, Venezuela) y la Universidad Central de Venezuela. Sin la suma de esfuerzos y conocimientos de sus grupos de investigadores multidisciplinarios no hubiera sido factible el alcance de los objetivos planteados en esta investigación.




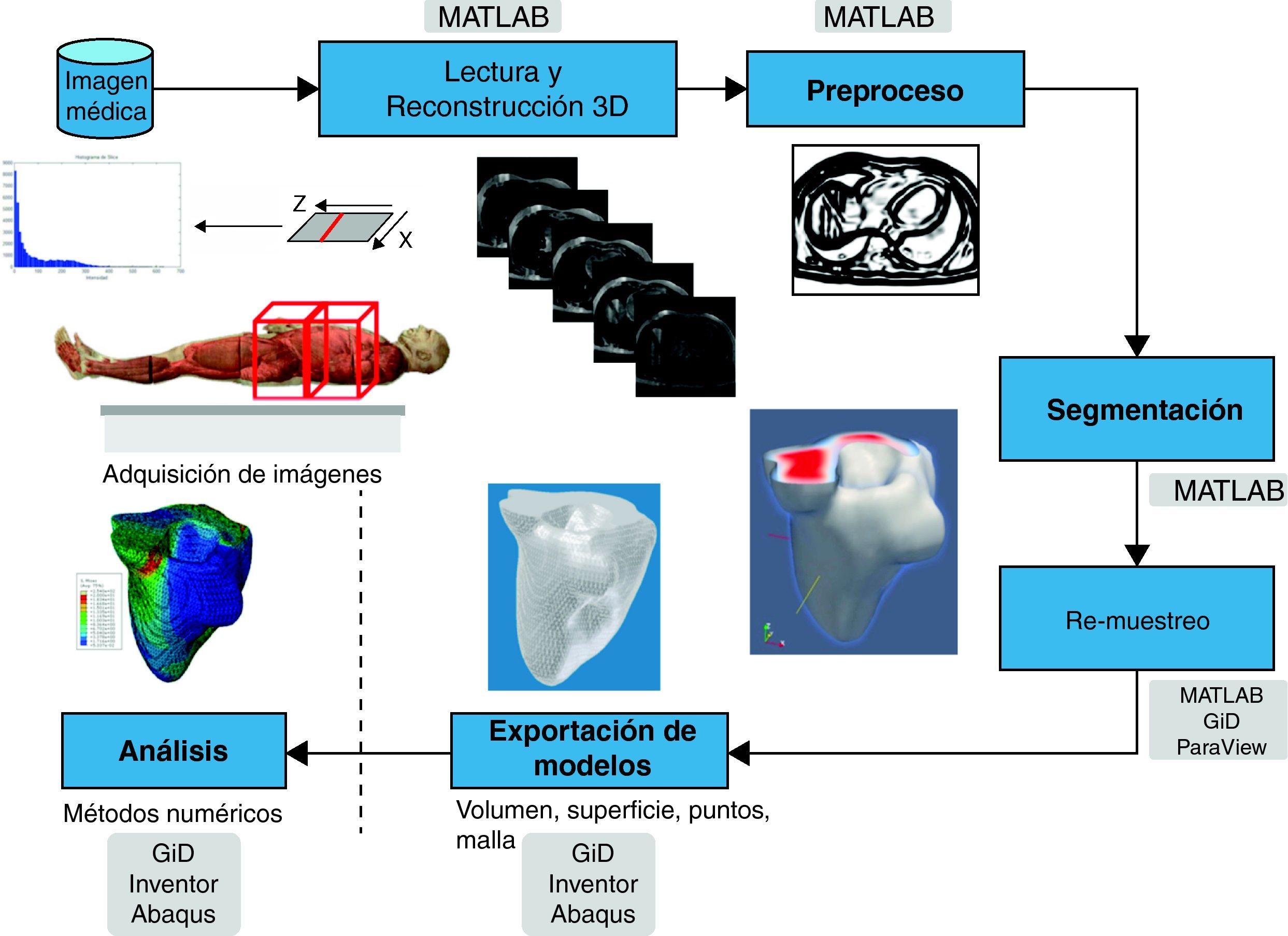
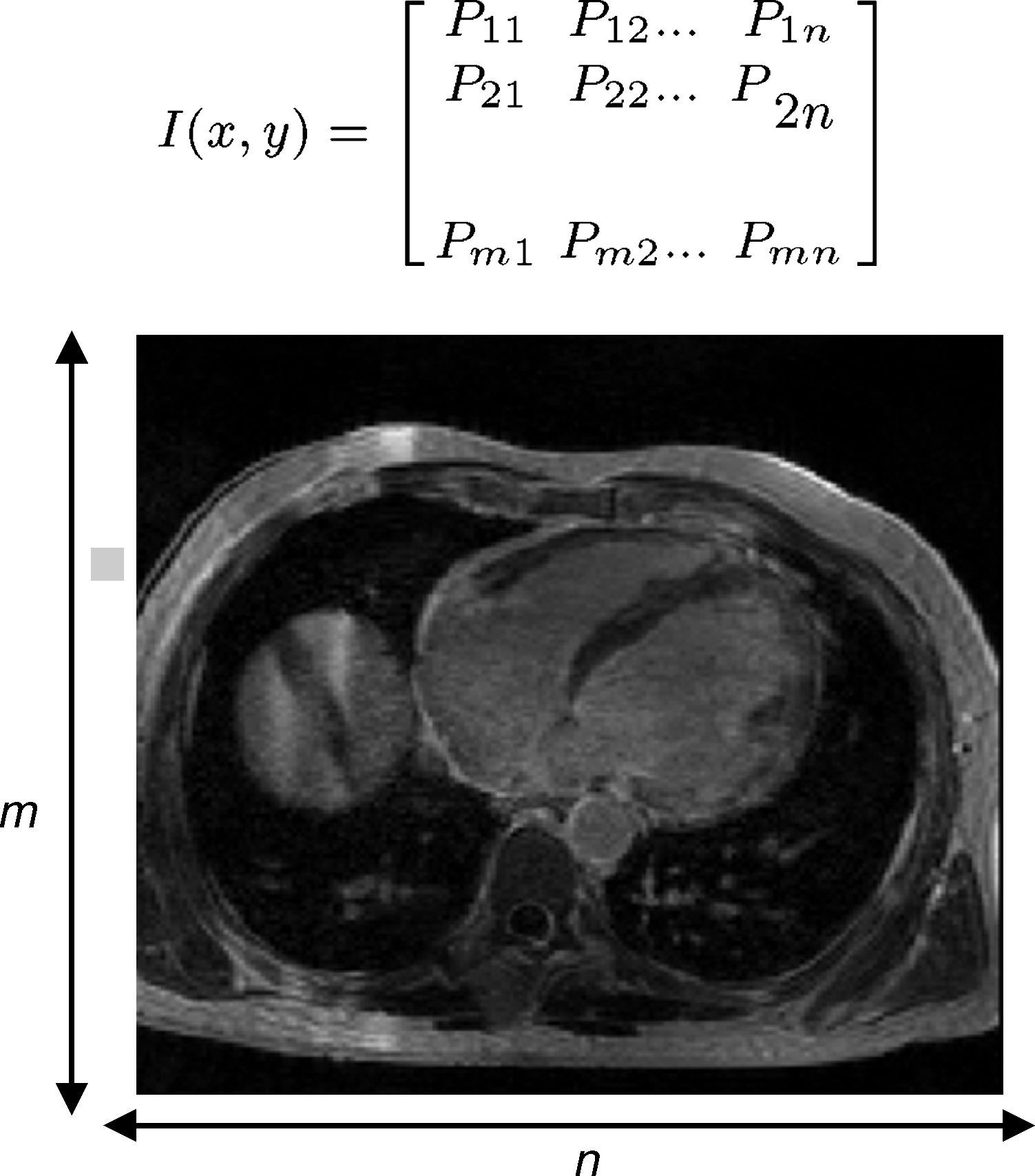
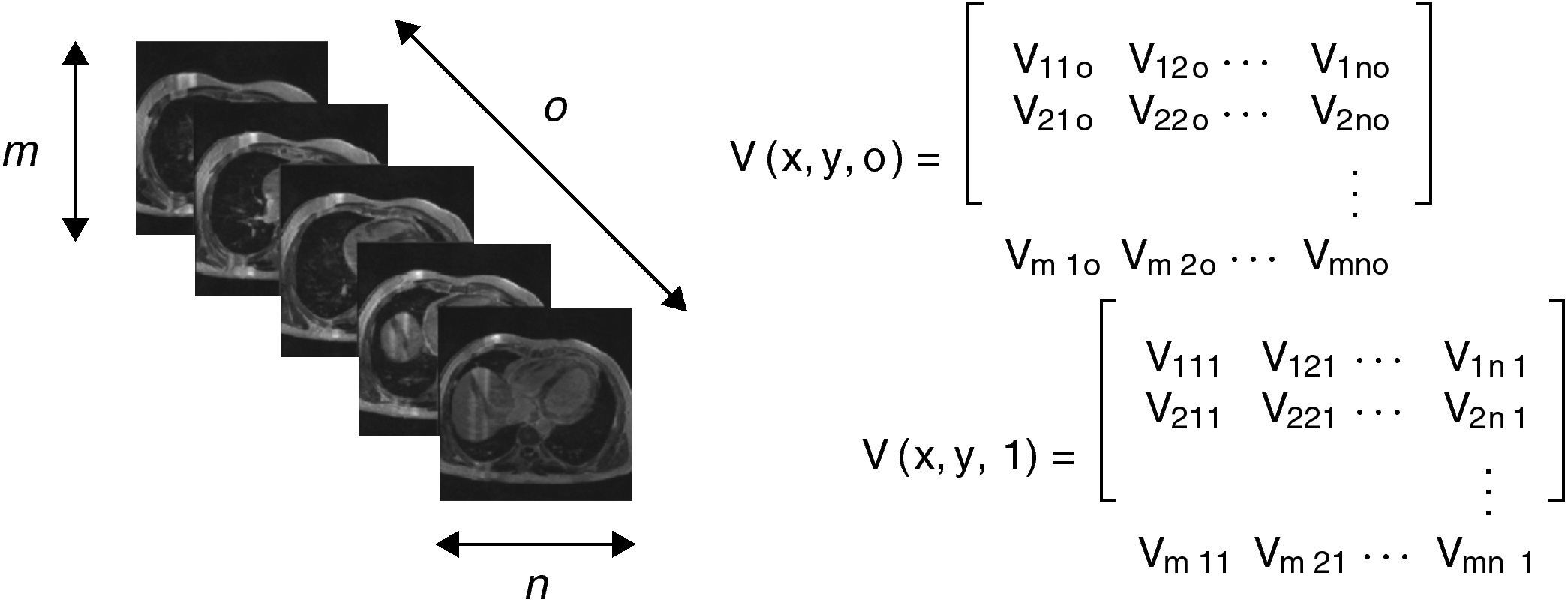
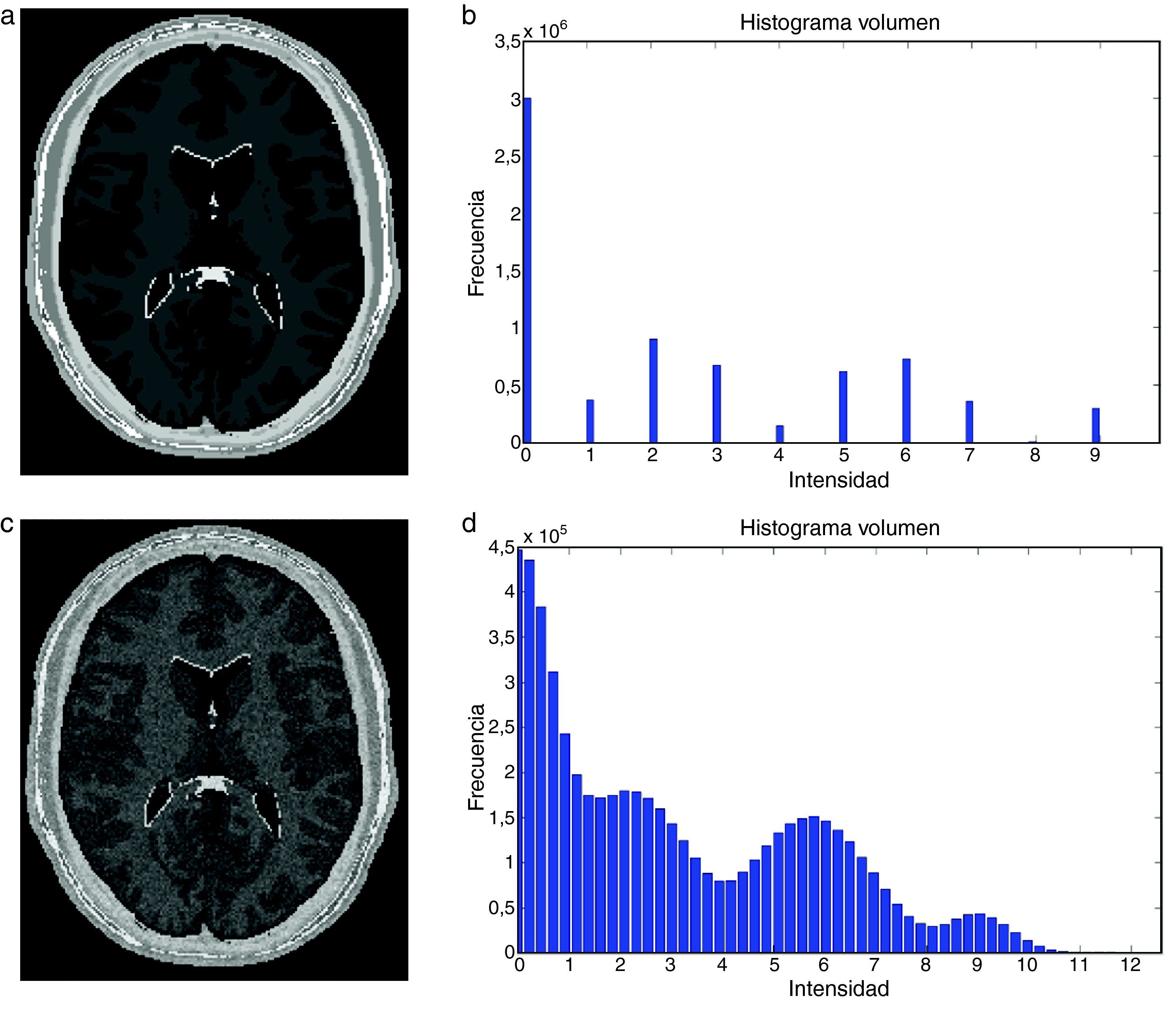
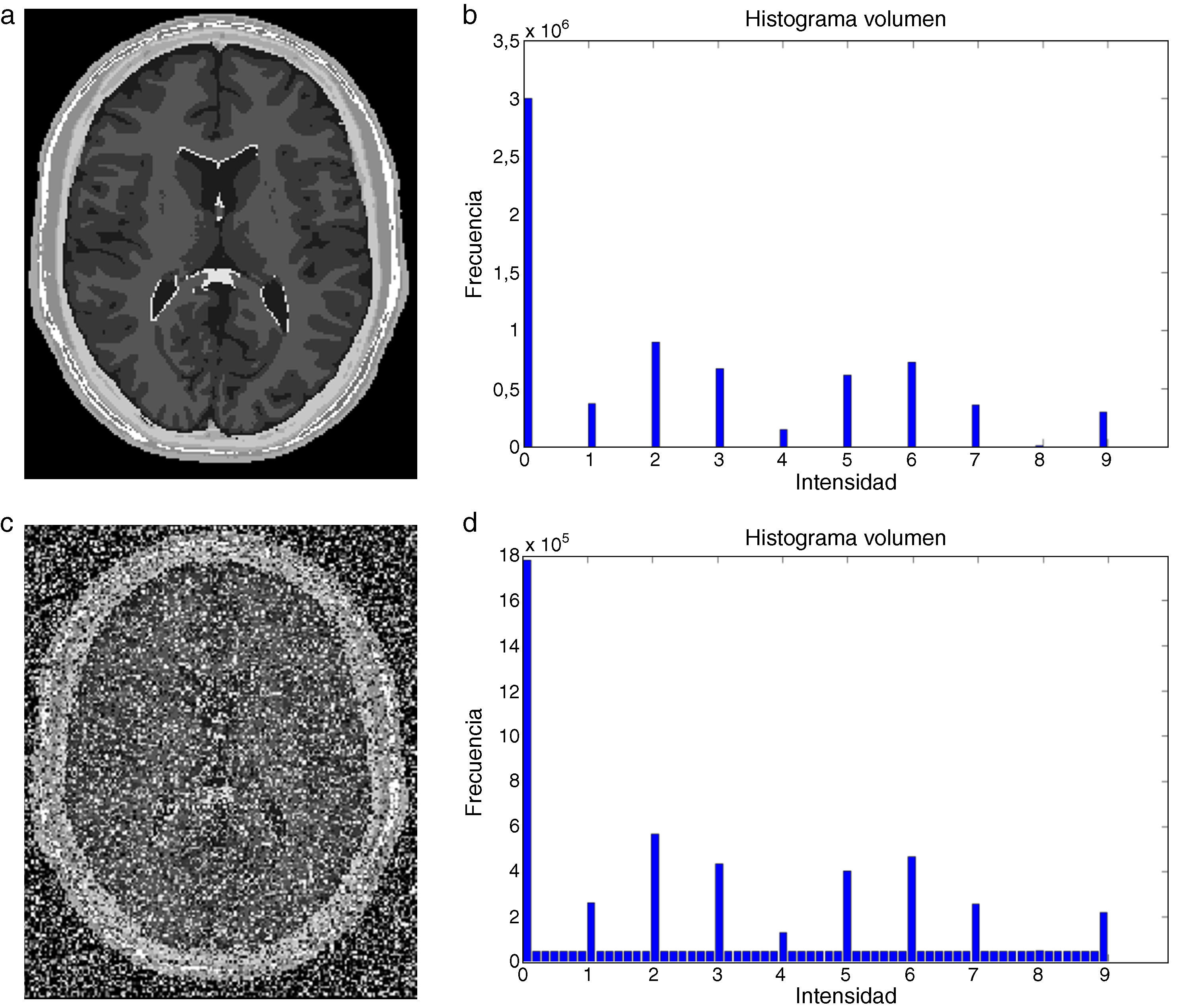
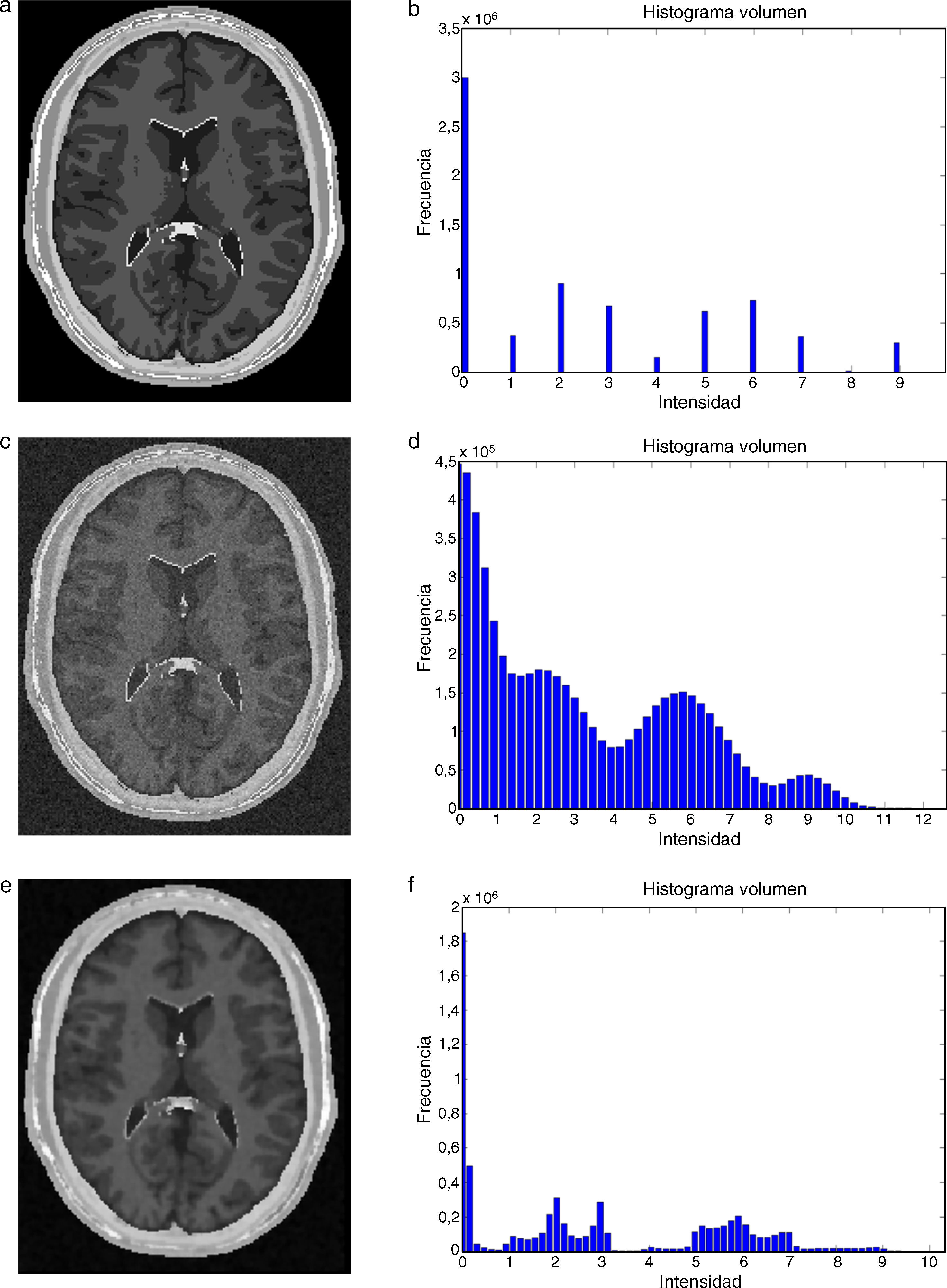
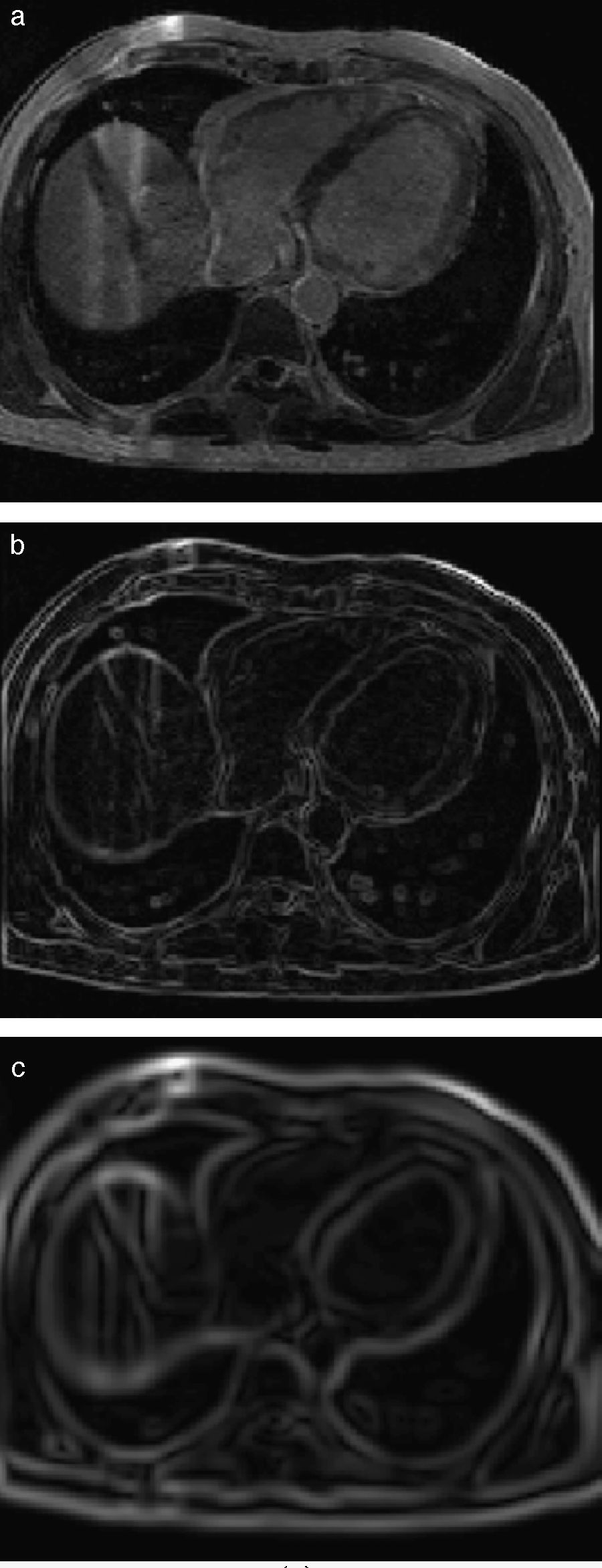
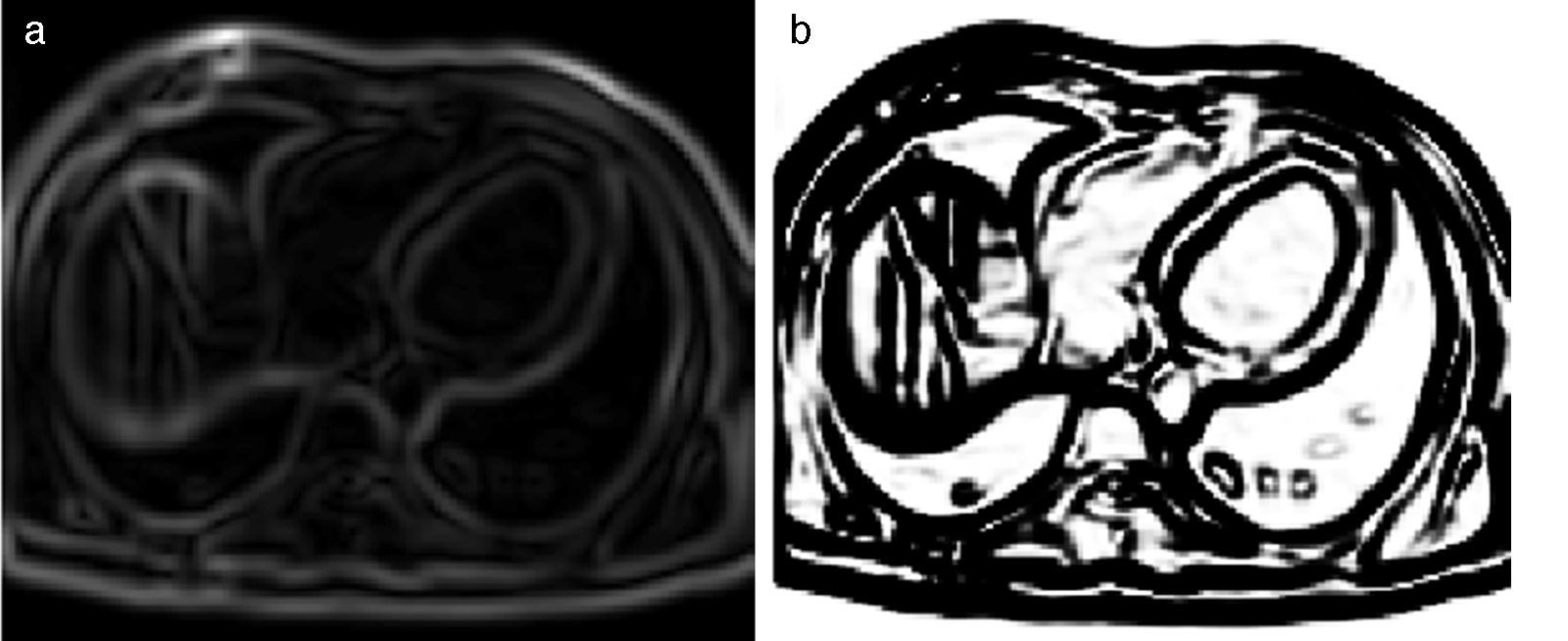
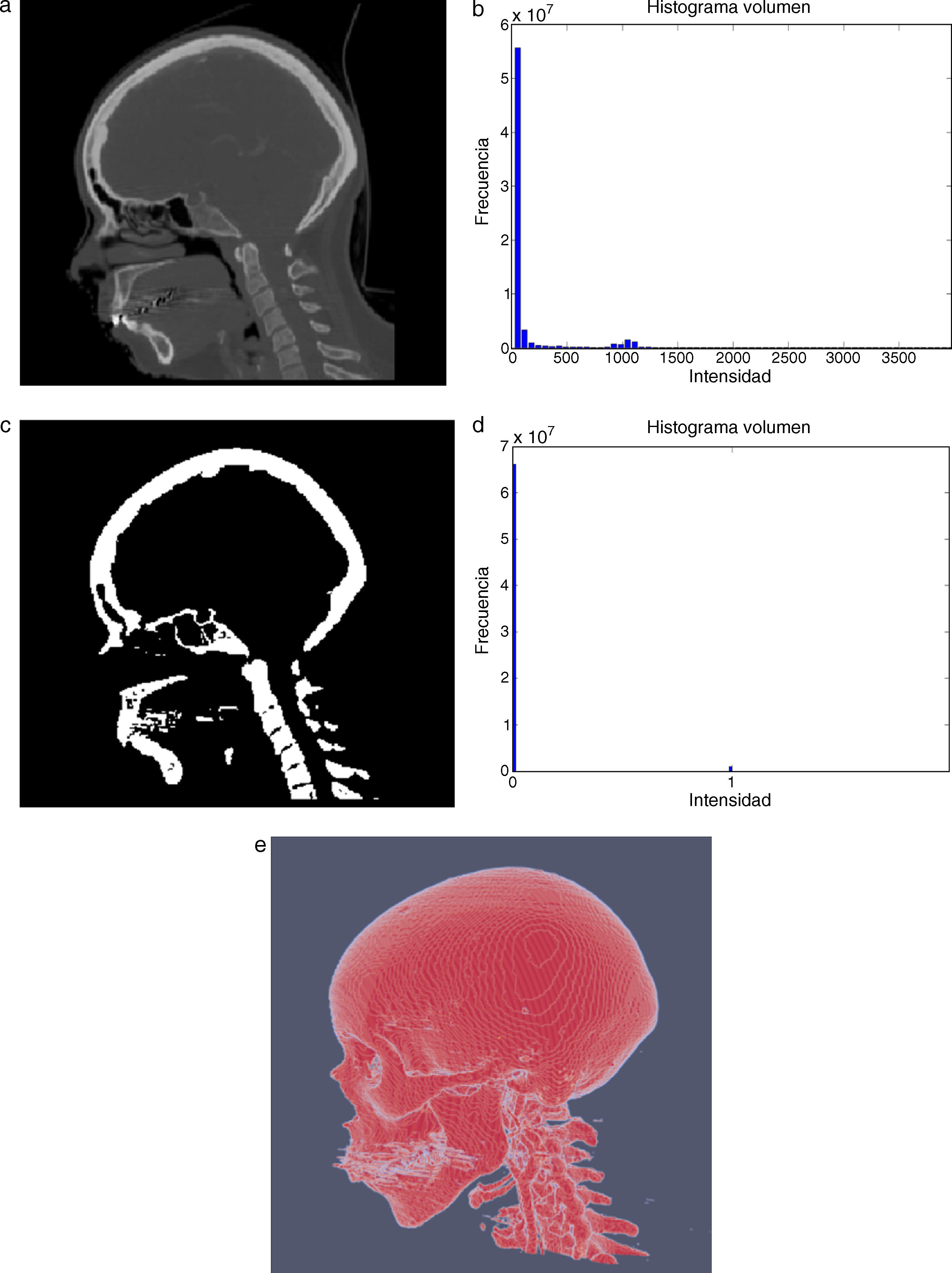
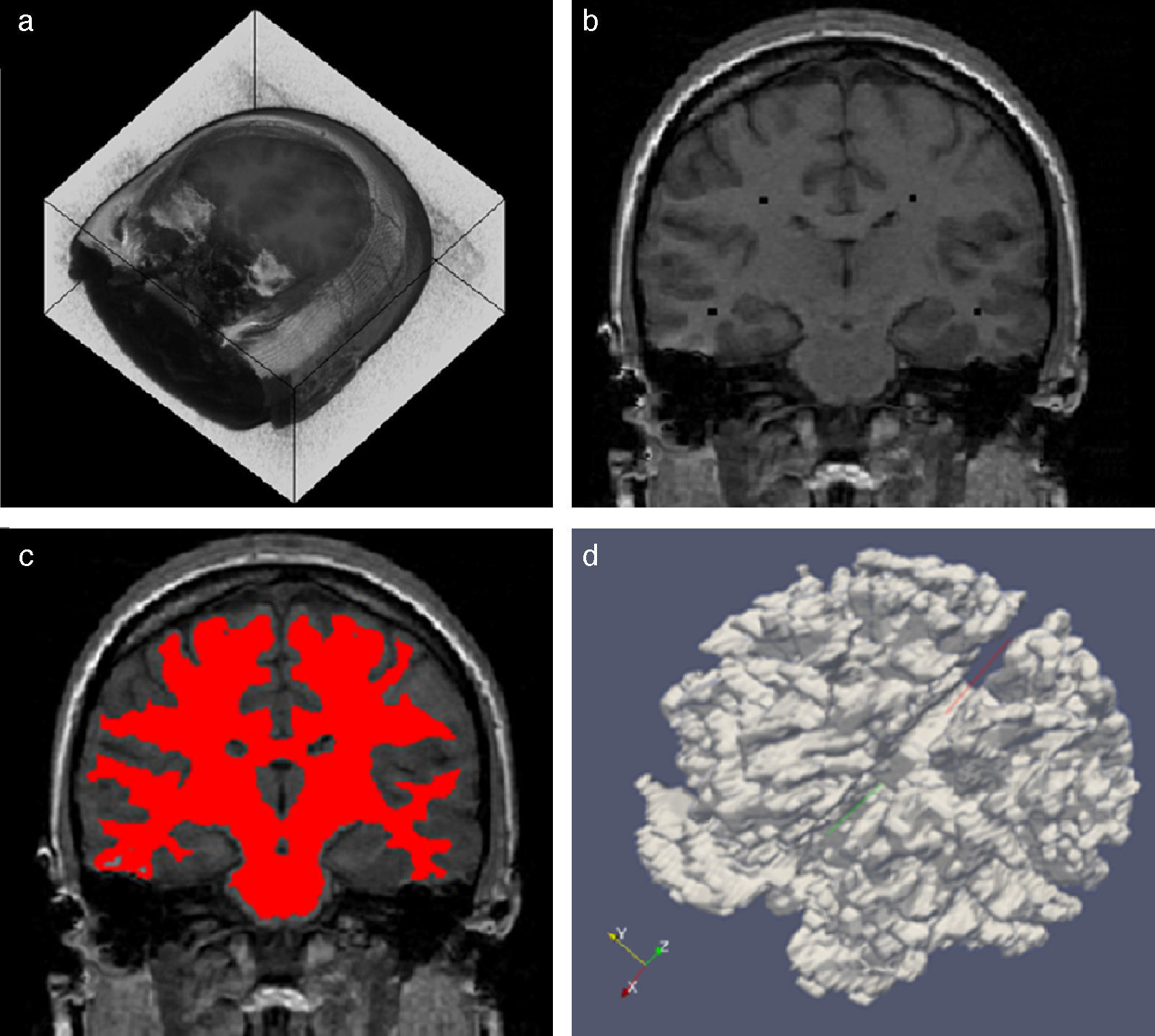
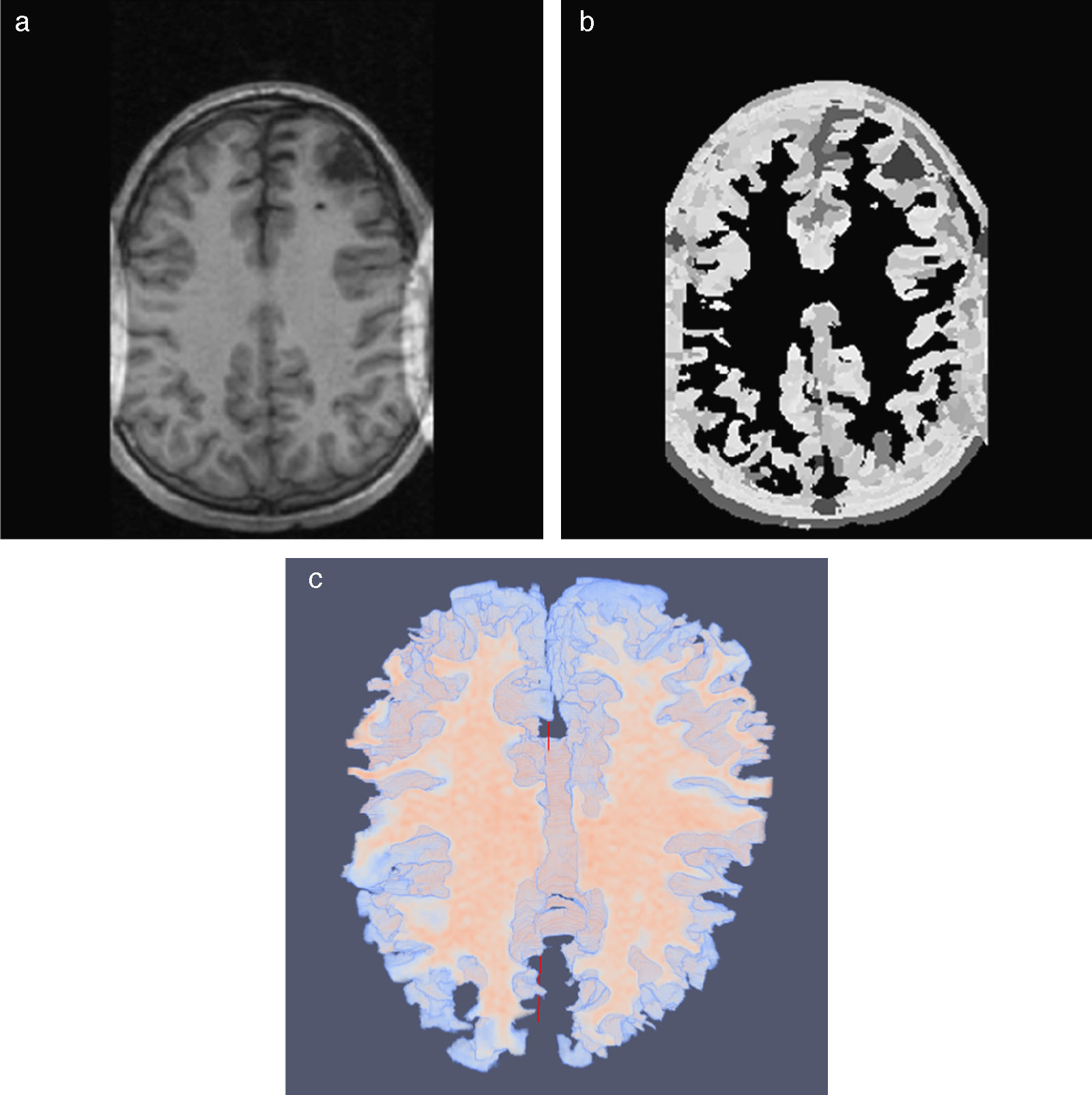
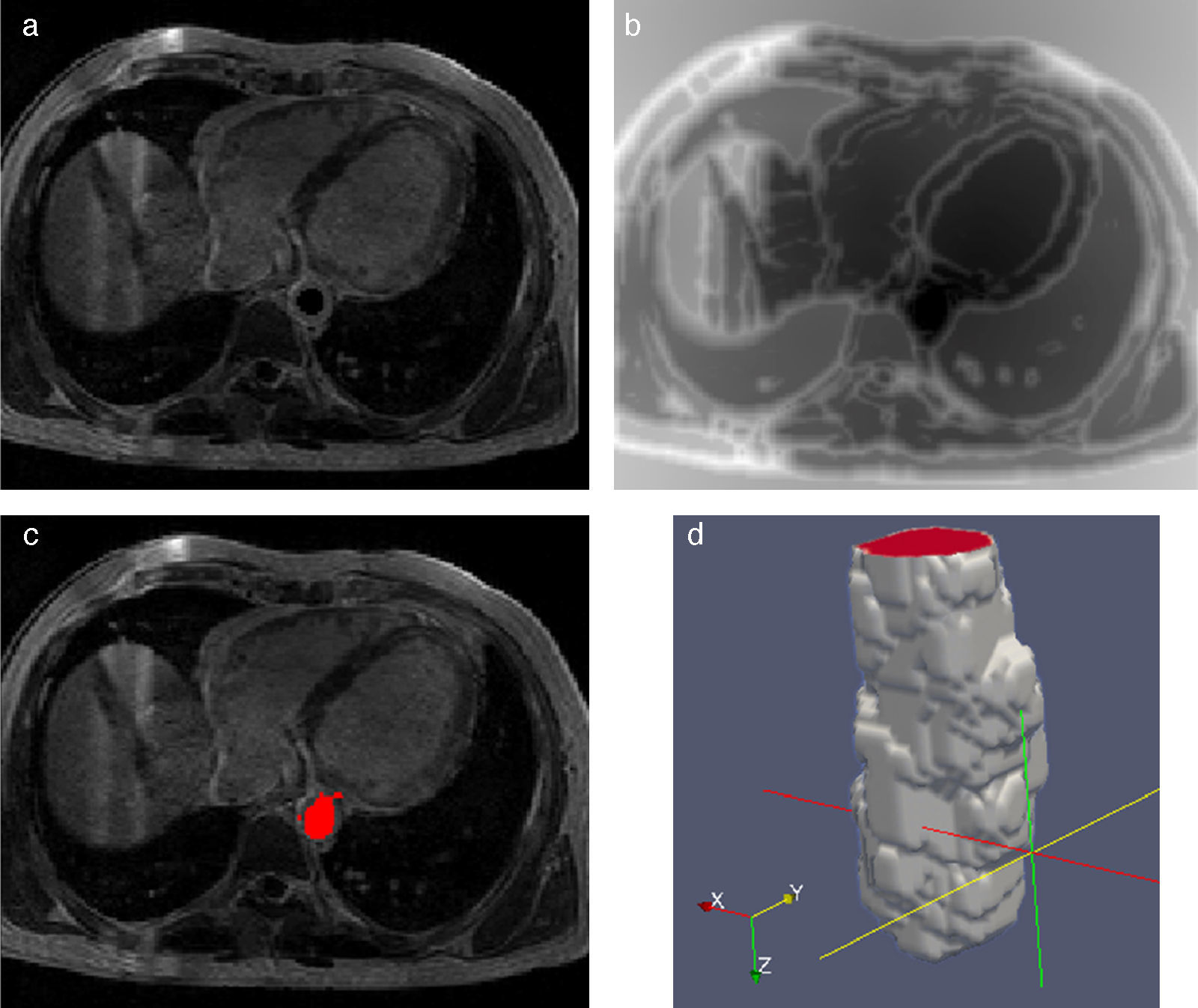
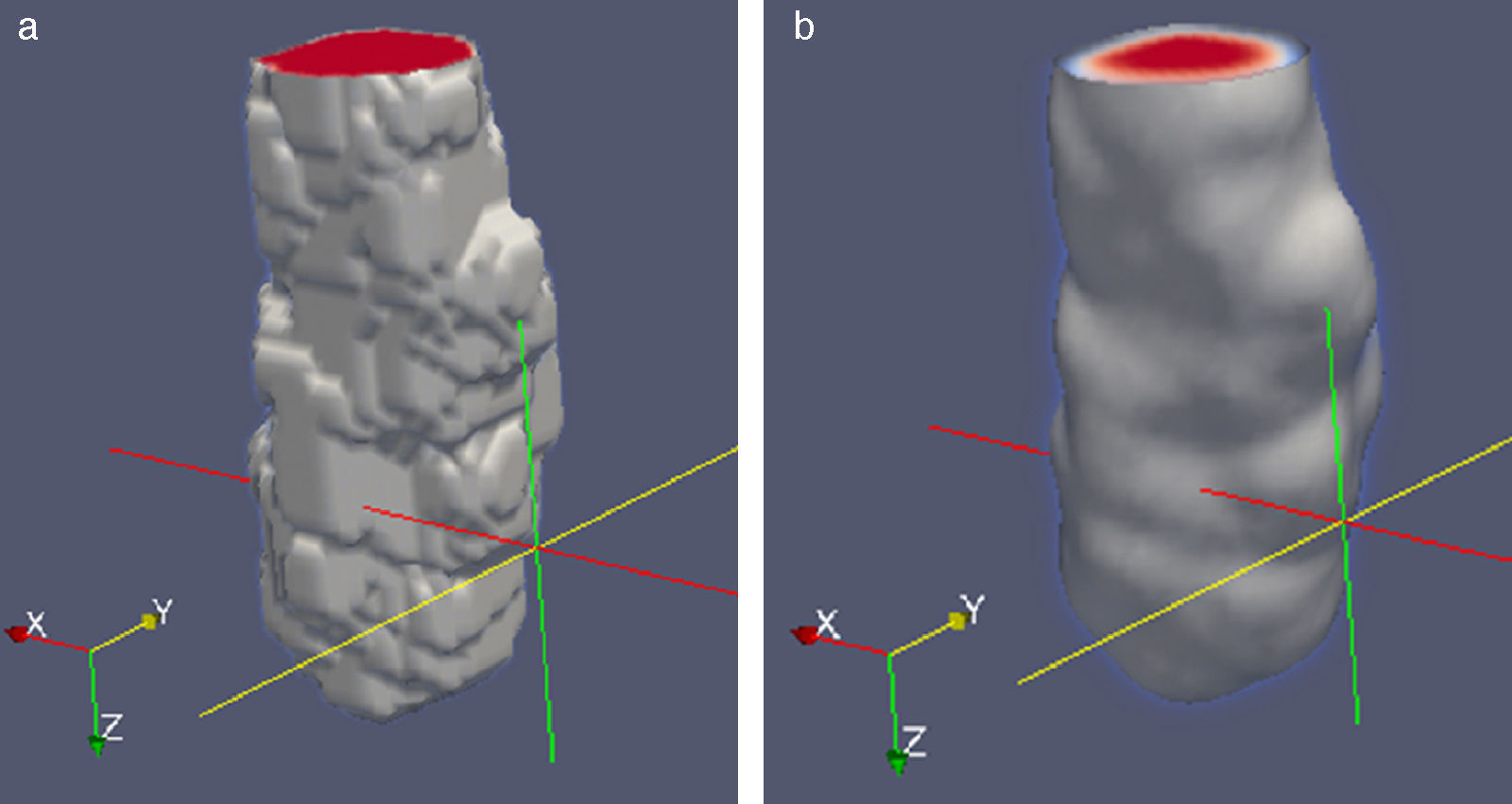
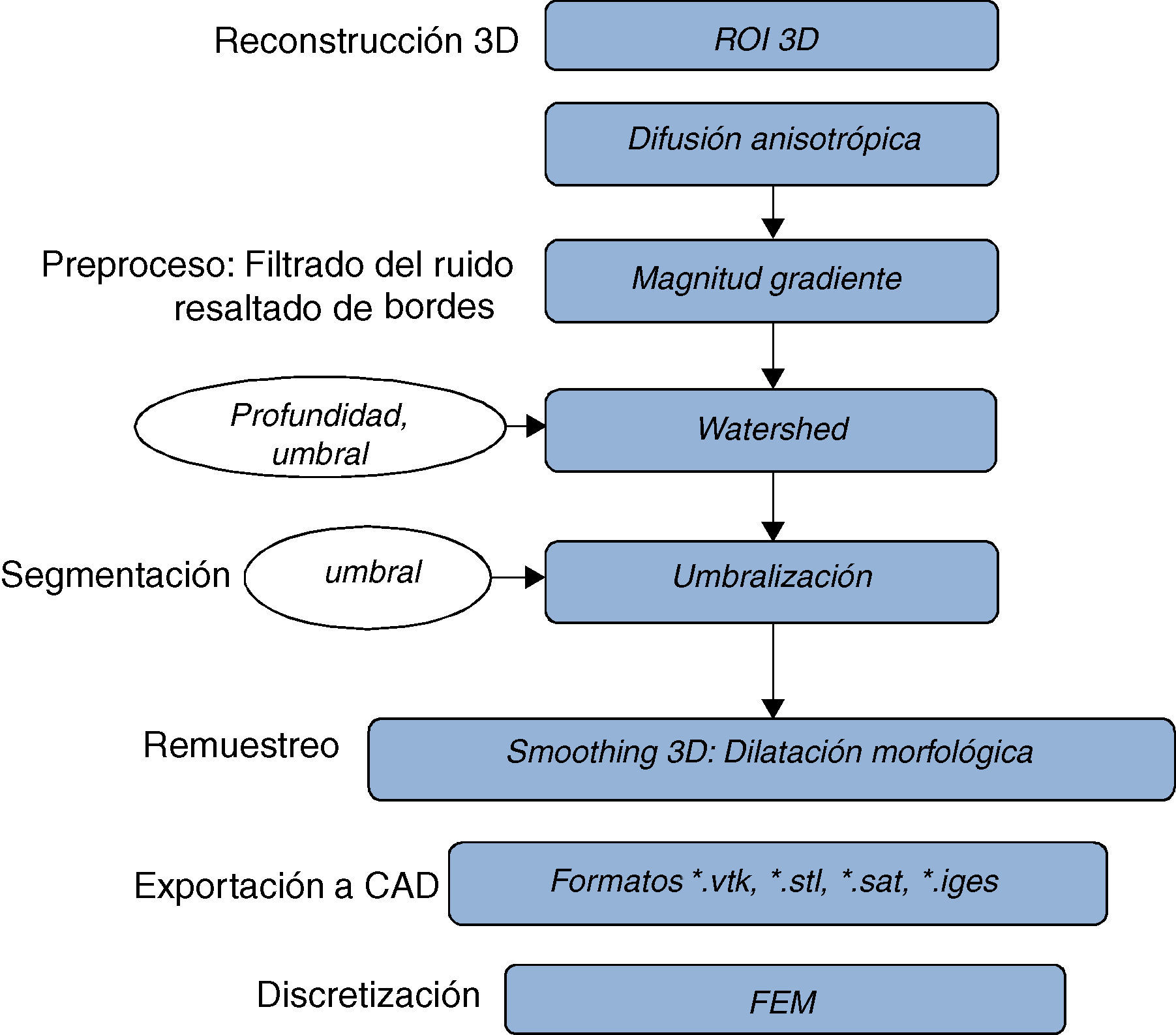
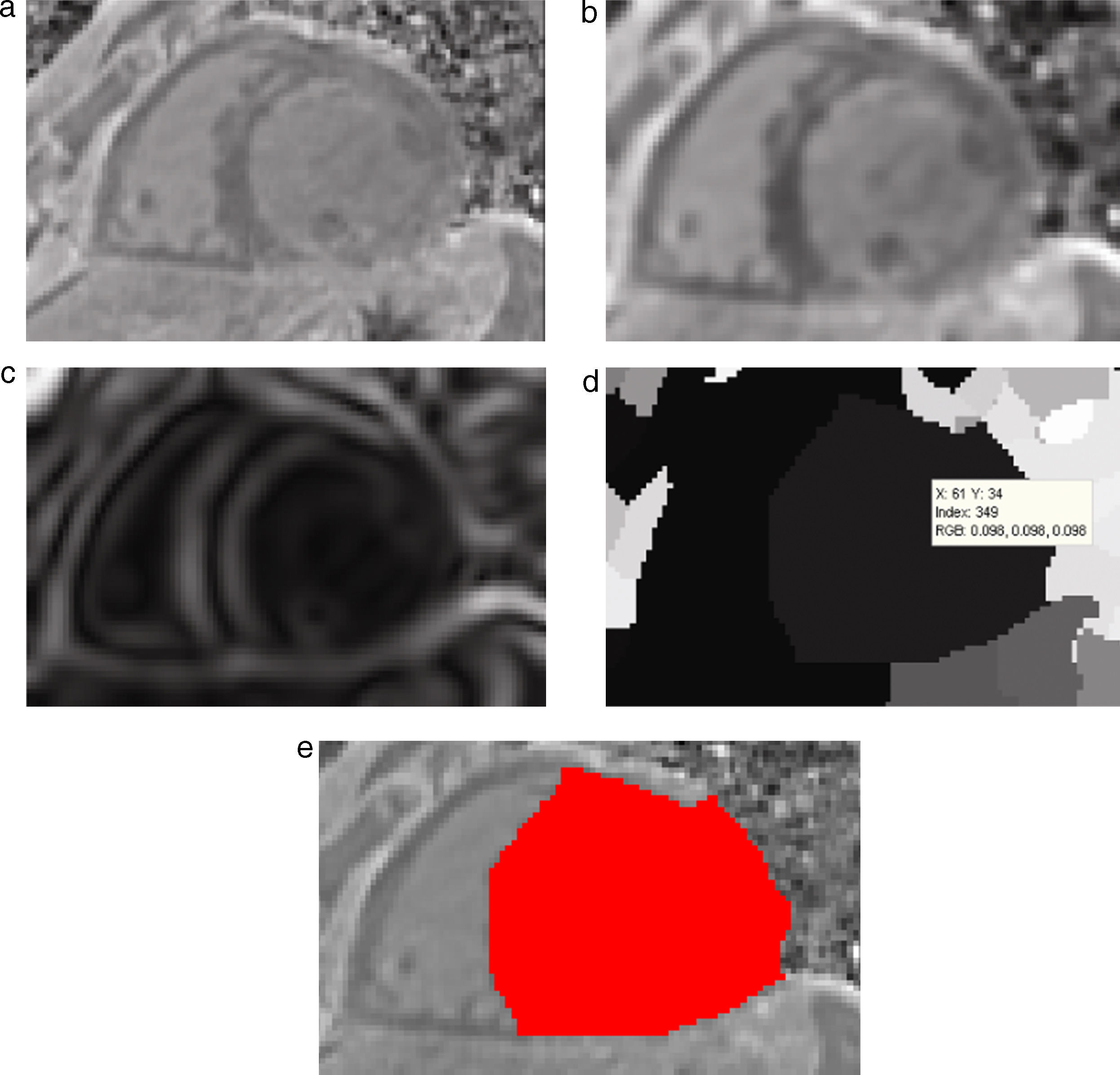
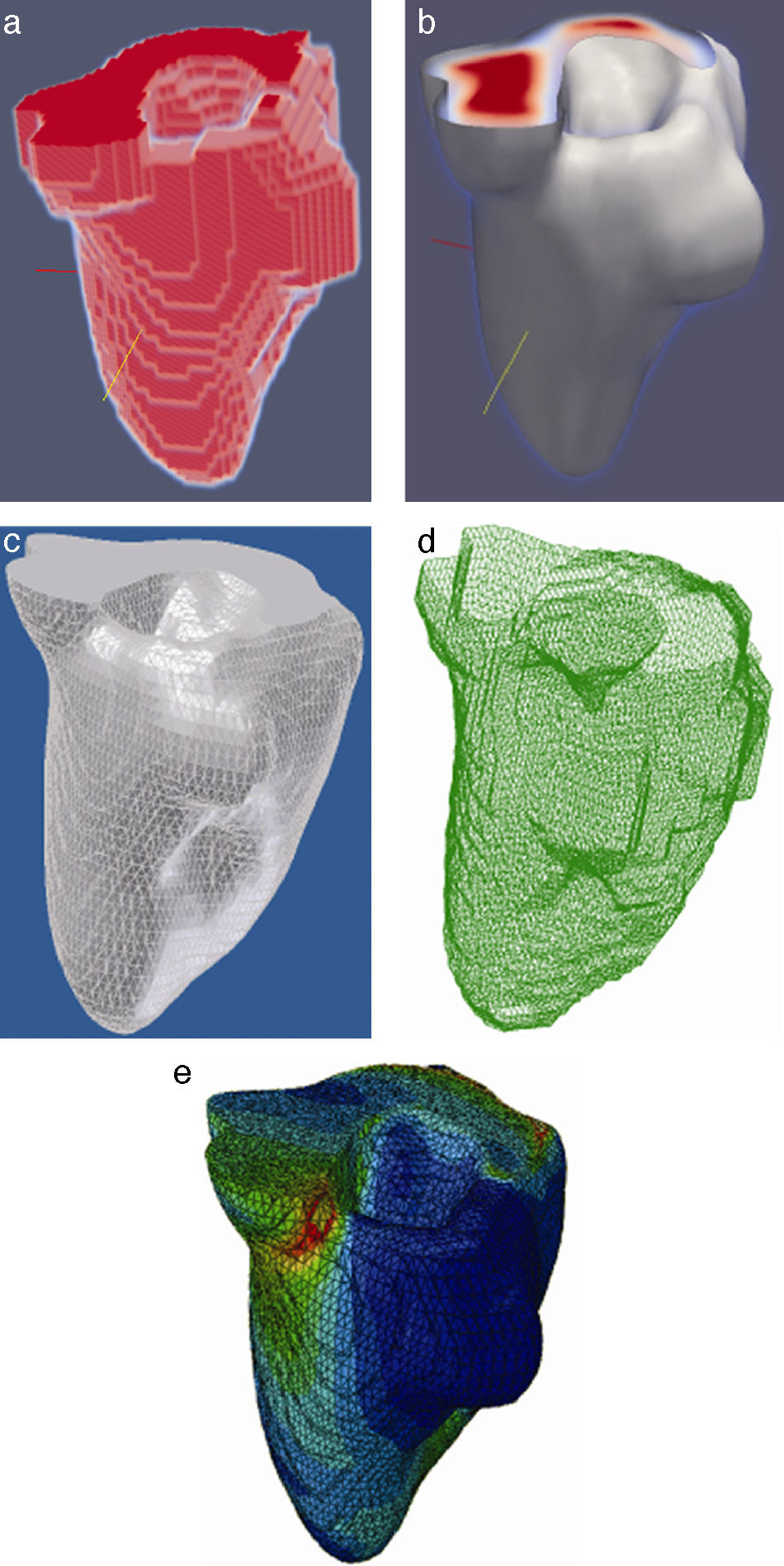
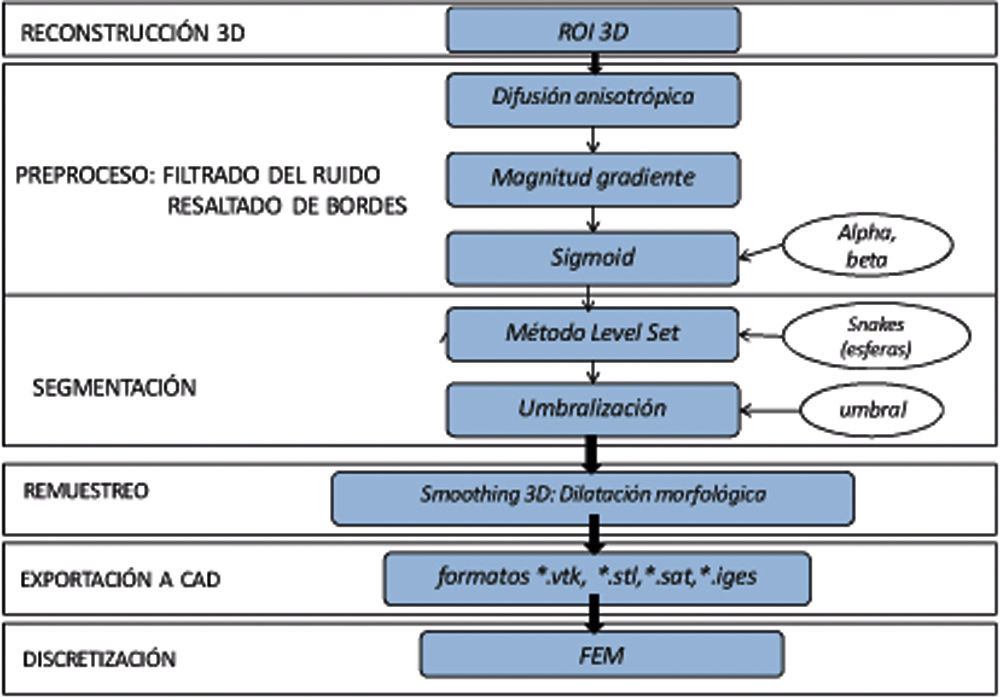
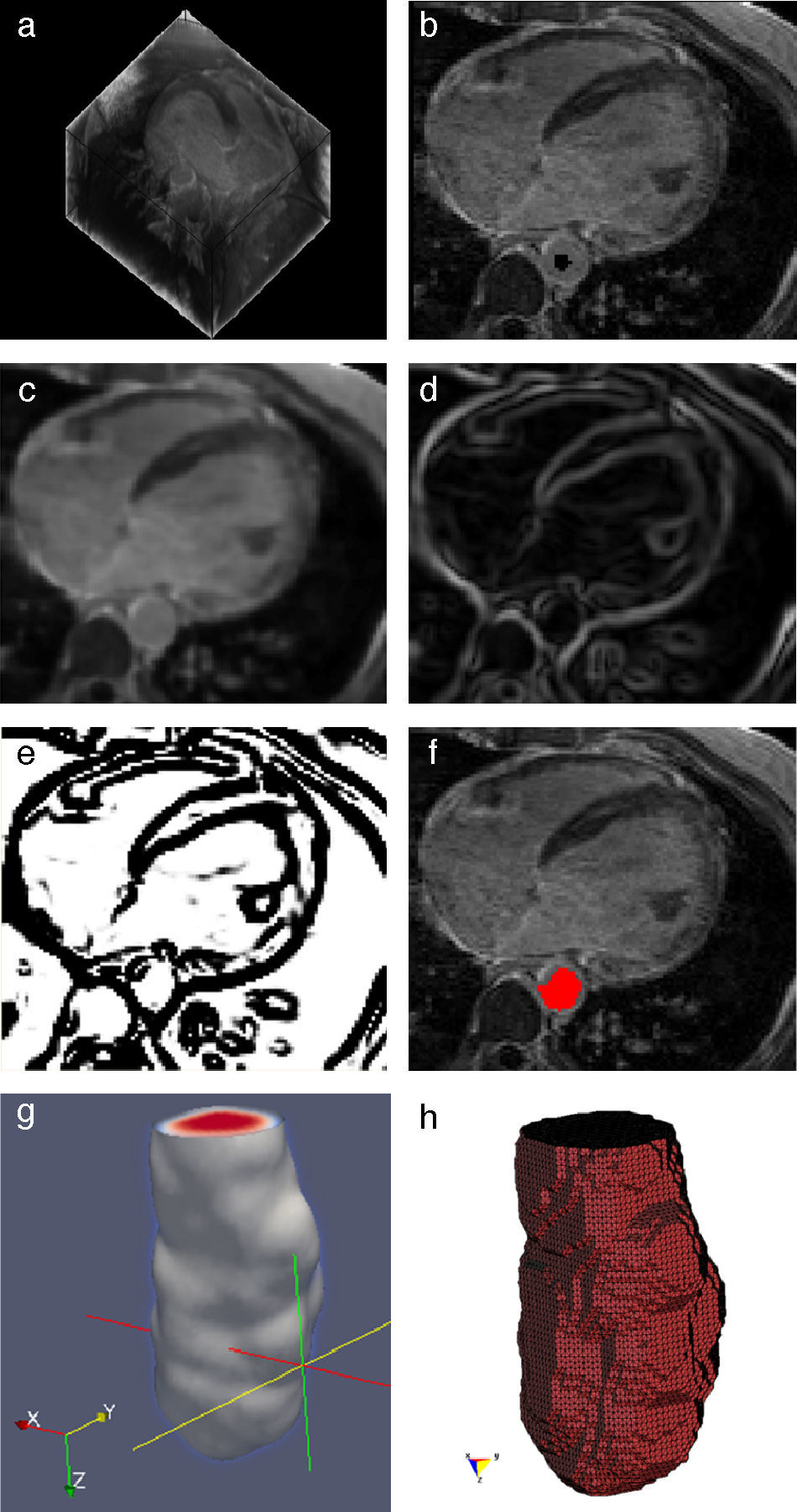
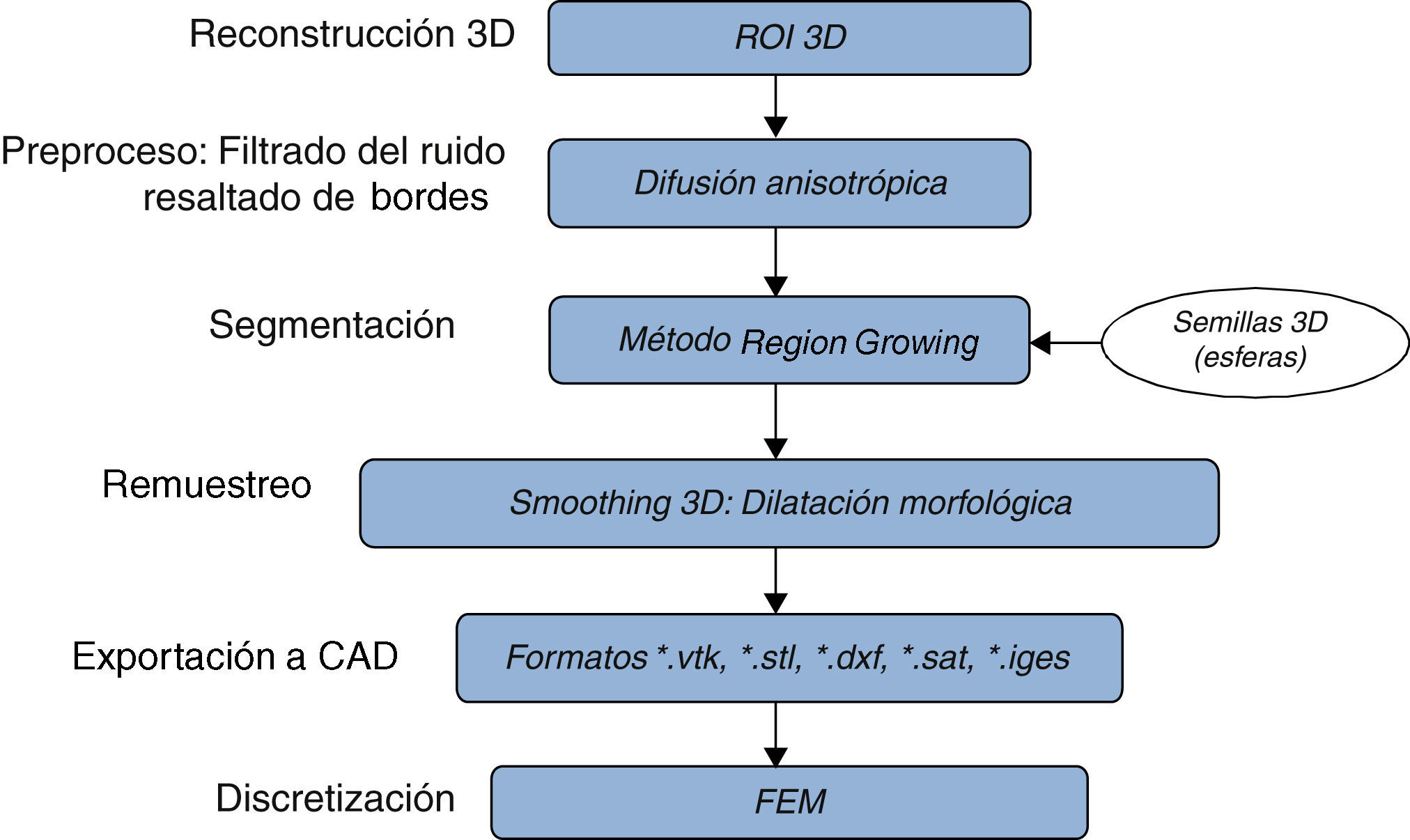
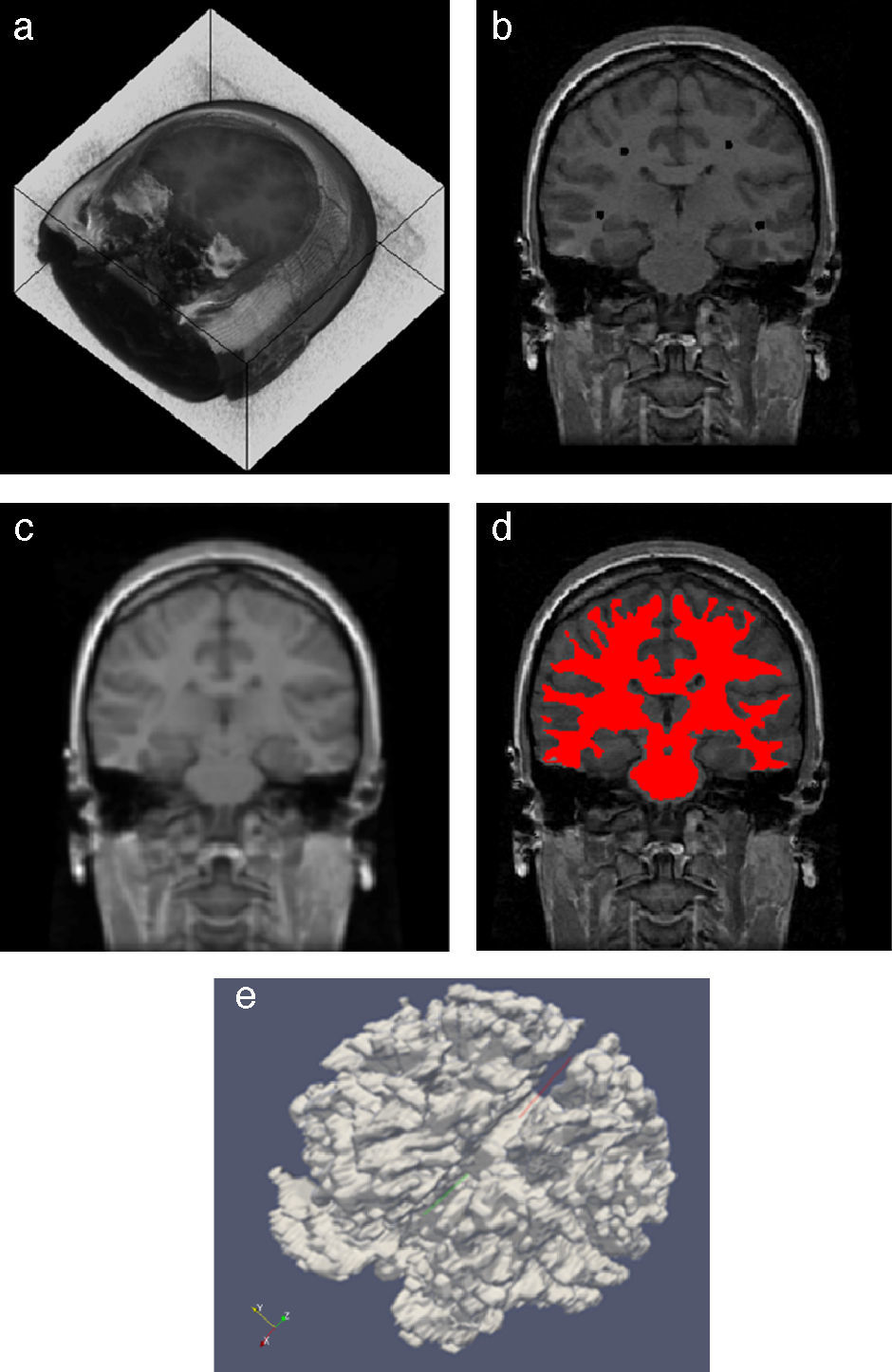
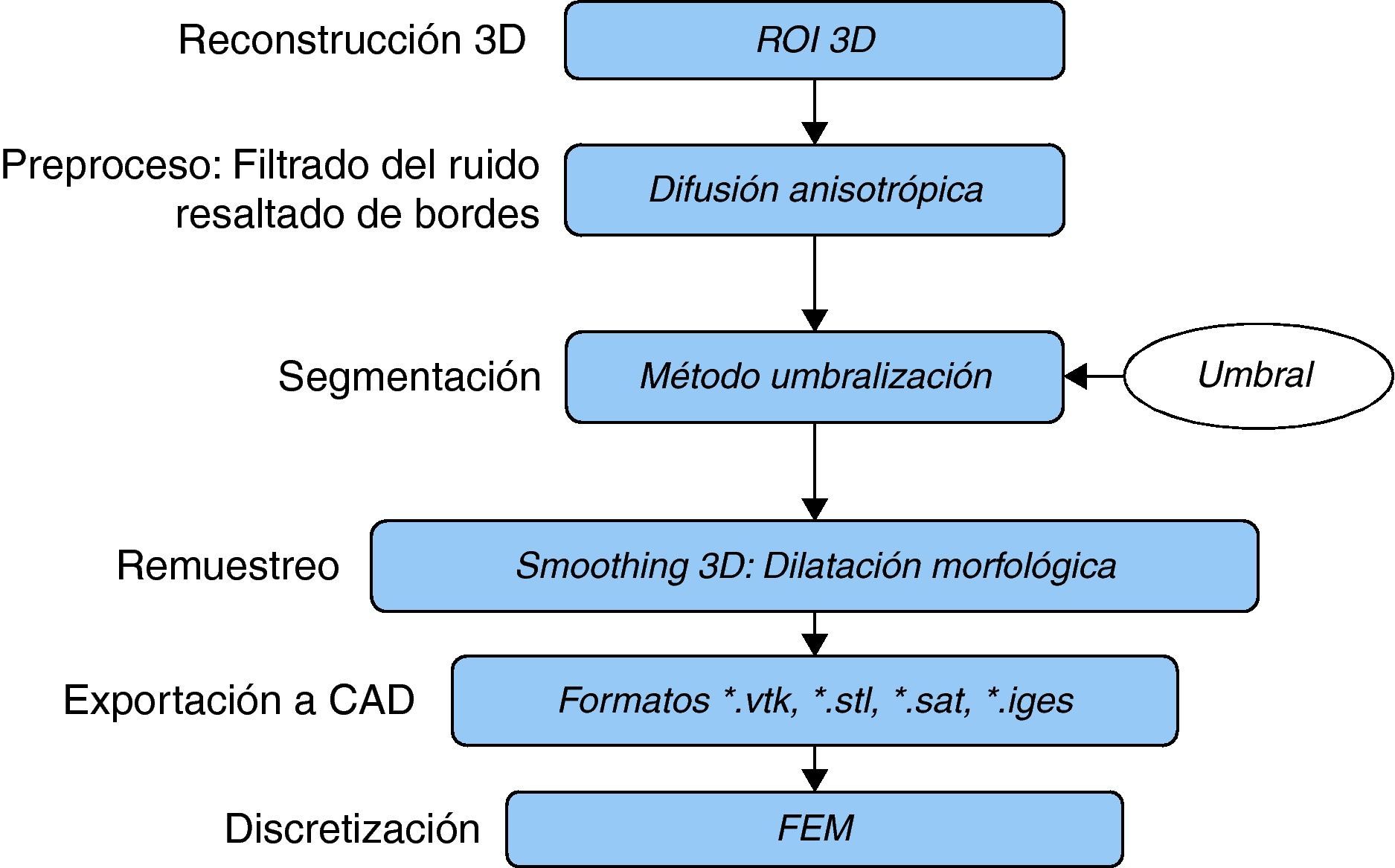
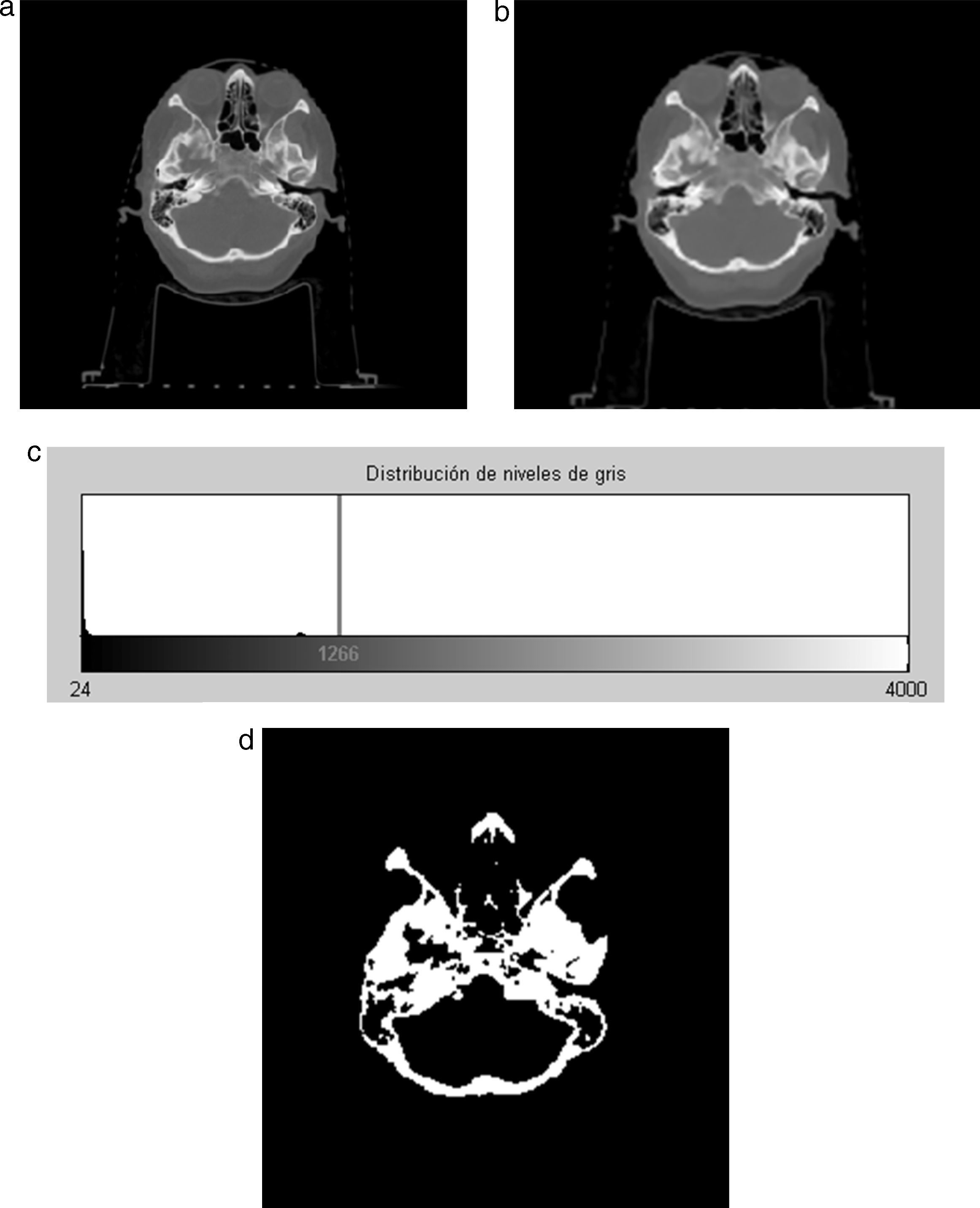
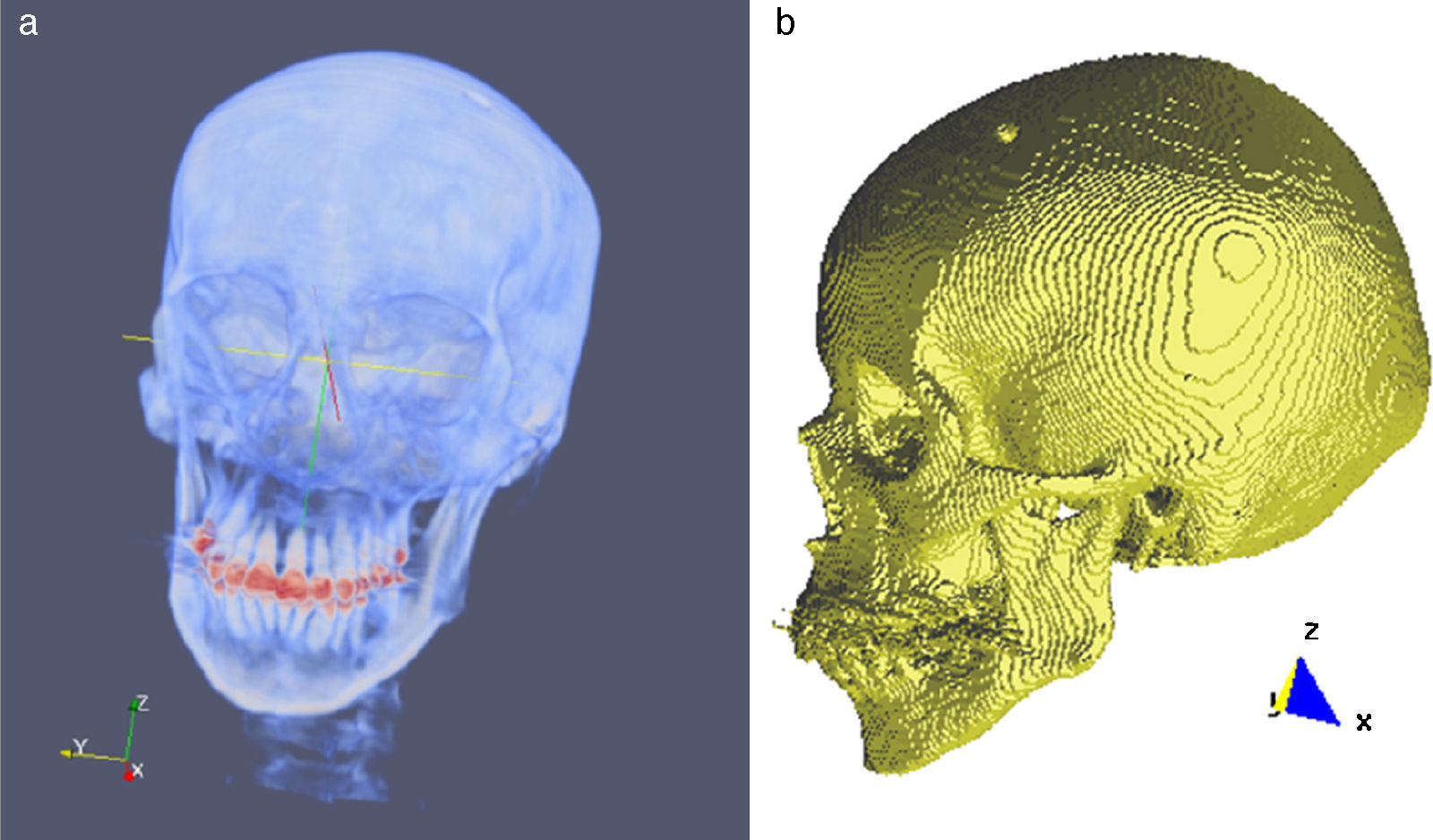
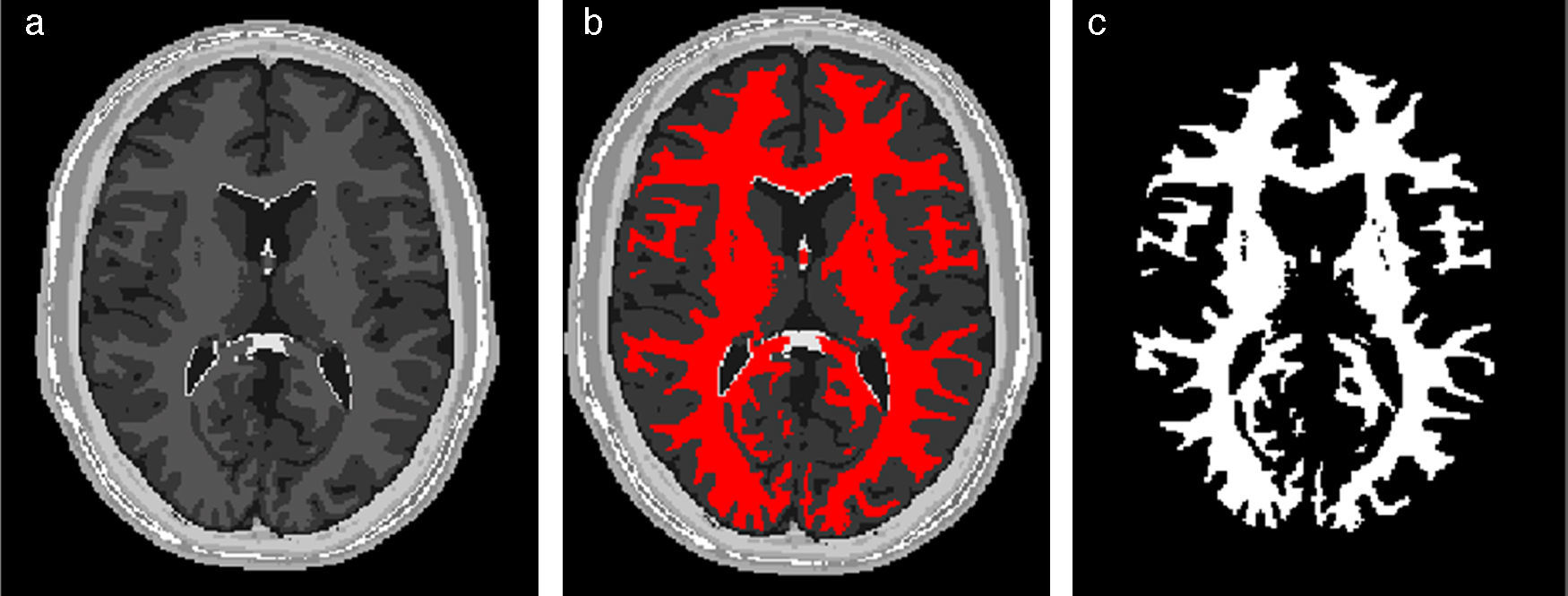
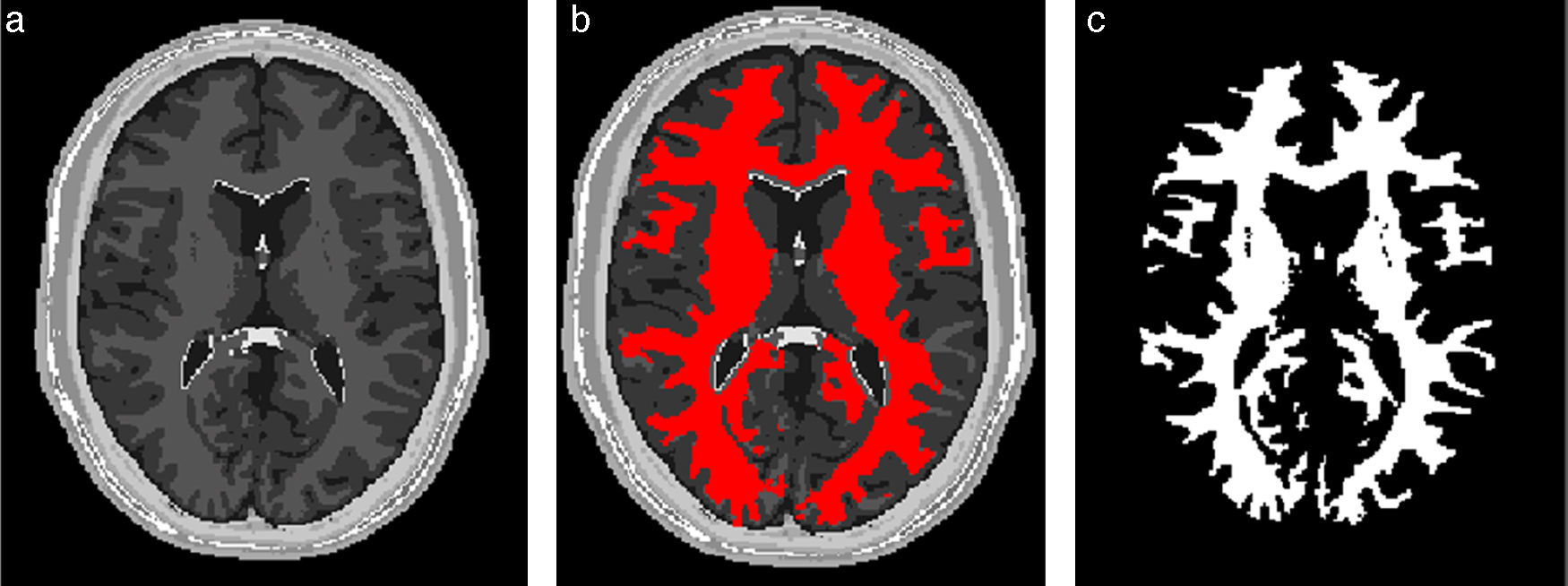
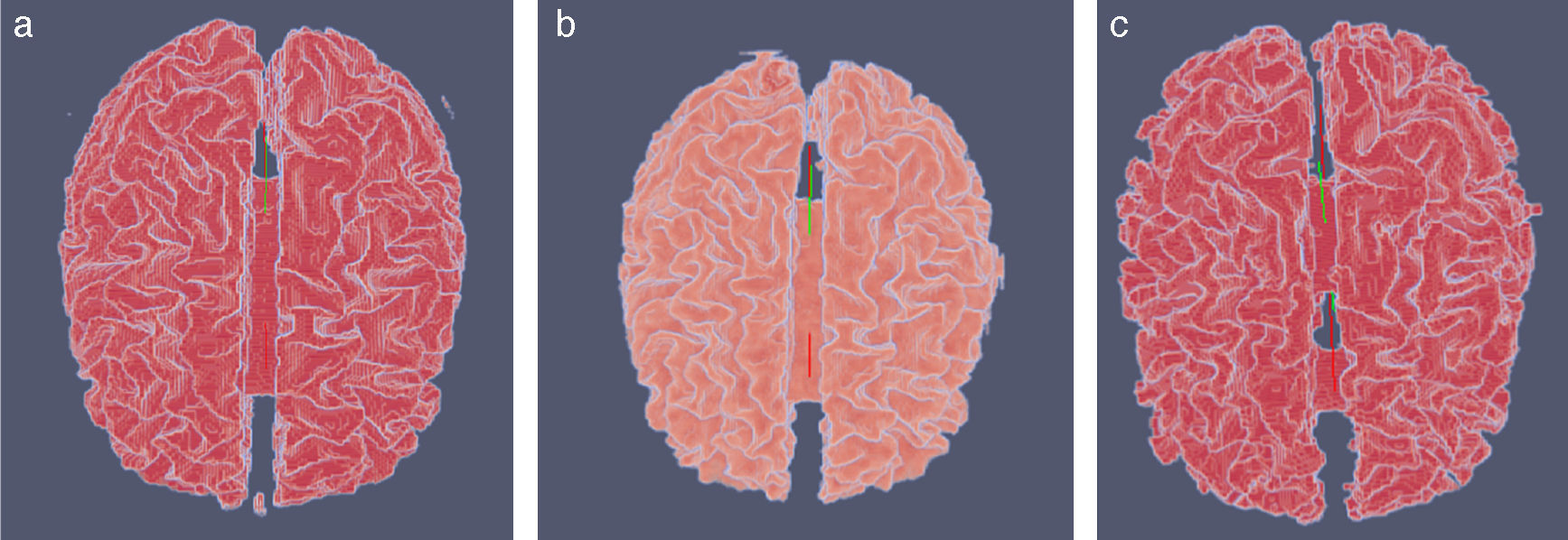
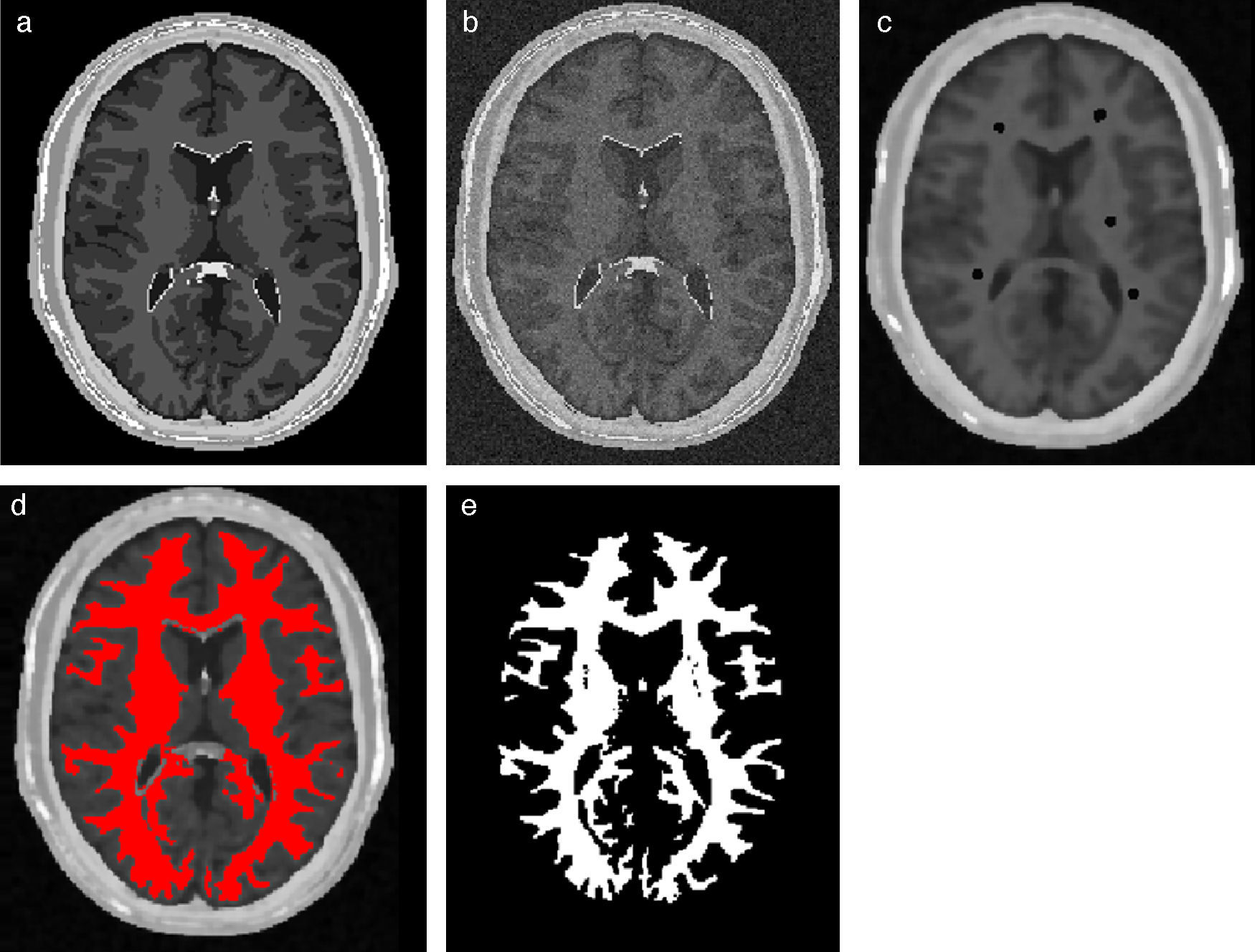
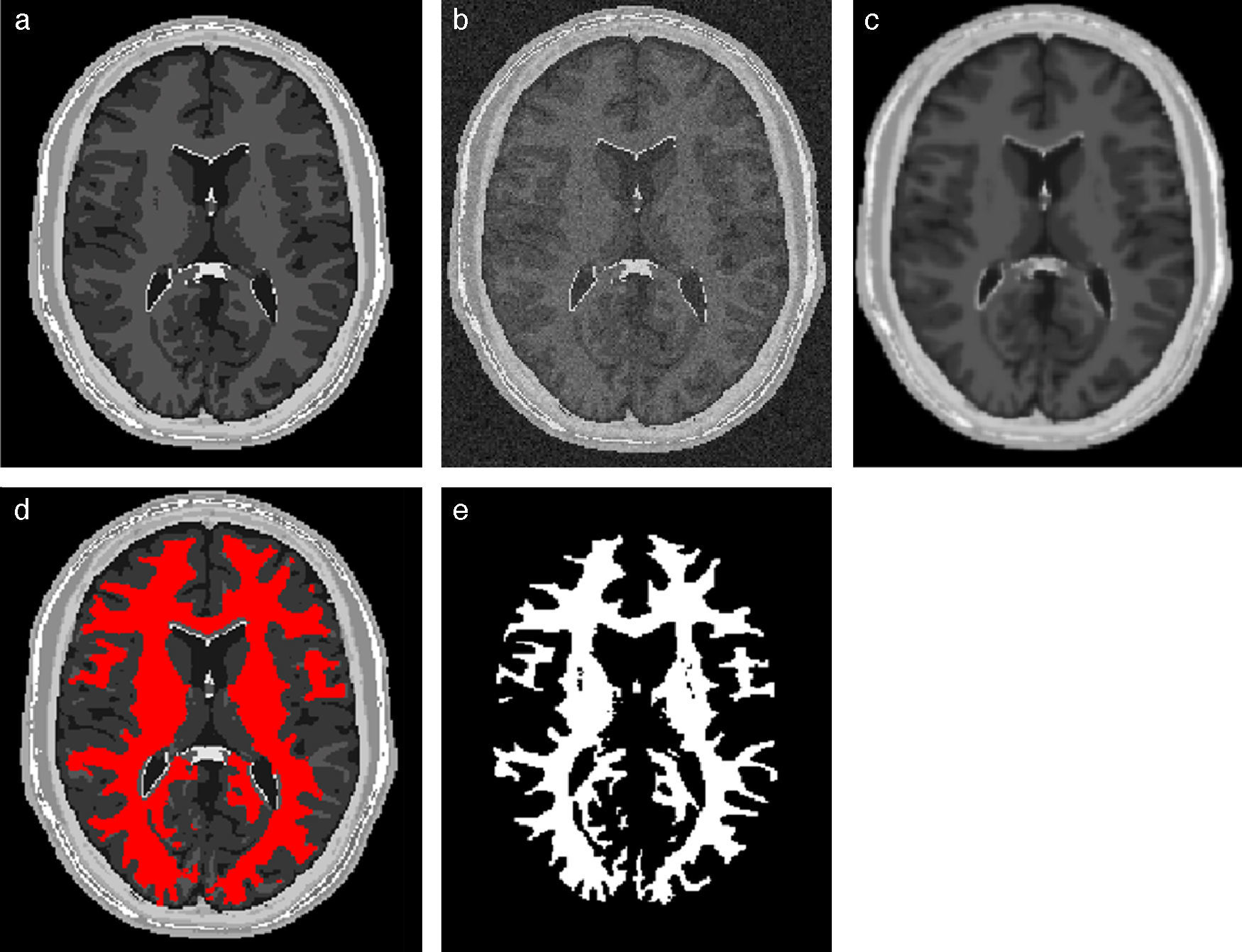
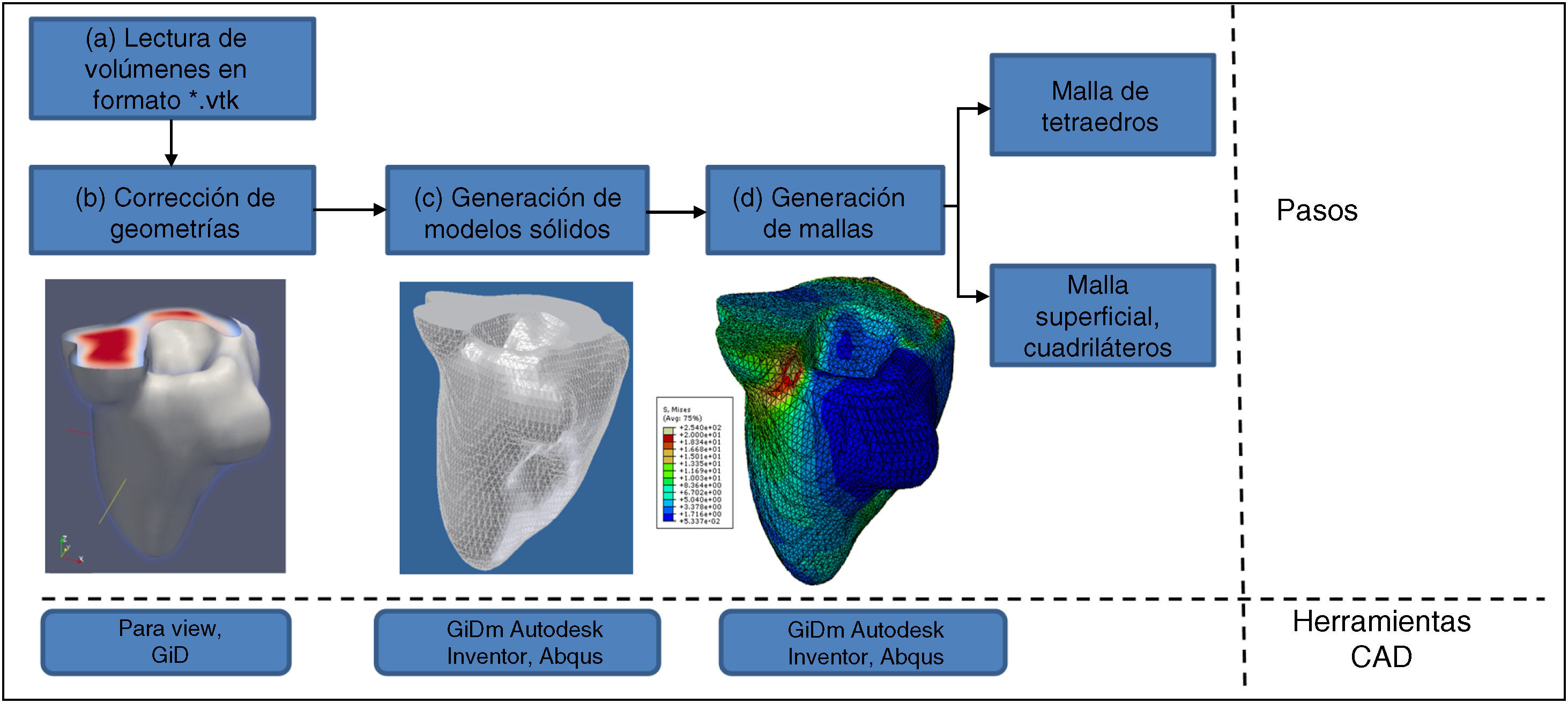
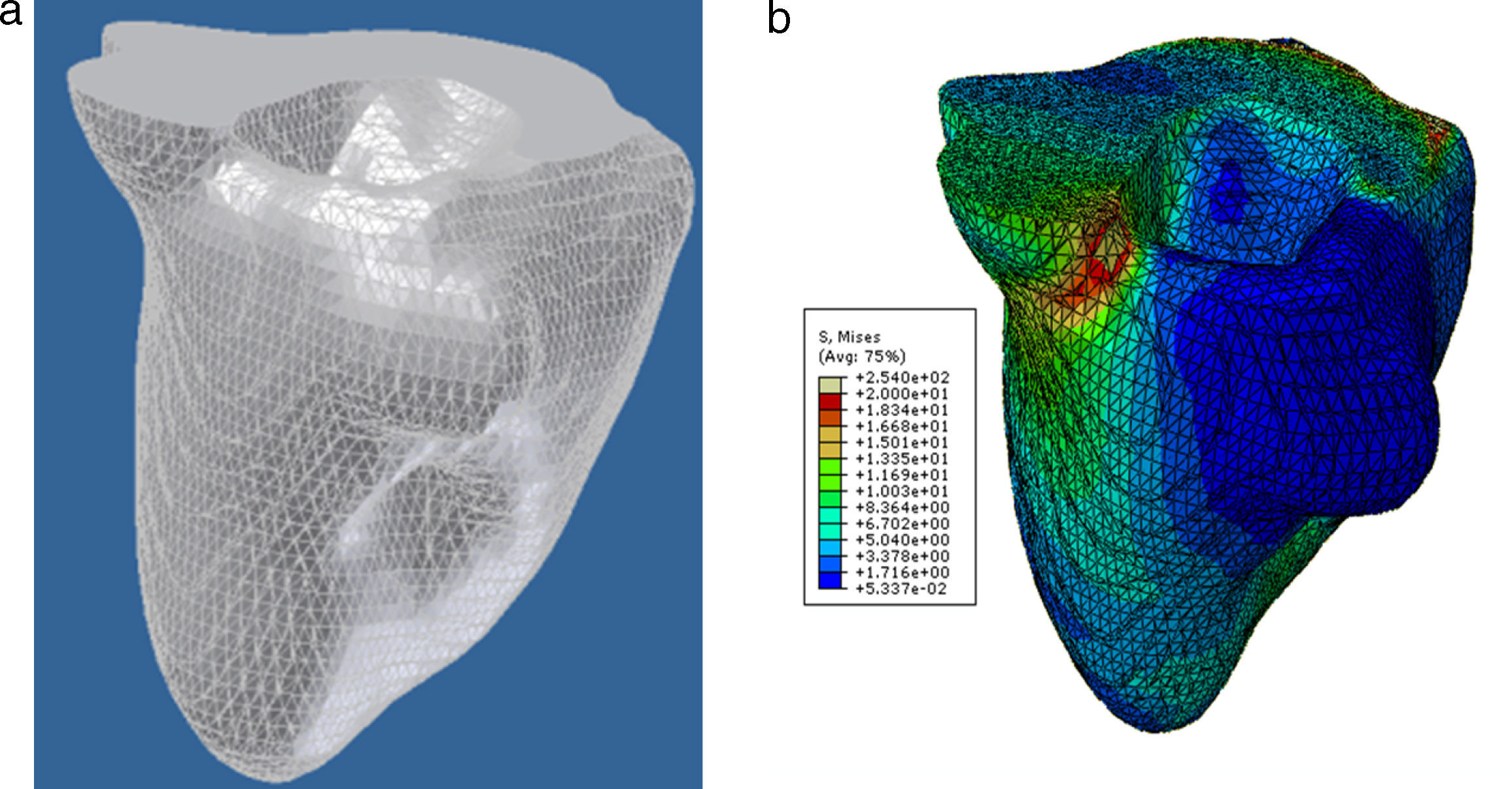
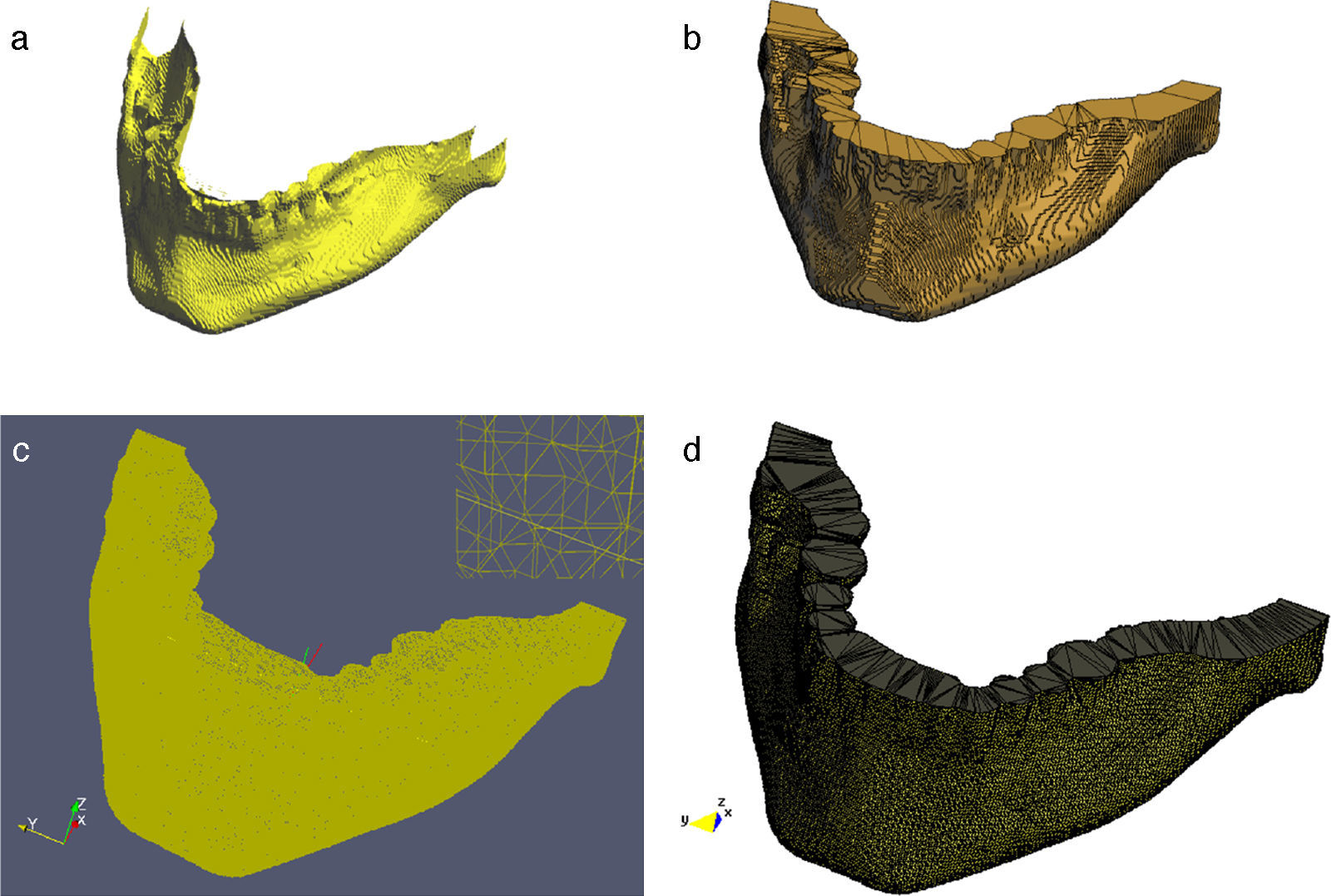
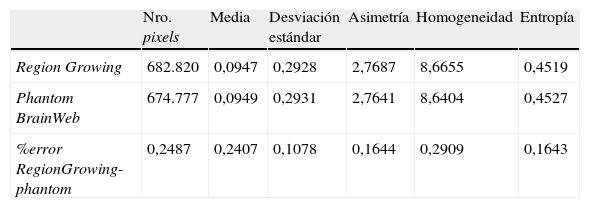
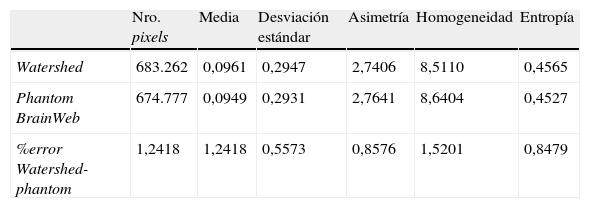
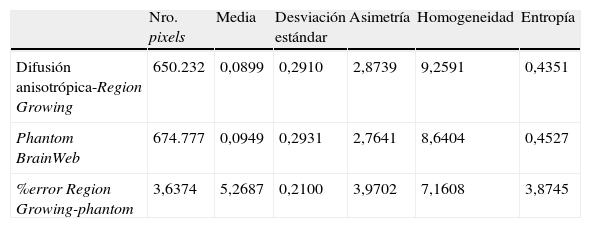
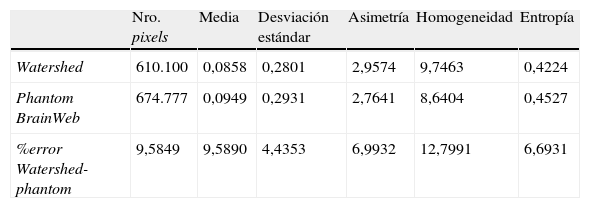
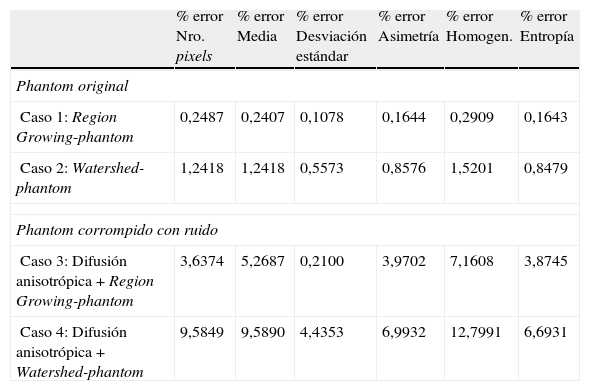
![Esquema de procesos y rutinas implementados en una herramienta de procesamiento de imágenes médicas desarrollada en MATLAB [13]. Esquema de procesos y rutinas implementados en una herramienta de procesamiento de imágenes médicas desarrollada en MATLAB [13].](https://static.elsevier.es/multimedia/02131315/0000002700000003/v1_201305061156/S0213131511000034/v1_201305061156/es/main.assets/thumbnail/gr1.jpeg?xkr=ue/ImdikoIMrsJoerZ+w96p5LBcBpyJTqfwgorxm+Ow=)