Se presenta un inventario de especies de Elateridae (Insecta: Coleoptera: Elateroidea) en la Estación de Biología Chamela, Jalisco, México. La identificación se realizó con base en evidencia morfológica y molecular (ADN mitocondrial del gen citocromo oxidasa subunidad I [COI]). El trabajo de campo se realizó entre 2009 y 2012. Se recolectaron 544 individuos pertenecientes a 46 especies, 25 géneros, 7 subfamilias y 11 tribus; se obtuvieron 106 secuencias de varias de las morfoespecies discriminadas. Treinta y ocho especies fueron discriminadas con información molecular, y se encontraron 10 casos de discordancia entre evidencia molecular y morfológica. La subfamilia Elaterinae presentó la mayor riqueza de especies (18 spp.), seguida de Agrypninae (16 spp.) y Cardiophorinae (6 spp.). Las subfamilias con menor número de especies fueron Cebrioninae (2 spp.), Lissominae (2 spp.), Semiotinae (1 sp.) y Thylacosterninae (1 sp.). Los géneros con mayor riqueza de especies fueron Conoderus (6 spp.), Megapenthes (5 spp.) y Esthesopus (4 spp.). Se obtuvieron 27 nuevos registros de especies para Jalisco, 5 nuevos registros para México y 7 especies potencialmente nuevas para la ciencia. Este estudio resalta la importancia de emplear diferentes fuentes de caracteres para obtener un mejor conocimiento de la riqueza de especies en estudios faunísticos, especialmente en grupos altamente diversos.
A species inventory of Elateridae (Insecta: Coleoptera: Elateroidea) from the Estación de Biología Chamela, Jalisco, Mexico, is presented. The identification was made based on morphological and molecular evidence (mtDNA gene cytochrome oxidase subunit I (COI)).. Fieldwork was carried out between 2009 and 2012. We obtained 544 individuals belonging to 46 species, 25 genera, 7 subfamilies and 11 tribes, and 106 sequences of several discriminated morphospecies were obtained. Thirty eight species were discriminated with molecular information, finding 10 cases of discordance between molecular and morphological evidence. The subfamily Elaterinae had the highest species richness (18 spp.), followed by Agrypninae (16 spp.) and Cardiophorinae (6 spp.). The subfamilies with lowest number of species were Cebrioninae (2 spp.), Lissominae (2 spp.), Semiotinae (1 sp.) and Thylacosterninae (1 sp.). The genera with the highest number of species were Conoderus (6 spp.), Megaphenthes (5 spp.) and Esthesopus (4 spp. each). Twenty-seven of the species collected represent new records for Jalisco, 5 for Mexico, and 7 more are potentially new to science. This study highlights the importance of employing different character sources to have a better knowledge about the species richness in faunistic studies, especially in groups with high number of species.
El bosque tropical caducifolio (BTC), también conocido como bosque tropical seco, es uno de los ecosistemas más diversos en el neotrópico y uno de los más amenazados (Janzen, 1988). Este tipo de bosque representa casi el 42% del área continental mundial con clima tropical y subtropical (Murphy y Lugo, 1995). En México es el ecosistema tropical mejor representado (Dirzo y Ceballos, 2010; Noguera et al., 2012). Posee una distribución característica de la vertiente del Pacifico mexicano, desde el norte en Sonora hasta la frontera con Guatemala en el estado de Chiapas (Varela-Espinosa, 2005; Rzedowski, 1978); cubre alrededor del 8% de su territorio y alberga un gran número de especies endémicas (Ceballos y García, 1995; Flores y Gerez, 1994; Noguera et al., 2012; Rzedowski, 1991; Toledo y Ordoñez, 1993). Trabajos previos han evidenciado la extraordinaria riqueza de especies de varios grupos de coleópteros en el BTC, incluyendo Cantharidae (Zaragoza-Caballero, 2000a,b, 2002); Lycidae, Phengodidae, Lampyridae y Cantharidae (Zaragoza-Caballero et al., 2003); Elateridae (Zurita-García, 2004) y Cantharidae, Lampyridae, Lycidae, Phengodidae y Telegeusidae (Zaragoza-Caballero y Ramírez-García, 2009).
Elateridae es una familia de coleópteros con gran diversidad de hábitos alimentarios; algunos géneros xilófagos son de importancia agrícola (Zurita-García, Johnson y Zaragoza-Caballero, 2014). A escala mundial, la familia está representada por más de 10,000 especies y 400 géneros (Costa, Lawrence y Rosa, 2010). De acuerdo con Bouchard et al. (2011), en México se han registrado 69 géneros agrupados en 15 subtribus, 29 tribus y 9 subfamilias. Varios estudios taxonómicos han descrito un gran número de especies de elatéridos para el país (ver referencias en Martínez-Luque, 2014), aunque hasta la fecha solo se ha realizado un estudio faunístico sistemático en la Reserva de la Biosfera Sierra de Huautla (RBSH) (Zurita-García, 2004). Posteriormente, se ha investigado la sistemática de la subtribu Agriotina (Zurita-García, 2007). Recientemente, Zurita-García et al. (2014) han presentado una síntesis de los registros de elatéridos mexicanos.
En el presente trabajo presentamos un inventario de las especies de Elateridae presentes en la Estación de Biología Chamela (EBCH), Jalisco, empleando evidencia morfológica y molecular (código de barras de la vida; Hebert, Cywinska, Ball y DeWaard, 2003; Hebert, Ratnasingham y deWaard, 2003). Esta reserva se encuentra dentro de la Reserva de la Biosfera de Chamela-Cuixmala (RBCC), la cual representa una de las regiones del país con mayor cobertura de BTC (Noguera, Vega-Rivera, García-Aldrete y Quesada-Avendaño, 2002).
Materiales y métodosEntre 2009 y 2012 se realizaron 7 salidas de campo. Se trató de incluir las épocas de lluvias y de secas, características del BTC. Las recolectas se realizaron dentro de la EBCH, propiedad del Instituto de Biología, Universidad Nacional Autónoma de México (IBUNAM). Esta estación está situada dentro de la RBCC, que se localiza en la costa del Pacífico mexicano, en el estado de Jalisco (entre 19°29’ y 19°34’ N, 104°58’ y 105°04’ O). Se utilizaron 6 técnicas de muestreo: trampas Malaise, recolectas directas, redes aéreas, trampas de luz negra y blanca, trampas funnel y trampas con cebo. El material fue preservado en etanol absoluto o al 96% y mantenido a −20°C hasta su procesamiento.
Los ejemplares se identificaron mediante claves y trabajos taxonómicos específicos para los diferentes grupos de elatéridos (Becker, 1973; Casari, 2002a,b,c; Champion, 1894-1896; Johnson, 2002; Muona, 1995; Vahtera, Jyrki y Lawrence, 2009; Wells, 2000). Los ejemplares recolectados, además, se compararon con ejemplares previamente determinados y depositados en la Colección Nacional de Insectos (CNIN) del IBUNAM. Se utilizó un microscopio marca Zeiss® con una lente de 2× para la observación de las estructuras de la morfología externa e interna. El material recolectado se encuentra depositado en la CNIN del IBUNAM.
Para la secuenciación de ADN se tomó una muestra de tejido (un trozo de pata) de un individuo de cada especie determinada morfológicamente. Cada muestra se colocó en etanol al 96% en uno de los 96 pocillos de placas de lisis. Las placas con muestras se enviaron después al Canadian Center for DNA Barcoding de la Universidad de Guelph en Ontario, Canadá, para su extracción de and, y amplificación y secuenciación del marcador genético seleccionado de acuerdo con los protocolos de laboratorio de Smith et al. (2008). El marcador genético secuenciado corresponde a un fragmento de 615-658 pares de bases del gen mitocondrial citocromo oxidasa subunidad I (COI). Este fragmento fue amplificado usando los primers LepF1/LepRI (Hebert, Penton, Burns, Janzen y Hallwachs, 2004) (LEP-F1: 5¿ATT CAA CCA ATC ATA AAG ATA T-3¿; LEP-R1: 5¿- TAA ACT TCT GCA TGT CCA AAA A-3¿). Las secuencias se editaron con el programa Sequencher® versión 4.9 y se alinearon de forma manual.
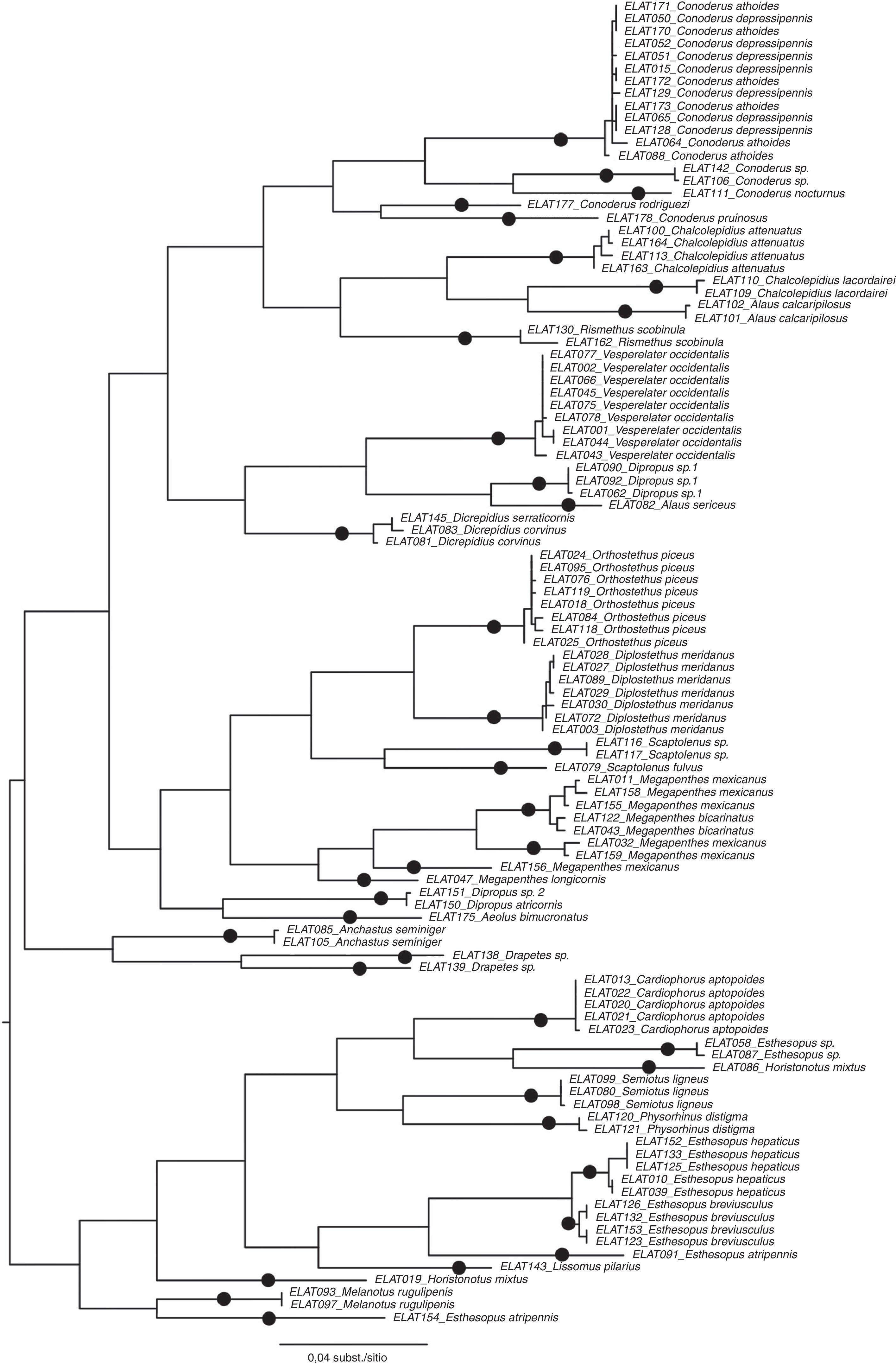
La delimitación de especies con base en las secuencias de COI se exploró con el enfoque del 2% de divergencia genética (2%DG; Hebert et al., 2003a; Hebert et al., 2003b). Las distancias entre cada par de secuencias se corrigieron empleando el modelo de evolución de Kimura-2 parámetros (K2P; Kimura, 1980) y se visualizaron construyendo un fenograma (fig. 3) con el método de neighbor-joining (NJ) implementado en BOLD (www.boldsystems.org). Los resultados obtenidos con la información molecular se compararon luego con las identificaciones morfológicas. Todas las secuencias generadas, así como los datos de recolecta y fotografías digitales de los especímenes secuenciados, están disponibles en el proyecto “Elateridae de Chamela, México” (ELAT project), en la sección de proyectos de la página web del Barcode of Life Data Systems (BOLD; www.barcodinglife.org).
Se recolectaron 544 ejemplares de Elateridae durante el trabajo de campo realizado entre 2009 y 2012. Se identificaron morfológicamente 46 especies agrupadas en 25 géneros (figs. 1 y 2), 7 subfamilias y 11 tribus. Se montaron 165 machos, de los cuales se extrajeron los genitales. El resto de los ejemplares se conservó en etanol al 96%.
Hábito dorsal de las especies que representan nuevos registros para Jalisco. 1, Rismethus scobinula Candèze, 1857; 2, Vesperelater occidentalis Champion, 1895; 3, Aeolus melliculus Candèze, 1859; 4, Conoderus pruinosus Champion, 1895; 5, C. rodriguezi Candèze, 1881; 6, C. depressipennis Candèze, 1859; 7, C. nocturnus Candèze, 1859; 8, Heteroderes gibbulus Champion, 1895; 9, Cardiophorus aptopoides Candèze, 1865; 10, Esthesopus hepaticus Eschscholtz, 1829; 11, E. breviusculus Champion, 1896; 12, E. atripennis Candèze, 1860; 13, Horistonotus mixtus Champion, 1895; 14, Drapetes sp.; 15, Lissomus sp.; 16, Agriotes mixtus Champion, 1896.
Hábito dorsal de las especies que representan nuevos registros para Jalisco (17-24) y para México (25-32). 17, Dicrepidius serraticornis Champion, 1894; 18, Diplostethus medirianus Champion, 1895; 19, Orthostethus piceus Candèze, 1853; 20, Melanotus rugulipenis Champion, 1895; 21, Physorhinus distigma Candèze, 1859; 22, Megapenthes mexicanus Champion, 1895; 23, M. cincticollis Champion, 1895; 24, M. longicornis Schaeffer, 1926; 25, Lacon pollinaria Candèze, 1857; 26, Aeolus bimucronatus Champion, 1895; 27, Conoderus athoides LeConte, 1863; 28, Scaptolenus fulvus Chevrolat 1874; 29, Anchastus seminiger Champion, 1895; 30, Dipropus atricornis Champion, 1895; 31, Megapenthes bicarinatus Lewis, 1894; 32, M. texanus Becker, 1971.
La subfamilia representada por un mayor número de especies es Elaterinae, con 18 especies (tabla 1). La subfamilia que sigue en mayor número de especies es Agrypninae con 16 especies, seguida por Cardiophorinae con 6, Cebrioninae y Lissominae con 2, y Semiotinae y Thylacosterninae con una. Cinco registros de especies son nuevos para el territorio mexicano: Lacon pollinaria (Candeze, 1857), Aeolus bimucronatus Champion, 1895, Conoderus athoides (LeConte, 1863), Scaptolenus fulvus Chevrolat, 1874, Anchastus seminiger Champion, 1895, Dipropus atricornis (Champion, 1895), Megapenthes bicarinatus Lewis, 1894 y M. texanus Becker, 1971. Se obtuvieron 7 especies probablemente no descritas, una correspondiente a los géneros Conoderus Eschscholtz, 1829, Esthesopus Eschscholtz, 1829 y Scaptolenus LeConte, 1854, así como 2 especies para Dipropus Germar, 1839 y 2 especies para Anchastus LeConte, 1853.
Lista de las especies de Elateridae registradas en la Estación de Biología, Chamela, Jalisco, México, de acuerdo con la clasificación taxonómica de Bouchard et al. (2011). Incluye nuevos registros para el estado de Jalisco con un asterisco (*) y con un punto (●) los nuevos registros para México. De igual manera muestra el número de ejemplares delimitados con evidencia molecular (2% de divergencia genética) por especie, el número de ejemplares delimitados con evidencia morfológica, el número de congruencias obtenidas entre ambas especies, y el número de ejemplares de los que se obtuvieron secuencias genéticas (COI), y entre paréntesis el número total de ejemplares secuenciados por especie.
| Especies | Evidencia molecular (2% DG) (Spp.) | Evidencia morfológica (SPP) | Número de congruencias (Spp.) | Número de ejemplares sec. por Spp. |
|---|---|---|---|---|
| SUBFAMILIA Agrypninae Candèze 1857 TRIBU Agrypnini Candèze 1857 | ||||
| Rismethus Fleutiaux, 1947 | 1 | 1 | 1 | |
| *Rismethus scobinula Candèze, 1857 | 2 (9) | |||
| Lacon Laporte 1836 | - | 1 | - | |
| ●Lacon pollinaria Candèze, 1857 | 0 (3) | |||
| TRIBU Pyrophorini Candèze, 1863 | ||||
| SUBTRIBU Pyrophorina | ||||
| Vesperelater Costa, 1975 | 1 | 1 | 1 | |
| *Vesperelater occidentalis Champion, 1895 | 9 (10) | |||
| TRIBU Oophorini Gistel 1856 [= Conoderini Fleutiaux 1919 (1859)] | ||||
| Aeolus Candèze, 1859 | 1 | 2 | 1 | |
| ●Aeolus bimucronatus Champion, 1895 | 1 (2) | |||
| *Aeolus melliculus Candèze, 1859 | 0 (1) | |||
| Conoderus Eschscholtz, 1829 | 5 | 6 | 4 | |
| *Conoderus athoides LeConte, 1863 | 8 (11) | |||
| *Conoderus pruinosus Champion, 1895 | 1 (1) | |||
| *Conoderus rodriguezi Candèze, 1881 | 1 (3) | |||
| *Conoderus depressipennis Candèze, 1859 | 9 (10) | |||
| *Conoderus nocturnus Candèze, 1859 | 1 (3) | |||
| Conoderus sp. | 2 (2) | |||
| Heteroderes Latreille, 1834 | - | 1 | - | |
| *Heteroderes gibbulus Champion, 1895 | 0 (1) | |||
| TRIBU Hemirhipini Candèze 1857 | ||||
| Chalcolepidius Eschscholtlz, 1829 | 2 | 2 | 2 | |
| Chalcolepidius lacordairii Candèze, 1857 | 2 (2) | |||
| Chalcolepidius attenuatus Erichson, 1841 | 4 (4) | |||
| Alaus Eschscholtz, 1829 | 2 | 2 | 1 | |
| Alaus sericeus Casari, 2003 | 1 (1) | |||
| Alaus calcaripilosus Casari, 1996 | 4 (4) | |||
| SUBFAMILIA Cardiophorinae Candèze, 1859 | ||||
| Cardiophorus Candèze, 1860 | 1 | 1 | 1 | |
| *Cardiophorus aptopoides Candèze, 1865 | 5 (5) | |||
| Esthesopus Eschscholtz, 1829 | 5 | 4 | 3 | |
| *Esthesopus hepaticus Eschscholtz, 1829 | 5 (5) | |||
| *Esthesopus breviusculus Champion, 1896 | 3 (4) | |||
| *Esthesopus atripennis Candèze, 1860 | 2 (2) | |||
| Esthesopus sp. | ||||
| Horistonotus Candèze, 1860 | 2 | 1 | 1 | |
| *Horistonotus mixtus Champion, 1895 | 2 (6) | |||
| SUBFAMILIA Semiotinae Jakobson, 1913 | ||||
| Semiotus Eschschltz, 1829 | 1 | 1 | 1 | |
| Semiotus ligneus Linnaeus, 1758 | 4 (6) | |||
| SUBFAMILIA Cebrioninae Latreille, 1802 | ||||
| Scaptolenus LeConte, 1854 | 2 | 2 | 2 | |
| ●Scaptolenus fulvus Chevrolat 1874 | 1 (5) | |||
| Scaptolenus sp. | 2 (2) | |||
| SUBFAMILIA Lissominae Laporte, 1835 | ||||
| Drapetes Redtenbacher, 1849 | 2 | 1 | 1 | |
| *Drapetes sp. | 2 (2) | |||
| Lissomus Dalman, 1824 | 1 | 1 | 1 | |
| *Lissomus sp. | 1 (1) | |||
| SUBFAMILIA Elaterinae Leach, 1815 TRUBU Agriotini Laporte, 1840 SUBTRIBU Agriotina Laporte, 1840 | ||||
| Agriotes Eschscholtz, 1829 | - | 1 | - | 0 (1) |
| *Agriotes mixtus Champion, 1896 | 0 (1) | |||
| TRIBU Physorhinini Candèze, 1859 | ||||
| Anchastus LeConte, 1853 | 1 | 3 | 1 | |
| ●Anchastus seminiger Champion, 1895 | 2 (2) | |||
| Anchastus sp. 1 | 0 (1) | |||
| Anchastus sp. 2 | 0 (1) | |||
| TRIBU Dicrepidiini Thomson, 1858 | ||||
| Dicrepidius Eschscholtz, 1829 | 1 | 2 | 2 | |
| *Dicrepidius serraticornis Champion, 1894 | 2 (2) | |||
| *Dicrepidius corvinus Candèze, 1859 | 3 (4) | |||
| Dipropus Germar, 1839 | 2 | 3 | 2 | |
| ●Dipropus atricornis Champion, 1895 | 1 (2) | |||
| Dipropus sp. 2 | 1 (3) | |||
| Dipropus sp. 1 | 4 (13) | |||
| TRIBU Elaterini Leach, 1815 | ||||
| Diplostethus Schwarz, 1907 | 1 | 1 | 1 | |
| *Diplostethus medirianus Champion, 1895 | 8 (13) | |||
| Orthostethus Lacordaere, 1857 | 1 | 1 | 1 | |
| *Orthostethus piceus Candèze, 1853 | 8 (9) | |||
| TRIBU Melanotini Candèze, 1859 | ||||
| Melanotus Eschscholtz, 1829 | 1 | 1 | 1 | |
| *Melanotus rugulipenis Champion, 1895 | 2 (2) | |||
| TRIBU Physorhinini Candèze, 1859 | ||||
| Physorhinus Germar, 1840 | 1 | 1 | 1 | |
| *Physorhinus distigma Candèze, 1859 | 2 (2) | |||
| TRIBU Megaphenthini Gurjeva, 1973 | ||||
| Megapenthes Kiesenwetter, 1863 | 4 | 5 | 1 | |
| ●Megapenthes bicarinatus Lewis, 1894 | 2 (4) | |||
| *Megapenthes mexicanus Champion, 1895 | 6 (6) | |||
| *Megapenthes cincticollis Champion, 1895 | 0 (1) | |||
| *Megapenthes longicornis Schaeffer, 1926 | 2 (8) | |||
| ●Megapenthes texanus Becker, 1971 | 0 (2) | |||
| SUBFAMILIA Thylacosterninae Fleutiaux, 1920 | ||||
| Thylacosternus Bonvouloir, 1870 | - | 1 | - | |
| *Thylacosternus sp. | 0 (1) | |||
| TOTAL= | 38 | 46 | 30 | 114 (169) |
Se obtuvo un total de 114 secuencias de COI, 106 de las cuales pudieron ser editadas e incluidas en el análisis de distancias. Las secuencias analizadas correspondieron a 30 de las 46 especies originalmente identificadas con base en la evidencia morfológica. En la tabla 2 se presentan las divergencias genéticas obtenidas para las especies delimitadas con el criterio de 2% de divergencia genética. En la figura 3 se presenta el árbol de NJ en donde se visualizan las distancias corregidas obtenidas con el modelo K2P.
Porcentajes de divergencia genética del locus del código de barras (COI) para las 27 especies de Elateridae con más de un ejemplar secuenciado.
| Especie | DG% |
|---|---|
| Alaus calcaripilosus | 0.1 |
| Anchastus seminiger | 0.1 |
| Cardiophorus aptopoides | 0-0.1 |
| Chalcolepidius attenuatus | 0-0.6 |
| Chalcolepidius lacordairei | 0.3 |
| Conoderus athoides | 0-0.3 |
| Conoderus depressipennis | 0-0.5 |
| Conoderus sp. | 0.1 |
| Drapetes sp. | 17.5 |
| Dicrepidius corvinus | 0.2 |
| Dicrepidius meridianus | 0.2 |
| Dipropus sp. 1 | 0 |
| Diplostethus meridanus | 0-0.2 |
| Esthesopus atripennis | 24.8 |
| Esthesopus breviusculus | 0-0.4 |
| Esthesopus hepaticus | 0-0.9 |
| Esthesopus sp. | 0.2 |
| Horistonotus mixtus | 23.9 |
| Megapenthes bicarinatus | 0.6 |
| Megapenthes mexicanus | 0.1-11.9 |
| Melanotus rugulipenis | 0 |
| Orthostethus piceus | 0-0.2 |
| Physorhinus distigma | 0.1 |
| Rismethus scobinula | 0.1 |
| Scaptolenus sp. | 0 |
| Semiotus ligneus | 0-0.1 |
| Vesperelater occidentalis | 0-0.4 |
Treinta y ocho especies fueron delimitadas molecularmente empleando el criterio de 2% de divergencia genética propuesto por Hebert et al., 2003b. De éstas, 30 resultaron corroboradas con las identificaciones morfológicas, per hubo 10 incongruencias tanto por la fusión de 2 especies identificadas con morfología como por la división de una especie en 2 o más especies (tablas 1 y 2). La congruencia encontrada entre la información morfológica y la molecular permitió corroborar la existencia de 6 de las 7 especies aparentemente nuevas para la ciencia pertenecientes a los géneros Conoderus (1 sp.), Scaptolenus (1 sp.), Dipropus (1 sp.), Esthesopus (1 sp.) y Anchastus (2 spp.). Entre las incongruencias encontradas con evidencia morfológica y molecular, algunas especies delimitadas con caracteres de morfología externa y genitales de machos aparecieron fusionadas con evidencia molecular. Tal es el caso de C. athoides y C. depressipennis (Candèze, 1859), cuyos ejemplares secuenciados aparecieron con distancias genéticas muy bajas (hasta 0.5%). La evidencia molecular también muestra que Dipropus atricornis y Dipropus sp. 2 representan una misma especie, con una divergencia genética de 0.1%. Por otra parte, la información molecular separó a los 2 organismos asignados a Horistonotus mixtus Champion, 1895 y a los 2 asignados a E. atripennis Candeze, 1860 en 2 especies distintas cada una, con una divergencia genética entre ellas de 23.9 y 24.8%, respectivamente, mientras que Drapetes sp. resultó dividida en 2 especies, con una divergencia genética observada de 17.5% entre sus 2 ejemplares secuenciados.
Durante las salidas al campo se observó que los especímenes de algunas especies y géneros se recolectaron específicamente con algún tipo de trampa. Por ejemplo, Semiotus ligneus Linnaeus, 1767 y Vesperelater occidentalis (Champion, 1896) se recolectaron únicamente con trampas de atracción luminosa (luz blanca). Las especies de Conoderus se recolectaron solo con luz negra y trampas Malaise, mientras que Chalcolepidius lacordairii Candèze, 1857 y C. attenuatus Erichson, 1841 se recolectaron siempre de manera directa. Rismethus scobinula (Candèze, 1857), así como las especies de Cardiophorus Eschscholtz, 1829, Megapenthes Kiesenwetter, 1858, Thylacosternus Bonvouloir, 1875, Drapetes Redtenbacher, 1849 y Esthesopus Eschscholtz, 1829 se recolectaron con red aérea en los arbustos localizados a los lados de los caminos y arroyos. El empleo de diferentes tipos de trampas maximizó la obtención de especies de elatéridos.
DiscusiónRiqueza de especies de ElateridaeLas Elateridae poseen gran variedad de hábitos alimentarios, lo que se expresa en la gran riqueza de especies y especialización en tamaños, ornamentaciones, estructuras morfológicas en antenas y patas, así como coloraciones corporales (Zurita-García et al., 2014), lo que les ha permitido que se encuentren en gran variedad de ecosistemas, altitudes y en casos extremos en condiciones desfavorables como sequías extremas utilizando microclimas (Deloya y Ordóñez, 2006). Sin embargo, el conocimiento taxonómico de esta familia es aún escaso. Durante los últimos 50 años, los trabajos realizados en México se han restringido a algunas zonas, y principalmente se ha enfocado a la revisión de material depositado en pocas colecciones entomológicas.
Con respecto a los estudios faunísticos para Elateridae en América, los trabajos se han restringido a ciertas zonas de estudio asociadas con los sitios visitados por los especialistas (Aguirre, 2009; Aguirre, Carrejo y Pardo-Locarno, 2010; Arias, 2000, 2013; Arias y Elgueta, 2012; Becker, 1966, 1979; Casari, 2002a,b; Champion., 1894-1896; Correa, Casari y Mermudes, 2011; Costa, Vanin y Casari, 1988; Lawrence y Arias, 2009; Silva, De Faria e Silva, Boieiro, Aguiar y Serrano, 2006; Rosa y Costa, 2009; Stibick, 1979). Estos estudios generalmente se han basado en caracteres de morfología externa en adultos, aunque algunos trabajos han empleado caracteres de morfología interna, incluyendo caracteres de edeagos y placas genitales de hembras, así como caracteres larvales (p. ej., Cate, 2007; Sánchez-Ruiz y Löbl, 2007). El presente trabajo representa el primer estudio faunístico para Elateridae que emplea evidencia de morfología interna y externa en adultos, además de evidencia molecular para tomar decisiones en la delimitación de especies.
Delimitación de especiesEl empleo del criterio de 2% DG se ha cuestionado, ya que asume que las tasas de sustitución del gen COI varían de manera uniforme en el tiempo en todos los taxones animales (Papadopoulou, Anastasiou y Vogler, 2010). No obstante, este procedimiento se emplea con frecuencia para la determinación de unidades taxonómicas, y ha demostrado ser una herramienta útil para la identificación de especies en la mayoría de los grupos de animales (Hajibabaei, Singer, Hebert y Hickey, 2007; Hebert, Cywinska, Ball y DeWaard, 2003a, b; Jones, Ghoorah y Blaxter, 2011). En particular, se ha observado que este criterio es útil para delimitar más del 95% de especies en estudios de varios grupos de insectos, incluyendo coleópteros (Chang et al., 2012). Actualmente, el uso de herramientas moleculares, incluyendo el locus del código de barras de la vida, ayuda a la toma de decisión para la delimitación de especies, ya que en ocasiones algunas especies no son morfológicamente diferenciables o solo presentan variaciones morfológicas que no serían referentes para la delimitación de especies. Por ejemplo, Etzler, Wanner, Morales-Rodriguez y Ivie (2014) utilizaron secuencias de COI para asociar estadios larvales de elatéridos del género Selatosomus Stephens, 1830 de importancia económica, los cuales son difíciles de determinar como especie. Basándose en sus resultados, los autores concluyeron que S. destructor Brown, 1935 es una especie distinta a S. aeripennis Kirby, 1837, ambas consideradas con problemas de delimitación taxonómica.
Con respecto a las incongruencias observadas entre la información morfológica y molecular para la delimitación de especies, estas podrían deberse a que la evidencia morfológica no esté bien sustentada, tomando en cuenta que posiblemente se han tomado caracteres taxonómicos poco útiles. Otra posible explicación es la existencia de diversidad críptica aún no descubierta, en donde la evolución morfológica con base en los caracteres diagnósticos podría ser lenta (Ornelas-García, Domínguez-Domínguez y Doadrio, 2008). Las incongruencias obtenidas entre la información morfológica y molecular apoyan la idea de que la evidencia taxonómica no debe estar basada únicamente en un tipo de carácter en casos problemáticos. En lo que se refiere a la evidencia molecular, la evidencia que otorga el gen COI puede verse afectada por fenómenos intrínsecos al genoma mitocondrial, tales como la separación incompleta de linajes o hibridación (Funk y Omland, 2003). Estos fenómenos pueden afectar la estructura de las topologías obtenidas con distintos métodos de reconstrucción filogenética, y causan que las especies aparezcan como parafiléticas empleando genes mitocondriales. Esto resalta la importancia de combinar distintos tipos de evidencia (p. ej. información morfológica, molecular, ecológica, etológica, ambiental, etc.) con un enfoque integrador (Padial, Miralles, De la Riva y Vences, 2010) y de esta manera tener más elementos para corroborar o refutar decisiones taxonómicas.
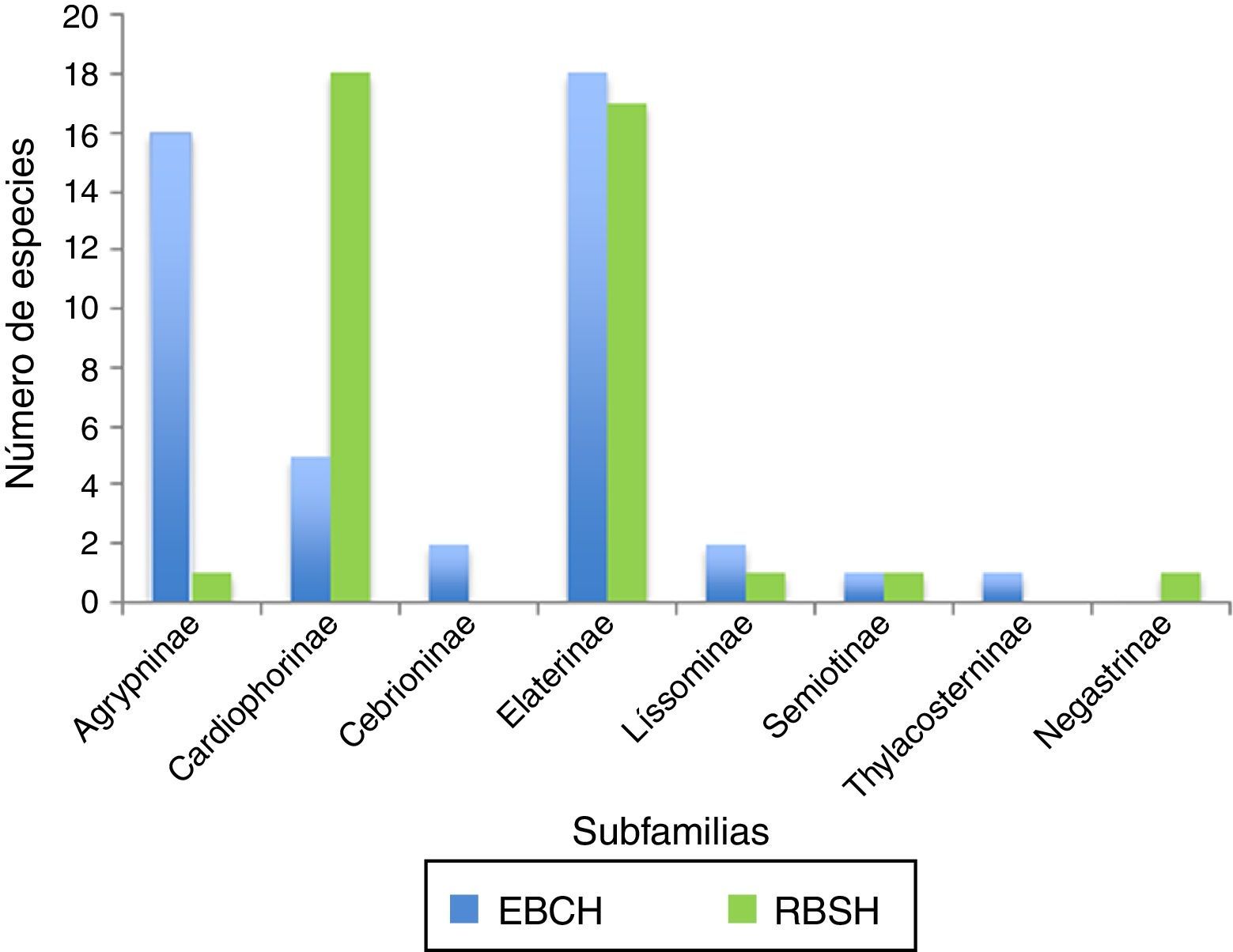
Con anterioridad a este estudio, solo se había realizado uno faunístico sistemático para Elateridae en México, llevado a cabo en la RBSH, en el estado de Morelos (Zurita-García, 2004). La vegetación en dicha reserva también está representada en su mayor parte por BTC. La comparación entre ambas regiones indica que comparten un total de 19 géneros y 6 subfamilias. Una subfamilia, Negastriinae, solo se encontró en la RBSH (fig. 4); 3 géneros fueron exclusivos para la RBSH (Meristhus Candèze, 1857, Aptopus Eschscholtz, 1829 y Paradonus Stibick, 1971), y 3 para la EBCH (Orthostethus Lacordaire, 1857, Agriotes Eschscholtz, 1829 y Cardiophorus Eschscholtz, 1829). A diferencia del trabajo realizado en la RBSH, para el presente estudio se consideró que Cebrioninae (Scaptolenus) y Thylacosterninae (Drapetes y Thylacosternus) eran subfamilias de Elateridae.
A Santiago Zaragoza, Adolfo Ibarra y Oscar Pérez por su ayuda para realizar el trabajo de campo, a Carlos Pedraza por su asesoría en el trabajo de laboratorio y el análisis de la información molecular, a Andrea Jiménez y Laura Márquez por la ayuda prestada en el laboratorio, a Cristina Mayorga y Guillermina Ortega por su apoyo en el trabajo realizado en la CNIN y a Susana Guzmán por la asistencia técnica en las fotografías digitales. Este estudio formó parte de la tesis de licenciatura de E. O. M. L., como parte de un proyecto de A. Z. R. financiado por el Consejo Nacional de Ciencia y Tecnología (Conacyt; Red temática del código de barras de la vida).
La revisión por pares es responsabilidad de la Universidad Nacional Autónoma de México.