Se empleó una muestra de 30 individuos de una población de Milla biflora de la Reserva Ecológica del Pedregal de San Ángel, en México, D. F., para analizar la arquitectura y comportamiento de los cromosomas mitóticos en células de meristemos radiculares. Se empleó una metodología de extendido en superficie y secado al aire (splash) que confirmó un número cromosómico 2n=6x=42 (x=7), permitió determinar la fórmula cariotípica de 6m+24sm+12st, la obtención de la longitud cromosómica total haploide (106.92μm), la talla cromosómica promedio (5.09μm) y el rango cromosómico (2.54−9.07μm). El índice de asimetría (TF%=32.45) corresponde a un cariotipo asimétrico, mismo que exhibe 3 tipos de cromosomas diferenciados por su talla y forma (trimodal). Las regiones NOR fueron asociadas a cromosomas st portadores de constricción secundaria y satélite. La presencia de rompimientos, fragmentos cromosómicos y heterocromosomas evidencian un mecanismo activo de rearreglos cromosómicos que moldean el cariotipo de Milla biflora y juegan un papel importante en la evolución de la especie. Su naturaleza autohexaploide se discute en función de la presencia de subgrupos cromosómicos, la ausencia de dominancia nucleolar y el papel de la redundancia génica.
Thirty individuals from one population of Milla biflora from Reserva Ecológica del Pedregal de San Ángel, Distrito Federal, Mexico were sampled to analyze the architecture and behavior of mitotic chromosomes in meristematic root cells. The use of the splash technique allowed the corroboration of the chromosome number 2n=6x=42 (x=7), and to propose the karyotype formula (6m+24sm+12st), to obtain the total haploid chromosome length (106.92μm) mean chromosome size (5.09μm) and chromosomal length range (2.54−9.07μm). The asymmetry index (TF%=32.45) correspond to asymmetric karyotype exhibiting 3 differentiated chromosomal types by their shape and size (trimodal). The NOR regions were associated to st chromosomes bearing a secondary constriction and satellite. Chromosome fragments and heterochromosomes show an active mechanism of chromosomal rearrangements that mold the karyotype of M. biflora and play an important role in the evolution of the species. Its autohexaploide origin is discussed in function of the presence of chromosome subgroups, absence of nucleolar dominance and the role of gene redundancy.
Milla Cav. es un género americano cormoso alguna vez ubicado dentro de las familias Liliaceae (Cronquist, 1981), Alliaceae (Dahlgren et al., 1985) o Themidaceae (Fay y Chase, 1996; Pires y Sytsma, 2002). Actualmente está situado en Asparagaceae, dentro del clado Milla de la subfamilia Brodiaeoideae Traub (APG III, 2009). El clado Milla incluye a los géneros Bessera, Dandya, Jaimehintonia, Milla y Petronymphe (Moore, 1953; Pires y Sytsma, 2002). El género Milla se distribuye desde el sur de Estados Unidos hasta América Central (Espejo-Serna y López-Ferrari, 2003) y agrupa 10 especies, todas representadas en México y con excepción de M. biflora, todos son taxa endémicos del país (Moore, 1953; Lenz, 1971; Howard, 1999; Pires et al., 2001).
Milla biflora Cavanilles, conocida también como estrellita, estrella mexicana, Mexican star, azúcar de campo, guije-cana, flor de San Juan, flor de mayo, esh-pi-sua, San Nicolás, es una hierba criptofita (geófita) y hermafrodita, localmente abundante en el suroeste de Estados Unidos, Arizona y Nuevo México, y con poblaciones que se extienden hasta Guatemala. En México se localiza en la mayoría de los estados del norte y centro del país en elevaciones medias desde 1 200 a 2 200m snm y, en ocasiones, hasta 2 700m snm en áreas montañosas asociadas a pastizales desérticos y chaparral. Posee flores blancas brillantes, actinomorfas, aromáticas, en forma de estrella y hojas lineares delgadas. El pedículo floral puede alcanzar hasta 60cm de altura, con flores de 6cm de ancho. M. biflora puede producir hasta 6 flores por cormo en especímenes excepcionales, aunque usualmente son sólo 2 flores que abren en secuencia. Dependiendo de las lluvias y probablemente de otros factores florece de junio a septiembre. El fruto es una cápsula con 3 ángulos. Los cormos son superficiales o poco profundos y crecen en áreas rocosas con buen drenaje y en pleno sol. Encuentra usos en la medicina tradicional, pues con la flor se prepara un té para calmar la tos y también se le usa como ornato (Martínez, 1979; Rojo y Rodríguez, 2002; Espejo-Serna y López-Ferrari, 2003; McNeal, 2003).
En la literatura especializada no se encuentra información detallada sobre la arquitectura cromosómica en especies del género Milla y sólo se registran recuentos cromosómicos donde sobresalen algunas especies con números diploides variables como M. mexicana con 2n=16 y 54, y M. potosina con 2n=18, 32 y 42, en tanto, que otras exhiben un solo número diploide como es el caso de M. oaxacana, 2n=16 y M. rosea, 2n=20 (Howard, 2001). En relación con M. biflora se han registrado recuentos, tales como 2n=14, 28 y 42, que corresponden a una serie poliploide con número base x=7, con diploides, tetraploides y hexaploides. En otros 2 recuentos se ha observado un 2n=16 y 48 (Lenz, 1971).
En la presente investigación se realizó un análisis citogenético empleando una metodología de extendido y secado al aire del tejido meristemático para estudiar y describir el cariotipo, establecer la fórmula cariotípica y otras características cuantitativas del complemento cromosómico de una muestra de individuos de M. biflora de la Reserva Ecológica del Pedregal de San Ángel (REPSA) en México, D. F. y, de esta manera, obtener datos citológicos fundamentales del sistema genético de esta especie, la cual presenta la mayor distribución geográfica dentro del género y es común en dicha reserva.
Materiales y métodosLos frutos de M. biflora se recolectaron durante los meses de octubre de 2007 y 2008 en un área de amortiguamiento; A5, Paseo de las Esculturas, de la Reserva Ecológica del Pedregal de San Ángel (REPSA) de Ciudad Universitaria, en el sur de la cuenca del valle de México; entre 19°20'2” y 19°13'45” N, 99°08'26” y 99°14'3” O; 2 250 a 2 350m snm. Semillas seleccionadas al azar provenientes de al menos 30 individuos fueron puestas a germinar a temperatura ambiente y en luz natural en cajas de Petri con algodón humedecido con agua destilada. Las raíces de 2–4mm de largo fueron cortadas y pretratadas en 8-hidroxiquinoleína 0.002M durante 5h a temperatura ambiente y en oscuridad. Posteriormente, fueron fijadas en solución Farmer; etanol-ácido acético, 3:1. Para la obtención de los cromosomas en metafase se siguió el método de secado al aire propuesto por Tapia-Pastrana y Mercado-Ruaro (2001), el cual se basa en la maceración enzimática (pectinasa 20%+celulasa 2%) de los meristemos apicales durante 2h a 37° C, seguido de la transferencia de los protoplastos a una solución fresca de KCl 0.075M durante 15–20 minutos a 37° C. Posteriormente, se realizaron 2 lavados con la misma solución y el botón celular fue fijado en solución Farmer. Dos gotas del botón celular se colocaron sobre portaobjetos limpios y la tinción de los cromosomas se realizó con Giemsa al 10%. Las preparaciones se hicieron permanentes empleando Entellán Nuevo (Merck) o resina sintética (Sigma) como medios de montaje. Los mejores campos fueron fotografiados con un microscopio óptico Zeiss Axioscop, usando película Kodak Technical Pan.
Se utilizó un vernier digital (Mitutoyo Digimatic Caliber CD-G”BS) para establecer las tallas cromosómicas y otras medidas morfométricas sobre 5 fotografías de placas en metafase típica con similar grado de condensación y amplificadas con la misma magnificación. Para la obtención de la fórmula cariotípica y nomenclatura, se aplicó el sistema propuesto por Levan et al. (1964). La proporción de brazos, r, se obtuvo mediante el cociente q/p, valor que muestra la relación del brazo largo entre el brazo corto de los cromosomas individuales. La proporción de la suma total de longitudes de brazos cortos respecto a la suma total de longitudes cromosómicas como indicador de simetría o asimetría de un cariotipo (TF%) se realizó según Sinha y Roy (1979).
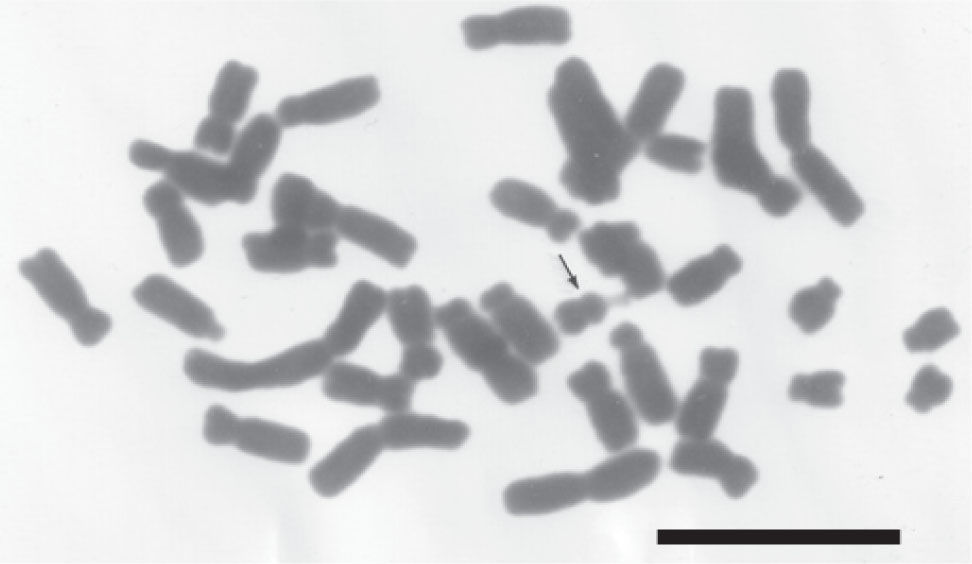
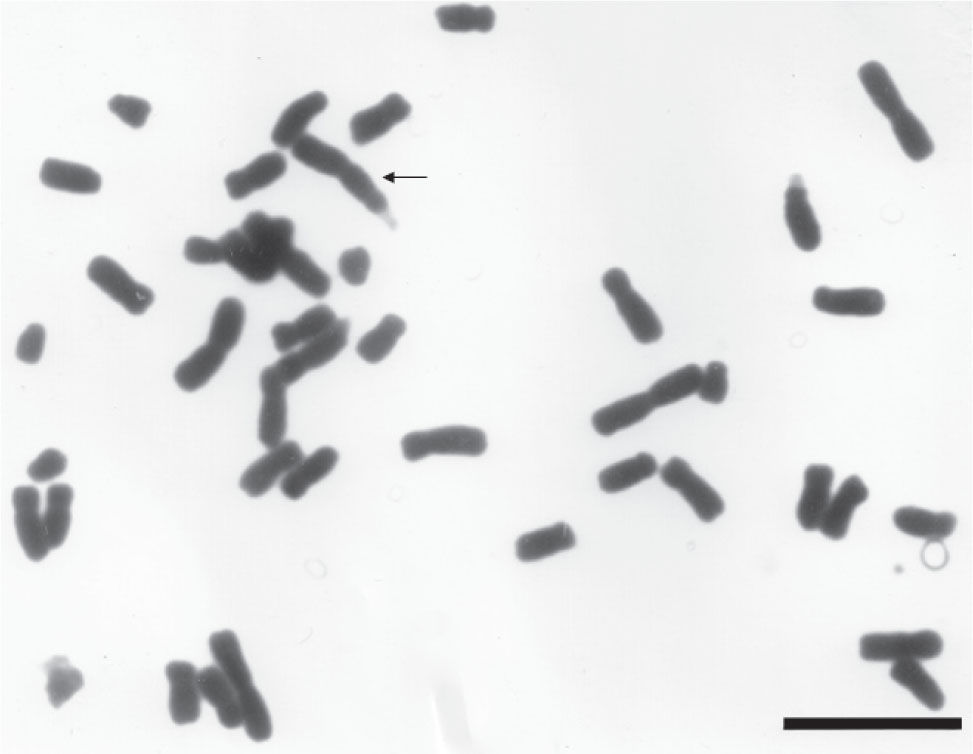
ResultadosSe observaron un total de 223 células en metafase, de las cuales 208 exhibieron un 2n=6x=42 (Fig. 1); 4 mostraron un 2n=41 (Fig. 2); en 4 células se observó un 2n=43 (Fig. 3) y en 7 células se apreciaron 1 o 2 fragmentos cromosómicos de diversos tamaños (Fig. 4). La fórmula cariotípica obtenida fue 6m+24sm+12st (Fig. 5A, B; Cuadro 1). La longitud cromosómica total haploide media (LCTH) fue 106.92±5.87μm y el tamaño cromosómico promedio se estableció como 5.09±0.55μm, con un rango (diferencia de longitudes entre los cromosomas mayor y menor) de 6.53μm. El índice de asimetría (TF%=32.45) señala un complemento cromosómico asimétrico. El complemento cromosómico de M. biflora es claramente trimodal. En su cariotipo resaltan 3 pares de cromosomas metacéntricos (m) notoriamente grandes (ca. 9μm) y 3 pares de cromosomas comparativamente más pequeños con centrómero desplazado (sm) de entre 2 y 3μm (Cuadro 1). Un grupo intermedio está conformado por cromosomas submetacéntricos (sm) y subtelocéntricos (st) con tallas de entre 3.5–6.0μm (Figs. 5A, B, 6). Asimismo y sin ambigüedad, fue posible reconocer la presencia de una a 6 constricciones secundarias asociadas a satélites de diferentes tamaños, todas situadas en los brazos cortos de cromosomas st (Fig. 7). Las células en prometafase exhibieron un número máximo de 2 nucleolos de apariencia grande, cada uno asociado exclusivamente con un cromosoma st, cuya constricción secundaria y satélite se encontraron adyacentes, e incluso inmersos en la matriz nucleolar (Fig. 8).
Medidas cromosómicas promedio en Milla biflora obtenidas de 6 células en metafase mitótica. Abreviaturas: PC= par cromosómico; LC= longitud cromosómica en μm; LBL= longitud brazo largo en μm; LBC= longitud brazo corto en μm; r= proporción del brazo largo (q) entre el brazo corto (p) de los cromosomas; N= Nomenclatura; *= cromosoma con constricción secundaria y satélites; m= metacéntrico; sm= submetacéntrico; st= subtelocéntrico
| PC | LC | LBL(q) | LBC(p) | R | N |
| 1 | 9.07 | 4.76 | 4.30 | 1.10 | m |
| 2 | 8.63 | 4.57 | 4.11 | 1.11 | m |
| 3 | 8.26 | 4.40 | 3.85 | 1.14 | m |
| 4 | 6.06 | 4.16 | 1.90 | 2.18 | sm |
| 5 | 5.71 | 3.89 | 1.82 | 2.13 | sm |
| 6 | 5.58 | 4.52 | 1.04 | 4.34 | st* |
| 7 | 5.50 | 3.71 | 1.79 | 2.07 | sm |
| 8 | 5.19 | 3.61 | 1.66 | 2.17 | sm |
| 9 | 5.03 | 4.08 | 0.93 | 4.32 | st |
| 10 | 4.98 | 3.36 | 1.63 | 2.06 | sm |
| 11 | 4.87 | 3.34 | 1.53 | 2.18 | sm |
| 12 | 4.77 | 3.19 | 1.59 | 2.00 | sm |
| 13 | 4.72 | 3.85 | 0.86 | 4.47 | st* |
| 14 | 4.57 | 3.15 | 1.43 | 2.20 | sm |
| 15 | 4.36 | 3.06 | 1.35 | 2.26 | sm |
| 16 | 4.12 | 3.25 | 0.86 | 3.77 | st |
| 17 | 3.85 | 2.96 | 0.89 | 3.32 | st |
| 18 | 3.50 | 2.75 | 0.74 | 3.71 | st* |
| 19 | 2.94 | 2.07 | 0.89 | 2.32 | sm |
| 20 | 2.67 | 1.87 | 0.80 | 2.33 | sm |
| 21 | 2.54 | 1.79 | 0.73 | 2.45 | sm |
Por otra parte, se identificaron con claridad sitios de rompimientos en cromosomas de diferentes tamaños (Figs. 9, 10); asimismo, se reconocieron cromosomas que no correspondieron con la fórmula cariotípica propuesta en este estudio, esto es, no coincidieron con la morfología o talla de alguno de los 3 tipos básicos que conforman el complemento cromosómico. Tales cromosomas excepcionales o heterocromosomas fueron semejantes a cromosomas metacéntricos chicos o grandes que ocasionalmente portaron constricciones secundarias (Figs. 10, 11, 12), y a submetacéntricos ligeramente mayores a los 6 más pequeños del complemento (Fig. 13).
El número cromosómico 2n=42 registrado en estas investigaciones para M. biflora coincide con recuentos obtenidos anteriormente para esta especie y corresponde al de una población hexaploide con un número básico x=7, lo que confirma la existencia de una serie poliploide exhibida dentro del amplio intervalo de distribución de este taxón (Lenz, 1966, 1971). Asimismo, concuerda con x=7 que ha sido reconocido como el número básico de las angiospermas basales y predominante en las monocotiledóneas (Raven, 1975; Dahlgren et al., 1985).
La fórmula cariotípica de 6m+24sm+12st para la población de M. biflora estudiada aquí, representa la primera que se obtiene para cualquiera de sus poblaciones y primera también para cualquiera de las especies del género. En relación con las tallas cromosómicas, una LCTH de 106.92±5.87μm con un tamaño cromosómico promedio de 5.09±0.55μm, señala una característica ya conocida en las monocotiledóneas cormosas, es decir, la presencia de genomas particularmente grandes con probable significado nucleotípico. La fórmula cariotípica, así como la obtención del rango cromosómico (6.53μm) y del índice de asimetría (TF%=32.79) añaden datos cuantitativos al sistema genético de M. biflora, tan escasos en las descripciones cariotípicas de monocotiledóneas.
El carácter trimodal, es decir, la presencia de 3 grupos de cromosomas bien caracterizados, con tallas claramente diferentes, observado en el cariotipo de M. biflora, ha sido descrito con anterioridad en Asparagales en los géneros Aspidistra, Galtonia, Pseudogaltonia, Maianthemum y otros géneros pertenecientes a otros órdenes y familias (Craspedolobium, Fabaceae) y el significado genético y taxonómico de este fenómeno queda por resolver (Forrest y Jong, 2004; Meng et al., 2005; Lin et al., 2010; Xu et al., 2011).
La identificación clara de grupos de 6 cromosomas homólogos observada en los cromosomas metacéntricos (m) grandes y en los submetacéntricos (sm) chicos (Figs. 5A, B) se consideró aquí como una evidencia de la naturaleza autohexaploide de la población estudiada, donde se observaron 6 homólogos para cada grupo de cromosomas del complemento. La idea anterior se reforzó con la observación frecuente de 6 cromosomas subtelocéntricos (st) portadores de constricciones secundarias y satélites (Fig. 7) que demuestra, además, la ausencia de supresión de la función nucleolar por amfiplastía o dominancia nucleolar, habitualmente manifiesta en especies alopoliploides (Navashin, 1928; Preuss y Pikaard, 2007; Tapia-Pastrana, 2012).
En general, la presencia de constricciones secundarias en un complemento se considera indicativo de regiones NOR activas (Tapia-Pastrana y Mercado-Ruaro, 2001; Tapia-Pastrana, 2007), sin embargo, en la población analizada el número máximo de nucleolos observados en prometafase fue de 2, lo que indica por una parte, que los cromosomas st asociados a éstos son los cromosomas NOR (Fig. 8) y, por otra, que la redundancia genética por copias extras de ciertas secuencias no compromete funciones esenciales en los poliploides.
En estas investigaciones, la identificación de 3 tipos básicos de cromosomas en M. biflora a partir de la ubicación del centrómero, descarta la existencia de cromosomas holocéntricos o con centrómero difuso, fenómeno que la investigación citogenética registra como una característica más común en monocotiledóneas (Leitch et al., 2010) que en el resto de las angiospermas: Cyperaceae (Heilborn, 1924), Juncaceae (Malheiros-Gardé et al., 1947; Heckmann et al., 2011) y el género Chionographis de Melanthiaceae (Tanaka y Tanaka, 1977), ejemplifican lo anterior, aunque la frecuencia del evento aún se desconoce (Greilhuber, 1995).
Al analizar el complemento cromosómico en algunos individuos de la población de M. biflora estudiada, se observó la presencia de fragmentos cromosómicos de diferentes tamaños, la existencia de rompimientos y de heterocromosomas, mismos que se consideraron como evidencia de rearreglos cromosómicos donde la probable participación de rompimientos y translocaciones desiguales es un mecanismo frecuente, cuyo resultado es la falta de homología en algunos grupos de cromosomas y la consecuente dificultad para aparearlos por semejanza al construir el cariotipo de esta especie (Figs. 5A, B). Los resultados de estas investigaciones apoyan la opinión de Stebbins (1971) sobre el papel de las translocaciones desiguales progresivas entre cromosomas, que al favorecer el acortamiento de algunos cromosomas y el alargamiento de otros, resultaron en nuevas combinaciones alélicas que han explicado el origen bimodal en los cariotipos de las monocotiledóneas. En efecto, las especies poliploides frecuentemente muestran gran cantidad de cambios cromosómicos que producen nuevas combinaciones de ADN en sus genomas (Song et al., 1995; Arnold, 1997).
Es importante señalar que la información derivada de los estudios citogenéticos en algunas familias de monocotiledóneas, tales como Poaceae, Commelinaceae y Asparagaceae ha contribuido de manera sustancial en la estimación de las relaciones filogenéticas (Raven, 1975; Dahlgren et al., 1985) y que el número cromosómico por sí solo ha sido útil para analizar la posición taxonómica de especies incluidas en otros géneros de la subfamilia Brodiaeoideae como Bloomeria y Triteleia. En este taxón, especies polimórficas exhiben poliploidía y números básicos de x=7 y x=8, lo cual sugiere que los cambios en el número cromosómico juegan un papel significativo en la evolución de estas especies (Burbank, 1941, 1944).
Si bien, el análisis citogenético tiene poca aplicación en niveles superiores de la taxonomía, debido a la plasticidad evolutiva y a datos morfológicos limitados, en las monocotiledóneas puede suministrar información útil particularmente por abajo del nivel de género (Greilhuber, 1995). De esta forma, es claro que el estudio cromosómico detallado en especies incluidas dentro del género Milla puede sumarse ahora a la lista de caracteres morfológicos confiables para la construcción de hipótesis evolutivas en Brodiaeoideae, particularmente si se considera que los intentos por resolver sus relaciones filogenéticas empleando secuencias de cloroplasto, tales como trnL-F, rp116 y ndhF han presentado escasa variación (Pires y Sytsma, 2002).
Finalmente, este estudio apoya la opinión de Greilhuber (1995), quien señala que en la era posgenómica de la secuenciación a gran escala y los análisis comparativos, la disponibilidad de grandes cantidades de información de secuencias junto con cantidades crecientes de información citogenética derivada del estudio cuidadoso de los cromosomas, suministrará nueva luz sobre las hipótesis de relación o filogenias.