¿ INTRODUCCIÓN
El cáncer de próstata (CaP) es la segunda neoplasia maligna más frecuente en el varón y ocasiona cerca de 6% de las muertes por cáncer a nivel mundial (OMS, GLOBOCAN, 2008).1 El CaP según su agresividad clínica y grado de invasión se puede clasificar en no agresivo (crecimiento lento, poca o nula actividad metastásica y alta supervivencia) y agresivo (progreso rápido, metastásico y escasa o nula supervivencia después de cinco años de su diagnóstico). Los estadios iniciales de esta patología representan un reto clínico, debido a la carencia de biomarcadores disponibles para definir una posible evolución tumoral, para definir el pronóstico y para guiar las decisiones terapéuticas.
¿ OBJETIVO
Identificar loci asociados con una evolución agresiva o no agresiva de CaP detectable en tumores primarios para el desarrollo de un perfil genómico de biomarcadores del tejido prostático que permitan la clasificación de CaP en agresivo o no agresivo en pacientes con CaP en etapas iniciales y de diagnóstico reciente.
¿ MÉTODOS
Se realizó un análisis retrospectivo de ganancias y pérdidas de regiones cromosómicas en muestras de ADN genómico aislado de tejido prostático embebido en parafina de pacientes con CaP agresivo (con metástasis mínimo a hueso) (n = 15) y no agresivo (más de cinco años de diagnóstico de CaP sin evidencia aparente de metástasis) (n = 8) usando la técnica de microarreglos de hibridación genómica comparativa (aCGH). La plataforma de hibridación está constituida por 3052 clonas de BACs únicas diseñadas para detectar desbalances en el número de copias a lo largo del genoma nuclear humano. Para el análisis de resultados se recurrió al agrupamiento no supervisado (centroides k-promedio) y supervisado (centroides reducidos) por medio del software R, función r- bioconductor y para validar los resultados se implementó la prueba de validación cruzada.
¿ RESULTADOS
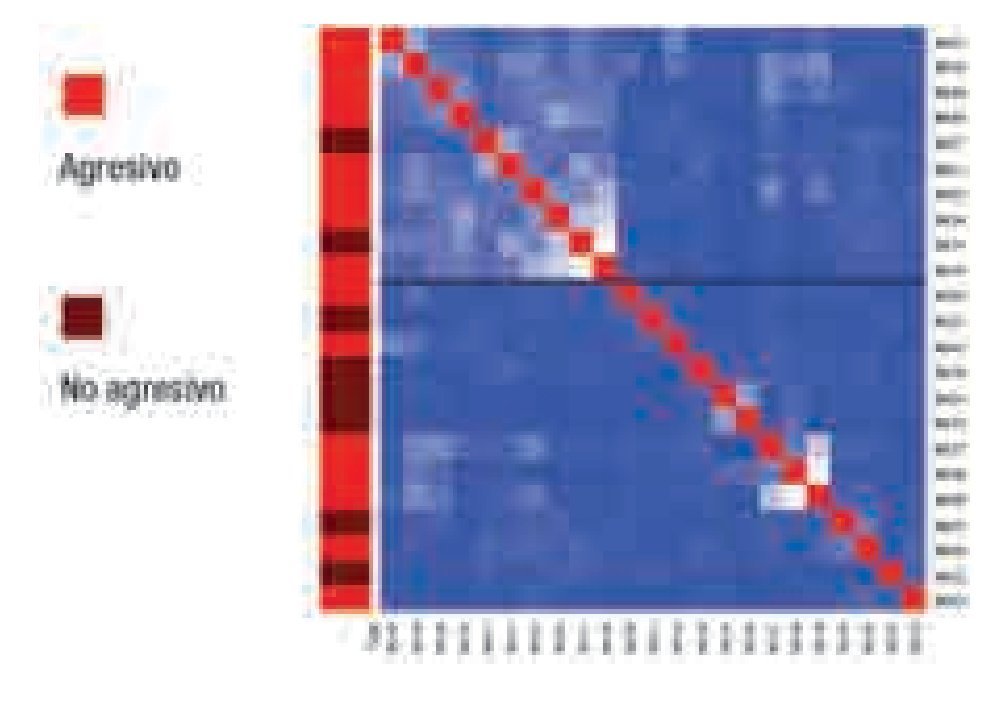
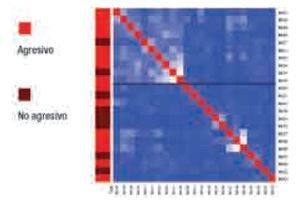
El agrupamiento no supervisado no evidenció correlación alguna entre los datos obtenidos por aCGH y la agresividad del CaP (Imagen 1).
Imagen 1. Agrupamiento no supervisado sin evidencia de correlación entre aCGH y la agresividad de CaP.
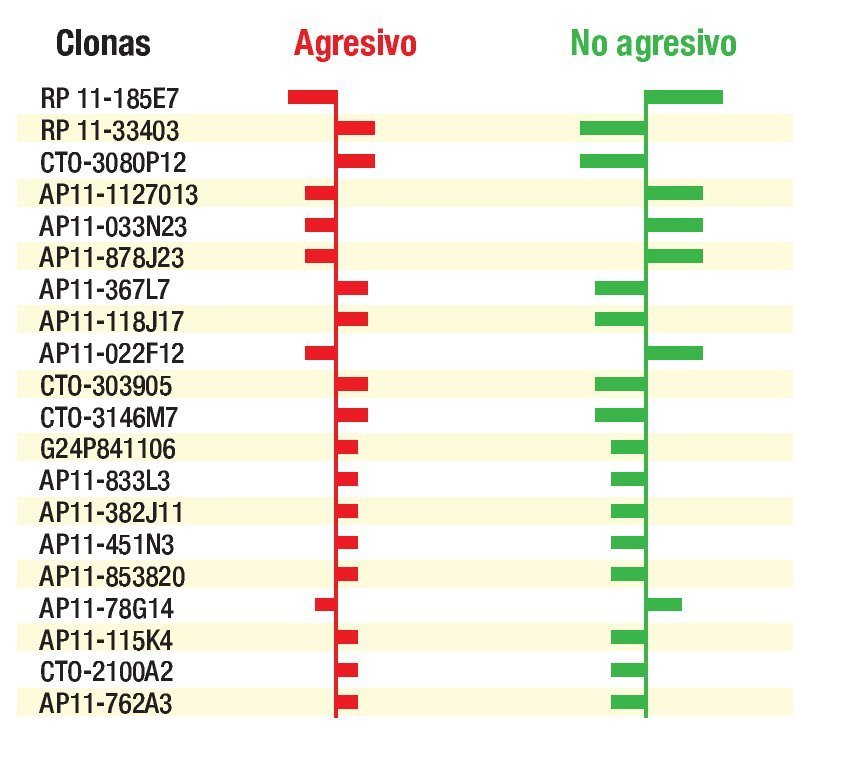
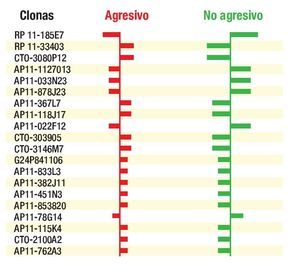
Con el agrupamiento supervisado se pudieron observar loci anteriormente reportados asociados a CaP agresivo, como ganancias en 5p15.33, 8q24.21 y 16p13.3, donde se localizan los genes TERT, MYC y SSTR5 respectivamente y regiones perdidas donde se localizan genes como CAV-1, EPB41L3, DACH1; entre otras regiones que aún no han sido reportadas. La mejor clasificación es obtenida en un grupo de aproximadamente 150 regiones cromosómicas; en la Imagen 2 se observan 20 regiones representativas.
Imagen 2. Clonas de CaP, agresivo y no agresivo.
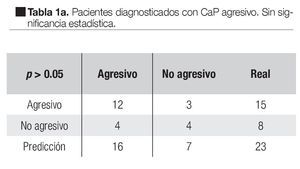
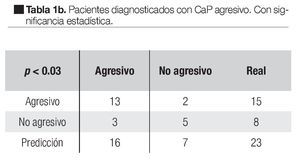
La prueba de validación cruzada revela valores elevados en el error de clasificación (30%) y la tasa de falsos positivos (20%). Sin embargo, es importante recalcar que el tamaño de muestra es pequeño y como consecuencia, la mala clasificación de sólo un paciente durante el proceso de validación cruzada viraría los resultados de significativos (p <0 03 a no significativos p >0.05). En la Tabla 1 se observa un ejemplo característico de una corrida de clasificación sin ( Tabla 1a) y con ( Tabla 1b) significancia estadística. Nuestro sistema clasificador calcula la probabilidad de que el CaP sea agresivo (P[CaPa]) y no agresivo (P[CaPna]) para cada uno de los pacientes, de esta forma si P(CaPa) >0.05 el paciente se clasifica como CaP agresivo y si P(CaPna) >0.05 el paciente se clasifica como CaP no agresivo donde P(CaPa) + P(CaPna) = 1. En la parte izquierda de la Tabla 1 se encuentran todos los pacientes diagnosticados con CaP agresivo y su clasificación determinada por nuestro sistema; por ejemplo, en la Tabla 1b de 15 muestras de CaP agresivo, nuestro clasificador detecta 13 muestras como CaP agresivo y dos como no agresivo (parte izquierda de la imagen), la prueba exacta de Fisher confirma una p <0 03 a diferencia de la Tabla 1a cuya p >0.05
¿ DISCUSIÓN
Los resultados de nuestro estudio concuerdan con reportes previos de ganancia y pérdida de genes asociados a CaP agresivo, como en los trabajos de Baird (2010) y Marian (2010) quienes proponen la ganancia del gen TERT y Sun (2007) e Ishkanian (2009), quienes postulan que existe una asociación en la ganancia del protooncogen MYC y la pérdida del gen supresor de tumor NKX3-1 con CaP de agresividad moderada y alta.2-5
Tambien se encontraron ganancias en varios loci donde se localizan genes cuya sobreexpresión ha sido asocianda a una modalidad más agresiva y metastásica de CaP como SSTR5, FAM84B, DDEF1: Moricheti (2010); Halmos (2000); Ghoussaini (2008); Lin (2008),6- pérdida de loci con genes asocciados a CaP agresivo como CAV-1, EPB41L3, DACH1: Di Vizio (2009); Wong (2007); Bernkopf (2008); Wu K (2009)10-13 y pérdida del loci 14q32, región rica en genes también relacionados con la respuesta inmune (Rose 2010).14 También se corroboraron los resultados obtenidos por Ghoussaini (2008) y van Duin (2005),15 quienes postulan a la región 8q como una de las regiones más frecuentemente alteradas en CaP, encontrándose amplificada especialmente en CaP agresivo. Por lo anterior, podemos inferir que a pesar del alto grado de heterogeneidad del CaP, existen ciertas regiones de pérdida y ganancia dentro del genoma que se pueden asociar con su agresividad. Se está ampliando el número de muestras de este trabajo para confirmar los hallazgos mencionados.
¿ CONCLUSIÓN
El uso de aCGH permitió una caracterización global inicial de las alteraciones genómicas presentes en tejido prostático tumoral. El tamaño de la muestra es pequeño para generar un clasificador significativo que discrimine entre CaP agresivo y no agresivo. Un análisis más extenso podrá confirmar las asociaciones que se sugieren en este estudio y la definición de posibles marcadores genómicos que identifiquen tumores agresivos, para ajustar las modalidades terapéuticas a las necesidades del paciente y mejorar su calidad de vida.
Correspondencia: R Garza-Cortez.
Av. Fco. I. Madero y Gonzalitos s/n 1er- piso No. 18. Col. Mitras centro. C. P. 64460, Monterrey, N. L. México.
Teléfono: 81 8333 1713, 8389 1111, Ext.: 2136, 3136.
Correo electrónico: jrcor77@yahoo.com, jeff.cortes-gonzalez@ki.se