Vaccines are administered worldwide to control on-going coronavirus disease-19 (COVID-19) pandemic caused by SARS-CoV-2. Vaccine efficacy is largely contributed by the epitopes present on the viral proteins and their alteration might help emerging variants to escape host immune surveillance. Therefore, this study was designed to study SARS-CoV-2 Nsp13 protein, its epitopes and evolution.
MethodsClustal Omega was used to identify mutations in Nsp13 protein. Secondary structure and disorder score was predicted by CFSSP and PONDR-VSL2 webservers. Protein stability was predicted by DynaMut webserver. B cell epitopes were predicted by IEDB DiscoTope 2.0 tools and their 3D structures were represented by discovery studio. Antigenicity and allergenicity of epitopes were predicted by Vaxijen2.0 and AllergenFPv.1.0. Physiochemical properties of epitopes were predicted by Toxinpred, HLP webserver tool.
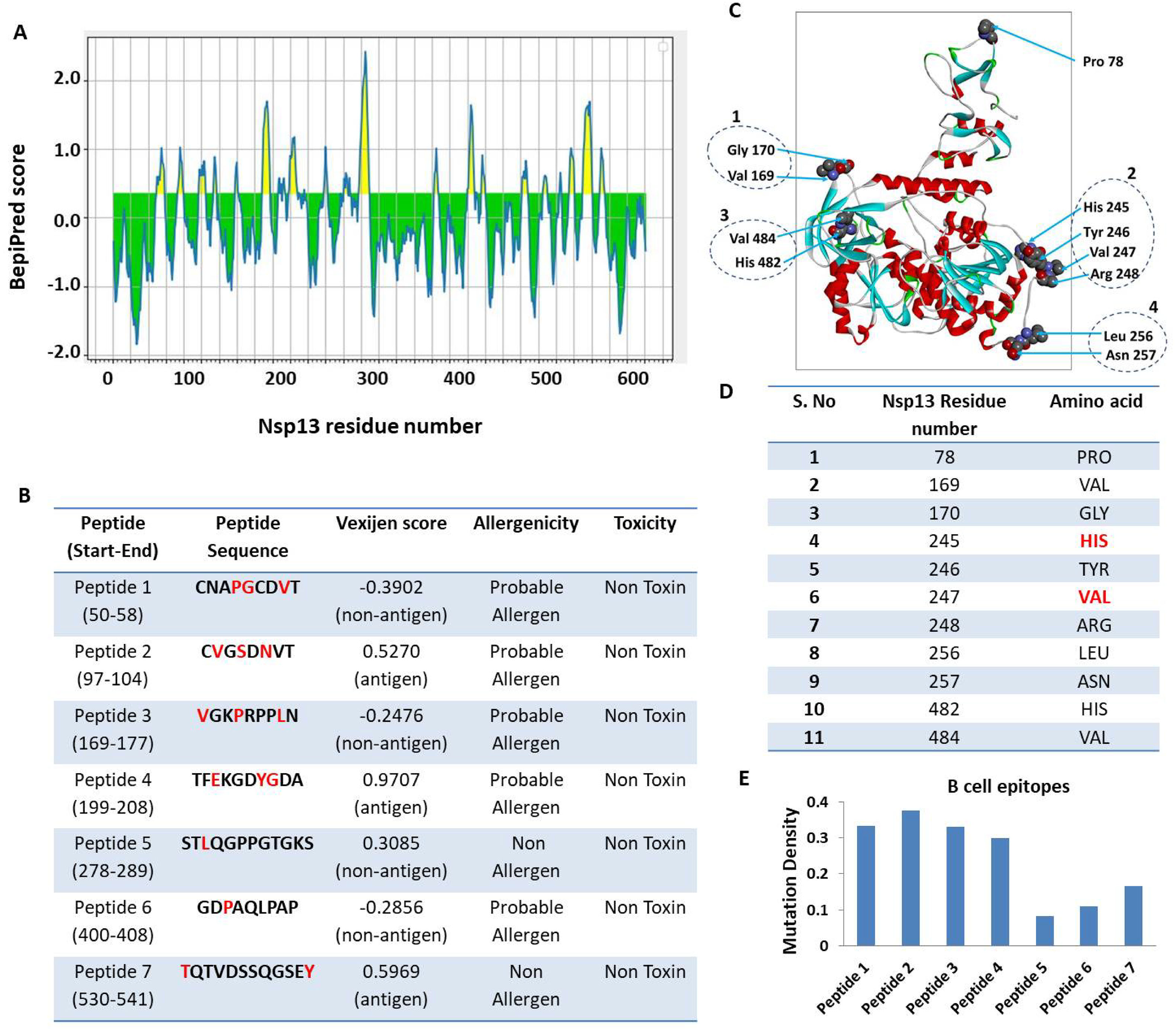
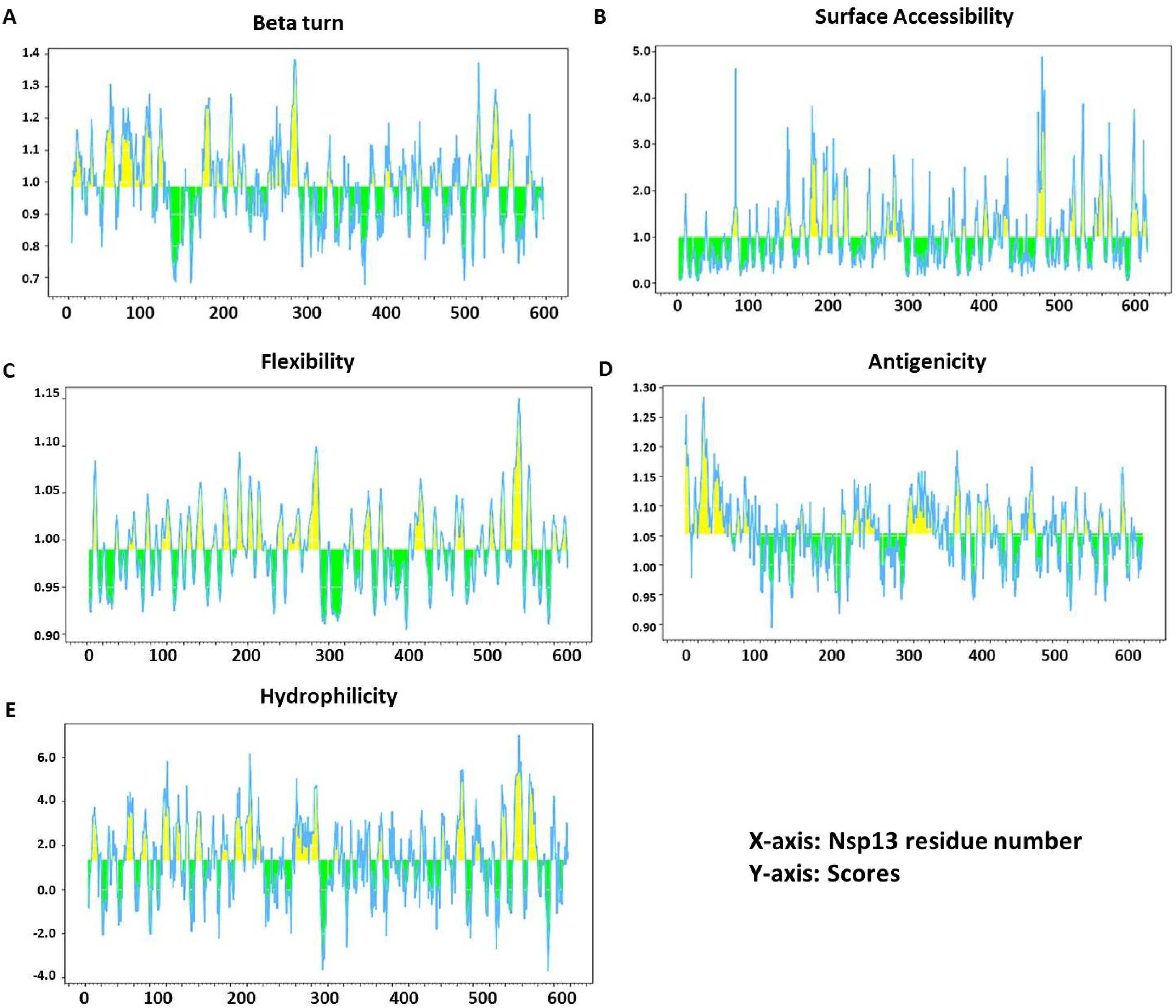
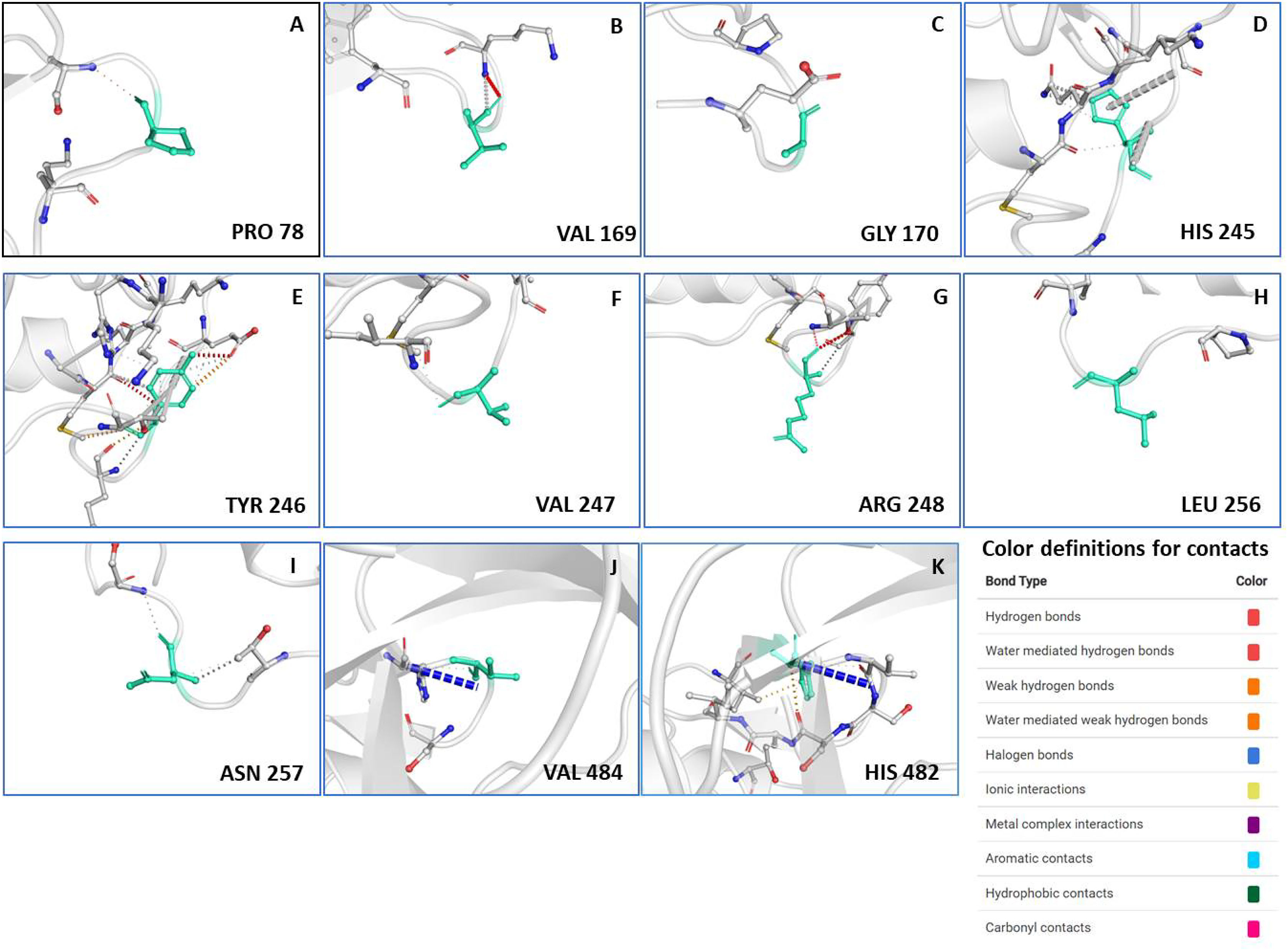
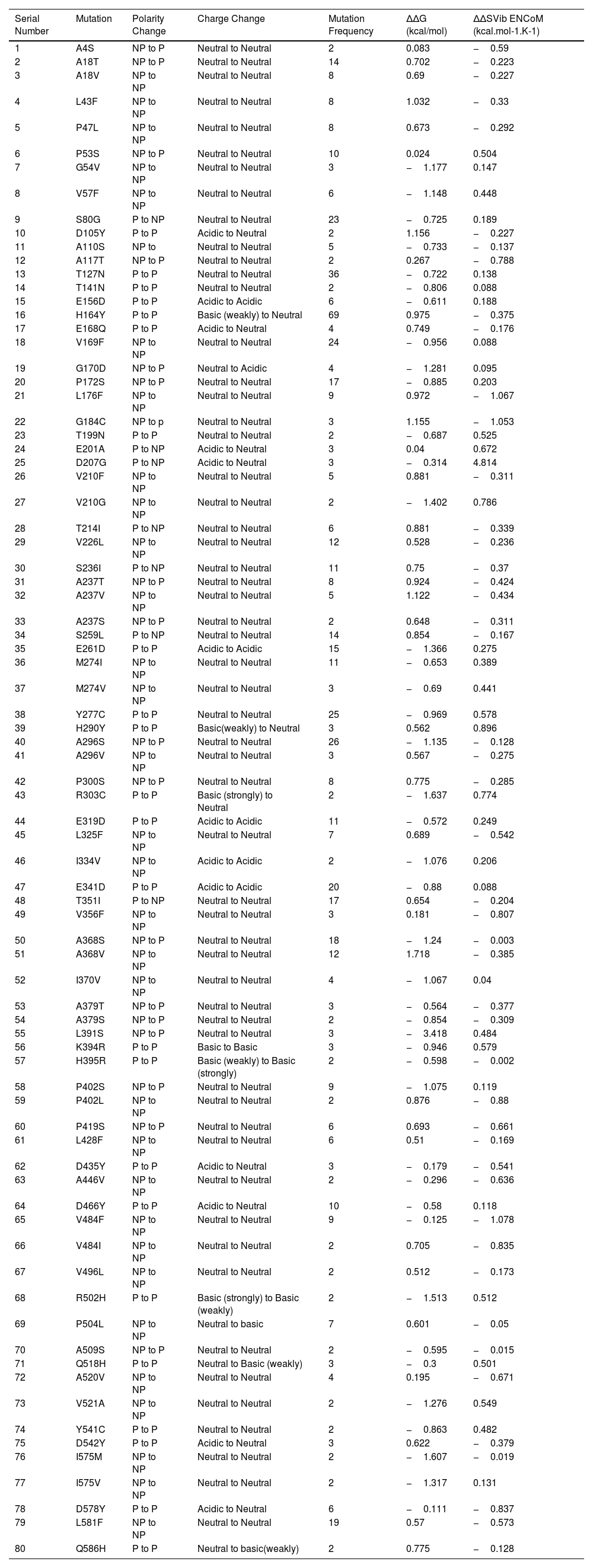
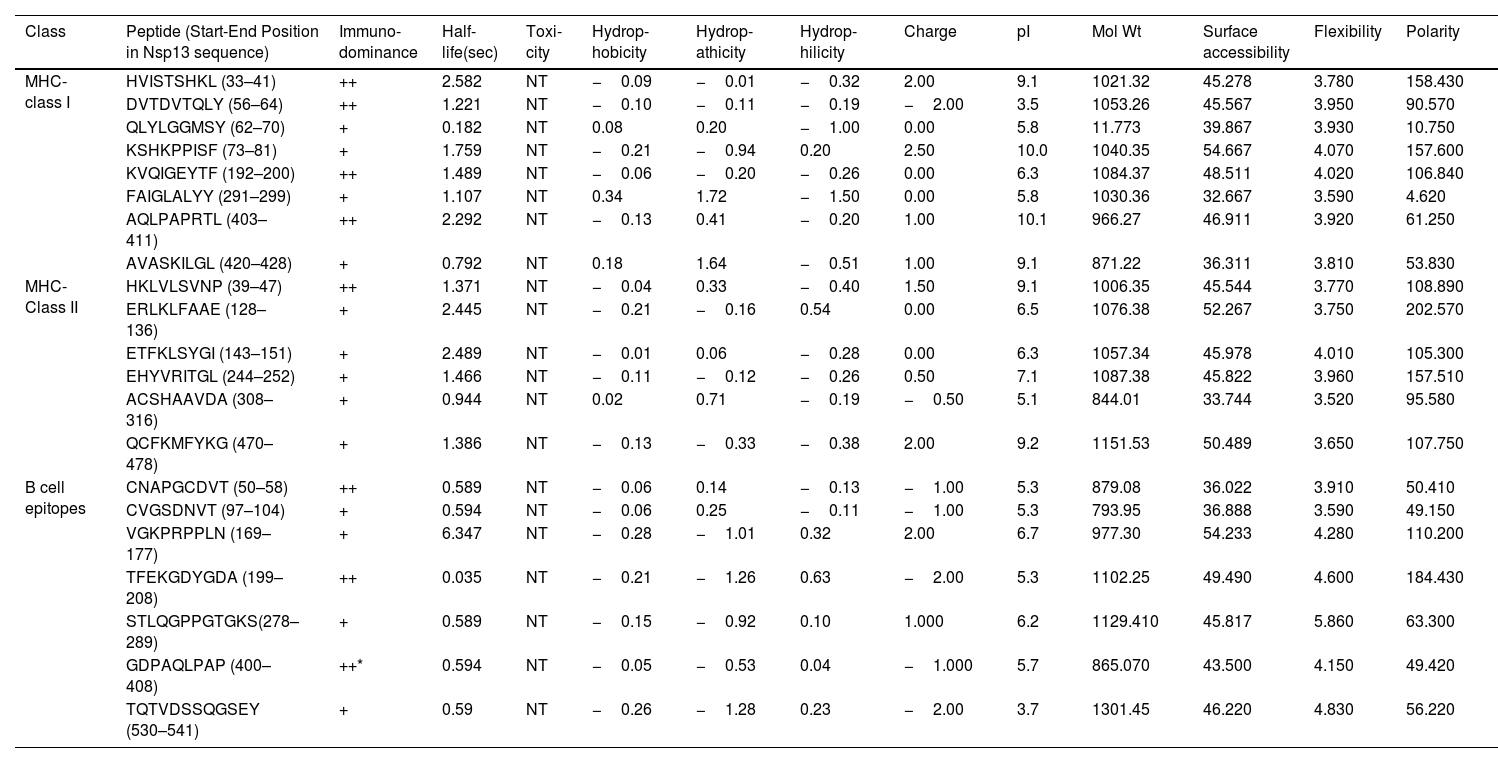
ResultsOur data revealed 182 mutations in Nsp13 among Indian SARS-CoV-2 isolates, which were characterised by secondary structure and per-residue disorderness, stability and dynamicity predictions. To correlate the functional impact of these mutations, we characterised the most prominent B cell and T cell epitopes contributed by Nsp13. Our data revealed twenty-one epitopes, which exhibited antigenicity, stability and interactions with MHC class-I and class-II molecules. Subsequently, the physiochemical properties of these epitopes were analysed. Furthermore, eighteen mutations reside in these Nsp13 epitopes.
ConclusionsWe report appearance of eighteen mutations in the predicted twenty-one epitopes of Nsp13. Among these, at least seven epitopes closely matches with the functionally validated epitopes. Altogether, our study shows the pattern of evolution of Nsp13 epitopes and their probable implications.
Las vacunas se administran a nivel mundial para controlar la pandemia en curso de la enfermedad por coronavirus de 2019 (COVID-19) causada por SARS-CoV-2. A la eficacia de la vacuna contribuyen ampliamente los epítopes presentes en las proteínas virales, y su alteración puede contribuir a que las variantes emergentes se escapen de la vigilancia inmunológica del huésped. Por tanto, este estudio fue diseñado para estudiar la proteína Nsp13 de SARS-CoV-2, sus epítopes y su evolución.
MétodosSe utilizó Clustal Omega para identificar las mutaciones de la proteína Nsp13. La estructura secundaria y la tasa de desorden se predijeron mediante los servidores web CFSSP y PONDR-VSL2. La estabilidad de la proteína fue predicha mediante el servidor web DynaMut. Los epítopes de las células B fueron predichos mediante las herramientas DiscoTope 2.0 de IEDB, y sus estructuras en 3D fueron representadas mediante Discovery Studio.
La antigenicidad y alergenicidad de los epítopes fueron predichas mediante Vaxijen2.0 y AlergenFPv.1.0. Las propiedades fisioquímicas de los epítopes fueron predichas mediante Toxinpred, la herramienta del servidor web HLP.
ResultadosNuestros datos revelaron 182 mutaciones en Nsp13 entre los aislados indios de SARS-CoV-2, que fueron caracterizadas mediante las predicciones de la estructura secundaria y la capacidad de desorden por residuo, la estabilidad y la dinamicidad. Para correlacionar el impacto funcional de estas mutaciones, caracterizamos los epítopes más prominentes de las células B y las células T a los que contribuyó Nsp13. Nuestros datos revelaron veintiún epítopes, que exhibieron antigenicidad, estabilidad e interacciones con las moléculas MHC de clase I y clase II. Seguidamente se analizaron las propiedades fisioquímicas de estos epítopes. Además, en estos epítopes de Nsp13 residen ocho mutaciones.
ConclusionesReportamos el aspecto de ocho mutaciones en los veintiún epítopes de Nsp13 predichos. Entre estos, al menos siete epítopes concuerdan estrechamente con los epítopes funcionalmente validados. En su conjunto, nuestro estudio refleja el patrón evolutivo de los epítopes de Nsp13 y sus implicaciones probables.