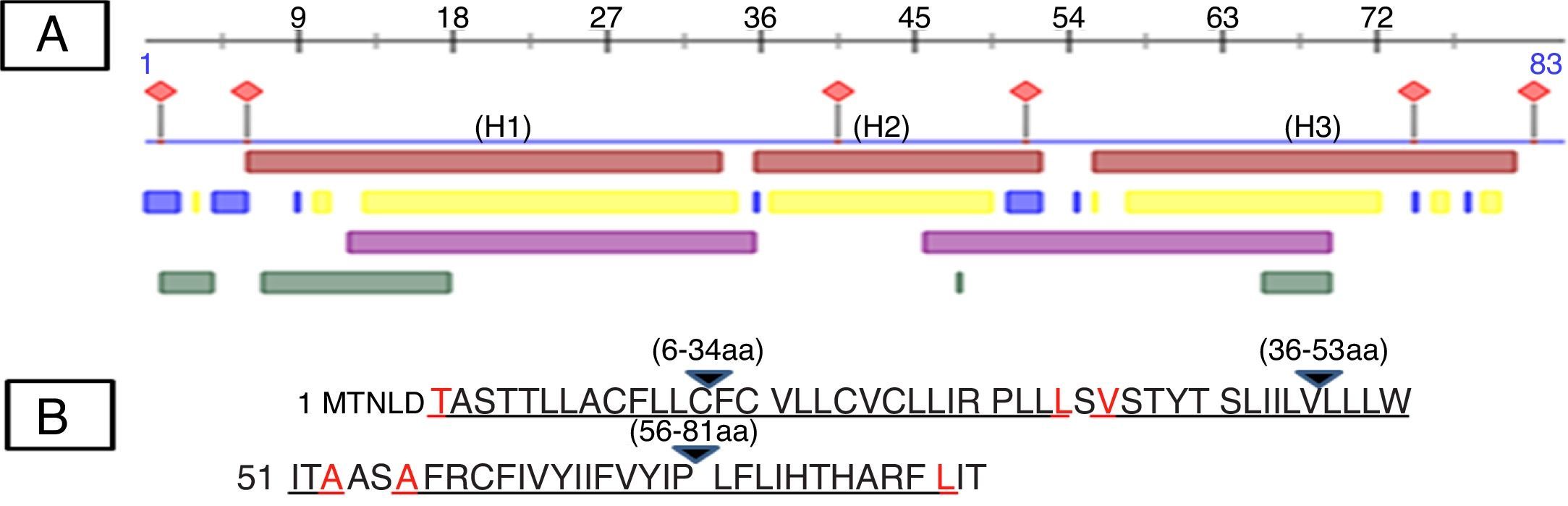
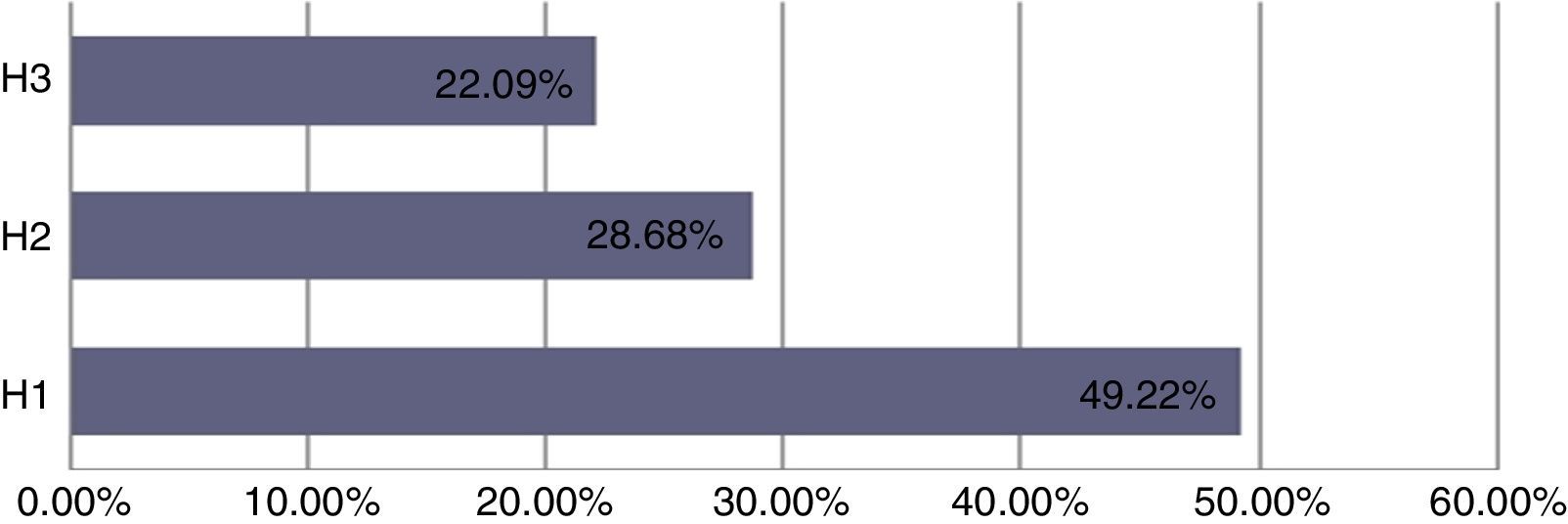
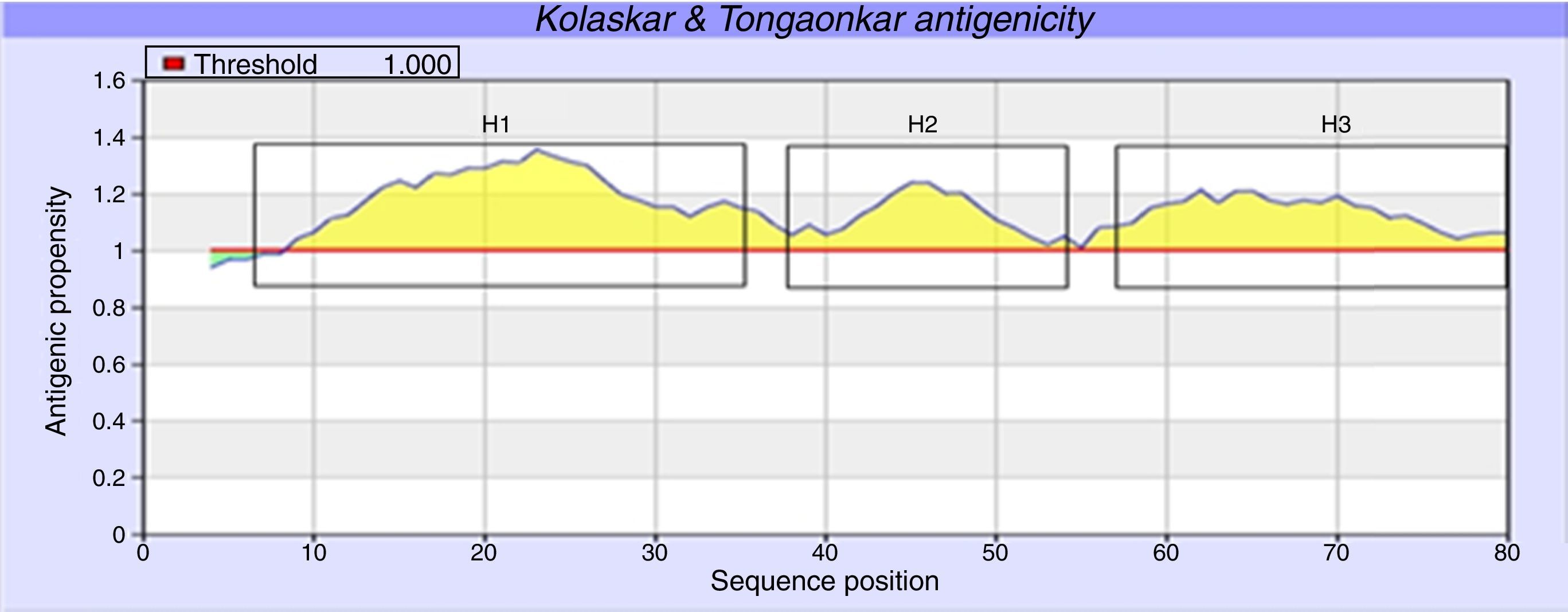
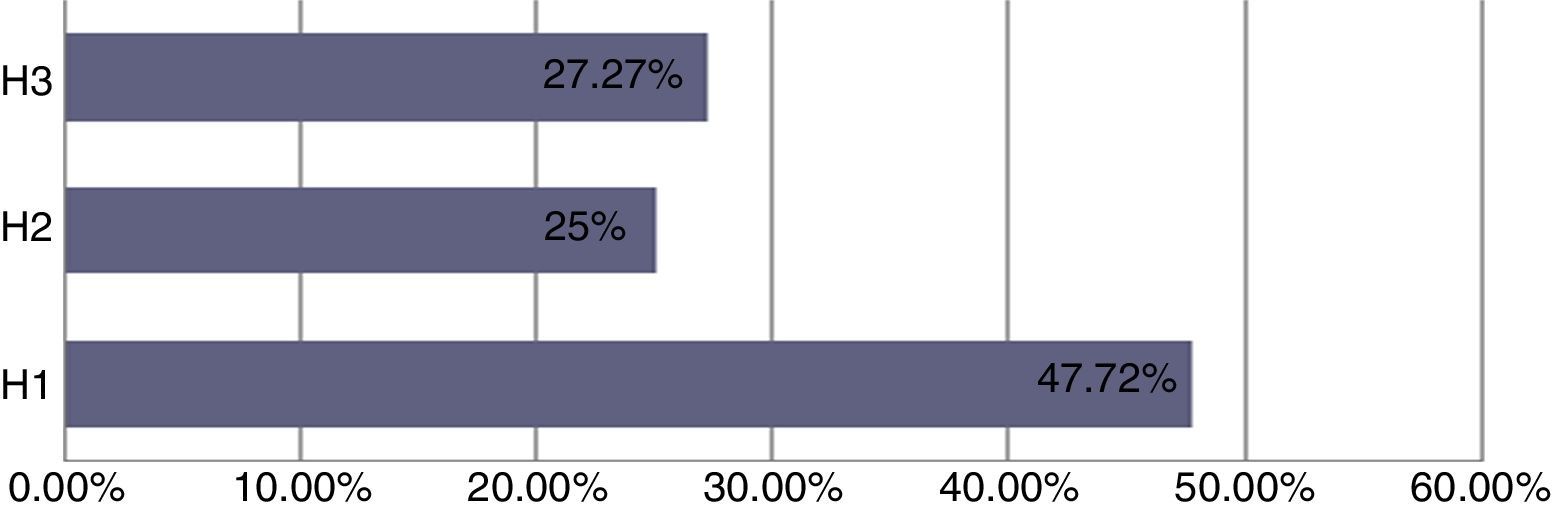
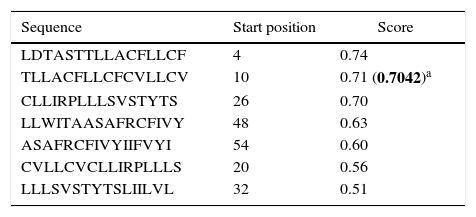
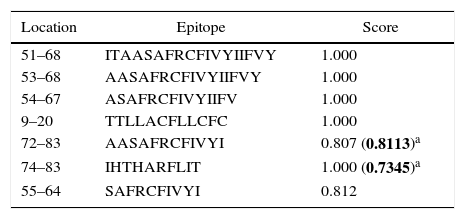
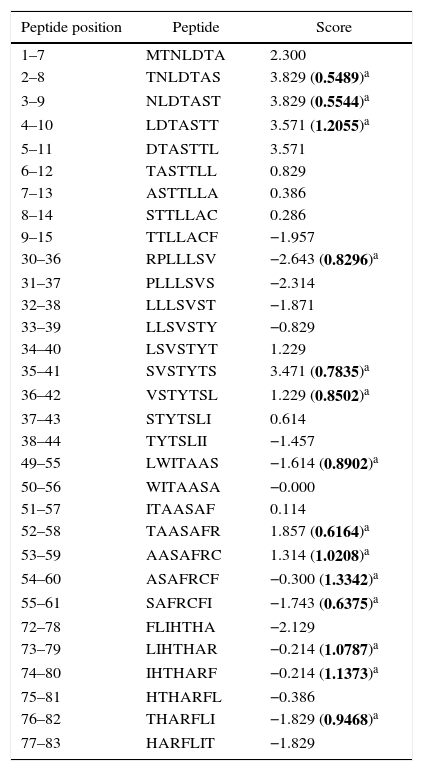
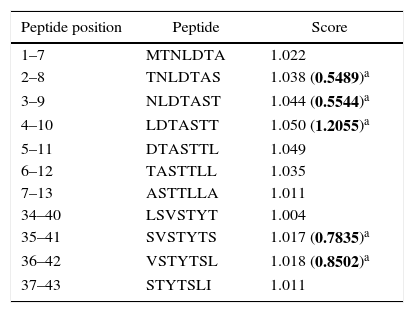
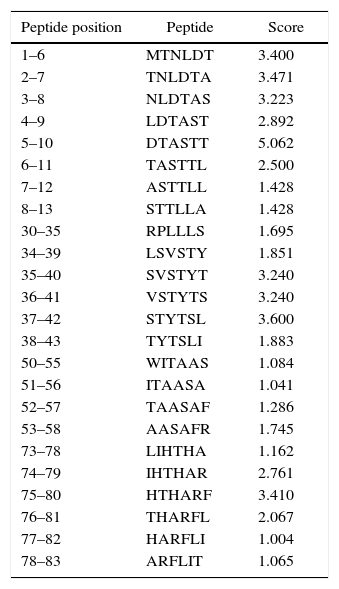
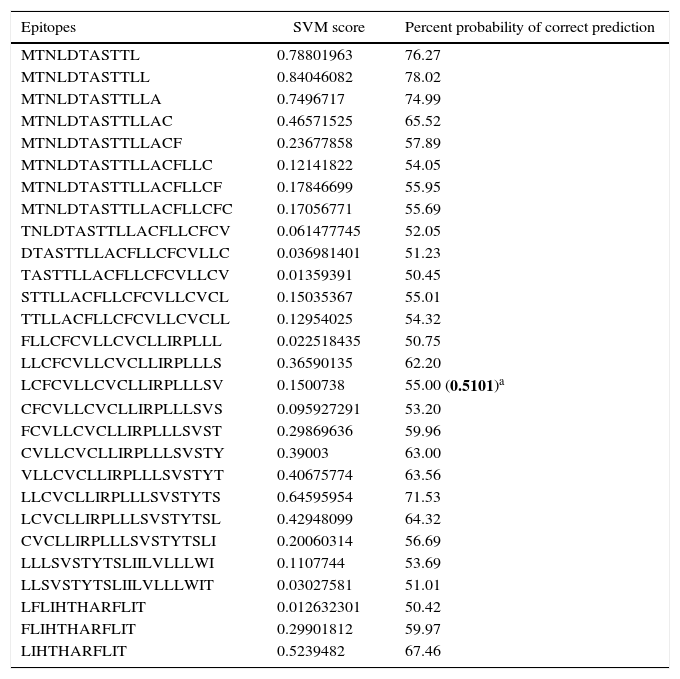
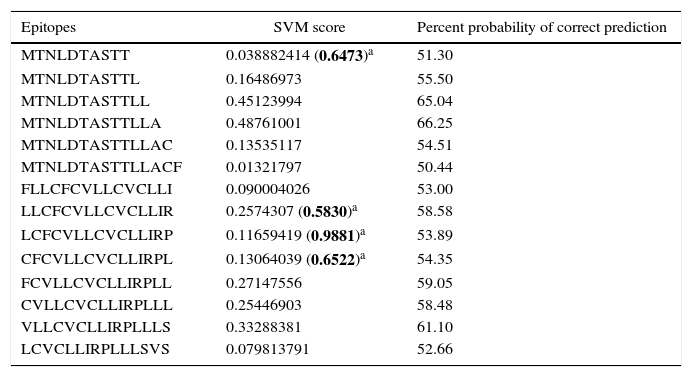
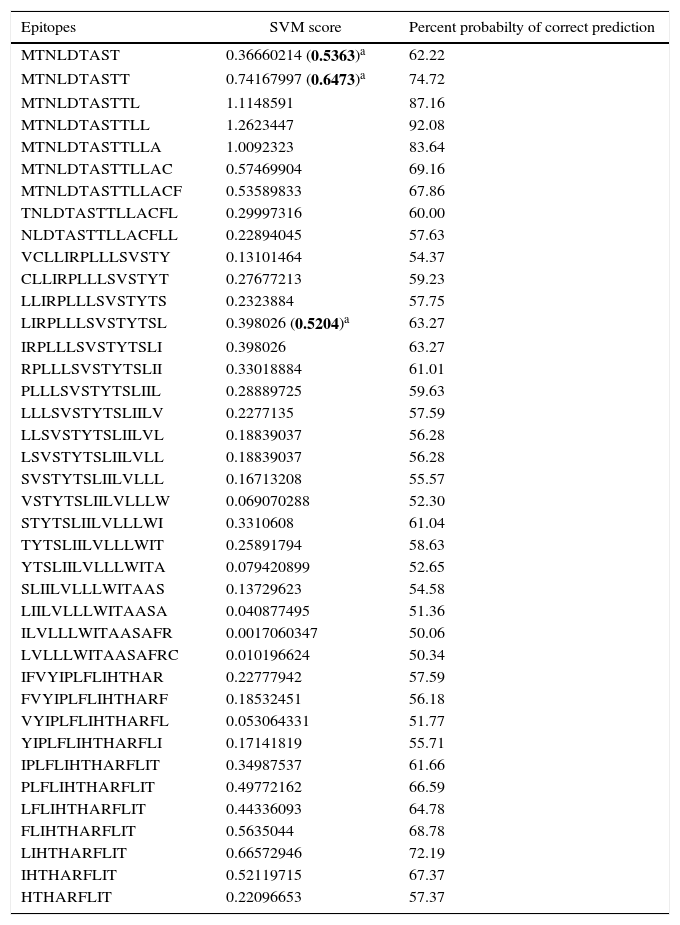
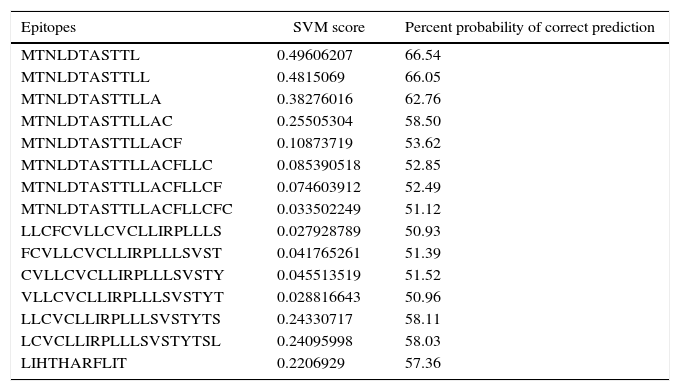
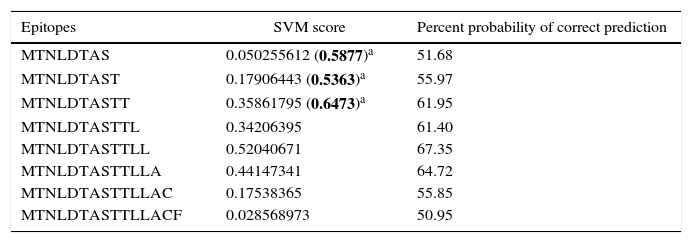
Human papillomavirus (HPV), one of the most common sexually transmitted infections worldwide, affects around 300 million new individuals each year. The HPV E5 gen appear to have coevolved with the major HPV oncogenes E6 and E7, and their presence in the viral genome correlates with risk of cancer. HPV-16 E5 is an oncoprotein with many functions during HPV-16 infection. The development of computational methods for epitope finding becomes an alternative to the wet experimental techniques, in order to save time and cost. In this work the bioinformatic tools: Predict Protein, BCPREDS (AAP, BCpreds and FBCPred), ABCpred, BepiPred, SVMTriP, LBtope, IEDB and LEPS were used in order to elucidate B-cell epitopes on HPV-16 E5. The HPV-16 E5 secondary structure was used to mapping the B-cell epitopes obtained in this study. A total of 202 B-cell epitopes and 44 B-cell epitopes with tumor activity were obtained with all together bioinformatics tools. Differences in ratio of these epitopes with the three α-helices of the secondary structure determined by Predict Protein were observed. The predicted epitopes in this study could facilitate the design of future therapeutic vaccines and the understanding of the immunological mechanisms of HPV-16.
El virus del papiloma humano (VPH) es una de las infecciones de transmisión sexual más comunes en todo el mundo, que afecta a unos 300 millones de personas cada año. El gen E5 del VPH parece haber coevolucionado con los principales oncogenes E6 y E7 y su presencia en el genoma viral se correlaciona con el riesgo de cáncer. VPH-16 E5 es una oncoproteína con muchas funciones durante la infección del VPH-16. El desarrollo de los métodos computacionales para el descubrimiento de epítopos se convierte en una alternativa a las técnicas experimentales húmedas, con el fin de ahorrar tiempo y costes. En este trabajo las herramientas bioinformáticas: Predict Protein, BCPREDS (AAP, BCpreds y FBCPred), ABCpred, BepiPred, SVMTriP, LBtope, IEDB y LEPS se utilizaron con el fin de elucidar epítopos de células B en la proteína E5 del VPH-16 (VPH-16 E5). La estructura secundaria de la VPH-16 E5 se utilizó para mapear los epítopos de células B predichos en este estudio. Un total de 202 epítopos de células B y 44 epítopos de células B con actividad tumoral se obtuvieron con todas las herramientas bioinformáticas en conjunto. Se observaron diferencias en la proporción de estos epítopos en las 3 α-hélices de la estructura secundaria determinada por Predict Protein. Los epítopos predichos en este estudio podrían facilitar el diseño de futuras vacunas terapéuticas y la comprensión de los mecanismos inmunológicos de VPH-16.
Artículo
Comprando el artículo el PDF del mismo podrá ser descargado
Precio 19,34 €
Comprar ahora