Los métodos de identificación bacteriana usados habitualmente en Microbiología Clínica, aunque miniaturizados y automatizados, se siguen basando en principios similares a las pruebas de identificación clásicas. Sin embargo, se van produciendo avances tecnológicos que pueden modificar radicalmente estas metodologías. En el presente estudio se determina la utilidad de la mass spectrometry (MS, ‘espectrometría de masas’) matrix-assisted laser desorption ionization time-of-flight (MALDI-TOF) para la identificación bacteriana habitual en el laboratorio de Microbiología Clínica.
MétodosSe analizaron 294 aislamientos bacterianos aerobios y anaerobios facultativos (65 grampositivos y 229 gramnegativos) obtenidos a partir de diferentes muestras clínicas mediante metodología microbiológica habitual (Wider, Francisco Soria Melguizo, Madrid, España; Vitek-2, API Staph, API 20 Strep, API Coryne y API NH, BioMérieux, Marcy L’Etoile, Francia) y mediante un dispositivo de MS MALDI-TOF Autoflex III (Bruker Daltonics GmbH, Leipzig, Alemania). Los aislamientos de Salmonella se tipificaron con sueros específicos. Los aislamientos que no ofrecieron una fiabilidad en la identificación superior al 95% en Vitek-2 o Wider se corroboraron mediante API. Los aislamientos en los que los sistemas API no ofrecieron un perfil fiable se descartaron. La identificación mediante MS MALDI-TOF se puntúa automáticamente por el software del sistema de 0–3 en función de su fiabilidad. Los aislamientos con puntuaciones inferiores a 1,5 se consideraron no fiables. La correlación entre ambas identificaciones se clasificó como correlación en la especie, el género o la ausencia de correlación.
ResultadosLa correlación en la especie en los grampositivos estudiados fue del 100%. La correlación en los gramnegativos fue del 87,7% en la especie y del 97,7% en el género. Solo hubo ausencia de correlación en un 2,2% de los aislamientos. Las identificaciones fallidas se dieron en los géneros Raoultella y Acinetobacter, en Stenotrophomonas maltophilia y en Francisella tularensis.
ConclusiónLa identificación de aislamientos bacterianos clínicos mediante MS MALDI-TOF muestra una excelente correlación con la identificación mediante la metodología convencional. A ello hay que añadir que se trata de una tecnología que permite la identificación de los microorganismos a partir de colonias crecidas en placa de cultivo en apenas unos minutos con una metodología extremadamente simple y sin apenas consumibles, aunque los costes de amortización de los equipos pueden ser considerables.
The methods routinely used for bacterial identification in Clinical Microbiology Laboratory, although miniaturized and automated, are still based on the same basic principles as classical identification methods. Nevertheless, technological advances are emerging which could modify these routine methods. We report a comparative study between conventional identification methods and mass spectrometry MALDI-TOF (MS MALDI-TOF) for bacterial identification in the Clinical Microbiology Laboratory.
MethodsWe analysed 294 facultative anaerobic and aerobic isolates (65 Gram positives and 229 Gram negatives), obtained from different clinical samples, using conventional microbiological methods (Wider, Fco. Soria Melguizo, Madrid, Spain; Vitek-2, APIStaph, API 20 Strep, API Coryne and API NH, bioMérieux, Marcy L’Etoile, France) and an Autoflex III MS with a MALDI-TOF device (Bruker Daltonics GmbH, Leipzig, Germany). Salmonella isolates were also typed by using specific sera. Isolates identified with a confidence rate <95% were checked by using API systems. Isolates which were not accurately identified by API systems were rejected. MS MALDI-TOF identification is automatically scored by the system software between 1 and 3 points. Isolates with scores <1.5 were classified as unreliable. Correlation between both identifications was classified as correlation at the species level, at the genus level or no correlation.
ResultsCorrelation at the species level in Gram positives was 100%. Correlation in Gram negatives was 87.7% at the species level and 97.7% at the genus level. There was no correlation in 2.2% of Gram negatives studied. Identification failures occurred in the genera Raoultella and Acinetobacter, in Stenotrophomonas maltophilia and in Francisella tularensis.
ConclusionBacterial clinical isolates identification obtained by MS MALDI-TOF shows excellent correlation with identification obtained by conventional microbiological methods. Moreover, MS MALDI-TOF allows the identification of bacteria from colonies grown on agar culture plates in just a few minutes, with a very simple methodology and hardly any consumable costs, although the financial costs of purchasing the device can be high.
La identificación bacteriana en el laboratorio de Microbiología Clínica se realiza de manera habitual a partir de las características morfológicas y tintoriales de los microorganismos, de su crecimiento en distintos medios de cultivo y atmósferas, de diversas pruebas que determinan características bioquímicas de las bacterias y, en algunos casos, a partir de determinadas características de sensibilidad a los antimicrobianos. Aunque la identificación basada en las características bioquímicas y metabólicas de las bacterias ha evolucionado tecnológicamente en cuanto a la miniaturización de las series de pruebas y a la automatización de su realización y lectura, se siguen basando en buena parte en los mismos principios y recursos que las pruebas en uso hace 25–30 años. Ello hace que la identificación del microorganismo se retrase horas o incluso días desde su crecimiento en los medios de cultivo, y que esta identificación sea en ocasiones dificultosa cuando se trata de microorganismos con escasa actividad bioquímica y enzimática. Pese a que hace ya años que se han ido desarrollando diferentes tecnologías basadas sobre todo en técnicas genómicas dirigidas a salvar estas limitaciones, no son técnicas que hayan conseguido hasta el momento una amplia implantación. Sin embargo, una identificación rápida y fiable de los microorganismos aislados de muestras clínicas es un objetivo irrenunciable, que tendría una repercusión evidente en el tratamiento del paciente infectado.
La identificación bacteriana basada en el perfil de proteínas obtenido mediante la mass spectrometry (MS, ‘espectrometría de masas’) denominada matrix-assisted laser desorption ionization time-of-flight (MALDI-TOF) fue ya propuesta hace varias décadas1–4. Sin embargo, sólo recientemente ha empezado a usarse como un método rápido y fiable para la identificación bacteriana5. Hasta el momento se han efectuado estudios parciales sobre su efectividad para la identificación de determinados microorganismos en condiciones controladas6–8, pero son muy escasos los trabajos que han estudiado su efectividad en la identificación de aislamientos clínicos sometidos a este test directamente desde los medios de cultivo habituales y sin condiciones especiales4. En el presente estudio se analiza la eficacia de la MS MALDI-TOF para la identificación de aislamientos clínicos de bacterias grampositivas y gramnegativas de diversos orígenes directamente a partir de su crecimiento en medios de cultivo habituales.
Material y métodosSe analizaron 294 aislamientos bacterianos aerobios y anaerobios facultativos obtenidos durante 2 semanas a partir de muestras clínicas de orina, coprocultivos, exudados vaginales y uretrales, exudado faríngeo, esputo, lavados broncoalveolares, exudados y hemocultivos. Las muestras se sembraron en medios de cultivo habituales (agar sangre, agar chocolate, agar MacConkey y agar chocolate bacitracina) y se incubaron en atmósfera aerobia o en el 10% de CO2 a 37°C, según el tipo de muestra procesada de acuerdo con la metodología microbiológica habitual.
Las muestras se identificaron de acuerdo con la metodología microbiológica habitual. Los cocos gramnegativos, Moraxella y Haemophilus se identificaron mediante API NH (BioMérieux, Marci L’Étoile, Francia). Las enterobacterias y los bacilos gramnegativos no fermentadores (BGNNF) se identificaron inicialmente mediante Wider panel MIC/ID gramnegativos (Francisco Soria Melguizo, Madrid, España) o mediante tarjetas Vitek-2 GNB (BioMérieux, Marci L’Étoile, Francia), y cuando esta identificación no fue fiable con porcentajes superiores al 95%, se ratificó mediante API 20E o API 20NE (BioMérieux, Marci L’Étoile, Francia). Los cocos y los bacilos grampositivos se identificaron inicialmente mediante Wider panel MIC/ID grampositivos (Francisco Soria Melguizo, Madrid, España), y cuando este sistema no ofreció una identificación con un porcentaje de fiabilidad superior al 95%, se ratificó mediante API Staph, API 20 Strep o API Coryne (BioMérieux, Marci L’Étoile, Francia). Los aislamientos de Salmonella se serotipificaron mediante sueros específicos. La Francisella tularensis se identificó, asimismo, mediante aglutinación con anticuerpos específicos. En aquellos casos en que ninguno de los sistemas ofreció una información fiable, el microorganismo se descartó.
Espectrometría de masas matrix-assisted laser desorption ionization time-of-flight- 1.
Preparación de la muestra. Se depositó una pequeña cantidad de una colonia directamente sobre la placa ground steel del espectrómetro de masas, y se formó una delgada película. Sobre ella se aplicó 1μl de la solución matriz (solución saturada de ácido alfa-ciano-4-hidroxicinámico en acetonitrilo al 50% y ácido trifluoroacético al 2,5%) y se dejó secar a temperatura ambiente.
- 2.
MS. Las medidas se realizaron en un espectrómetro de masas MALDI-TOF-TOF Autoflex III (Bruker Daltonics GmbH, Leipzig, Alemania). Se obtuvo el espectro entre 2–20kD de forma automática, y se trabajó en modo lineal positivo a una frecuencia de 200 Hz. Los parámetros que se fijaron para el espectrómetro fueron IS1 a 20kV, IS2 a 18,6kV, lente a 6kV y PIE a 40ns. El espectro obtenido se comparó de manera automática a partir de algoritmos integrados en el software del sistema con la base de datos MALDI Biotyper. El protocolo de trabajo del MALDI Biotyper proporciona la adquisición óptima de la muestra (acumulación de 500 disparos del láser en diferentes lugares de la muestra). Los espectros se calibraron externamente mediante la utilización de una mezcla de calibrante estándar (extracto de Escherichia coli DH5-α más 2 proteínas adicionales: ARNasa A y mioglobina para cubrir un intervalo de 4–17kD).
- 3.
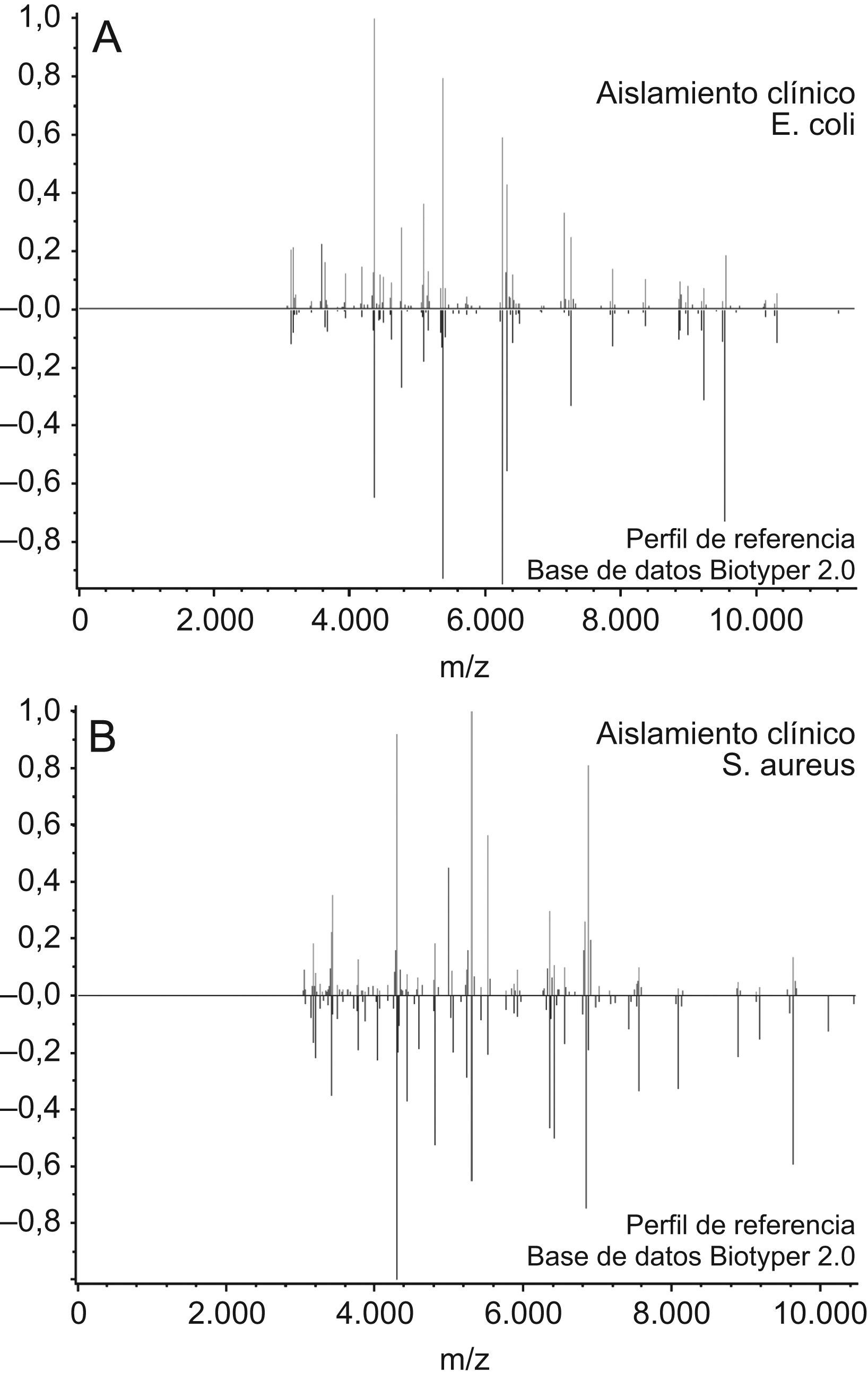
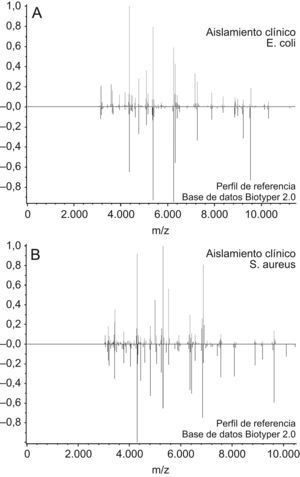
Análisis de los espectros (Bruker Daltonics GmbH, Leipzig, alemania). Para la identificación de los microorganismos, el espectro obtenido de los microorganismos problema se procesó con el programa MALDI Biotyper 1.1. La lista de picos generada se comparó con la biblioteca de referencia del MALDI Biotyper 2.0 mediante la utilización de un algoritmo de comparación integrado en el software (fig. 1). Tras importar el espectro al programa, todo el proceso, desde su análisis hasta su identificación, se lleva a cabo de forma automática, sin intervención alguna por parte del usuario.
Figura 1.A) Comparación del perfil de un aislamiento clínico de Escherichia coli con los recogidos en la base de datos Biotyper 2.0. B) Comparación del perfil de un aislamiento clínico de Staphylococcus aureus con los recogidos en la base de datos Biotyper 2.0. Aparecen en azul los perfiles de referencia de la base de datos, en verde los picos de la cepa clínica coincidentes con éstos, en amarillo los de coincidencia moderada y en rojo los no coincidentes.
(0.2MB). - 4.
Valoración de resultados. Las identificaciones mediante MALDI-TOF se clasificaron como buena (puntuación en la comparación entre el perfil del microorganismo problema y la base de datos ≥2), probable (puntuación ≥1,5 y <2) y no fiable (puntuación<1,5). Las identificaciones que entraron en esta última categoría se consideraron fallidas. En cuanto a la comparación entre MALDI-TOF y los sistemas clásicos, se diferenció según hubiera correlación de género y especie, correlación solo de género (bien porque el sistema solo identificara a este nivel o porque hubiera discrepancia en la especie identificada) o no hubiera correlación de especie ni género. En este último caso, la identificación se consideró fallida.
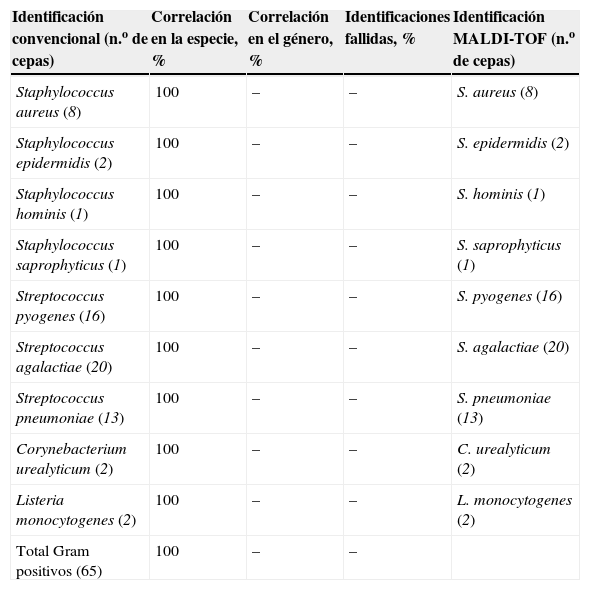
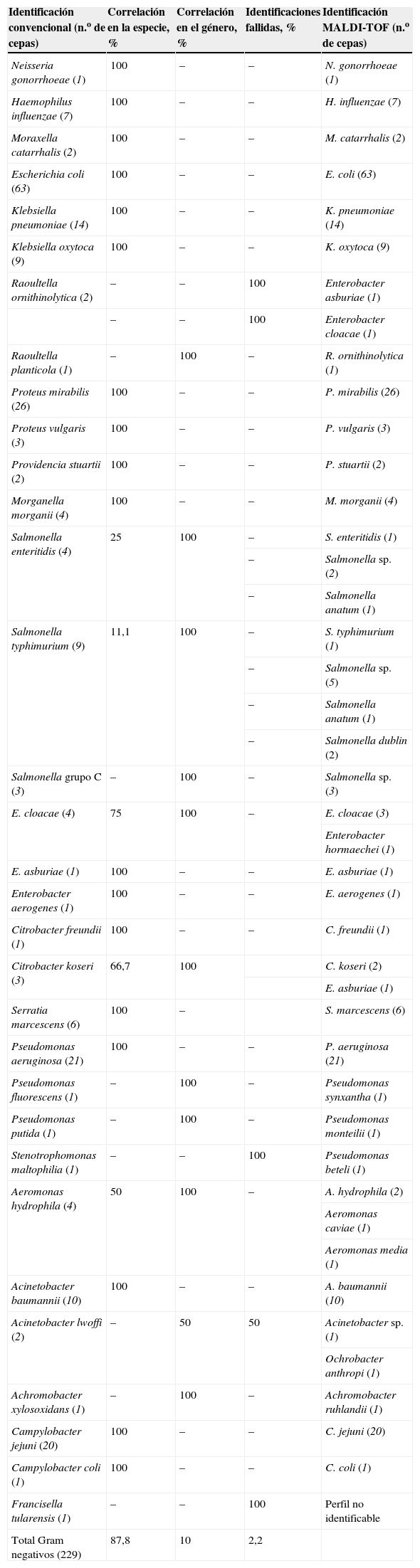
Se incluyeron en el estudio 65 microorganismos grampositivos y 229 microorganismos gramnegativos. Los resultados obtenidos aparecen en las tablas 1 y 2. Como puede observarse en estas tablas, la correlación en la especie de todos los grampositivos estudiados fue del 100%. La correlación en gramnegativos fue excelente en el género (97,7%), aunque inferior en la especie (87,8%). Esta caída de la correlación en la especie está vinculada fundamentalmente a los problemas de identificación a este nivel de los aislamientos de Salmonella no typhi probados, que suponen las dos terceras partes de los aislamientos con identificación coincidente en el género, pero no en la especie. Sólo hubo ausencia de correlación en un 2,2% de los aislamientos. Las identificaciones fallidas se dieron en los géneros Raoultella y Acinetobacter, en Stenotrophomonas maltophilia y en F. tularensis.
Comparación de la identificación de 65 bacterias grampositivas mediante métodos microbiológicos convencionales y mediante espectrometría de masas matrix-assisted laser desorption ionization time-of-flight
| Identificación convencional (n.o de cepas) | Correlación en la especie, % | Correlación en el género, % | Identificaciones fallidas, % | Identificación MALDI-TOF (n.o de cepas) |
| Staphylococcus aureus (8) | 100 | – | – | S. aureus (8) |
| Staphylococcus epidermidis (2) | 100 | – | – | S. epidermidis (2) |
| Staphylococcus hominis (1) | 100 | – | – | S. hominis (1) |
| Staphylococcus saprophyticus (1) | 100 | – | – | S. saprophyticus (1) |
| Streptococcus pyogenes (16) | 100 | – | – | S. pyogenes (16) |
| Streptococcus agalactiae (20) | 100 | – | – | S. agalactiae (20) |
| Streptococcus pneumoniae (13) | 100 | – | – | S. pneumoniae (13) |
| Corynebacterium urealyticum (2) | 100 | – | – | C. urealyticum (2) |
| Listeria monocytogenes (2) | 100 | – | – | L. monocytogenes (2) |
| Total Gram positivos (65) | 100 | – | – |
MALDI-TOF: matrix-assisted laser desorption ionization time-of-flight.
Comparación de la identificación de 229 bacterias gramnegativas mediante métodos microbiológicos convencionales y mediante espectrometría de masas matrix-assisted laser desorption ionization time-of-flight
| Identificación convencional (n.o de cepas) | Correlación en la especie, % | Correlación en el género, % | Identificaciones fallidas, % | Identificación MALDI-TOF (n.o de cepas) |
| Neisseria gonorrhoeae (1) | 100 | – | – | N. gonorrhoeae (1) |
| Haemophilus influenzae (7) | 100 | – | – | H. influenzae (7) |
| Moraxella catarrhalis (2) | 100 | – | – | M. catarrhalis (2) |
| Escherichia coli (63) | 100 | – | – | E. coli (63) |
| Klebsiella pneumoniae (14) | 100 | – | – | K. pneumoniae (14) |
| Klebsiella oxytoca (9) | 100 | – | – | K. oxytoca (9) |
| Raoultella ornithinolytica (2) | – | – | 100 | Enterobacter asburiae (1) |
| – | – | 100 | Enterobacter cloacae (1) | |
| Raoultella planticola (1) | – | 100 | – | R. ornithinolytica (1) |
| Proteus mirabilis (26) | 100 | – | – | P. mirabilis (26) |
| Proteus vulgaris (3) | 100 | – | – | P. vulgaris (3) |
| Providencia stuartii (2) | 100 | – | – | P. stuartii (2) |
| Morganella morganii (4) | 100 | – | – | M. morganii (4) |
| Salmonella enteritidis (4) | 25 | 100 | – | S. enteritidis (1) |
| – | Salmonella sp. (2) | |||
| – | Salmonella anatum (1) | |||
| Salmonella typhimurium (9) | 11,1 | 100 | – | S. typhimurium (1) |
| – | Salmonella sp. (5) | |||
| – | Salmonella anatum (1) | |||
| – | Salmonella dublin (2) | |||
| Salmonella grupo C (3) | – | 100 | – | Salmonella sp. (3) |
| E. cloacae (4) | 75 | 100 | – | E. cloacae (3) |
| Enterobacter hormaechei (1) | ||||
| E. asburiae (1) | 100 | – | – | E. asburiae (1) |
| Enterobacter aerogenes (1) | 100 | – | – | E. aerogenes (1) |
| Citrobacter freundii (1) | 100 | – | – | C. freundii (1) |
| Citrobacter koseri (3) | 66,7 | 100 | C. koseri (2) | |
| E. asburiae (1) | ||||
| Serratia marcescens (6) | 100 | – | S. marcescens (6) | |
| Pseudomonas aeruginosa (21) | 100 | – | – | P. aeruginosa (21) |
| Pseudomonas fluorescens (1) | – | 100 | – | Pseudomonas synxantha (1) |
| Pseudomonas putida (1) | – | 100 | – | Pseudomonas monteilii (1) |
| Stenotrophomonas maltophilia (1) | – | – | 100 | Pseudomonas beteli (1) |
| Aeromonas hydrophila (4) | 50 | 100 | – | A. hydrophila (2) |
| Aeromonas caviae (1) | ||||
| Aeromonas media (1) | ||||
| Acinetobacter baumannii (10) | 100 | – | – | A. baumannii (10) |
| Acinetobacter lwoffi (2) | – | 50 | 50 | Acinetobacter sp. (1) |
| Ochrobacter anthropi (1) | ||||
| Achromobacter xylosoxidans (1) | – | 100 | – | Achromobacter ruhlandii (1) |
| Campylobacter jejuni (20) | 100 | – | – | C. jejuni (20) |
| Campylobacter coli (1) | 100 | – | – | C. coli (1) |
| Francisella tularensis (1) | – | – | 100 | Perfil no identificable |
| Total Gram negativos (229) | 87,8 | 10 | 2,2 |
MALDI-TOF: matrix-assisted laser desorption ionization time-of-flight.
Los resultados obtenidos muestran una excelente correlación entre las identificaciones llevadas a cabo mediante ambos sistemas. El 100% de los microorganismos grampositivos, entre los que se incluían aislamientos de todos los patógenos principales de este grupo (Staphylococcus aureus, Streptococcus pyogenes, Streptococcus agalactiae, Streptococcus pneumoniae, etc.), coincidió en su identificación en la especie. Estudios previos muestran una excelente correlación entre MALDI-TOF e identificación convencional en la especie para algunos microorganismos con cifras del 94,4% para S. aureus (el 100% en el género), aunque en otros casos la identificación es buena en el género (>99% para Corynebacterium y estafilococos coagulasa negativa), pero peor en la especie (el 53,3% para Corynebacterium y el 77,7% para estafilococos coagulasa negativa)5. Un estudio reciente sobre 234 aislamientos clínicos de estafilococos coagulasa negativa pertenecientes a 20 especies9 muestra que la correlación del MALDI-TOF con la identificación mediante secuenciación del gen sodA fue del 93,2 frente al 75–76% de los sistemas comerciales convencionales con que se comparó. Dentro de los gramnegativos, las identificaciones en la especie tuvieron una correlación algo menor (87,8%), pero la correlación en el género se sitúa en el 97,8%. Dentro de las discrepancias en el género se han incluido aquellos casos en los que MALDI-TOF indicaba en su identificación de un aislamiento de Salmonella, una serovariedad distinta de la que se observaba mediante aglutinación con anticuerpos específicos, o no indicaba serovariedad alguna. De haber considerado todos los aislamientos de Salmonella como una sola especie (Salmonella enterica), la correlación en la especie hubiera sido incluso mayor (94,3%). En cuanto a las identificaciones fallidas (error en el género), ha de tenerse en cuenta que la mayor parte de ellas se producen en géneros y especies relativamente poco frecuentes, como Raoultella, o en especies en las que la identificación por métodos clásicos también puede ser con frecuencia dificultosa y sólo relativamente fiable, como Acinetobacter lwoffi o Pseudomonas putida. De hecho, estudios recientes que comparan esta metodología con la secuenciación del ácido ribonucleico ribosómico 16-S para identificación de BGNNF10 muestran que esta última metodología en muchos casos tampoco es capaz de discriminar entre P. putida y Pseudomonas monteilii, como ocurre en el presente estudio. Por otra parte, la discrepancia observada en el caso de S. maltophilia ha de valorarse con precaución al tratarse de un solo aislamiento. Alguna publicación reciente corrobora los problemas de identificación de esta especie5, pero otros estudios muestran una excelente correlación entre MALDI-TOF y ácido ribonucleico ribosómico 16-S en un amplio grupo de BGNNF, incluida S. maltophilia10. Será necesario estudiar un mayor número de aislamientos de esta especie para determinar la fiabilidad real de este sistema en la identificación de esta especie. Tiene en cambio mayor importancia, a nuestro juicio, la identificación fallida de F. tularensis. El software Biotyper no fue capaz de hallar ningún perfil en la base de datos semejante al de F. tularensis para indicar una identificación. La revisión de la versión de la base de datos utilizada mostró que no aparece en la misma especie alguna del género Francisella, lo que explica la imposibilidad de identificación y obliga, por otra parte, a una revisión de esta base de datos para completarla con un microorganismo que en nuestro medio tiene suficiente importancia como para que su inclusión sea obligada.
Un estudio reciente llevado a cabo con un diseño similar al presente11 muestra un porcentaje de identificaciones correctas del 95,2% sobre 1.116 aislamientos clínicos habituales. La correlación de la identificación con sistemas convencionales fue del 95,5% en enterobacterias, del 79,7% en BGNNF, del 99,5% en estafilococos, del 100% en enterococos y del 93,7% en estreptococos.
A diferencia de trabajos más antiguos, que medían proteínas con pesos moleculares más bajos12, los sistemas más recientes, incluido el usado en este estudio, utilizan un intervalo de 2.000–20.000kD. Este intervalo representa de forma predominante las proteínas ribosómicas obtenidas de extractos bacterianos completos13. Estas proteínas son abundantes en la célula bacteriana y están cargadas positivamente, lo que favorece su medida mediante MALDI-TOF y confiere mayor consistencia a la identificación10. Finalmente, el desarrollo de un software que permite comparar los patrones de MS con una amplia base de datos de microorganismos en función de los picos obtenidos y la intensidad de estos (se le asigna a cada identificación un nivel de fiabilidad entre 0–3) ha sido importante en el desarrollo de su uso clínico. Un ejemplo de los perfiles de un aislamiento clínico de E. coli y otro de S. aureus comparados con los perfiles de referencia de la base de datos aparece en la figura 1. De hecho, alguno de los trabajos más recientes muestra que existe una correlación entre los microorganismos para los que hay menos de 10 espectros en la base de datos y la aparición de errores de identificación5.
Estudios recientes muestran a la MS como un método extraordinariamente prometedor para la identificación de hongos patógenos. Un estudio reciente sobre 267 aislamientos de hongos, 250 de ellos pertenecientes al género Candida, obtuvo una identificación correcta en el 92,5% de los aislamientos14, y otros estudios muestran, asimismo, buenos resultados en identificación tanto de microorganismos del género Fusarium15 como de dermatofitos16.
De forma global, la identificación mediante MS tiene varias ventajas fundamentales:
- •
Sencillez. El único procedimiento por realizar es una extensión del microorganismo sobre una superficie metálica, similar a la que se realizaría sobre un porta para cualquier tinción, salvo que de un tamaño menor, y la adición sobre esta extensión de una gota de solución matriz.
- •
Rapidez. Todo el proceso de realización de la extensión, aplicación de solución matriz y tiempo de secado de ésta requiere apenas unos minutos. Posteriormente, la introducción de la placa en la que se realiza la extensión en el espectrómetro y la lectura de éste requiere un tiempo similar. En conjunto, se considera que se requieren alrededor de 11min para una sola muestra desde que se deposita en la placa MALDI hasta la obtención del resultado. Hay que tener en cuenta, no obstante, que es posible la identificación simultánea de hasta 96 microorganismos, en cuyo caso se optimizaría el tiempo de identificación por microorganismo y se reduciría a no más de 2–3min/microorganismo.
- •
Coste. El coste en fungibles es virtualmente nulo, ya que las placas metálicas de extensión y lectura son reutilizables, de modo que el único material desechable por usar es el asa con el que se realice la extensión y la solución matriz, de la que se usa 1μl por identificación. Por tanto, los únicos costes significativos serían los derivados de la amortización del espectrómetro de masas, que no obstante pueden ser importantes. El equipo MALDI-TOF Autoflex III utilizado en este estudio tiene un coste de alrededor de 250.000 euros, si bien no es el tipo de equipo que se suele recomendar para identificación habitual en Microbiología Clínica para la que pueden ser suficiente equipos más sencillos, cuyo coste se sitúa, no obstante, por encima de los 100.000 euros.
El resultado, en conjunto, es un sistema de identificación extremadamente fiable, que permite, además, disponer de esta identificación de manera prácticamente inmediata una vez que se dispone de un crecimiento suficiente en placa. Ello supone una ganancia de al menos 18–24h en la mayor parte de las identificaciones.
Se puede argumentar que con frecuencia la disponibilidad de la identificación sin datos de sensibilidad a antimicrobianos tiene una utilidad sólo relativa, y que al tener que esperar a disponer de la sensibilidad a antimicrobianos por métodos clásicos, el tiempo requerido para disponer del estudio completo no se modifica. Esto es así, en efecto, pero también es cierto que en otras ocasiones la posibilidad de disponer de manera inmediata de la identificación permite inferir perfiles probables de sensibilidad y ajustar tratamientos empíricos en consecuencia.
La utilidad de este sistema puede incluso incrementarse si, como indican algunas publicaciones, la caracterización del microorganismo se puede llevar más allá del nivel de especie, y es capaz incluso de discriminar la presencia de factores de patogenicidad o de mecanismos de resistencia. A este respecto, existen publicaciones que indican que la MS sería capaz, por ejemplo, de detectar la presencia de betalactamasas de espectro extendido17 y de diferenciar entre S. aureus sensible y resistente a meticilina18 o entre cepas productoras y no productoras de proteína de Panton-Valentine19.
Finalmente, cabría la posibilidad incluso de la utilización de este sistema directamente sobre muestras clínicas, aunque es probable que con importantes limitaciones. Por una parte, está la limitación de la sensibilidad. La MS puede ser un método muy específico desde el punto de vista de la identificación, pero no es un sistema particularmente sensible. La cantidad de microorganismos presentes en buena parte de las muestras clínicas probablemente sea insuficiente para proporcionar una identificación fiable. Por otra parte, muchas muestras contienen importantes cantidades de proteínas de origen humano que pueden interferir en los perfiles de identificación. Finalmente, incluso en ausencia de proteínas de origen humano, la presencia de más de un tipo de microorganismos originaría, asimismo, perfiles no identificables. Por tanto, probablemente solo algunos tipos de muestras de las que no es problemático obtener volúmenes grandes, en las que puede haber poblaciones importantes de microorganismos, las infecciones son habitualmente monobacterianas, y en las que las proteínas de origen humano sean escasas, como la orina, podrían ser susceptibles de identificación directa mediante este sistema.
En definitiva, los datos obtenidos en el presente estudio indican que la MS MALDI-TOF puede ser un método fiable y rápido para la identificación habitual de aislamientos clínicos bacterianos, y puede suponer el cambio más radical desde el punto de vista tecnológico que se produzca en esta área del laboratorio de Microbiología Clínica en décadas. La posibilidad de actuar directamente sobre muestras y de llevar la aplicación de esta tecnología a caracterizaciones de los microorganismos más allá del nivel de especie, e identificar factores de resistencia o de patogenicidad, abre importantes perspectivas futuras para esta nueva tecnología.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.