La detección de los mecanismos de resistencia en los microorgansimos gramnegativos tiene una gran repercusión clínica y epidemiológica, existiendo aún hoy en día una cierta discusión sobre cuál es la mejor técnica fenotípica para este fin, así como si se deben o no interpretar los resultados in vitro de sensibilidad. Se describen los fenotipos y mecanismos de resistencia a antibióticos betalactámicos, quinolonas y aminoglucósidos en bacilos gramnegativos, así como las diferentes herramientas fenotipicas disponibles para su detección e interpretación clínica. También se incluyen las betalactamasas de espectro extendido, las resistentes a los inhibidores, las de tipo AmpC y las carbapenemasas; las resistencias a quinolonas por mutaciones en los genes de la DNA girasa y la topoisomesasa IV o las mediadas por plásmidos; y los patrones de resistencia a aminoglucósidos debidos a la expresión de enzimas modificadoras. En un apartado específico se discute la detección fenotípica de la resistencia a los antibióticos betalactámicos en Neisseria spp. y Haemophilus influenzae.
Detecting resistance in gram-negative microorganisms has a strong clinical and epidemiological impact, but there is still a great deal of debate about the most sensitive phenotypic method and whether in vitro susceptibility results should be interpreted. The present work reviews the phenotypes and mechanisms of resistance to beta-lactams, quinolones and aminoglycosides in gram-negative bacilli and also revises the different phenotypic methods used for their detection. A clinical interpretation of in vitro susceptibility results is also discussed. Extended-spectrum and inhibitor resistant beta-lactamases, AmpC type beta-lactamases and carbapenemases are thoroughly reviewed. As regards quinolones, the resistance mediated both by plasmids and by mutations in the DNA gyrase and the topoisomerase IV genes is also reviewed. This report includes resistance patterns to aminoglycosides caused by modifying enzymes. Phenotypic detection of beta-lactam resistance in Neisseria spp. and Haemophilus influenzae is also reviewed in a separate section.
En la presente revisión, se describen una serie de herramientas fenotípicas útiles para la detección de determinados mecanismos de resistencia en los microorganismos gramnegativos que pueden implicar diferentes actitudes terapéuticas o tener interés epidemiológico1. La práctica totalidad de los mecanismos descritos se refieren a las enterobacterias, Pseudomonas aeruginosa y puntualmente también a otros bacilos gramnegativos no fermentadores, entre los que destaca el género Acinetobacter. La resistencia a betalactámicos en Neisseria spp. y Haemophilus spp. se tratan en un apartado específico por presentar mecanismos de resistencia peculiares.
En esta revisión se han recogido diferentes opiniones, criterios y recomendaciones aparecidas en la literatura, destacando las del Clinical and Laboratory Standards Institute (CLSI)2,3, Comité de l’Antibiogramme de la Sociedad Francesa de Microbiología (CASFM)4, European Committee on Antimicrobial Susceptibility Testing (EUCAST)5, Sociedad Española de Enfermedades Infecciosas y Microbiología Clínica (SEIMC), del grupo GEMARA (Grupo de estudio de los mecanismos de acción y resistencia a los antimicrobianos) y antiguas recomendaciones de MENSURA (Mesa Española de Normalización de las Pruebas de Sensibilidad a los Antimiocrobianos)6,7. Por último, es necesario aclarar que en la mayoría de los casos (excepto en los apartados en los que se hace mención expresa a las recomendaciones terapéuticas), los términos de sensible o resistente no se refieren necesariamente a las categorías clínicas de interpretación basadas en los puntos de corte sino a consideraciones microbiológicas de pertenencia a las poblaciones salvajes (sin mecanismos de resistencia y por tanto sensible) o la expresión de un mecanismo de resistencia.
Betalactamasas resistentes a los inhibidores (IRT, OXA)La asociación amoxicilina-ácido clavulánico presenta actividad frente a un gran número de enterobacterias incluyendo las resistentes a la amoxicilina por producción de betalactamasas de amplio espectro como TEM-1, TEM-2 o SHV-1. Estas betalactamasas hidrolizan penicilinas (aminopenicilinas [ampicilina, amoxicilina], carboxipenicilinas [ticarcilina]) y, como se ha mencionado, son sensibles a los inhibidores de betalactamasa. A partir de estas enzimas y mediante mutaciones puntuales aparecieron las betalactamasas resistentes a la inhibición por los inhibidores de betalactamasas. Estas enzimas se han denominado IRT (inhibitor-resistant TEM mutant) porque en su mayoría derivan de TEM-1 y TEM-2, aunque también se han descrito derivadas de SHV-1. Además, algunas oxacilinasas como la OXA-1, también confieren un fenotipo similar al de las IRT, que se caracteriza por resistencia a aminopenicilinas, carboxipenicilinas y ureidopenicilinas siendo insensibles a la acción de los inhibidores de betalactamasa de clase A como el ácido clavulánico y, en su gran mayoría no tienen actividad sobre el resto de betalactámicos8-12.
Detección fenotípica de betalactamasas resistentes a los inhibidoresLa detección de estas enzimas es factible solo en enterobacterias naturalmente sensibles a la asociación amoxicilina-ácido clavulánico. Por tanto, y debido a la superposición de mecanismos de resistencia, su presencia no puede detectarse fenotípicamente en las enterobacterias naturalmente resistentes a esta asociación como Enterobacter cloacae, Enterobacter aerogenes, Citrobacter freundii, Morganella morganii, Serratia marcescens o los bacilos gramnegativos no fermentadores (P. aeruginosa) portadores de betalactamasa tipo AmpC inducible13,14. Así, para el resto de enterobacterias a partir de los antibiogramas realizados, bien por la técnica de disco-difusión o bien por la de microdilución, se puede sospechar la presencia de las enzimas IRT y OXA, aunque su confirmación definitiva debe ser realizada mediante técnicas moleculares dado que son varios los mecanismos que pueden dar patrones de resistencia similares. En la tabla 1 se muestran algunas características fenotípicas diferenciales de los distintos mecanismos implicados en la resistencia a las asociaciones betalactámico-inhibidor de betalactamasa15.
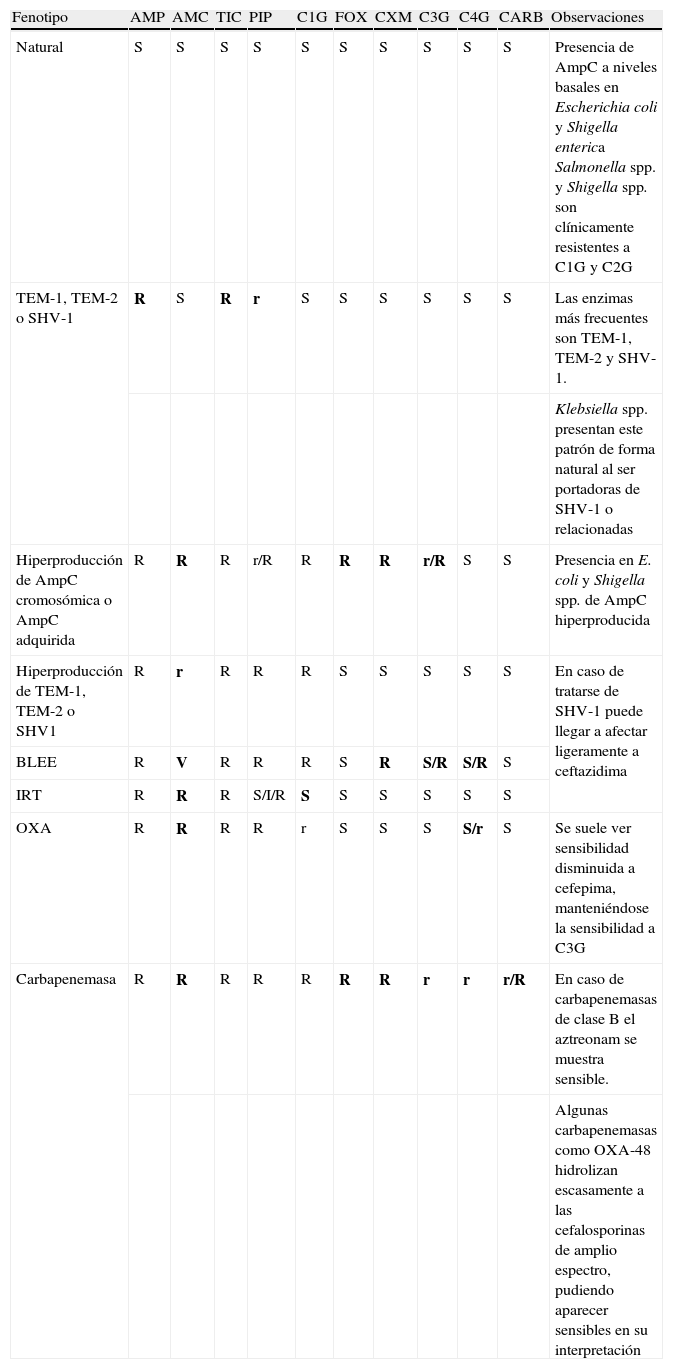
Principales patrones de resistencia a betalactámicos en función de la betalactamasa implicada en las principales enterobacterias que carecen de betalactamasa tipo AmpC cromosómica inducible.
| Fenotipo | AMP | AMC | TIC | PIP | C1G | FOX | CXM | C3G | C4G | CARB | Observaciones |
| Natural | S | S | S | S | S | S | S | S | S | S | Presencia de AmpC a niveles basales en Escherichia coli y Shigella enterica Salmonella spp. y Shigella spp. son clínicamente resistentes a C1G y C2G |
| TEM-1, TEM-2 o SHV-1 | R | S | R | r | S | S | S | S | S | S | Las enzimas más frecuentes son TEM-1, TEM-2 y SHV-1. |
| Klebsiella spp. presentan este patrón de forma natural al ser portadoras de SHV-1 o relacionadas | |||||||||||
| Hiperproducción de AmpC cromosómica o AmpC adquirida | R | R | R | r/R | R | R | R | r/R | S | S | Presencia en E. coli y Shigella spp. de AmpC hiperproducida |
| Hiperproducción de TEM-1, TEM-2 o SHV1 | R | r | R | R | R | S | S | S | S | S | En caso de tratarse de SHV-1 puede llegar a afectar ligeramente a ceftazidima |
| BLEE | R | V | R | R | R | S | R | S/R | S/R | S | |
| IRT | R | R | R | S/I/R | S | S | S | S | S | S | |
| OXA | R | R | R | R | r | S | S | S | S/r | S | Se suele ver sensibilidad disminuida a cefepima, manteniéndose la sensibilidad a C3G |
| Carbapenemasa | R | R | R | R | R | R | R | r | r | r/R | En caso de carbapenemasas de clase B el aztreonam se muestra sensible. |
| Algunas carbapenemasas como OXA-48 hidrolizan escasamente a las cefalosporinas de amplio espectro, pudiendo aparecer sensibles en su interpretación |
En negrita se resaltan los antibióticos clave para la sospecha de cada una de las betalactamasas implicadas.
AMC: amoxicilina-ácido clavulánico; AMP: ampicilina; CARB: carbapenémicos; C1G: cefalosporinas de primera generación; C3G: cefalosporinas de tercera generación y monobactámicos; C4G: cefalosporinas de cuarta generación; CXM: cefuroxima; FOX: cefoxitina; PIP: piperacilina; R: resistente; r: halos reducidos o CMI elevadas con respecto al fenotipo salvaje, pero habitualmente dentro del rango de sensibilidad; S: sensible; TIC: ticarcilina; V: variable.
Generalmente, una cepa que expresa una betalactamasa de tipo IRT presentará resistencia a aminopenicilinas, carboxipenicilinas, ureidopenicilinas (en mayor o menor medida) y sensibilidad disminuida o resistencia a amoxicilina-ácido clavulánico9,10,13. Estas cepas muestran además sensibilidad a las cefalosporinas, incluyendo las de primera generación. También se afecta la asociación ampicilina-sulbactam y generalmente suele mantenerse o disminuir levemente la sensibilidad a piperacilina-tazobactam, posiblemente por la acción intrínseca de la piperacilina. Este patrón se puede ver alterado ante la presencia añadida de otros mecanismos de resistencia15.
Las cepas portadoras de las betalactamasas tipo OXA comentadas previamente (principalmente OXA-1) suelen presentar patrones similares a las portadoras de IRT. Así presentan resistencia a aminopenicilinas, carboxipenicilinas, ureidopenicilinas y sensibilidad disminuida o resistencia a amoxicilina-ácido clavulánico, ampicilina-sulbactam y piperacilina-tazobactam. Una característica de las enzimas de tipo OXA es que generalmente presentan una menor sensibilidad a cefepima. Debido a una cierta actividad del ácido clavulánico y a la menor sensibilidad de cefepima, es frecuente observar (especialmente mediante la técnica de disco-difusión) sinergia entre el ácido clavulánico y cefepima, siendo este patrón característico de las enzimas tipo OXA-1.
Información de los resultados y recomendaciones terapéuticasAnte la presencia de enzimas tipo IRT u OXA, deberíamos prescindir de tratamientos con las asociaciones de antibióticos betalactámicos e inhibidores de betalactamasa como amoxicilina-ácido clavulánico y ampicilina-sulbactam, presentándose como válida cualquier otra alternativa en función del antibiograma, del foco infeccioso y de la situación clínica del paciente. En el caso de la asociación piperacilina-tazobactam, debido a la propia actividad de la piperacilina, podría plantearse como alternativa siempre que se observe sensibilidad in vitro. En el caso de enterobacterias portadoras de betalactamasas tipo OXA debe plantearse también la conveniencia de evitar el uso de cefepima, especialmente si esta betalactamasa está hiperproducida y se observa disminución de la sensibilidad in vitro.
Betalactamasas de espectro extendido (BLEE)Un grupo importante de enzimas son las betalactamasas de espectro extendido (BLEE) que tienen capacidad de hidrolizar y causar resistencia o sensibilidad disminuida a penicilinas, oximino-cefalosporinas (cefotaxima, ceftriaxona, ceftazidima, cefepima) y monobactámicos (aztreonam), pero no a cefamicinas (cefoxitina) ni carbapenémicos (imipenem, meropenem y ertapenem), siendo inhibidas por el ácido clavulánico16. Estas betalactamasas pertenecen a la clase molecular A de Ambler y entre ellas se encuentran las de tipo TEM y SHV (derivadas de enzimas con menor espectro de hidrólisis), la familia CTX-M (procedente de betalactamasas cromosómicas del género Kluyvera), y otras menos prevalentes como PER, VEB, BES, GES, TLA y SFO, incluidas todas ellas en el grupo funcional 2be de Bush y Jacoby17-20.
Otras enzimas BLEE también pertenecientes a la clase A, aunque del subgrupo 2ber son las betalactamasas CMT (complex mutant TEM) como la TEM-50 que combinan una cierta resistencia a la inhibición por el ácido clavulánico junto a una mayor actividad frente a oximino-cefalosporinas9. Algunas enzimas de la familia OXA (clase D de Ambler y grupo funcional 2de), se consideran también betalactamasas de espectro extendido y se han descrito con mayor frecuencia en P. aeruginosa.
Desde su descripción inicial, se han identificado más de 300 BLEE diferentes, y la mayoría pertenece a las familias TEM, SHV y CTX-M (http://www.lahey.org/webt.asp).
Detección fenotípica de betalactamasas de espectro extendidoLa detección de las BLEE en el laboratorio no siempre es fácil, ya que depende de su expresión fenotípica y esto viene condicionado por la cantidad de enzima producida por la bacteria, y de la presencia o no de otros mecanismos de resistencia. Su detección se basa en la capacidad de estas enzimas de hidrolizar las cefalosporinas de tercera y cuarta generación y los monobactámicos, disminuyendo por tanto la sensibilidad de la bacteria a estos antibacterianos. Otra de las características de estas enzimas es que son inhibidas por el ácido clavulánico (tabla 1)13,17.
Se han desarrollado diversas pruebas fenotípicas para la detección de BLEE, la mayoría basadas en la actividad inhibitoria del ácido clavulánico15. Entre ellas destaca la técnica de disco-difusión en la que la presencia de una BLEE se sospecha no solo por la resistencia o disminución de los halos de inhibición de algunos o todos los sustratos sino también por el efecto sinérgico producido entre las cefalosporinas de amplio espectro o los monobactámicos y el ácido clavulánico, cuando previamente se han situado de forma estratégica los discos15,17,18,21.
Otras técnicas basadas en el mismo principio son la utilización de discos combinados de cefalosporinas con ácido clavulánico y su variante en las técnicas de microdilución que permiten conocer las CMI de las cefalosporinas solas y en presencia de inhibidor. La técnica de difusión en gradiente (Etest) con tiras combinadas de cefalosporinas con y sin inhibidor es también de utilidad para la detección de BLEE15.
Todas estas pruebas requieren como mínimo 48 horas desde que el producto patológico llega al laboratorio. Se han buscado nuevos métodos para acortar este tiempo por lo que se han diseñado medios cromogénicos para el aislamiento selectivo y la identificación presuntiva de enterobacterias productoras de BLEE. Entre ellos se encuentra el ChromID ESBL (bioMérieux), Brilliance ESBL agar (Oxoid) y el CHROMagar™ ESBL (CHROMagar).
Otro método cromogénico rápido es el Cica-beta-Test (Kanto Chemical) que se utiliza para la detección rápida de BLEE directamente de la colonia de enterobacteria aislada. El método utiliza una cefalosporina cromogénica (HMRZ-86) y el ácido clavulánico como inhibidor para detectar rápidamente si el aislado es portador o no de una BLEE. Esta técnica permite asimismo detectar metalobetalactamasas y AmpC hiperproducidas mediante el uso de EDTA y ácido borónico, respectivamente.
Es importante recordar que algunas enterobacterias poseen betalactamasas cromosómicas que hidrolizan las cefalosporinas y son inhibidas por el ácido clavulánico. Cuando se hiperproducen dan lugar a un patrón fenotípico de resistencia compatible con la presencia de una BLEE. Entre ellas se encuentra la betalactamasa K1 de Klebsiella oxytoca, la SHV-1 de Klebsiella pneumoniae y las cefalosporinasas CepA de Proteus vulgaris y Proteus penneri.
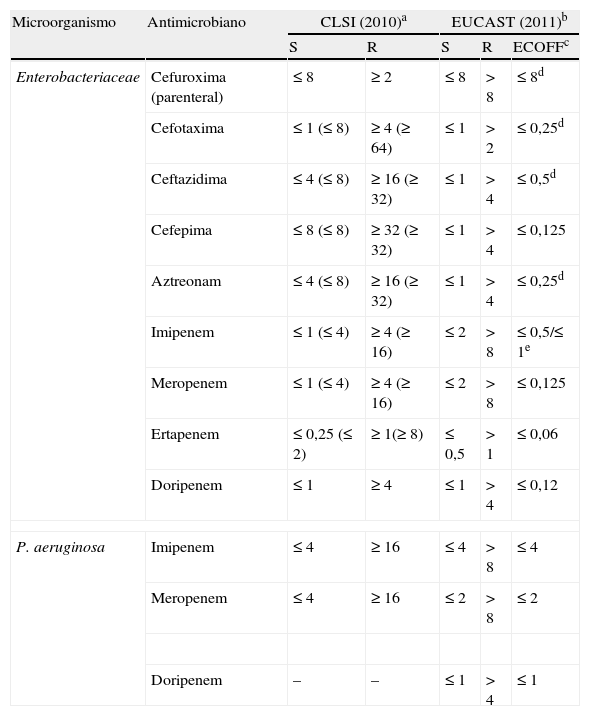
Información de los resultados y recomendaciones terapéuticasEl grado de hidrólisis frente a cefalosporinas de tercera y cuarta generación y monobactámicos puede variar según el tipo de BLEE y el nivel de producción, pudiendo aparecer sensibles in vitro a algunos de estos antibacterianos22. El CLSI antes del año 20102 recomendaba informar las cepas con fenotipo de BLEE como resistentes a penicilinas, cefalosporinas y aztreonam indistintamente del valor de la CMI o del halo de inhibición mientras que el EUCAST5 recomendaba interpretar como intermedio un resultado sensible y como resistente un resultado intermedio. En el año 2010 ambos comités modificaron los puntos de corte de las cefalosporinas y aztreonam basándose en estudios PK/PD y efectuaron una nueva recomendación consistente en informar la sensibilidad de los aislados con BLEE según los resultados obtenidos en las pruebas de sensibilidad in vitro independientemente del mecanismo de resistencia3,5 (tabla 2).
Puntos de corte actuales (mg/L) para Enterobacteriaceae y Pseudomonas aeruginosa del CLSI y EUCAST
| Microorganismo | Antimicrobiano | CLSI (2010)a | EUCAST (2011)b | |||
| S | R | S | R | ECOFFc | ||
| Enterobacteriaceae | Cefuroxima (parenteral) | ≤ 8 | ≥ 2 | ≤ 8 | > 8 | ≤ 8d |
| Cefotaxima | ≤ 1 (≤ 8) | ≥ 4 (≥ 64) | ≤ 1 | > 2 | ≤ 0,25d | |
| Ceftazidima | ≤ 4 (≤ 8) | ≥ 16 (≥ 32) | ≤ 1 | > 4 | ≤ 0,5d | |
| Cefepima | ≤ 8 (≤ 8) | ≥ 32 (≥ 32) | ≤ 1 | > 4 | ≤ 0,125 | |
| Aztreonam | ≤ 4 (≤ 8) | ≥ 16 (≥ 32) | ≤ 1 | > 4 | ≤ 0,25d | |
| Imipenem | ≤ 1 (≤ 4) | ≥ 4 (≥ 16) | ≤ 2 | > 8 | ≤ 0,5/≤ 1e | |
| Meropenem | ≤ 1 (≤ 4) | ≥ 4 (≥ 16) | ≤ 2 | > 8 | ≤ 0,125 | |
| Ertapenem | ≤ 0,25 (≤ 2) | ≥ 1(≥ 8) | ≤ 0,5 | > 1 | ≤ 0,06 | |
| Doripenem | ≤ 1 | ≥ 4 | ≤ 1 | > 4 | ≤ 0,12 | |
| P. aeruginosa | Imipenem | ≤ 4 | ≥ 16 | ≤ 4 | > 8 | ≤ 4 |
| Meropenem | ≤ 4 | ≥ 16 | ≤ 2 | > 8 | ≤ 2 | |
| Doripenem | – | – | ≤ 1 | > 4 | ≤ 1 | |
aDocumento M100-S20-U, junio de 2010, y documento M100-S20. Entre parentesis los puntos de corte anteriores a estos documentos; bversión 1.3, enero 2011; epunto de corte epidemiológico; dE. coli; eE.coli//K. pneumoniae.
A pesar de la disminución de los puntos de corte, que parecen dar una buena prediccion de la evolución clínica al tratamiento, sigue existiendo discusión sobre la influencia del inóculo bacteriano en el foco de infección. Por lo tanto, la decisión de seguir esta recomendación dependerá de criterios locales atendiendo a criterios epidemiológicos y de política de antimicrobianos y a la realización de estudios clínicos que aseguren la eficacia terapéutica de estos antibióticos en el tratamiento de infecciones producidas por bacterias productoras de BLEE en diferentes situaciones clínicas.
Betalactamasas tipo AmpCLas betalactamasas de la clase molecular C de Ambler (grupo 1 de la clasificación de Bush-Jacoby-Medeiros)18 hidrolizan cefalosporinas de primera y segunda generación, incluidas las cefamicinas y, en menor medida las de tercera generación, mientras que generalmente son muy poco eficaces hidrolizando las cefalosporinas de cuarta generación y los carbapenémicos13,23. Este espectro de hidrólisis puede ampliarse y afectar además a cefalosporinas de cuarta generación (AmpC de espectro extendido), pero se desconoce cuál es la prevalencia y la relevancia clínica y epidemiológica de estas variantes de AmpC. La cloxacilina y el aztreonam, así como el ácido borónico y sus derivados (ácido fenil-borónico), inhiben a las betalactamasas de tipo AmpC, mientras que el ácido clavulánico, sulbactam y tazobactam no son buenos inhibidores (tabla 1).
La producción de AmpC puede ser constitutiva o inducible, siendo los niveles de producción dependientes del grado de expresión del gen blaAmpC23. Cuando el gen blaAmpC se expresa de forma constitutiva (ausencia de genes reguladores del tipo ampD o ampR) puede hacerlo a niveles basales bajos, confiriendo un fenotipo de resistencia natural o salvaje característico de la especie bacteriana (tabla 3), o puede hacerlo a unos niveles muy superiores al basal (sobreexpresión de blaAmpC mediada por mutaciones en el atenuador y/o promotor de blaAmpC, adquisición de promotores fuertes para la expresión de blaAmpC) produciendo cantidades elevadas de AmpC (hiperproducción de AmpC). En determinadas especies bacterianas como E. cloacae, M. morganii, P. aeruginosa etc. el gen blaAmpC se expresa de forma inducible. En los aislados que tienen un gen blaAmpC inducible, su expresión puede estar desreprimida establemente de forma parcial o total (mutaciones en genes reguladores de tipo ampD y ampR) dando lugar a la producción estable de grandes cantidades de AmpC (hiperproducción parcial o total de AmpC)13,18,23,24.
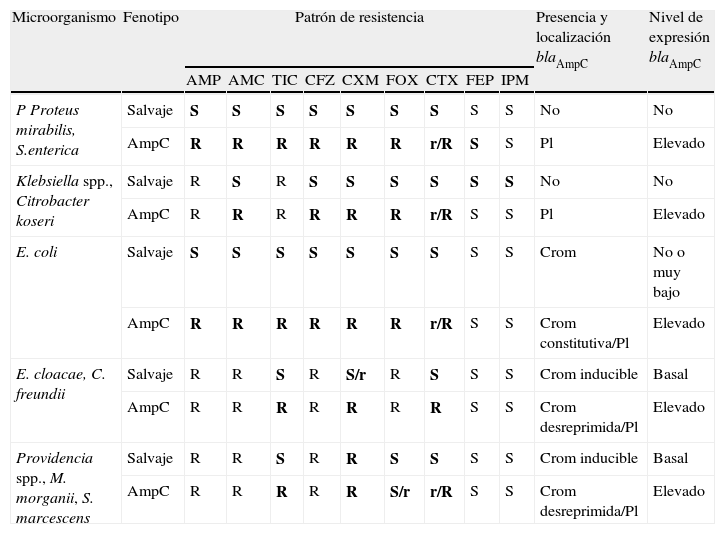
Patrones de resistencia a antibióticos betalactámicos en algunas enterobacterias de interés clínico y epidemiológico
| Microorganismo | Fenotipo | Patrón de resistencia | Presencia y localización blaAmpC | Nivel de expresión blaAmpC | ||||||||
| AMP | AMC | TIC | CFZ | CXM | FOX | CTX | FEP | IPM | ||||
| P Proteus mirabilis, S.enterica | Salvaje | S | S | S | S | S | S | S | S | S | No | No |
| AmpC | R | R | R | R | R | R | r/R | S | S | Pl | Elevado | |
| Klebsiella spp., Citrobacter koseri | Salvaje | R | S | R | S | S | S | S | S | S | No | No |
| AmpC | R | R | R | R | R | R | r/R | S | S | Pl | Elevado | |
| E. coli | Salvaje | S | S | S | S | S | S | S | S | S | Crom | No o muy bajo |
| AmpC | R | R | R | R | R | R | r/R | S | S | Crom constitutiva/Pl | Elevado | |
| E. cloacae, C. freundii | Salvaje | R | R | S | R | S/r | R | S | S | S | Crom inducible | Basal |
| AmpC | R | R | R | R | R | R | R | S | S | Crom desreprimida/Pl | Elevado | |
| Providencia spp., M. morganii, S. marcescens | Salvaje | R | R | S | R | R | S | S | S | S | Crom inducible | Basal |
| AmpC | R | R | R | R | R | S/r | r/R | S | S | Crom desreprimida/Pl | Elevado | |
En negrita se resalta los betalactámicos en los que existen diferencias entre el fenotipo natural o salvaje y el fenotipo AmpC.
AMC: amoxicilina-ácido clavulánico; AMP: ampicilina; CFZ: cefazolina; Crom: cromosómica; CXM: cefuroxima; FEP: cefepima; FOX: cefoxitina; IPM: imipenem; Pl: plasmídica; R: resistencia; r: sensibilidad intermedia; S: sensible; TIC: ticarcilina.
Independientemente del mecanismo que conduce a una hiperproducción de AmpC, los aislados hiperproductores de AmpC presentan un fenotipo de resistencia (fenotipo AmpC) a las penicilinas, las asociaciones de betalactámicos con inhibidores de betalactamasa, cefalosporinas de primera y segunda generación, incluidas generalmente las cefamicinas, así como a las de tercera generación, pero en grado variable, dependiendo del nivel de hiperproducción. Los aislados con este fenotipo AmpC suelen ser además sensibles a cefalosporinas de cuarta generación y a los carbapenémicos, aunque dicha sensibilidad se reduce significativamente si se produce la pérdida de alguna porina relacionada con la resistencia antimicrobiana13,23.
Un grupo de betalactamasas de tipo AmpC están codificadas por genes blaAmpC asociados a integrones, como los de clase 1, o transposones localizados en plásmidos conjugativos (AmpC plasmídicas). Estos genes blaAmpC plasmídicos proceden del cromosoma bacteriano y se clasifican en 6 familias que se diferencian por la homología de sus genes: CIT (derivadas de AmpC cromosómica de C. freundii), DHA (derivadas de AmpC cromosómica de M. morganii), ACC (cuyo origen está relacionado con AmpC cromosómica de Hafnia alvei), FOX (derivadas probablemente de AmpC cromosómica de Aeromonas media), MOX (presumiblemente derivadas de AmpC cromosómica de Aeromonas caviae), EBC (derivadas de las AmpC cromosómicas de E. cloacae y/o Enterobacter asburiae)13,18,23,24.
Las betalactamasas de tipo AmpC plasmídicas pueden causar fracasos terapéuticos, similares a los descritos en infecciones causadas por aislados hiperproductores de AmpC cromosómica inducible (selección de mutantes con desrepresión estable) en tratamientos con betalactámicos.
Detección fenotípica de betalactamasas tipo AmpCLos métodos fenotípicos para la detección de AmpC plasmídicas son sencillos y económicos, pero solo resultan de utilidad en aislados que no tienen una AmpC cromosómica natural (Kebsiella spp., S. enterica, P. mirabilis) o que la expresan constitutivamente a muy bajo nivel, como ocurre en E. coli. La presencia de AmpC plasmídica debe sospecharse cuando estos aislados presenten un patrón de resistencias a betalactámicos (fenotipo AmpC) diferente al de su respectivo fenotipo salvaje o de resistencia natural (tabla 3), siendo los marcadores de mayor utilidad la sensibilidad intermedia o resistencia a amoxicilina-ácido clavulánico y a algunas de las cefalosporinas de tercera generación13,15,23.
Los métodos fenotípicos más rentables por su eficacia, su sencillez y su bajo coste económico, son el método de sinergia de doble disco (usando discos de cloxacilina o ácido fenil-borónico y discos de cefotaxima y ceftazidima) y el método de discos combinados con inhibidores15. Existen otros métodos fenotípicos bastante sensibles, pero son más complejos (test en 3D) o más caros que los anteriores (agar cefoxitina, Etest de cefotetán/cefotetán más cloxacilina)23. En el caso concreto de E. coli, la utilización del método de inducción de AmpC puede resultar útil en la detección de AmpC plasmídica puesto que un resultado positivo solo es posible si media la adquisición de una AmpC plasmídica inducible y descarta sin lugar a dudas la hiperproducción de la AmpC cromosómica, dado que ésta no es inducible23. Se ha descrito otro método simple para diferenciar las AmpC plasmídicas de las AmpC cromosómicas que puede ser de utilidad. Los aislados productores de AmpC plasmídicas suelen presentar colonias dispersas por el borde de los halos de inhibición con discos de cefoxitina, cefotaxima, ceftazidima y aztreonam25.
Los métodos fenotípicos de detección de betalactamasas de tipo AmpC plasmídicas tienen varias limitaciones importantes que deben ser consideradas para poder realizar una interpretación fiable de los resultados obtenidos. Estos métodos aún no han sido estandarizados por ningún comité u organización de expertos (CLSI, EUCAST, CASFM)2-5. Un marcador fenotípico muy utilizado para diferenciar la producción de AmpC de la de BLEE es la cefoxitina. Salvo algunas excepciones, los aislados con fenotipo AmpC son generalmente resistentes a cefoxitina, mientras que los aislados productores de BLEE suelen ser sensibles, excepto cuando se produce la pérdida o una disminución en la expresión de alguna porina15.
Información de los resultados y recomendaciones terapéuticasEn los aislados en los que se ha detectado la producción de una AmpC plasmídica, tanto si éstos no tienen una AmpC cromosómica, como Klebsiella spp., S. enterica y P. mirabilis, como si la tienen, pero ésta no es inducible, como ocurre por ejemplo con E. coli, los valores de sensibilidad obtenidos in vitro deben informarse sin que sea necesaria la realización de una lectura interpretada de los mismos. En estos casos es aconsejable recomendar el uso de antimicrobianos alternativos a las cefalosporinas de tercera generación, aunque no existen criterios unificados ni consensuados sobre esta recomendación13,23.
El CASFM4 recomienda que cuando las pruebas de sensibilidad antimicrobiana indican que el aislado presenta sensibilidad disminuida o resistencia a algunas de las cefalosporinas de tercera generación se informen todas ellas como resistentes (si presentan sensibilidad intermedia) o con sensibilidad intermedia (si son sensibles), independientemente de que el microorganismo produzca una AmpC cromosómica o una AmpC plasmídica. En el caso que sean sensibles a todas las cefalosporinas, se aconseja informar, particularmente con Enterobacter spp. y C. freundii, la posibilidad de que se produzca un fracaso terapéutico si el tratamiento se realiza con cefalosporias de tercera generación, por la selección de mutantes AmpC establemente desreprimidos4,13. Si después de 3-4 días de tratamiento antimicrobiano continúa aislándose la misma especie bacteriana se recomienda repetir las pruebas de sensibilidad para determinar si se ha producido un incremento en la resistencia a betalactámicos.
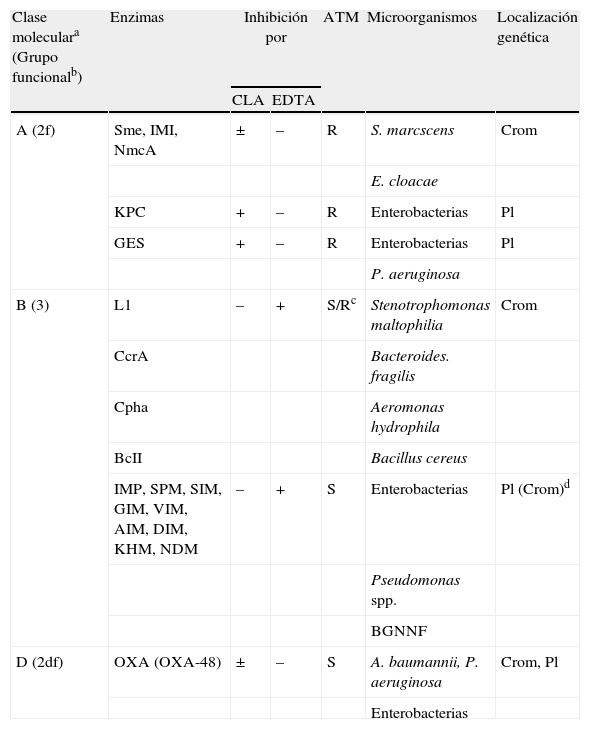
CarbapenemasasEn los últimos años se ha producido una gran alarma y preocupación por la gran dispersión de los bacilos gramnegativos resistentes a los carbapenémicos por producción de betalactamasas capaces de hidrolizar este grupo de antimicrobianos y que se han asociado a elementos genéticos trasferibles26-29. Estas enzimas se denominan genéricamente carbapanemasas y se agrupan en las diferentes clases moleculares de Ambler que se corresponden con diferentes grupos funcionales de la clasificación de Bush y Jacoby del año 201018. En la tabla 4 se recogen estas enzimas, indicando las más relevantes y los microorganismos en los que se encuentran habitualmente. Asimismo, en las tablas 1 y 4 se muestran sus características más sobresalientes y que pueden utilizarse para su reconocimiento fenotípico.
Clasificación general de las carbapenemasas
| Clase moleculara (Grupo funcionalb) | Enzimas | Inhibición por | ATM | Microorganismos | Localización genética | |
| CLA | EDTA | |||||
| A (2f) | Sme, IMI, NmcA | ± | – | R | S. marcscens | Crom |
| E. cloacae | ||||||
| KPC | + | – | R | Enterobacterias | Pl | |
| GES | + | – | R | Enterobacterias | Pl | |
| P. aeruginosa | ||||||
| B (3) | L1 | – | + | S/Rc | Stenotrophomonas maltophilia | Crom |
| CcrA | Bacteroides. fragilis | |||||
| Cpha | Aeromonas hydrophila | |||||
| BcII | Bacillus cereus | |||||
| IMP, SPM, SIM, GIM, VIM, AIM, DIM, KHM, NDM | – | + | S | Enterobacterias | Pl (Crom)d | |
| Pseudomonas spp. | ||||||
| BGNNF | ||||||
| D (2df) | OXA (OXA-48) | ± | – | S | A. baumannii, P. aeruginosa | Crom, Pl |
| Enterobacterias | ||||||
aSegún la clasificación de Ambler; bSegún la clasificación de Bush y Jacoby, 2010; cPuede aparecer resistente por la coexistencia con otros mecanismos de resistencia; dOcasionalmente de codificación cromosómica.
ATM: aztreonam; BGNNF: bacilos gramnegativos no fermentadores; CLA: ácido clavulánico; Crom, cromosómica; Pl, plasmídica
El grupo más importante de carbapenemasas lo constituyen las metalo-betalactamasas pertenecientes a la clase B o grupo 3 de Bush y Jacoby. Las enzimas principales son las IMP y VIM que tienen un perfil hidrolítico que incluye todos los antibióticos betalactámicos con la excepción del aztreonam y no se inhiben por el ácido clavulánico, sulbactan o tazobactam. Sin embargo, se inhiben por agentes quelantes de cationes divalentes como el EDTA, compuestos tiólicos como el ácido 2-mercaptopropiónico, o el ácido dipicolínico26,30. Con características similares se han descrito enzimas de los grupos SPM, GIM, SIM, AIM, DIM y KHM, y más recientemente el enzima NDM-1 que ha creado una importante alarma mediática debido al perfil mutirresistente o panresistente de los aislados que la producen28,31. El perfil hidrolítico que presenta esta enzima es similar a la de otras metalo-betalactamasas32.
Otro grupo importante de carbapenemasas son las de clase A (grupo 2f). Estas enzimas, cuyo primer representante fue la betalactamasa SME, confieren un fenotipo con pérdida marcada de sensibilidad a los carbapenémicos y un perfil hidrolítico que incluye el aztreonam y en menor medida a las cefalosporinas de tercera y cuarta generación. No son inhibidas por el EDTA, pero como peculiaridad destaca la inhibición parcial por ácido clavulánico (mejor con tazobactam). Otras enzimas relacionadas son las de los grupos IMI (IMI-1 y -2) y NMC-A. No obstante, dentro de las carbapenemasas de clase A, las que tienen mayor importancia epidemiológica son las denominas KPC. Son de naturaleza plasmídica asociadas al trasposón Tn4401. Asimismo, y aunque no de manera exclusiva, se han encontrado mayoritariamente ligadas a la secuencia tipo (ST) 258 de K. pneumoniae. Las enzimas KPC se han descrito no solo en Enterobacteriaceae sino también en P. aeruginosa y en A. baumannii. En España su aparición ha sido más tardía y no está ligada al clon ST25833,34.
Desde el punto de vista fenotípico, las enzimas KPC hidrolizan de forma eficiente penicilinas, cefalosporinas y carbapenémicos. Como excepción tendrían una menor tasa de hidrólisis de las cefamicinas aunque los valores de CMI que se obtienen suelen estar por encima del punto de corte de sensibilidad. No se inhiben por el ácido clavulánico, pero sí por el ácido borónico, inhibidor que se utiliza para su reconocimiento fenotípico. No obstante, la inhibición por el ácido borónico no es exclusiva de las enzimas KPC ya que también es un inhibidor eficiente de las betalactamasas de tipo AmpC y que con la excepción del enzima CMY-10 no hidrolizan carbapenémicos15.
Dentro de las carbapenemasas de clase A deben citarse también algunas variantes de las BLEE de tipo GES como GES-4 encontrada en P. aeruginosa, Acinetobacter spp. y en enterobacterias que hidroliza de forma eficiente penicilinas y cefalosporinas y muy débilmente a los carbapenémicos.
En el grupo de las OXA (clase D de Ambler y 2df de Bush y Jacoby) también se encuentran variantes que hidrolizan los carbapenémicos27. Entre ellas destacan las variantes de los subgrupos OXA-23, OXA-24, OXA-58, OXA-143 y, en menor medida, OXA-51 descritas en Acinetobacter spp. y sobre todo la OXA-48 descrita en enterobacterias en países del entorno mediterráneo. La detección fenotípica de OXA-48 es compleja ya que la hidrólisis de los carbapenémicos es poco eficiente y prácticamente inexistente para las cefalosporinas de tercera y cuarta generación15. El perfil de sensibilidad que confieren mantiene las características generales de las OXA al ser poco inhibida por el ácido clavulánico, sulbactam o tazobactam. Por ello, en un antibiograma habitual de K. pneumoniae o E. coli, enterobacterias en las que mayoritariamente se ha encontrado la OXA-48, se mostrarían resistentes a las penicilinas y sus asociaciones con los inhibidores de betalactamasas de clase A, sensibles a las cefalosporinas y con pérdida de sensibilidad a los carbapenémicos.
Detección fenotípica de carbapenemasasPara la detección fenotípica de las carbapenemasas se debe tener en cuenta el perfil hidrolítico general que confiere cada una de sus clases y de manera específica cada una de las enzimas incluidas en estas clases, la posible inhibición por los diferentes inhibidores de betalactamasas, la epidemiología local y la identidad del microorganismo en el que se pretende detectar o inferir la producción de estas enzimas. En este último punto es esencial valorar la posible presencia de otros mecanismos de resistencia que puedan «enmascarar» el fenotipo que confieren las cabapenemasas, entre ellos la alteración de la permeabilidad, la presencia de bombas de expulsión, afectación de las PBPs o presencia simultánea de otras betalactamasas. En este sentido, no es igual la expresión de una carbapenemasa en P. aeruginosa o en A. baumannii que en E. coli, K. pneumoniae o en una cepa de E. cloacae. Cada una de estas especies tiene sus peculiaridades fenotípicas naturales que deben ser contempladas13,26,28,30.
Desde un punto de vista práctico y una vez observado en el antibiograma, la expresión de un fenotipo compatible con la presencia de una carbapenemasa, generalmente ilustrado por la sensibilidad disminuida o resistencia a las cefalosporinas de amplio espectro y a alguno de los carbapenémicos, es importante verificar que existe un mecanismo de inactivación de los carbapenémicos. Se recomienda investigar este hecho en las cepas en las que los valores de CMI de los carbapenémicos se incrementan por encima de los correspondientes puntos de corte epidemiológicos (separan las poblaciones salvajes de aquellas que presentan mecanismos de resistencia). El método de referencia no siempre al alcance de todos los laboratorios, sería el ensayo espectrofotométrico, por lo que se han propuesto métodos biológicos (bioensayos) sencillos que permiten su detección15. La prueba modificada de Hodge35 fue recomendada por vez primera por el CLSI en el año 2009 como test fenotípico de confirmación. Tiene una elevada sensibilidad, pero no sirve para la diferenciación del tipo de carbapenemasa. A pesar de su sencillez, el test de Hodge modificado tiene detractores por las discordancias observadas con algunas cepas. Se han observado resultados falsos negativos por la baja expresión de la carbapenemasa, sobre todo con cepas con metalo-betalactamasa y oxacilinasas. No obstante, los falsos negativos pueden evitarse añadiendo sulfato de zinc al medio que incrementan la expresión del enzima (en el caso de las metalo-betalactamasas). También se han comunicado falsos positivos con cepas productoras de CTX-M-15 y pérdida de porinas y con las cepas hiperproductoras de AmpC. Asimismo, se ha discutido cuál es el carbapenémico más adecuado para el test de Hodge modificado, recomendándose la utilización de meropenem y ertapenem15,26,35.
Para la detección de microorganismos productores de carbapenemasas directamente de muestras clínicas se ha propuesto la utilización de medios cromogénicos, entre ellos los que se emplean para la detección de BLEE y los que específicamente se han diseñado para la detección de KPC. Los medios cromogénicos para BLEE detectan la presencia de microorganismos con carbapenemasas de las clases A y B, pero no las cepas que tienen OXA-48. No obstante, tienen el inconveniente de ser poco específicos dado que en ellos también crecen los microorganismos productores de BLEE. Los medios cromogénicos específicos para carbapenemasas como el CHROMagar™ KPC (CHROMagar) tienen una elevada sensibilidad y especificidad para la detección de cepas con enzimas tipo VIM y KPC directamente de muestras rectales, si bien no discrimina el tipo de carbapenemasa.
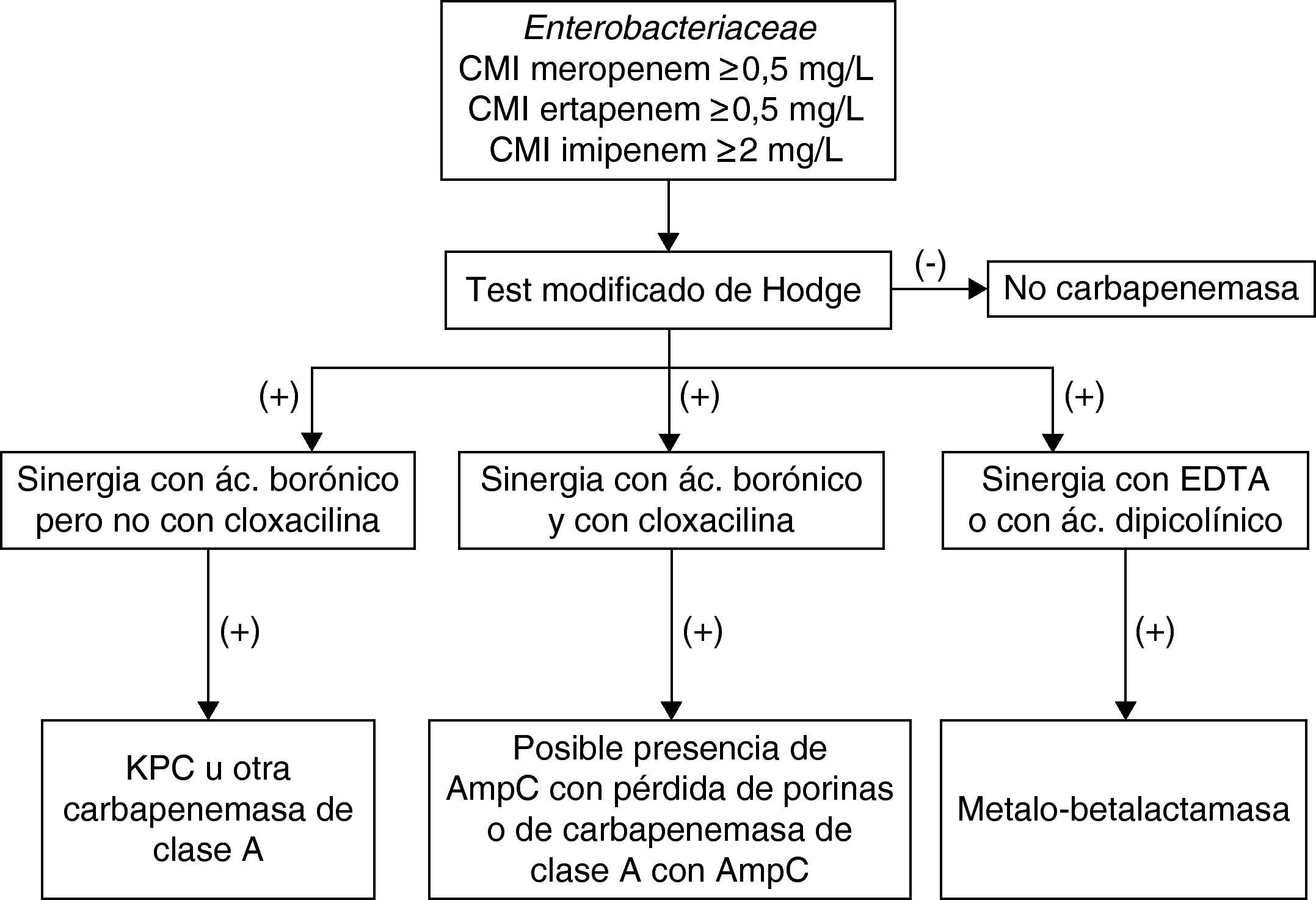
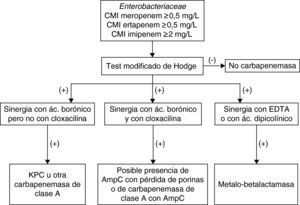
Diferenciación fenotípica de las diferentes carbapenemasasUna vez confirmado que la cepa problema produce una enzima que inactiva los carbapenémicos es preciso diferenciar el tipo de carbapenemasa. En Enterobacteriaceae, utilizando diferentes inhibidores puede seguirse el esquema que se incluye en la figura 1.
En el caso de las metalo-betalactamasa, la sensibilidad al aztreonam nos orienta hacia este tipo de enzimas que puede confirmarse por la sinergia entre los carbapenémicos y EDTA o entre ceftazidima y EDTA. No obstante, se han detactado falsos positivos con P. aeruginosa y particularmente con A. baumannii por la actividad intrínseca del EDTA, aunque puede evitarse añadiendo Zn2+ al medio de cultivo. Se han diseñado pruebas de aproximación de discos, discos de carbapenémicos combinados con EDTA y tiras de Etest con un carbapenémico y EDTA para identificar fenotípicamente estas enzimas. En algunos casos se añade además fenantrolina o se sustituye EDTA por compuestos tiólicos como el mercaptopropiónico para mejorar la sensibilidad de la prueba15. También se ha ensayado el ácido dipicolínico con buenos resultados26.
En el método de aproximación de discos es importante «acertar» con la distancia entre los discos del carbapenémico y el inhibidor, sobre todo en las cepas con baja expresión de la carbapenemasa en las que los halos de inhibición son amplios. Por este motivo, existen métodos que incluyen directamente el inhibidor en el mismo disco que el carbapenémico y se compara el halo de inhibición resultante con el que se produce con el carbapenémico solo. Este mismo principio se utiliza con las tiras de Etest que en un extremo contienen imipenem y en el otro imipenem con EDTA. Por diferencia entre los valores de CMI de imipenem sin y con inhibidor se puede inferir la presencia de las carbapenemasas. Asimismo, los sistemas expertos de determinados sistemas automáticos son también útiles para la detección de la metalo-betalactamasa, incluida la NDM-115,32.
Las KPC confieren resistencia al aztreonam y no se inhiben por el EDTA, pero sí por el ácido borónico y discretamente por el ácido clavulánico. No se recomienda utilizar ácido clavulánico por su baja sensibilidad. La utilización del ácido borónico tiene como inconveniente el ser también un buen inhibidor de AmpC, circunstancia que dificulta la detección de las KPC cuando está presente esta enzima (por ejemplo en E. cloacae). Se ha propuesto utilizar simultáneamente una prueba de discos combinados con cloxacilina para demostrar la presencia de estas betalactamasas tipo AmpC. La sinergia con ácido borónico en las cepas productoras de KPC se puede demostrar con los carbapenémicos y también con cefalosporinas de amplio espectro, preferentemente con cefepima. Se han diseñado pruebas que añaden ácido borónico directamente a los discos y comparan los halos de inhibición con los del carbapenémico sin el ácido borónico26. También se puede observar esta sinergia con pruebas de aproximación de discos. Aunque se han propuesto diferentes compuestos derivados del borónico, se prefiere el ácido fenilborónico al 3’-aminofenilborónico por su mayor capacidad inhibitoria. Asimismo, como sustrato se recomienda preferentemente utilizar meropenem o imipenem ya que con ertapenem pueden observarse resultados falsos positivos cuando el microorganismo estudiado produce AmpC, incluidas las AmpC plasmídicas15.
Para las carbapenemasas de tipo OXA no es posible utilizar un método fenotípico como el propuesto con las carbapenemasas de clase A o B ya que no existen inhibidores específicos de enzimas de clase D. Por este motivo, se recomienda confirmar la presencia de estas enzimas por métodos moleculares.
Información de los resultados y recomendaciones terapéuticasLos microorganismos productores de carbapenemasas suelen tener un perfil multirresistente que incluye los aminoglucósidos, las fluoroquinolonas y el cotrimoxazol, circunstancia que restringe sus posibilidades terapéuticas36,37. Como opciones alternativas se ha sugerido la tigecilina y la colistina, aunque debe confirmarse su sensibilidad con un antibiograma. Se ha recomendado también fosfomicina o nitrofurantoina, sobre todo en el caso de las infecciones urinarias, que debe guiarse por el antibiograma y su interpretación por los puntos de corte correspondientes.
Con respecto a los antibióticos betalactámicos existen controversias acerca de su utilización cuando los valores de CMI (o halos de inhibición) se encuentran por debajo del punto de corte de sensibilidad, circunstancia bastante habitual para los carbapenémicos y las carbapenemasas de tipo VIM y en menor medida para las KPC38. El CLSI3 y EUCAST5 recomiendan taxativamente informar los carbapanémicos en función de los puntos de corte de sensibilidad establecidos, algo menores para el CLSI, sin modificar la interpretación. No obstante, los nuevos puntos de corte de carbapenémicos, sobre todo en Enterobacteriaceae, están muy cercanos a los puntos de corte de vigilancia epidemiológica, circunstancia que facilita la detección fenotípica de las carbapenemasas (tabla 2). A pesar de ello, y debido a que la información clínica es todavía muy escasa, la variabilidad de la expresión de las enzimas, la baja reproducibilidad en las pruebas de sensibilidad (sobre todo con los métodos automáticos) y la muy dispar prevalencia de este tipo de microorganismos, en muchos laboratorios se siguen criterios interpretativos propios que trasforman la categoría sensible de los carbapenémicos a resistente cuando se identifica la presencia de una carbapenemasa (como se hacía con las cefalosporinas de amplio espectro en las cepas productoras de BLEE con anterioridad al cambio de los puntos de corte).
En el caso de las metalo-betalactamasa y dado que no hidrolizan el aztreonam podría sugerirse este antibiótico como tratamiento de elección. Un caso particular lo constituirían las carbapenemasas de tipo OXA, esencialmente OXA-48, ya que presentan una ligera pérdida de sensibilidad a los carbapenémicos (suficiente en muchos casos para que se categoricen como resistentes) pero que por su perfil hidrolítico aparecen sensibles a las cefalosporinas de amplio espectro, por lo que se deberían informar como sensibles en espera de estudios clínicos que avalen la modificación de las interpretaciones.
Resistencia a quinolonasNormalmente la resistencia clínica en enterobacterias se alcanza por una acumulación de mutaciones en los genes de las topoisomerasas, fundamentalmente gyrA y parC. Estas mutaciones se concentran en una región denominada QRDR (quinolone-resistance-determining-region), que codifican aminoácidos próximos al sitio activo de ambas enzimas39,40.
Otros mecanismos de resistencias como la hiperexpresión de bombas de expulsión activa o las alteraciones de las porinas causan un nivel de resistencia bajo. Se han descrito varios sistemas de expulsión activa, de los que AcrAB-TolC en enterobacterias, MexAB-OprM, MexCD-OprJ, MexEF-OprN y MexXY-OprM en P. aeruginosa son los más conocidos13,14,39,40.
Desde 1998 se empezaron a describir mecanismos de resistencia de origen plasmídico, como la protección de la diana por proteínas Qnr, la modificación de las quinolonas por la acetiltransferasa AAC(6’)-Ib-cr, o las bombas de expulsión activa QepA y OqxAB. Aunque la resistencia a quinolonas mediada por genes plasmídicos es de bajo nivel, se ha observado (tanto in vitro como in vivo) que facilitan la selección de mecanismos adicionales de resistencia, que contribuirán a un mayor nivel de resistencia13,39-43.
Detección fenotípica de la resistencia a quinolonasLos mecanismos cromosómicos de resistencia van apareciendo secuencialmente, y el uso de quinolonas es uno de los factores más importantes en la selección de aislados con resistencia de alto nivel a fluoroquinolonas. En cuanto a la detección de determinantes plasmídicos de resistencia, no existen marcadores fenotípicos claros para reconocerlos y su detección debe hacerse por métodos moleculares, no siempre accesibles. En los últimos años se ha observado que algunas enterobacterias presentan sensibilidad disminuida a las fluoroquinolonas siendo sensibles a ácido nalidíxico, situación que en muchos casos se ha relacionado con la presencia de genes plasmídicos de resistencia a quinolonas15,40,41.
Los resultados de sensibilidad al ácido nalidíxico y ciprofloxacino son suficientes para el estudio de mecanismos de resistencias a quinolonas en enterobacterias, Haemophilus y Neisseria spp15.
Información de los resultados y recomendaciones terapéuticas- a)
Resistencia de alto nivel a ácido nalidíxico y sensibilidad a ciprofloxacino. Presumiblemente presentan ya una mutación en gyrA, por lo que es importante informar el riesgo de seleccionar mutantes con resistencia a fluoroquinolonas tras tratamiento con las mismas en este tipo de cepas. El CLSI2 solo alerta del fracaso del tratamiento con fluoroquinolonas en caso de infecciones extraintestinales por S. enterica. Asimismo EUCAST5, recomienda en los aislados de salmonela resistentes al ácido nalidíxico informar como resistentes todas las fluoroquinolonas, pero muchos autores opinan que esta regla debe extenderse al resto de enterobacterias, o al menos interpretarse como «intermedio» sobre todo en situaciones en las que por el lugar de la infección no accedan bien las fluoroquinolonas.
- b)
Resistencia a ácido nalidíxico de alto nivel (CMI > 32mg/L) y sensibilidad intermedia o resistencia a ciprofloxacino (CMI > 1mg/L). Muy probablemente son aislados con al menos dos mutaciones en gyrA o gyrA+parC. Ello implica resistencia a todas las fluoroquinolonas, independientemente de su posible sensibilidad in vitro a alguna de ellas. La resistencia a una fluoroquinolona invariablemente conlleva una sensibilidad disminuida al resto de las fluoroquinolonas.
- c)
Sensibilidad disminuida al ácido nalidíxico (CMI 16-32mg/L) y ciprofloxacino (CMI 0,25-1mg/L). Este fenotipo sugiere con alta probabilidad la presencia de genes qnr y/o de otros genes plasmídicos, sin alteraciones adicionales en las topoisomerasas. No hay consenso al respecto, pero debido al riesgo de selección de mutantes con alto nivel de resistencia, algunos autores han propuesto informarlos como sensibilidad intermedia a quinolonas.
El principal mecanismo de resistencia a los aminoglucósidos es la inactivación enzimática, habiéndose descrito tres tipos de enzimas inactivantes: las acetiltransferasas (AAC) que acetilan un grupo amino del antibiótico, las fosfotransferasas (APH) que fosforilan un grupo hidroxilo y las nucleotidiltransferasas (ANT) que adenilan también un grupo hidroxilo. Cada enzima reconoce un cierto número de aminoglucósidos, lo cual se traduce en un fenotipo de resistencia concreto13,44,45.
Desde 2003 se han descrito distintos genes como responsables de la metilación postranscripcional del RNA ribosómico (armA, rmt o npmA) y de momento con una prevalencia moderada y geográficamente dependiente. En este caso la resistencia observada suele ser de alto nivel a todos los aminoglucósidos excepto neomicina13. Además, una bacteria puede disminuir su sensibilidad a los aminoglucósidos mediante mutaciones que afectan la difusión pasiva a través de la membrana externa, porinas o estructura del polisacárido.
Para la detección de los distintos fenotipos de resistencia adquiridos es importante una correcta elección de los aminoglucósidos en estudio. Puede hacerse un antibiograma completo, por ejemplo para el estudio epidemiológico de los genes de resistencia de las cepas, o bien un antibiograma reducido donde solo se incluya los aminoglucósidos de uso en terapéutica. Para el antibiograma completo se recomienda el estudio de la amikacina, estreptomicina, gentamicina, kanamicina, neomicina, netilmicina y tobramicina. El estudio de la estreptomicina puede ser optativo, dado su reducido uso en clínica, sin embargo, en un estudio epidemiológico es el único marcador de la presencia de las enzimas APH (3″) y ANT (3″)-Ia. En cambio para el antibiograma reducido es suficiente el estudio de la amikacina, gentamicina y tobramicina13,15.
Un determinado patrón de resistencia puede sugerir la presencia de una determinada enzima inactivante de aminoglucósidos. De todos modos, estos patrones se pueden desdibujar ante la presencia de otras enzimas y/o otros mecanismos tal como se ha comentado13. Además, para interpretar el patrón de sensibilidad a los aminoglucósidos se debe estar alerta ante situaciones donde puede haber una débil expresión de la enzima. En este contexto debe tenerse en cuenta que:
- a)
Ante una cepa sensible a amikacina, pero con sensibilidad intermedia o resistente a tobramicina o netilmicina y sensible a gentamicina debería interpretarse sensibilidad intermedia a amikacina, ya que puede tratarse de la producción de la enzima AAC (6’).
- b)
Cuando se observa una disminución del halo de inhibición solo de la gentamicina (comprendido entre 16 y 19mm), debe considerarse sensibilidad intermedia a gentamicina por producción de la enzima AAC (3)-I.
- c)
Si la gentamicina es resistente o presenta un halo de inhibición reducido y en la tobramicina también se observa reducción del halo de inhibición (16-19mm), debe interpretarse como sensibilidad intermedia a la tobramicina pues puede estar presente la enzima ANT (2”).
- d)
Debe interpretarse como sensibilidad intermedia a la netilmicina cuando haya una reducción del diámetro de inhibición (comprendido entre 19 y 22mm), si también aparecen reducidos los halos de la gentamicina y la tobramicina, pues puede estar presente la enzima AAC (3)-II o AAC (3)-IV.
Clásicamente los betalactámicos son el tratamiento de primera línea de las infecciones causadas por este microorganismo46. Dos mecanismos principales son la causa de la resistencia a las penicilinas: hidrólisis enzimática del antibiótico por betalactamasas plasmídicas, y alteraciones en la proteína PBP3 por mutaciones en el gen ftsI46,47. Las betalactamasas son el mecanismo más común, son mayoritariamente de tipo TEM-1, y más raramente TEM-2 o ROB-146,48. Estas enzimas confieren un perfil de sensibilidad similar con resistencia de alto nivel a amino-, carboxi- y ureido-penicilinas, y son eficazmente inhibidas por inhibidores de betalactamasas como el ácido clavulánico (fenotipo BLPAR). Sin embargo, puede haber variaciones en los niveles de resistencia en función del promotor asociado al gen TEM-1, estos promotores pueden tener diferentes afinidades por la RNA polimerasa, dando lugar a una mayor o menor producción de enzima que podría incluso afectar a amoxicilina-ácido clavulánico y cefalosporinas de segunda generación46.
Mientras que la resistencia debida a la producción de betalactamasa está asociada a CMI altas de ampicilina (≥ 4mg/L), la resistencia por alteraciones en la PBP3 se asocia mayoritariamente a cepas con bajas CMI de ampicilina (≤ 2mg/L)46,48. El fenotipo del mecanismo de resistencia debido a sustituciones de aminoácido en la PBP3 se conoce como «betalactamasa negativa ampicilina resistente (BLNAR)», y se caracteriza por bajos niveles de resistencia a los betalactámicos y a sus combinaciones con inhibidores de betalactamasas.
Por último, cada vez son más frecuentes las cepas BLPACR (betalactamasa positiva amoxicilina-ácido clavulánico resistente) que producen betalactamasa y poseen alteraciones en la PBP3 y se caracterizan por altos niveles de resistencia a ampicilina junto a disminución de la sensibilidad a amoxicilina-ácido clavulánico. No se detectan diferencias en los valores de CMI para las cefalosporinas de tercera generación entre las cepas BLNAR y las cepas BLPACR.
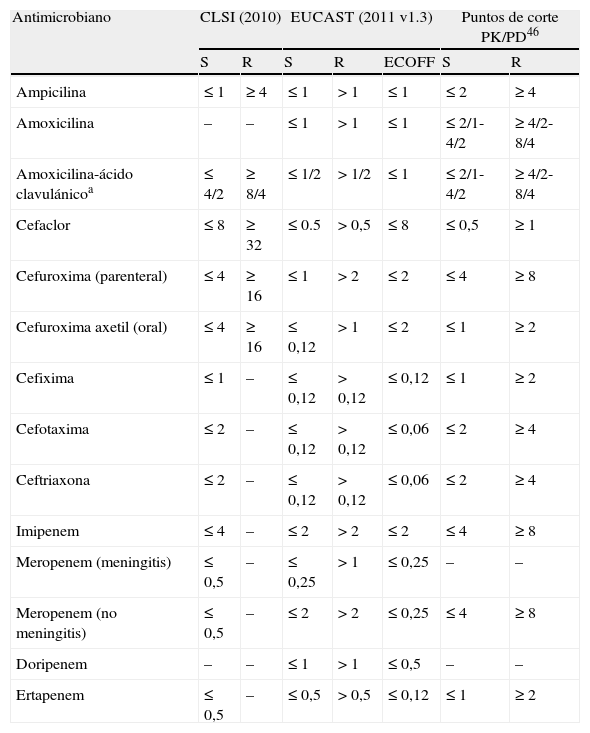
Detección fenotípica de la resistenciaPor desgracia, no hay consenso en los puntos de corte de sensibilidad entre los diferentes comités de estandarización. Como se observa en la tabla 5, hay grandes diferencias entre los puntos de corte de CLSI2 y EUCAST5 para amoxicilina-ácido clavulánico y las cefalosporinas. EUCAST5, además de los cortes clínicos de sensibilidad, aporta puntos de corte epidemiológicos que podrían ser más eficaces a la hora de detectar cepas con bajos niveles de resistencia. El punto de corte para ampicilina propuesto tanto por CLSI2 como EUCAST5 (≤ 1mg/L) no es útil ya que clasificaría como sensibles a numerosas cepas BLNAR que presentan valores bajos de CMI para ampicilina (low-BLNAR), que son mayoritarias en España46,48.
Puntos de corte (mg/L) para Haemophilus influenzae según CLSI y EUCAST
| Antimicrobiano | CLSI (2010) | EUCAST (2011 v1.3) | Puntos de corte PK/PD46 | ||||
| S | R | S | R | ECOFF | S | R | |
| Ampicilina | ≤ 1 | ≥ 4 | ≤ 1 | > 1 | ≤ 1 | ≤ 2 | ≥ 4 |
| Amoxicilina | – | – | ≤ 1 | > 1 | ≤ 1 | ≤ 2/1-4/2 | ≥ 4/2-8/4 |
| Amoxicilina-ácido clavulánicoa | ≤ 4/2 | ≥ 8/4 | ≤ 1/2 | > 1/2 | ≤ 1 | ≤ 2/1-4/2 | ≥ 4/2-8/4 |
| Cefaclor | ≤ 8 | ≥ 32 | ≤ 0.5 | > 0,5 | ≤ 8 | ≤ 0,5 | ≥ 1 |
| Cefuroxima (parenteral) | ≤ 4 | ≥ 16 | ≤ 1 | > 2 | ≤ 2 | ≤ 4 | ≥ 8 |
| Cefuroxima axetil (oral) | ≤ 4 | ≥ 16 | ≤ 0,12 | > 1 | ≤ 2 | ≤ 1 | ≥ 2 |
| Cefixima | ≤ 1 | – | ≤ 0,12 | > 0,12 | ≤ 0,12 | ≤ 1 | ≥ 2 |
| Cefotaxima | ≤ 2 | – | ≤ 0,12 | > 0,12 | ≤ 0,06 | ≤ 2 | ≥ 4 |
| Ceftriaxona | ≤ 2 | – | ≤ 0,12 | > 0,12 | ≤ 0,06 | ≤ 2 | ≥ 4 |
| Imipenem | ≤ 4 | – | ≤ 2 | > 2 | ≤ 2 | ≤ 4 | ≥ 8 |
| Meropenem (meningitis) | ≤ 0,5 | – | ≤ 0,25 | > 1 | ≤ 0,25 | – | – |
| Meropenem (no meningitis) | ≤ 0,5 | – | ≤ 2 | > 2 | ≤ 0,25 | ≤ 4 | ≥ 8 |
| Doripenem | – | – | ≤ 1 | > 1 | ≤ 0,5 | – | – |
| Ertapenem | ≤ 0,5 | – | ≤ 0,5 | > 0,5 | ≤ 0,12 | ≤ 1 | ≥ 2 |
aPara EUCAST el ácido clavulánico se estudia con una concentración fija de 2mg/L y para CLSI se estudia a la mitad de la concentración de amoxicilina.
ECOFF: punto de corte epidemiológico.
Numerosos autores han propuesto diferentes puntos de corte para detectar las cepas BLNAR, aunque la mayoría se inclina por valores de CMI ≥1mg/L para ampicilina, mientras que valores ≤ 0,25mg/L descartarían cualquier mecanismo de resistencia48. Valores de CMI ≥ 0,5mg/L o diámetros de inhibición ≤ 29mm para cefixima son muy indicativos de cepas BLNAR con mutaciones en el motivo SSN que conllevan una menor sensibilidad a las cefalosporinas de tercera generación, que podría tener un impacto en el tratamiento de infecciones graves como meningitis.
Además del antibiograma convencional, se debe realizar siempre la prueba cromogénica de la nitrocefina para caracterizar las cepas productoras de betalactamasa (BLPAR) y se caracterizan por valores altos de CMI para ampicilina (> 4mg/L)15. Por último, valores de CMI ≥ 2mg/L para amoxicilina-ácido clavulánico en cepas productoras de betalactamasa nos indica con alta probabilidad un fenotipo BLPACR. Si utilizamos el método de Etest, valores de 0,5-1mg/L también pueden detectarse en las cepas BLPACR. En cambio con valores < 0,5mg/L para amoxicilina-ácido clavulánico se puede descartar la presencia de mutaciones en el gen ftsI por ambas metodologías (microdilución y Etest)15.
El método de difusión con discos se ha demostrado que tiene menos capacidad de discriminación entre cepas BLNAR y cepas sin modificaciones en la PBP3. Si se siguen los puntos de corte establecidos por el CLSI2 para ampicilina (disco 10μg), la mayoría de las cepas BLNAR identificadas en nuestro país se identificarían como sensibles. Se han propuesto diferentes alternativas con mejor sensibilidad para detectar BLNAR, EUCAST5 propone discos de ampicilina 2μg (< 16mm), cefaclor 30μg (< 19mm) o fenoxymetilpenicilina 10μg (< 15mm). Otras propuestas publicadas incluyen la utilización de discos de amoxicilina-ácido clavulánico de baja carga (2/1μg), cefalotina, cefalexina, cefsulodina, etc. Desafortunadamente, aún no hay resultados concluyentes sobre un método óptimo que pueda ser utilizado por la generalidad de los laboratorios clínicos15.
Información de los resultados y recomendaciones terapéuticasLas cepas productoras de betalactamasa deben informarse como resistentes a todas las penicilinas.
Las cepas BLNAR además de ser resistentes a ampicilina, tienen sensibilidad disminuida al resto de los betalactámicos, particularmente las cefalosporinas.
Según el CLSI2 y EUCAST5, las cepas BLNAR deben informarse como resistentes a amoxicilina-ácido clavulánico, ampicilina-sulbactam, cefaclor, cefamandol, cefetamet, cefonicid, cefprozil, cefuroxima, loracarbef y piperacilina-tazobactam, con independencia del resultado de su sensibilidad in vitro.
Comentario aparte merece la utilidad clínica de las cefalosporinas orales de segunda generación como cefaclor y cefuroxima axetil independientemente si son cepas BLNAR o sin mecanismos de resistencia. Hay una disparidad de criterios entre CLSI2 y EUCAST5 al respecto (tabla 5), aunque el CLSI2 comenta que los puntos de corte que aporta para las cefalosporinas orales pueden no ser útiles para el tratamiento de los pacientes. EUCAST5 propone puntos de corte clínicos para cefaclor y cefuroxima oral muy por debajo al punto de corte epidemiológico (tabla 5), que en la práctica eso se traduce en informar casi todos los aislados como resistentes a cefaclor e intermedios a cefuroxima axetil. Ello equivale a diámetros del halo de inhibición ≥ 50mm como punto de corte de sensibilidad, aunque proponen diámetros < 19mm con un disco de cefaclor de 30μg como screening para detectar cepas BLNAR.
Las mutaciones en el gen fitsI (BLNAR) provoca un aumento importante en las CMI de las cefalosporinas de tercera generación, que es variable dependiendo de las mutaciones detectadas y otros factores aún no conocidos. Desgraciadamente, no hay consenso en los puntos de corte de sensibilidad y no se conoce suficientemente el impacto clínico de estas mutaciones. En nuestro entorno, aún no son frecuentes las cepas BLNAR, y mucho menos las infecciones graves por estos aislados, por lo que no se han informado fracasos terapéuticos con las cefalosporinas de tercera generación.
Resistencia a betalactámicos en NeisseriaNeisseria gonorrhoeaeLa penicilina ha sido la base del tratamiento durante varias décadas, pero poco después de su introducción, N. gonorrhoeae comenzó a desarrollar resistencia de bajo nivel a penicilina49. Esta disminución gradual de la sensibilidad es el resultado del efecto aditivo de múltiples mutaciones cromosómicas que provocan alteración en las PBPs, hiperexpresión de las bombas de expulsión y disminución de la entrada de los antibióticos a través de la membrana externa. Al menos 5 genes cromosómicos están implicados en esta resistencia: ponA, penA, penB, pilQ y mtr. Los genes ponA y penA codifican las PBP1 y PBP2 respectivamente, cuyas alteraciones afectan únicamente a la actividad de las penicilinas. Mutaciones en penB llevan a alteraciones de una porina que también causa resistencia a otras familias de antibióticos como las tetraciclinas y a una pequeña disminución de la sensibilidad a quinolonas. La hiperexpresión de la bomba de expulsión MtrCDE favorece la resistencia a penicilinas, macrólidos, rifampicina, tetraciclinas y quinolonas, y puede estar también implicada en la disminución de sensibilidad a cefalosporinas. Mutaciones en pilQ (antiguamente denominado penC, que codifica una porina) provoca la resistencia de alto nivel a penicilina en cepas con mutaciones de penA, penB y mtr. Así, las diferentes CMI que se observan son producto de la acción sinérgica de estos diferentes mecanismos, pudiendo provocar resistencia de alto nivel a penicilina.
Además de los mecanismos cromosómicos descritos, la penicilina puede afectarse también por una betalactamasa plasmídica de tipo TEM-1, y puede estar presente en diferentes tipos de plásmidos con una frecuencia geográfica variable. Esa resistencia se considera de alto nivel aunque su expresión in vitro es variable (1 a 64mg/L), y se inhibe por ácido clavulánico, aunque generalmente no es de utilidad terapéutica. En España, alrededor del 90% de las cepas tienen sensibilidad disminuida o resistencia a penicilina, y se detecta la presencia de penicilinasa plasmídica en alrededor de un 20%.
El método de dilución en agar se considera el método de referencia para la determinación de la CMI, sin embrago el método de difusión con tiras de gradiente de antibiótico como el Etest es una alternativa aceptable para la determinación de CMI en la rutina del laboratorio. Hay coincidencia en los puntos de corte de sensibilidad para penicilina del CLSI y EUCAST (S ≤ 0,064mg/L; R >1mg/L).
El método de difusión con discos en cambio presenta resultados más aleatorios sobre todo a la hora de diferenciar los aislados sensibles de aquéllos con sensibilidad disminuida a la mayoría de los antibióticos pero particularmente los betalactámicos.
El antibiograma debe acompañarse además de la prueba cromogénica de nitrocefina para detectar la posible producción de betalactamasa, lo cual implicará resistencia a amino-, carboxi- y ureidopenicilinas, con independencia de su resultado de sensibilidad in vitro, mientras que las cefalosporinas no se ven afectadas por este mecanismo15.
Neisseria meningitidisLa penicilina ha sido históricamente el tratamiento de elección, y desde los años 80 se empezaron a describir aislados con sensibilidad disminuida a penicilina (CMI de 0,12-0,25mg/L) en todo el mundo, siendo España uno de los países con mayores tasas, superiores al 50%. El mecanismo de resistencia de este fenotipo denominado PenI se debe sobre todo a alteraciones en la PBP2 (codificado por el gen penA). El fenotipo afecta con mayor o menor intensidad a las penicilinas, cefalosporinas de primera y segunda generación, imipenem y aztreonam, y se mantiene la actividad de las cefalosporinas de tercera generación50,51.
El método de dilución en agar o la microdilución en caldo se consideran los métodos de elección, aunque también es aceptable el método de Etest o similar, mientras que el método de difusión con discos no es fiable para el estudio de sensibilidad a betalactámicos15,50,51. Se observa una alta tasa de errores menores (sensible o resistente por el método de referencia o la difusión con discos pero intermedio por el otro método) cuando se usan discos de penicilina y ampicilina, incluso con discos de baja carga como penicilina 1 U o ampicilina 2μg. CLSI2 y EUCAST5 proponen los mismos puntos de corte de sensibilidad para penicilina, ampicilina, cefotaxima, ceftriaxona y meropenem.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.