Actualmente, las enfermedades infecciosas siguen causando una elevada mortalidad y morbilidad. Los métodos de diagnóstico microbiológico se basan en el cultivo seguido de la identificación fenotípica del microorganismo una vez aislado, y el tiempo necesario para su obtención puede variar de 24 a 48h. Dado que la identificación microbiológica repercute directamente en el manejo del paciente y su pronóstico, son necesarias nuevas herramientas diagnósticas capaces de detectar e identificar cualquier microorganismo de manera rápida y fiable. A lo largo de los últimos años se han desarrollado diferentes técnicas moleculares basadas en la amplificación genética con el objetivo de reducir el tiempo necesario para la identificación de los microorganismos implicados en diferentes tipos de procesos infecciosos. Por otro lado, la espectrometría de masas ha surgido como una alternativa rápida y eficaz a los métodos convencionales para la identificación de microorganismos. En esta revisión se describe la tecnología en sus dos formas más utilizadas —desorción/ionización por láser asistida por matriz (MALDI-TOF) e ionización por electrospray (ESI-TOF)— para el análisis tanto de las proteínas como de los ácidos nucleicos microbianos, así como las diferentes plataformas comerciales disponibles. Así mismo, se hace una revisión de los trabajos de mayor interés en microbiología clínica.
Infectious diseases are still a cause of high mortality and morbidity rates. Current microbiological diagnostic methods are based on culture and phenotypic identification of isolated microorganisms, which can be obtained in about 24-48h. Given that the microbiological identification is of major importance for patient management, new diagnostic methods are needed in order to detect and identify microorganisms in a timely and accurate manner. Over the last few years, several molecular techniques based on the amplification of microbial nucleic acids have been developed with the aim of reducing the time needed for the identification of the microorganisms involved in different infectious processes. On the other hand, mass spectrometry has emerged as a rapid and consistent alternative to conventional methods for microorganism identification. This review describes the most widely used mass spectrometry technologies —matrix-assisted laser desorption/ionization time-of-flight (MALDI-TOF) and electrospray ionization time-of-flight (ESI-TOF)—, both for protein and nucleic acid analysis, as well as the commercial platforms available. Related publications of most interest in clinical microbiology are also reviewed.
A pesar de los avances en el campo de la medicina, las enfermedades infecciosas siguen siendo la causa de una elevada mortalidad y morbilidad. Según el informe actualizado en 2011 de la Organización Mundial de la Salud, 4 de las 10 principales causas de muerte a nivel mundial están causadas por agentes infecciosos, siendo estas las infecciones de las vías respiratorias bajas (6,1%), la diarrea (4,3%), la infección por el VIH (3,1%) y la tuberculosis (2,4%). En países subdesarrollados, a estas se les suman la malaria (5,2%) y las infecciones neonatales (2,6%)1.
Para un óptimo manejo de los pacientes es indispensable obtener la identificación microbiológica del agente causal de la infección en el menor espacio de tiempo posible. Solo así será posible aplicar un tratamiento antibiótico efectivo o reducir el espectro del tratamiento administrado empíricamente, disminuyendo la aparición de resistencias2.
Sin embargo, la obtención de un diagnóstico microbiológico rápido resulta compleja utilizando los métodos convencionales considerados como gold standard, que se basan en el cultivo seguido de la identificación fenotípica del microorganismo una vez aislado3. Aunque la obtención de un cultivo puro del microorganismo implicado en una infección permite no solo la identificación del mismo, sino también el estudio de su sensibilidad a diferentes antibióticos y la determinación de marcadores epidemiológicos, el tiempo necesario para su obtención puede variar entre 24 y 48h, según los requerimientos del microorganismo. Así mismo, serán necesarias 24h adicionales para disponer de los resultados definitivos de la identificación y el antibiograma, lo que supone como mínimo un total de 48 a 72h desde la obtención de la muestra del paciente.
Este periodo de tiempo puede reducirse utilizando instrumentos que ofrecen una identificación automatizada basada en el perfil bioquímico de los microorganismos. Uno de los sistemas más utilizados de identificación automatizada es el Vitek 2 Compact (bioMérieux, Marcy l’Etoile, Francia), que permite tanto la identificación de bacterias y levaduras como la obtención de su patrón de susceptibilidad a diversos antibióticos y antifúngicos en unas 6-12h partiendo del cultivo puro4. Otros sistemas también utilizados para esta finalidad son MicroScan (MicroScan Systems, INC., Renton, EE. UU.) o Phoenix (Becton Dickinson, Franklin Lakes, EE. UU.), entre otros3.
A diferencia de otros laboratorios, como los de bioquímica o hematología, que se han beneficiado de la tecnología para simplificar el procesamiento de las muestras y poder obtener de esta manera resultados en un breve espacio de tiempo, la automatización del laboratorio de microbiología es más compleja, dada la gran diversidad de muestras clínicas a analizar y las características inherentes a los diferentes microorganismos.
Los requisitos ideales en una plataforma o equipo de detección e identificación microbiológica serían los siguientes: capacidad de detección de cualquier microorganismo, detección a partir de la muestra clínica directa, obtención de resultados en un periodo corto de tiempo (1-6h), detección de resistencias, factores de virulencia o toxinas, y una buena relación coste-efectividad.
Recientemente, la espectrometría de masas (EM) ha irrumpido en el laboratorio de microbiología ofreciendo una alternativa rápida y fiable para la identificación de microorganismos. A pesar de que esta tecnología se ha aplicado en diferentes campos durante los últimos años, la reciente creación de plataformas simples y fáciles de utilizar dirigidas al diagnóstico microbiológico ha conllevado su creciente incorporación a los laboratorios de microbiología. Las plataformas comercializadas hasta el momento utilizan la EM para la identificación de microorganismos mediante dos aproximaciones diferentes: la identificación basada en el perfil proteico específico de cada microorganismo (aproximación proteómica) o el análisis de sus ácidos nucleicos (aproximación genómica).
Esta revisión pretende explicar esta tecnología y hacer un resumen de sus aplicaciones actuales o futuras, centrándose en las plataformas comerciales existentes.
Espectrometría de masasBreve historiaLa EM fue concebida hace más de cien años. Sin embargo, no fue hasta la invención de las llamadas técnicas de ionización suave MALDI (por las siglas en inglés de Matrix-assisted laser desorption/ionization [desorción/ionización por láser asistida por matriz]) y ESI (por las siglas en inglés de Electrospray ionization [ionización por electrospray])5,6 que esta tecnología empezó a utilizarse en los laboratorios para aplicaciones rutinarias. Estas técnicas de ionización suave permitieron el análisis de proteínas de elevado peso molecular y a la vez facilitaron el análisis de mezclas, ya que mediante estas técnicas los componentes de una mezcla compleja se ionizan sin sufrir apenas fragmentaciones, lo cual simplifica los espectros de masas7. En los últimos 20 años, estas nuevas técnicas de EM se convirtieron en indispensables para el avance de la proteómica, sobre todo al ser combinadas con el analizador de masas tipo TOF (por sus siglas en inglés de Time-of-flight [tiempo de vuelo]). Aunque la separación de partículas cargadas basada en la masa y el tiempo de vuelo se conoce desde el 1897, la utilización del tiempo de vuelo como medio para obtener un espectro de masas tardó en aplicarse en la EM, y fue en 1946 cuando apareció la primera referencia de su empleo8.
El MALDI-TOF se utilizaba inicialmente para el análisis cualitativo de biopolímeros, como una herramienta para valorar la pureza de diferentes productos9. Debido a las características técnicas de los primeros espectrómetros de tipo MALDI, su utilización para el análisis de analitos de bajo peso molecular resultaba inapropiada debido a que el exceso de matriz enmascaraba la señal de estos. Además, resultaba difícil obtener una aproximación cuantitativa, debido a que la cristalización de la matriz no era uniforme, por lo que se obtenían señales de diferente intensidad según la zona. Actualmente se han subsanado estos inconvenientes, y se trata de una técnica ampliamente utilizada para el análisis y la cuantificación de proteínas, péptidos, toxinas, ácidos nucleicos y otros compuestos.
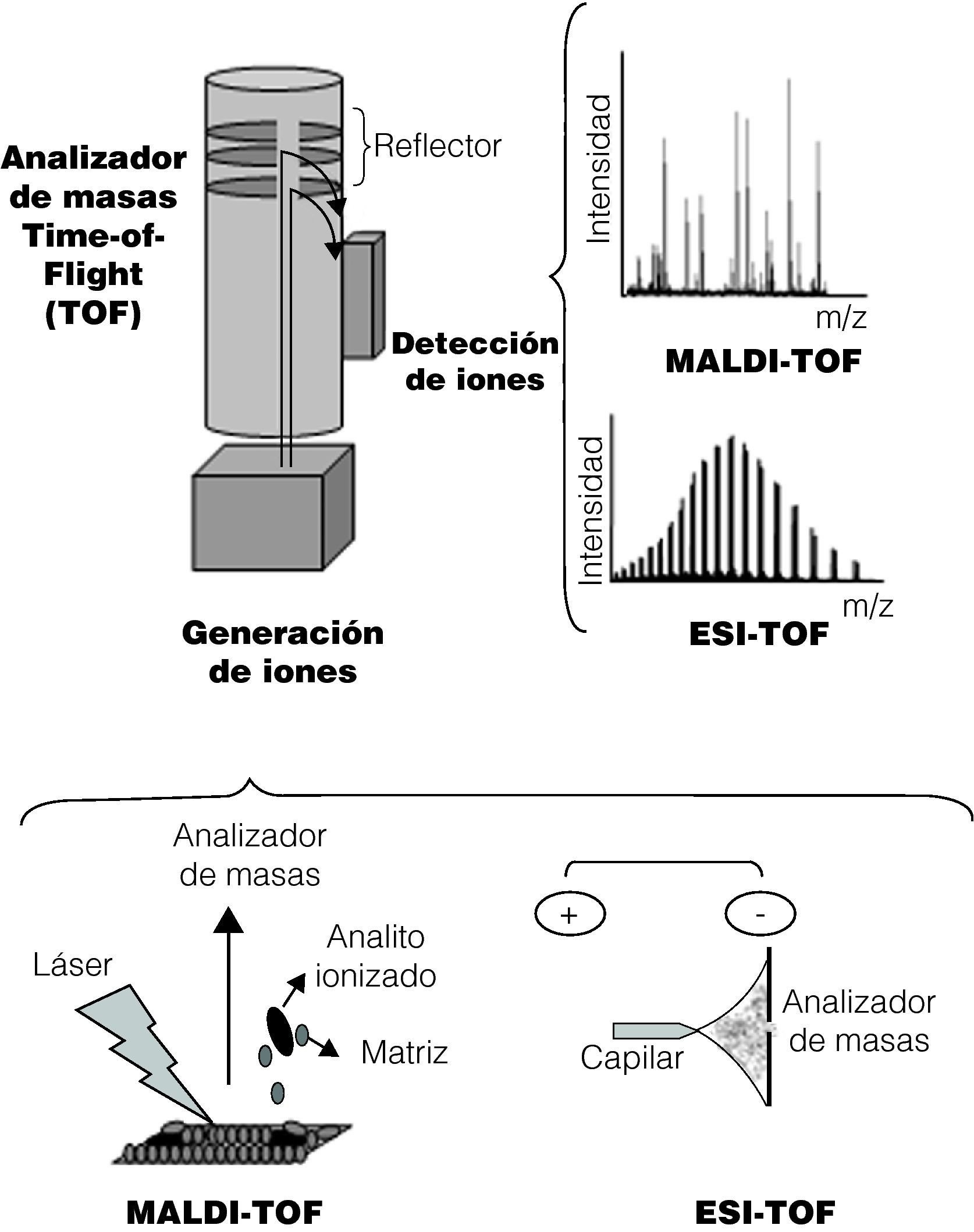
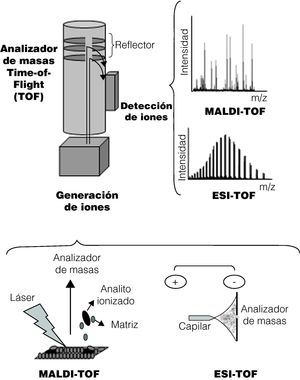
Fundamentos de la tecnologíaEl espectrómetro de masas produce, separa y detecta iones en fase gaseosa10. Los elementos principales que forman un espectrómetro de masas son tres: una fuente de ionización, un analizador de masas y un detector. Los tres elementos se mantienen en una atmósfera de vacío. En la figura 1 se esquematizan los componentes de un espectrómetro de masas.
El resultado de aplicar una fuente de ionización sobre una molécula es la formación de iones, generados por exceso o pérdida de electrones, por lo que dichas moléculas estarán cargadas eléctricamente8. En el caso del MALDI, la muestra es embebida en una matriz orgánica, la cual cristaliza en contacto con el aire. Esta mezcla se deposita en una tarjeta de un material conductivo y es irradiada por un láser. La energía del láser causa una desestructuración de la matriz cristalizada generando una nube de partículas. Los iones de dicha nube se extraen al ser sometidos a un campo eléctrico, a través del cual estos son acelerados debido a su carga. Los iones obtenidos son dirigidos hacia el analizador de masas y, posteriormente, al detector. El papel de la matriz es fundamental para los procesos electroquímicos que se producen. Generalmente se trata de sustancias orgánicas que absorben la energía del láser. Algunas de las matrices más utilizadas son el ácido α-ciano-4-hidroxi-trans cinámico, el ácido 2,5-dihidrobenzoico, o el ácido sinapinico.
En el caso del ESI, la muestra se encuentra disuelta en un solvente orgánico y es inyectada a través de un capilar. La diferencia de potencial se genera porque existe un electrodo en contacto con el capilar, mientras que el otro electrodo se sitúa en el detector del espectrómetro. Cuando la muestra sale del capilar se genera una nube de pequeñas gotas cargadas que da lugar a los iones en fase gaseosa. Con esta tecnología, aplicando una diferencia de potencial positiva o negativa se pueden generar cationes o aniones10.
Analizador de masasSe trata del componente principal del espectrómetro. A pesar de existir diferentes analizadores, el más utilizado en las aplicaciones de la EM dentro del campo de la microbiología es el analizador de tipo TOF. La estructura delimita una zona de vuelo a través de la cual los iones son acelerados adquiriendo una elevada energía cinética, y durante este trayecto se separarán según su ratio masa/carga (m/z). La mayor parte de iones generados poseen una sola carga (z=1), por lo que la ratio m/z equivale a m. El periodo de tiempo que tarda cada ión en llegar hasta el detector es denominado tiempo de vuelo y depende de dicha ratio8,11.
DetectorAl final de la zona de vuelo los iones impactan en el detector. En muchos casos, antes del detector encontramos un cuarto elemento, el reflector, cuyo objetivo es compensar las velocidades cinéticas antes de que los iones impacten contra el detector. Con ello se obtiene un aumento en la resolución12,13. A partir de la información recogida por el detector, se genera el espectro de masas en el cual se muestra la masa de los diferentes iones de la muestra calculada a partir del tiempo de vuelo. En el eje de las ordenadas del espectro de masas se representan los valores de la ratio m/z, mientras que en el eje de las abscisas se representa la intensidad, es decir, el número de iones de una determinada ratio m/z que ha impactado contra el detector.
En el caso del MALDI-TOF, como se ha comentado con anterioridad, la ratio m/z suele ser equivalente a la masa molecular (m) del analito, por lo que en el espectro típico de MALDI-TOF se observa un solo pico predominante, correspondiente a dicha masa. En el caso del ESI-TOF, en el espectro generado se ven representados todos los iones generados a partir del analito en sus diferentes intensidades, de manera que se observan múltiples picos. En este caso, la masa molecular del analito se obtiene calculando la media de todas las masas moleculares relativas de cada ión obtenido.
Primeras aplicaciones de la espectrometría de masas a la microbiologíaYa en 1975 se publicó un trabajo en que se utilizó una combinación de pirólisis con EM para la identificación directa de microorganismos liofilizados14. Mediante los espectros obtenidos pudieron diferenciarse inequívocamente 7 bacterias diferentes (2 grampositivas y 5 gramnegativas). La desventaja de la pirólisis es que el tamaño y el rango de las masas obtenidos por esta técnica son pequeños, por lo cual ofrece poca información estructural. Posteriormente se utilizó otra metodología, el bombardeo con átomos rápidos o FAB-MS (por las siglas en inglés Fast atom bombardement ionization mass spectrometry), la cual, a pesar de ofrecer mayor información estructural, solo permite analizar analitos de extractos celulares, como, por ejemplo, lípidos de membrana15. Posteriormente, Cain et al.16 diferenciaron diversas bacterias mediante EM tipo MALDI-TOF con una extracción previa de proteínas hidrosolubles. En 1996 se publicaron los primeros estudios en los que se analizaban células bacterianas intactas sin ninguna extracción previa, sino depositando las células directamente en la tarjeta de un instrumento MALDI-TOF y recubriéndola con una matriz orgánica; Holland et al.17 generaron los espectros de referencia para 5 bacterias gramnegativas para proceder posteriormente a su identificación a ciegas, pudiendo identificarlas correctamente. En el trabajo de Claydon et al.18 fueron 10 las bacterias (6 bacterias gramnegativas y 4 bacterias grampositivas) caracterizadas y posteriormente identificadas mediante la tecnología MALDI-TOF.
A partir de estos estudios iniciales, muchos otros utilizaron la EM para resolver problemas microbiológicos (identificación, genotipado, estudio de resistencias, etc.)19-22. Poco a poco las bases de datos se fueron ampliando con espectros de los microorganismos más relevantes obtenidos mediante MALDI-TOF.
La espectrometría de masas en el laboratorio de microbiología en la actualidadRecientemente han aparecido diversas plataformas que han permitido a los laboratorios de microbiología clínica acceder más fácilmente a esta tecnología, ligada en sus inicios a la investigación básica. Desde la comercialización de estas plataformas, el número de publicaciones en las que se describe la utilización de la EM para la identificación de microorganismos ha aumentado de manera exponencial.
Como se detalla a continuación, la EM tiene dos principales aplicaciones: la caracterización de proteínas y de ácidos nucleicos.
Espectrometría de masas dirigida a la detección de proteínasActualmente, las dos plataformas comerciales más utilizadas para esta finalidad son MALDI Biotyper (Bruker Daltonics, Leipzig, Alemania) y Axima@SARAMIS (AnagnosTec GmbH, Potsdam, Alemania). Bruker Daltonics ofrece tanto el espectrómetro de masas o hardware, como el software y la base de datos para la interpretación de los resultados (Biotyper 2.0 y Biotyper 3.0 en su nueva versión recientemente disponible). En el caso de Axima@SARAMIS, el espectrómetro de masas y el software de interpretación son fabricados por la compañía Shimadzu (Kioto, Japón), mientras que la base de datos SARAMIS ha sido desarrollada por AnagnosTec GmbH3,23.
Técnicamente hablando, ambos equipos son muy parecidos. Ambos utilizan la tecnología MALDI-TOF y tienen un rango de detección de proteínas que oscila entre 2.000 y 200.000Da. La identificación se consigue por la comparación del espectro o perfil proteico obtenido con la base de datos que ofrece cada casa comercial, en la que se encuentran representadas una gran variedad de bacterias, levaduras y hongos filamentosos. En el caso de la base de datos Biotyper 2.0 se encuentran representados hasta 1.960 microorganismos. Según la información ofrecida por Bruker Daltonics, el sistema MALDI Biotyper permite, además, la identificación a partir de esporas. En el caso de la base de datos SARAMIS, son 1.160 las especies representadas. Ambas bases de datos permiten la incorporación de nuevos espectros, permitiendo personalizarla según las necesidades de cada laboratorio.
Una vez obtenido el espectro de masas y comparado con los existentes en la base de datos, el software le adjudica una identificación y un valor indicativo de la fiabilidad de dicha identificación. En el caso de Biotyper 2.0, este valor se encuentra en un rango que va de 0 a 3, y se recomienda dividir el rango en tres intervalos: un valor ≥2 indica una identificación fiable a nivel de especie, un valor entre 1,7 y 2 indica un parentesco muy cercano y ofrece una identificación fiable a nivel de género, y un valor <1,7 ofrece una identificación poco fiable. En el caso de SARAMIS, el valor adjudicado indicativo de la fiabilidad de la detección tiene un solo punto de corte: un valor superior o igual al 70% indica una identificación con elevada fiabilidad a nivel de especie, mientras que en el caso de obtener valores inferiores se recomienda una segunda evaluación de la muestra24.
En cuanto a la matriz utilizada, el espectrómetro de masas de Bruker Daltonics utiliza el ácido α-ciano-4-hidroxi-trans cinámico, mientras que en el Axima@SARAMIS se utiliza el ácido 2,5-dihidrobenzoico. En ambos casos se recomienda utilizar 1μl de matriz para recubrir la muestra.
La tarjeta donde se colocan las muestras en el sistema de Bruker Daltonics consta de 96 pocillos, y éste tarda 1h en analizar 96 muestras. En el caso de Shimazdu, la tarjeta consta de 48 pocillos y es capaz de realizar 380 identificaciones en 5h. Ambos sistemas ofrecen la posibilidad de transferir los datos al sistema informático del laboratorio3.
En cuanto a la muestra de partida, se han descrito dos aproximaciones diferentes: análisis de células intactas o transferencia directa, y extracción previa de proteínas25. Los protocolos se describen brevemente a continuación.
- •
Análisis de células intactas. Se selecciona la colonia de interés y se aplica directamente sobre la tarjeta del instrumento mediante un asa de siembra o una punta de pipeta. Se necesitan aproximadamente 106-107 células. A modo de guía, la visualización de la masa microbiana sobre la tarjeta indicaría que la cantidad es suficiente para lograr una identificación. A continuación se cubre la muestra con la solución matriz y se deja secar al aire. Posteriormente se introduce la tarjeta en el instrumento para su análisis. Esta aproximación también puede utilizarse a partir de un cultivo líquido, previa centrifugación para concentrar los microorganismos. El pellet obtenido puede ser aplicado directamente a la tarjeta del MALDI.
- •
Extracción de proteínas. Tal y como se comenta más adelante, se ha descrito que una extracción previa de proteínas mejora la identificación, sobre todo cuando se trata de bacterias grampositivas y levaduras26.
Extracción mediante etanol-ácido fórmico. Este protocolo, indicado para el estudio de bacterias no formadoras de esporas, utiliza el etanol para inactivar las bacterias y el ácido fórmico para la disrupción de su pared. Brevemente, el protocolo es el siguiente: se resuspende el pellet o colonia de interés con agua bidestilada y etanol. Después de centrifugar y eliminar completamente los restos de etanol, se añade ácido fórmico al 70% y la misma cantidad de acetonitrilo para la disrupción de la pared celular. Una vez centrifugada la muestra, se recoge el sobrenadante y se deposita 1μl en la tarjeta del instrumento, se recubre con la matriz, se deja secar y se procede a su análisis.
Extracción mediante ácido trifluoroacético (TFA). Este protocolo está indicado para bacterias formadoras de esporas o de elevada patogenicidad. Mediante el TFA se consigue tanto la inactivación de la bacteria como la disrupción de la pared. Brevemente, se resuspende la muestra en TFA al 80% hasta su completa disolución y se deja incubar a temperatura ambiente. Tras añadir agua bidestilada, se le añade un volumen igual de acetonitrilo y se centrifuga. A continuación se deposita 1μl del sobrenadante en la tarjeta del instrumento, se recubre con la matriz, se deja secar y se procede a su análisis.
A continuación se describen las principales aplicaciones de las plataformas comerciales:
Identificación de microorganismos a partir de colonia- •
Bacterias. Las dos plataformas anteriormente comentadas ofrecen la posibilidad de identificación directamente a partir de una colonia. Se han publicado diversos trabajos en los que se compara la identificación obtenida por EM, especialmente utilizando el MALDI Biotyper, con los métodos microbiológicos clásicos. Los más relevantes se resumen en la tabla 1. En general se han obtenido valores de concordancia a nivel de especie con los métodos convencionales entre el 84 y el 95%.
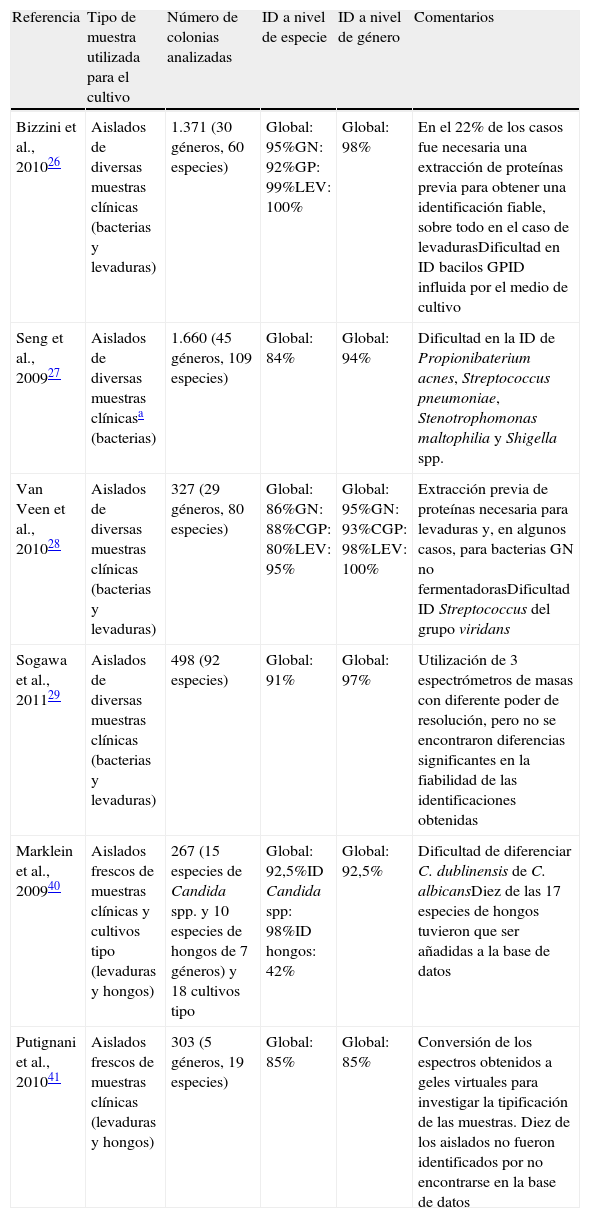
Tabla 1.Identificación a partir de colonia con la plataforma MALDI Biotyper (Bruker Daltonics)
Referencia Tipo de muestra utilizada para el cultivo Número de colonias analizadas ID a nivel de especie ID a nivel de género Comentarios Bizzini et al., 201026 Aislados de diversas muestras clínicas (bacterias y levaduras) 1.371 (30 géneros, 60 especies) Global: 95%GN: 92%GP: 99%LEV: 100% Global: 98% En el 22% de los casos fue necesaria una extracción de proteínas previa para obtener una identificación fiable, sobre todo en el caso de levadurasDificultad en ID bacilos GPID influida por el medio de cultivo Seng et al., 200927 Aislados de diversas muestras clínicasa (bacterias) 1.660 (45 géneros, 109 especies) Global: 84% Global: 94% Dificultad en la ID de Propionibaterium acnes, Streptococcus pneumoniae, Stenotrophomonas maltophilia y Shigella spp. Van Veen et al., 201028 Aislados de diversas muestras clínicas (bacterias y levaduras) 327 (29 géneros, 80 especies) Global: 86%GN: 88%CGP: 80%LEV: 95% Global: 95%GN: 93%CGP: 98%LEV: 100% Extracción previa de proteínas necesaria para levaduras y, en algunos casos, para bacterias GN no fermentadorasDificultad ID Streptococcus del grupo viridans Sogawa et al., 201129 Aislados de diversas muestras clínicas (bacterias y levaduras) 498 (92 especies) Global: 91% Global: 97% Utilización de 3 espectrómetros de masas con diferente poder de resolución, pero no se encontraron diferencias significantes en la fiabilidad de las identificaciones obtenidas Marklein et al., 200940 Aislados frescos de muestras clínicas y cultivos tipo (levaduras y hongos) 267 (15 especies de Candida spp. y 10 especies de hongos de 7 géneros) y 18 cultivos tipo Global: 92,5%ID Candida spp: 98%ID hongos: 42% Global: 92,5% Dificultad de diferenciar C. dublinensis de C. albicansDiez de las 17 especies de hongos tuvieron que ser añadidas a la base de datos Putignani et al., 201041 Aislados frescos de muestras clínicas (levaduras y hongos) 303 (5 géneros, 19 especies) Global: 85% Global: 85% Conversión de los espectros obtenidos a geles virtuales para investigar la tipificación de las muestras. Diez de los aislados no fueron identificados por no encontrarse en la base de datos ID: identificación (proporción de resultados obtenidos mediante el sistema BioTyper 2.0 concordantes con la identificación obtenida por los métodos microbiológicos convencionales); GN: bacterias gramnegativas; GP: bacterias grampositivas; CGP: cocos grampositivos; LEV: levaduras.
Cherkaoui et al. realizaron un estudio comparando ambas plataformas comerciales, MALDI Biotyper y Axima@SARAMIS24. De 720 colonias analizadas, el sistema MALDI Biotyper identificó correctamente y con una elevada confianza el 94,4% de ellas, en comparación al 88,8% obtenido por el sistema Axima@SARAMIS. En las muestras en las que se obtuvo una identificación diferente a la conseguida mediante los métodos convencionales se llevó a cabo la secuenciación del gen 16S para confirmar la identificación. De las identificaciones discrepantes obtenidas utilizando la plataforma MALDI Biotyper, la secuenciación del gen 16S confirmó los resultados obtenidos por dicha plataforma en el 75% de los casos. Este porcentaje fue del 78,3% en el caso de la plataforma de Axima@SARAMIS.
Además de la identificación a ciegas de aislados clínicos, la EM ha demostrado ser útil a la hora de identificar microorganismos a los que es difícil hacerlo por métodos clásicos, como por ejemplo las bacterias gramnegativas no fermentadoras30,31 o las diferentes especies del género Bacteroides32. Así mismo, esta tecnología también permite la identificación y tipificación de Listeria spp.33, Staphylococcus spp.34,35 y Mycobacterium spp.36.
La mayor limitación de esta técnica es que pueden existir errores en la identificación de bacterias genéticamente muy parecidas, ya que pueden presentar proteomas parecidos. Por ello, el criterio del microbiólogo clínico es crucial en la interpretación de los resultados. La identificación en estos casos puede mejorarse con una extracción proteica previa, pero se recomienda la confirmación por métodos bioquímicos o por secuenciación. Por otro lado, tal y como se explica en el apartado «Espectrometría de masas dirigida a la detección de proteínas», el software adjudica a cada identificación un valor indicativo de su fiabilidad. En el caso de Biotyper 2.0, se ha observado que con valores entre 1,7 y 2, que indicarían un parentesco muy cercano y una identificación fiable tan solo a nivel de género, de hecho se obtienen identificaciones correctas a nivel de especie. Algunos autores consideran estos intervalos demasiado restrictivos a la hora de validar los resultados. Así pues, dentro de dicho intervalo se debería poder adjudicar un elevado grado de fiabilidad a la identificación, sobre todo si se puede disponer de una morfología de colonia que la corrobore37. Por último, se ha observado una necesidad de completar la base de datos con más perfiles proteicos para mejorar la identificación correcta a nivel de especie, sobre todo en el caso de bacterias poco comunes29,38.
- •
Levaduras y hongos filamentosos. Dada la gravedad de las fungemias, sobre todo en pacientes immunodeprimidos, es necesaria una rápida identificación de las mismas. Bader et al.39 realizaron un estudio en el que compararon ambas plataformas comerciales (MALDI Biotyper y Axima@SARAMIS) para la identificación de levaduras patógenas. Se analizaron un total de 1.192 aislados, que incluyeron 36 especies diferentes. El porcentaje de identificaciones correctas obtenidas mediante la plataforma MALDI Biotyper fue del 97,6%, y en el caso de la plataforma Axima@SARAMIS el porcentaje fue de 96,1%. Los resultados de otros estudios de interés aparecen resumidos en la tabla 1.
Desde nuestro mejor conocimiento, a día de hoy solo se ha publicado un artículo en el que se utiliza una plataforma comercial, la MALDI Biotyper en su versión de software Biotyper 1.0 (Bruker Daltonics), para la identificación de hongos filamentosos del género Fusarium42. En este estudio se creó una base de datos para la identificación de este patógeno. Se incluyeron 60 cepas obtenidas a partir de aislados clínicos y dos cepas de aislados ambientales. La identificación de todos los aislados fue comprobada mediante secuenciación. Al analizar 49 aislados clínicos a ciegas, se obtuvo una identificación correcta en el 92% de los casos. Cuando no se obtuvo un resultado fiable fue debido a la presencia de especies del género Fusarium que no estaban incluidas en la base de datos creada.
A pesar de ser este un territorio poco explorado con la utilización plataformas comerciales, la EM se ha empleado durante años para la identificación de hongos filamentosos. Santos et al.43 han publicado recientemente una exhaustiva revisión referida al tema en cuestión.
En cuanto a la preparación de la muestra en el caso de las levaduras, en los trabajos revisados (tabla 2) se realiza una extracción de proteínas previa mediante etanol/ácido fórmico. En el caso de hongos filamentosos, Marinach-Patrice et al.42 realizan una extracción previa mediante ácido trifluoroacético/acetonitrilo. Sin embargo, en su trabajo de revisión Santos et al.43 comentan que, si bien la lisis era necesaria para la obtención del espectro en los primeros experimentos, en trabajos recientes se ha logrado evitar este paso.
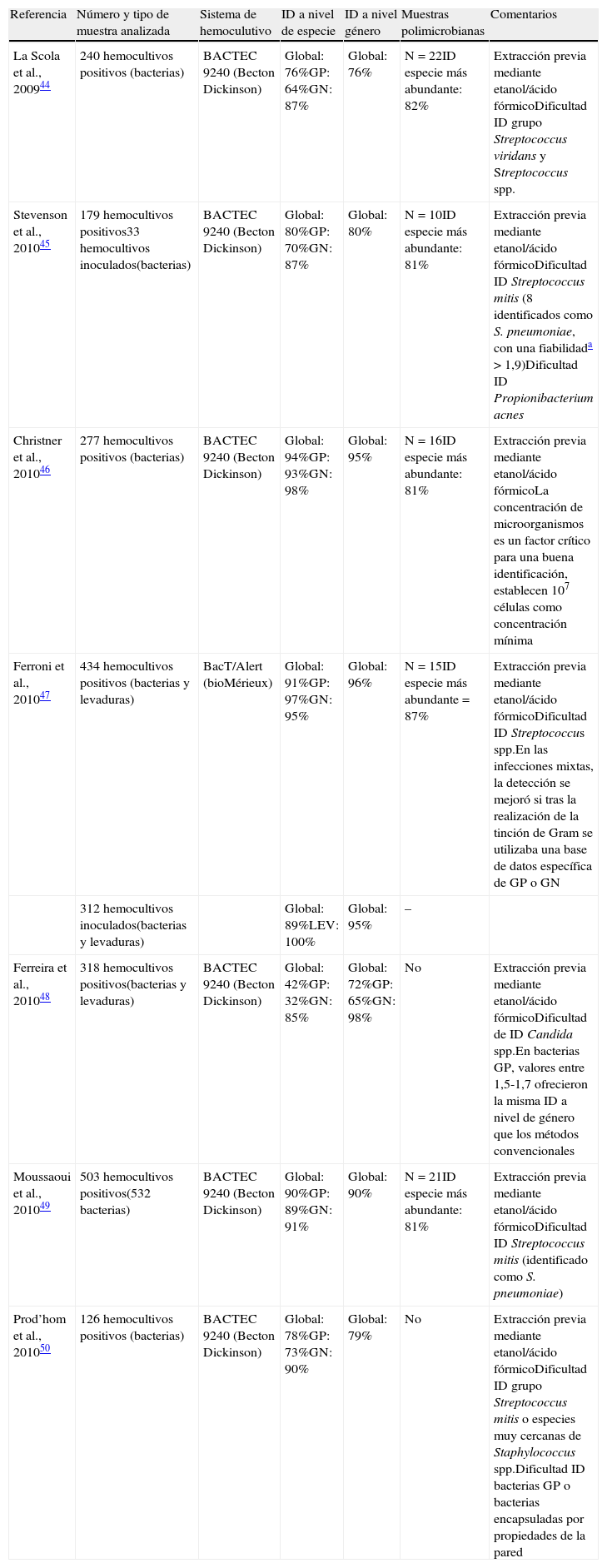
Identificación de bacterias y levaduras a partir de hemocultivos mediante la plataforma MALDI Biotyer (Bruker Daltonics)
| Referencia | Número y tipo de muestra analizada | Sistema de hemoculutivo | ID a nivel de especie | ID a nivel género | Muestras polimicrobianas | Comentarios |
| La Scola et al., 200944 | 240 hemocultivos positivos (bacterias) | BACTEC 9240 (Becton Dickinson) | Global: 76%GP: 64%GN: 87% | Global: 76% | N=22ID especie más abundante: 82% | Extracción previa mediante etanol/ácido fórmicoDificultad ID grupo Streptococcus viridans y Streptococcus spp. |
| Stevenson et al., 201045 | 179 hemocultivos positivos33 hemocultivos inoculados(bacterias) | BACTEC 9240 (Becton Dickinson) | Global: 80%GP: 70%GN: 87% | Global: 80% | N=10ID especie más abundante: 81% | Extracción previa mediante etanol/ácido fórmicoDificultad ID Streptococcus mitis (8 identificados como S. pneumoniae, con una fiabilidada >1,9)Dificultad ID Propionibacterium acnes |
| Christner et al., 201046 | 277 hemocultivos positivos (bacterias) | BACTEC 9240 (Becton Dickinson) | Global: 94%GP: 93%GN: 98% | Global: 95% | N=16ID especie más abundante: 81% | Extracción previa mediante etanol/ácido fórmicoLa concentración de microorganismos es un factor crítico para una buena identificación, establecen 107 células como concentración mínima |
| Ferroni et al., 201047 | 434 hemocultivos positivos (bacterias y levaduras) | BacT/Alert (bioMérieux) | Global: 91%GP: 97%GN: 95% | Global: 96% | N=15ID especie más abundante=87% | Extracción previa mediante etanol/ácido fórmicoDificultad ID Streptococcus spp.En las infecciones mixtas, la detección se mejoró si tras la realización de la tinción de Gram se utilizaba una base de datos específica de GP o GN |
| 312 hemocultivos inoculados(bacterias y levaduras) | Global: 89%LEV: 100% | Global: 95% | – | |||
| Ferreira et al., 201048 | 318 hemocultivos positivos(bacterias y levaduras) | BACTEC 9240 (Becton Dickinson) | Global: 42%GP: 32%GN: 85% | Global: 72%GP: 65%GN: 98% | No | Extracción previa mediante etanol/ácido fórmicoDificultad de ID Candida spp.En bacterias GP, valores entre 1,5-1,7 ofrecieron la misma ID a nivel de género que los métodos convencionales |
| Moussaoui et al., 201049 | 503 hemocultivos positivos(532 bacterias) | BACTEC 9240 (Becton Dickinson) | Global: 90%GP: 89%GN: 91% | Global: 90% | N=21ID especie más abundante: 81% | Extracción previa mediante etanol/ácido fórmicoDificultad ID Streptococcus mitis (identificado como S. pneumoniae) |
| Prod’hom et al., 201050 | 126 hemocultivos positivos (bacterias) | BACTEC 9240 (Becton Dickinson) | Global: 78%GP: 73%GN: 90% | Global: 79% | No | Extracción previa mediante etanol/ácido fórmicoDificultad ID grupo Streptococcus mitis o especies muy cercanas de Staphylococcus spp.Dificultad ID bacterias GP o bacterias encapsuladas por propiedades de la pared |
ID: identificación (proporción de resultados obtenidos mediante el sistema BioTyper 2.0 concordantes con la identificación obtenida por los métodos microbiológicos convencionales). GN: bacterias gramnegativas; GP: bacterias grampositivas; LEV: levaduras.
La identificación de bacterias mediante MALDI-TOF directamente a partir de un hemocultivo positivo permite reducir el tiempo de obtención del resultado en comparación con los métodos convencionales, ofreciendo la posibilidad de mejorar el manejo de los pacientes con sepsis. Recientemente se han publicado varios estudios cuyos resultados se resumen en la tabla 2. En la mayoría de los estudios se realizó la extracción previa con etanol/ácido fórmico previamente descrita, ya que como se demuestra en el trabajo publicado por La Scola et al.44, parece ser más efectiva para las bacterias grampositivas que la extracción mediante TFA. De los 322 hemocultivos analizados mediante la extracción con TFA, en el 59% de las muestras los resultados fueron concordantes con los obtenidos mediante los métodos convencionales. De estos, la concordancia para las bacterias gramnegativas fue del 94%, mientras que para las grampositivas fue del 37%. En una segunda fase del estudio se analizaron un total de 240 hemocultivos y se utilizó el protocolo de extracción con etanol/ácido fórmico, mediante el cual se mejoró la detección de las bacterias grampositivas, tal y como se refleja en la tabla 2. En el trabajo realizado por Ferreira et al.51 se compararon la identificación a partir de células intactas y la extracción mediante etanol/ácido fórmico. Mediante este segundo método la extracción de proteínas consiguió identificar un mayor número de microorganismos y con valores de fiabilidad más elevados.
En un estudio recientemente publicado, Schmidt et al.52 compararon los frascos de hemocultivo BD BACTEC Plus-aerobic (Becton Dickinson, Franklin Lakes, EE. UU.), que contienen resinas, con los frascos del sistema BacT/Alert (bioMérieux, Marcy l’Etoile, Francia) SA y FA, de los cuales solo los segundos contienen carbón activo. La plataforma de EM utilizada fue Axima@SARAMIS. Los frascos se inocularon con 103 microorganismos distintos. Para los frascos BD Bactec Plus-aerobic, BacT/Alert SA y BacT/Alert FA la concordancia general con los métodos convencionales fue, respectivamente, del 72, del 46 y del 23%. Para las bacterias gramnegativas la concordancia fue, respectivamente, del 87, del 69 y del 47%. Finalmente, la concordancia en la identificación de las bacterias grampositivas fue, respectivamente, del 60, del 29 y del 5%. Estos datos corroboran lo anteriormente publicado por Szabados et al.53, quienes demostraron que la sensibilidad de la identificación directa de hemocultivos positivos utilizando el sistema BacT/Alert era baja.
La identificación mediante esta técnica a partir de hemocultivo presenta las mismas limitaciones comentadas anteriormente en «Identificación de microorganismos a partir de colonia».
Identificación de microorganismos a partir de muestra directaDado que estas plataformas necesitan una concentración mínima de bacterias para lograr una identificación, resulta difícil la aplicación de la EM a la muestra directa sin pasar por el cultivo. Aun así, sí se han realizado estudios utilizando EM en muestras con una considerable concentración de microorganismos, como es la orina.
Ferreira et al.54 utilizaron el sistema MALDI Biotyper para la identificación de bacterias directamente a partir de orina mediante la aproximación de células intactas. Sin embargo, en el caso de no obtener una buena identificación, se realizó la extracción previa de proteínas mediante el protocolo de etanol/ácido fórmico. A nivel de especie, la concordancia obtenida con los métodos convencionales fue del 91,8%, y del 92% a nivel de género.
Otras aplicaciones del MALDI-TOF no ofrecidas por las plataformas comercialesA pesar de no existir aún publicaciones en las que se estudie la resistencia a antibióticos mediante las plataformas comerciales previamente mencionadas, sí se han publicado trabajos en los que se utiliza la EM para la discriminación de cepas de Staphylococcus aureus resistentes a la meticilina de aquellas sensibles, ya que presentan variaciones de picos en el espectro de masas22,55. De manera similar, en otro trabajo se describe la diferenciación entre cepas de Escherichia coli resistentes y sensibles a la ampicilina56.
Por otro lado, aunque la identificación de virus aún no ha sido puesta a punto por las casas comerciales descritas, también constituye una de las aplicaciones de la EM. Por ejemplo, la identificación de Norovirus se ha llevado a cabo a partir de la detección de las proteínas de la cápside57, y la identificación del virus de Newcastle, por la detección de un polipéptido vírico58.
Detección de ácidos nucleicosPCR y MALDI-TOFSEQUENOM (San Diego, EE. UU.) ofrece un método para la tipificación de bacterias con una precisión comparable al multilocus sequence typing (MLST). Esta metodología se basa en una amplificación inicial del DNA de la muestra utilizando un par de primers que incorporan en sus extremos 5′ los promotores T7 y SP6, respectivamente, lo que permite la posterior transcripción de ambas cadenas en dos reacciones diferentes. Una vez realizada la transcripción, se utiliza una enzima de restricción para cortar de manera específica el producto amplificado después de cada residuo de uracilo o citosina59,60. Estos fragmentos se analizan mediante EM tipo MALDI-TOF. Para la identificación se utiliza una base de datos previamente generada in silico de patrones de MLST generados a partir de secuencias de referencia.
Esta tecnología ha demostrado ser útil para la tipificación de Neisseria menigitidis59, para el genotipado de micobacterias61, así como para el genotipado de los virus de las hepatitis B y C62,63, entre otros.
PCR y ESI-TOFEsta tecnología, denominada comercialmente PLEX-ID (anteriormente T5000), ha sido propuesta por Ibis Biosciences Inc. (Carlsbad, EE. UU.), empresa subsidiaria de Abbott Molecular (Chicago, EE. UU.). Se basa en la amplificación de los ácidos nucleicos existentes en la muestra y en el posterior análisis del producto de PCR mediante EM tipo ESI-TOF64.
El principio básico de esta tecnología es que todos los microorganismos comparten características comunes codificadas en sus genomas, y que estas secuencias conservadas permiten el diseño de primers universales. Con ellos es posible amplificar regiones conservadas pero con suficiente variabilidad como para permitir la identificación de los diferentes microorganismos. Además, el uso de distintos pares de primers en diferentes pocillos promueve la amplificación de los diferentes microorganismos que puedan coexistir en una muestra clínica sin que haya competencia por los recursos de la PCR. Posteriormente, el producto se analiza por ESI-TOF, determinándose así la masa de los amplicones presentes en la reacción de PCR con tal precisión que proporciona información sobre su composición específica de bases, es decir, en qué proporción se encuentran los diferentes nucleótidos en el amplicón. Sin embargo, no se obtiene la secuencia nucleotídica de los productos de PCR. Esta composición de bases es única para cada gen y específica de cada microorganismo, permitiendo así su identificación gracias al software y a la base de datos proporcionados.
Potencialmente, esta tecnología es capaz de identificar todas las bacterias conocidas, así como las principales familias de hongos patógenos y de virus a partir de cualquier tipo de muestra, previa extracción de los ácidos nucleicos. Además, debido a su diseño, esta técnica también permite la identificación de microorganismos no descritos previamente. Dado que se basa en la amplificación genética a partir de muestra directa, permite además la identificación de microorganismos no cultivables65. Otra de las ventajas de esta tecnología es que permite la cuantificación, gracias a un calibrador interno de concentración conocida añadido a cada reacción de PCR, informando del número inicial de genomas del microorganismo identificado en la muestra.
- •
Identificación de bacterias. Los primers universales más utilizados son los primers ribosomales, que amplifican los genes 16S o 23S, pero existen otras regiones ampliamente conservadas, como el gen rpoB y otros genes housekeeping. El objetivo es utilizar varios pares de primers, cada uno dirigido a amplificar un grupo o división de bacterias concreto. De esta manera los diferentes tipos de bacterias que pueda haber en la muestra podrán ser amplificados sin presentar competencia, ya que si la bacteria menos abundante no consigue ser amplificada mediante los primers ribosomales, sí podría detectarse mediante los primers específicos de su división. El resultado final es la obtención de una mezcla de amplicones que reflejará la complejidad de la muestra inicial. La utilidad de esta tecnología para la vigilancia epidemiológica queda reflejada en diversos trabajos66-70. Algunos ejemplos considerados de especial interés se comentan a continuación.
Identificación de Mycobacterium tuberculosis multirresistente. El ensayo molecular descrito por Massire et al.70 consta de 16 pares de primers repartidos en 8 reacciones dúplex. Se analizaron un total de 1.340 aislados, cuya resistencia había sido previamente caracterizada. La sensibilidad y la especificidad en la detección de la resistencia a la isoniazida fueron del 89,3 y del 95,8%, respectivamente. Para la detección de la resistencia a la rifampicina, estas fueron del 96,3 y del 98,6%, respectivamente. Por último, en el caso de la resistencia al etambutol, los valores fueron del 73,6 y del 97,3%. Los valores predictivos positivos y negativos para la detección de estas resistencias fueron, respectivamente, del 97,3 y del 84,3% para la isoniazida; del 96,7 y del 98,5% para la rifampicina, y del 84,4 y del 94,9% para el etambutol.
El ensayo también demostró su utilidad para la especiación y distinción entre micobacterias pertenecientes al complejo M. tuberculosis y micobacterias no tuberculosas (NTM). La sensibilidad en la detección de las bacterias pertenecientes al complejo M. tuberculosis fue del 98%, y la especificidad, del 100%. Para las NTM, dichos valores fueron del 97 y del 99,9%. Los valores predictivos positivos y negativos para la identificación de estos dos grupos fueron, respectivamente, del 100 y del 99,5% para el complejo M. tuberculosis, y del 99,6 y del 99,6% para las NTM.
Vigilancia de patógenos respiratorios. En el estudio de Ecker et al.71 se presentan los resultados obtenidos tras el análisis de diferentes brotes de infecciones respiratorias ocurridos durante los años 2002 y 2003 en diversas instalaciones militares de Estados Unidos. En el ensayo desarrollado para este fin, se combinaron primers universales para la detección de bacterias y primers específicos para el genotipado de Streptococcus pyogenes basados en el gen emm, el cual codifica para la proteína M, un factor de virulencia. En 48 de las 51 muestras analizadas del brote del 2002 se detectó S. pyogenes perteneciente al genotipo emm3, el cual ha sido asociado a una elevada virulencia. Además, durante el análisis de las muestras se detectó la presencia de otros microorganismos, siendo Haemophilus influenzae y Neisseria meningitidis los más abundantes, además de S. pyogenes, lo cual demuestra que en la mayoría de casos las infecciones respiratorias tienden a ser polimicrobianas. Al analizar muestras de pacientes sanos, el patrón de microorganismos encontrados en la garganta de dichos voluntarios fue diferente, predominando una microbiota de estreptococos del grupo viridans. Durante el brote de 2003 se comprobó que la distribución de S. pyogenes había cambiado y que el genotipo virulento emm3 ya no predominaba.
Identificación de bacterias a partir de hemocultivo. Existen tres trabajos publicados en los que se utiliza esta tecnología para identificar bacterias y levaduras a partir del hemocultivo positivo mediante el ensayo BAC Assay (Abbott Sterile Fluid Bacteria and Candida assay). En dichos trabajos se comparó la identificación obtenida mediante el PLEX-ID con los resultados obtenidos mediante los métodos clásicos: cultivo e identificación mediante perfil bioquímico (Vitek 2 Compact, bioMérieux). En el primero de los dos estudios72 se obtuvo una concordancia a nivel de género del 94% y a nivel de especie del 93%, de un total de 170 hemocultivos positivos y 199 microorganismos analizados. En el segundo de los trabajos73 se obtuvo una concordancia a nivel de género del 98,7% y a nivel de especie del 96,5%, de un total de 234 hemocultivos y 211 microorganismos. Finalmente, el último trabajo74, que se trata de una comparación de esta tecnología con la plataforma comercial de EM por MALDI-TOF MALDI Biotyper, se comenta más adelante. Sin embargo, esta tecnología podría utilizarse a partir de muestra directa; en este caso, la sangre total del paciente antes de inocular el hemocultivo. En el caso de la sepsis, esto permitiría acortar el proceso en una media de 9h, tiempo necesario para la positivización del hemocultivo, acelerando el proceso diagnóstico y mejorando el manejo de los pacientes. Sin embargo, hasta el momento no existen publicaciones al respecto, aunque nuestro centro está actualmente realizando una evaluación.
- •
Identificación de virus. Mediante esta tecnología es posible identificar las principales familias víricas. Se han publicado varios trabajos que demostraban la versatilidad de esta técnica para la detección de virus, como por ejemplo el ensayo diseñado para la detección de los virus de la gripe A y B, con una sensibilidad y una especificidad superiores al 97% (determinadas tras el análisis a ciegas de 656 muestras clínicas)75. También se han descrito ensayos para la detección de Coronavirus, Bocavirus y Metapneumovirus, entre otros75-78.
Esta tecnología fue una de las primeras en identificar la nueva cepa H1N1 del virus de la gripe responsable de la pandemia del 2009, ya que el ensayo para la identificación de este virus fue capaz de detectar esta nueva cepa sin ninguna modificación previa del mismo79.
- •
Identificación de factores de virulencia y/o resistencia. Incluidos en el ensayo BAC Assay (Abbott Sterile Fluid Bacteria and Candida assay) se encuentran los genes mecA, el cual confiere resistencia a la meticilina en cepas de S. aureus, la detección de los genes vanA y vanB, los cuales confieren resistencia a la vancomicina, de especial interés cuando se trata del género Enterococcus spp., y la detección del gen kpc, de gran relevancia médica en el caso de Klebsiella pneumoniae y que le confiere resistencia al carbapenem. La detección de esta última resistencia también ha sido evaluada en Acinetobacter baumannii80. En el caso de S. aureus, también existe la posibilidad de detección de factores de virulencia, como por ejemplo la leucocidina de Panton-Valentine81,82.
Recientemente se ha publicado un trabajo en el que se comparan estas dos técnicas para la identificación de bacterias y levaduras a partir de un total de 273 hemocultivos positivos74.
- •
Concordancia entre los resultados obtenidos por EM y los métodos clásicos. Ambas técnicas mostraron una muy buena concordancia con la identificación obtenida mediante cultivo y pruebas bioquímicas (Vitek 2, bioMérieux). En el caso del PLEX-ID, la concordancia obtenida fue del 96,7% a nivel de género y del 95,6% a nivel de especie. En al caso del MALDI-TOF, la plataforma utilizada fue la de Bruker Daltonics, y la concordancia obtenida fue del 97,1% a nivel de género y del 94,9% a nivel de especie.
- •
Preparación, análisis y coste por muestra. En el caso del PLEX-ID, desde la obtención de la muestra del paciente son necesarias de 4 a 6h, que incluyen la extracción del DNA, la amplificación por PCR y el análisis de la muestra mediante el espectrómetro de masas. En el caso del sistema MALDI Biotyper, se realizó un subcultivo de los hemocultivos positivos en caldo de enriquecimiento, los cultivos enriquecidos se sembraron en diferentes medios y se utilizó una colonia para el análisis por MALDI-TOF. Como se menciona en «Espectrometría de masas dirigida a la detección de proteínas», esta plataforma permite el análisis de hasta 48 muestras en una hora.
Por otro lado, hay que tener en cuenta que el PLEX-ID necesita diversos reactivos y equipos, tanto para la extracción automatizada de DNA como para la realización de la PCR y el análisis por EM, mientras que para la utilización del MALDI-TOF los requerimientos son mínimos: tan solo es necesaria la matriz orgánica. Por ello, aunque ambas tecnologías requieren de una inversión inicial considerable en instrumentación, el coste por muestra mediante el PLEX-ID se encuentra entre 34 y 70 euros, mientras que el coste por muestra utilizando el MALDI-TOF es de 2 a 4 euros.
ConclusionesLa EM ha demostrado ser una técnica muy útil para la identificación y la tipificación de microorganismos. El reciente desarrollo de plataformas comerciales diseñadas para este fin ha puesto esta tecnología al alcance de los laboratorios de microbiología, con sistemas de fácil manejo y de tamaño y precio asequible. A pesar de necesitar una inversión previa en el instrumento, la rapidez en la obtención de los resultados es un beneficio a tener en cuenta.
La tecnología basada en la identificación del perfil proteico mediante MALDI-TOF ofrece una identificación fiable tanto de bacterias como de levaduras, con el inconveniente de que se necesita una concentración mínima de 105-106 células para obtener una buena identificación, con lo cual ha sido mayormente aplicado al análisis de cultivos. A pesar de esto, se ha demostrado su utilidad en el análisis directo de orina.
Durante el análisis de hemocultivos, es usual encontrar infecciones polimicrobianas. En estos casos, la tecnología MALDI-TOF generalmente identifica la bacteria más abundante, aunque se ha visto que si se observan manualmente los espectros y el listado de diferentes identificaciones con valores de fiabilidad más bajos, es posible identificar más de un microorganismo. Otra de las desventajas de esta tecnología es la incapacidad actual de detectar resistencias a los antibióticos, salvo las excepciones antes mencionadas.
La plataforma ofrecida por SEQUENOM ofrece una herramienta rápida y fiable de tipificación, pero es necesario conocer previamente el microorganismo analizado para el diseño de los primers. A pesar de resultar una herramienta útil para la epidemiología, no lo es para la identificación de microorganismos de manera rutinaria en el laboratorio de microbiología.
Finalmente, la tecnología PLEX-ID, basada en una PCR y ESI-TOF, permite la detección de cualquier microorganismo. Además, al utilizar primers universales no se necesita saber de antemano el microorganismo diana. El mayor potencial de esta tecnología es su capacidad de detección a partir de muestra directa, ofreciendo un resultado en 4 a 6h, sin ser necesario pasar por el cultivo, aunque de momento son pocos los trabajos en los que se analiza una muestra directa (principalmente, en la detección de virus). Son necesarios, por tanto, estudios clínicos que evalúen la sensibilidad y la especificidad de detección a partir de muestras directas.
A pesar de que la EM ofrece alternativas al cultivo para la identificación de los microorganismos, hay que considerar que, por el momento, aún no existe una tecnología alternativa para el estudio fenotípico de las resistencias y del antibiograma, por lo que de momento el cultivo clásico sigue siendo esencial.
FinanciaciónEste trabajo ha sido financiado por el contrato Miguel Servet del Ministerio de Ciencia e Innovación (MICINN), dentro del Plan Nacional de Investigación científica, Desarrollo e Innovación Tecnológica (I+D+I) (EM), y ha tenido el soporte de la Sociedad Española de Enfermedades Infecciosas y de Microbiología Clínica (SEIMC) (EJL).
Conflicto de interesesLos autores declaran que no existe ningún conflicto de intereses.
AgradecimientosLos autores agradecen al Dr. José María Hernández la revisión crítica del manuscrito.