Describir un brote por Klebsiella pneumoniae (KPN) productora de KPC-3 y determinar la eficacia diagnóstica de MALDI-TOF en su detección.
MétodosEstudio retrospectivo de las KPN-KPC-3 aisladas en 2 hospitales de Ciudad Real. Se buscó el pico a 11,109kDa±15 en el espectro proporcionado por MALDI-TOF para KPN.
ResultadosSe aislaron 156 cepas de KPN que portaban el gen blaKPC-3, con un único perfil perteneciente al ST512 (31 cepas estudiadas). Hubo un 25% de infectados. Un 84% tuvieron origen nosocomial o relacionado con la asistencia sanitaria. El 93% tenía alguna enfermedad de base (31% de exitus en el primer mes).
La detección del pico mostró una sensibilidad del 90% y una especificidad del 100%.
ConclusionesDetectamos la diseminación clonal de una cepa de KPN ST512 productora de KPC-3 en 3 hospitales de Ciudad Real. Además, evidenciamos la rentabilidad de MALDI-TOF en la detección precoz de KPN-KPC.
To describe an outbreak of KPC-3-producing Klebsiella pneumoniae (KPN) and determine the diagnostic efficacy of MALDI-TOF in its detection.
MethodsRetrospective study of the KPC-3-KPN isolated in 2 hospitals in Ciudad Real. The peak at 11,109kDa±15 was sought in the KPN spectra provided by MALDI-TOF.
ResultsWe isolated 156 KPN strains that carried the blaKPC-3 gene, with a unique profile belonging to ST512 (31 strains studied). There was 25% of infected patients, 84% were nosocomial or related to health care and 93% had some underlying disease (31% of exitus in the first month).
The detection of the peak showed 90% sensitivity and 100% specificity.
ConclusionsWe detected the clonal spread of a KPN ST512 strain producing KPC-3 in 3 hospitals in Ciudad Real. In addition, we show the profitability of MALDI-TOF in the early detection of KPC-KPN.
Desde el año 2005, en que se detectó la primera enterobacteria productora de carbapenemasa (EPC) en España, hemos presenciado una creciente diseminación de estas bacterias entre los diferentes niveles asistenciales, con un incremento de los hospitales afectados por brotes1,2. Asimismo, se han establecido reservorios de microorganismos resistentes en los distintos nieveles3.
El diagnóstico precoz de las infecciones causadas por EPC es fundamental para implantar medidas de control de la infección y administrar una antibioterapia eficaz. En los últimos años se han desarrollado distintas pruebas rápidas fenotípicas y moleculares para la detección de EPC, incluyendo la tecnología MALDI-TOF4–7. En 2014 se identificó en el espectro proporcionado por MALDI-TOF-MS un pico a 11,109-Da correspondiente a la proteína pKpQIL_p019, codificada en un plásmido que portaba el gen blaKPC8.
Nuestros objetivos fueron revisar los aislamientos de Klebsiella pneumoniae (KPN) productora de carbapenemasa KPC-3 procedentes de 2 hospitales castellano-manchegos, y determinar la eficacia diagnóstica de MALDI-TOF en su detección.
Material y métodosDiseño y variables recogidasEstudio retrospectivo de las cepas de KPN productoras de KPC-3 aisladas en 2 hospitales de Ciudad Real (HGMC y HGT) entre 2016 y junio de 2019 (un aislado por paciente).
Se revisaron las historias clínicas, recogiéndose los siguientes datos: nombre, edad, tipo de muestra, sensibilidad de la cepa, adquisición de la infección (hospitalaria, comunitaria y relacionada con la asistencia sanitaria: residencia o ingreso en los 6 meses anteriores), enfermedades subyacentes, antibioterapia recibida en los 6 meses previos, instrumentalización (sonda, nutrición parenteral/enteral, otras), antibioterapia recibida para tratar la infección y exitus al mes del aislamiento.
Estudio microbiológico y de clonalidadLa identificación bacteriana y el estudio de sensibilidad a los antimicrobianos se realizó mediante el sistema Vitek2®, Vitek-MS® (versión 3.2-IVD) y tiras de gradiente de difusión Etest (BioMérieux), según criterios EUCAST.
En los casos en los que detectamos una disminución de la sensibilidad a algún carbapenémico se comprobó la posible producción de carbapenemasas mediante el test beta-carba (Bio-Rad); en los casos positivos se amplió el estudio fenotípico mediante la sinergia de meropenem con ácido fenilborónico, cloxacilina, ácido dipicolínico y sensibilidad a temocilina (Rosco Diagnostica). Para la detección de la colonización intestinal por la cepa en estudio se utilizaron placas de cribado de agar chromIDTM Carba Smart (BioMérieux).
Se enviaron 31 aislados al Laboratorio de Resistencias a Antibióticos del Centro Nacional de Microbiología para la caracterización genotípica del mecanismo de resistencia implicado (PCR y secuenciación) y el estudio de la epidemiología molecular mediante PGFE tras digestión del ADN total con la enzima de restricción XbaI y MLST. Se seleccionaron cepas representativas (aislamientos de los 4 años de estudio, de distintas procedencias y localizaciones).
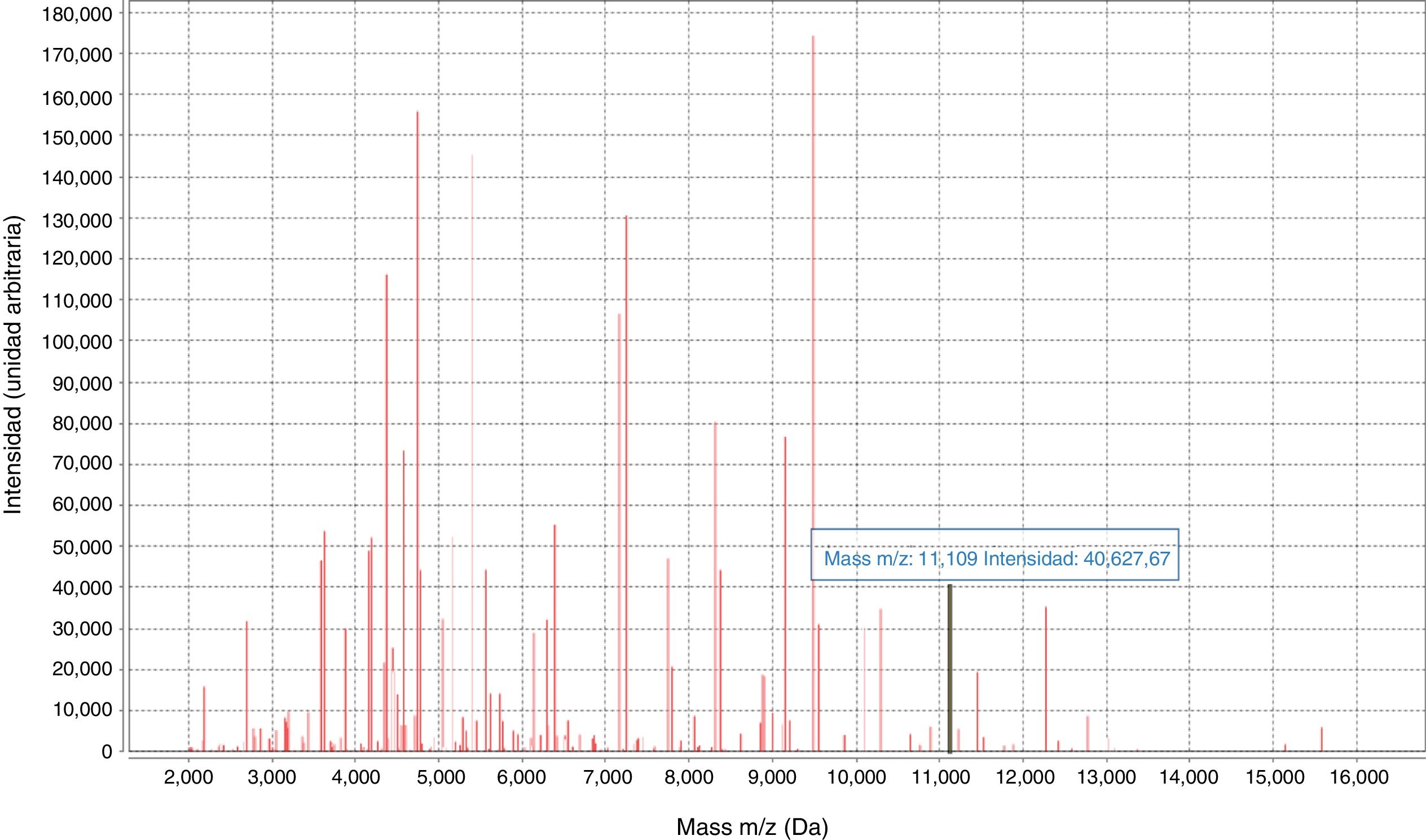
La detección del pico específico de KPC (11,109kDa±15)8 se hizo prospectivamente (enero de 2019) mediante la búsqueda visual en el espectro proporcionado por MALDI-TOF para aislados de KPN8. Los aislamientos procedieron de colonias y de hemocultivos positivos tras un subcultivo de corta duración (4h en agar chocolate). Únicamente se añadió la matriz a la colonia, procesándose una vez seca. El resultado se comparó con el test beta-carba y la sinergia con discos.
Estudio epidemiológicoSe realizó un estudio de colonización intestinal a todos los pacientes con muestras clínicas positivas y a sus compañeros de habitación en el momento del aislamiento. Para ello, se obtuvieron hisopos rectales semanales hasta el alta hospitalaria o hasta lograr 3 resultados de colonización negativos consecutivos.
En 2019 se hizo una búsqueda activa de un posible reservorio acuático (sifón, grifo y desagüe) y en los expendedores de papel en ambos hospitales.
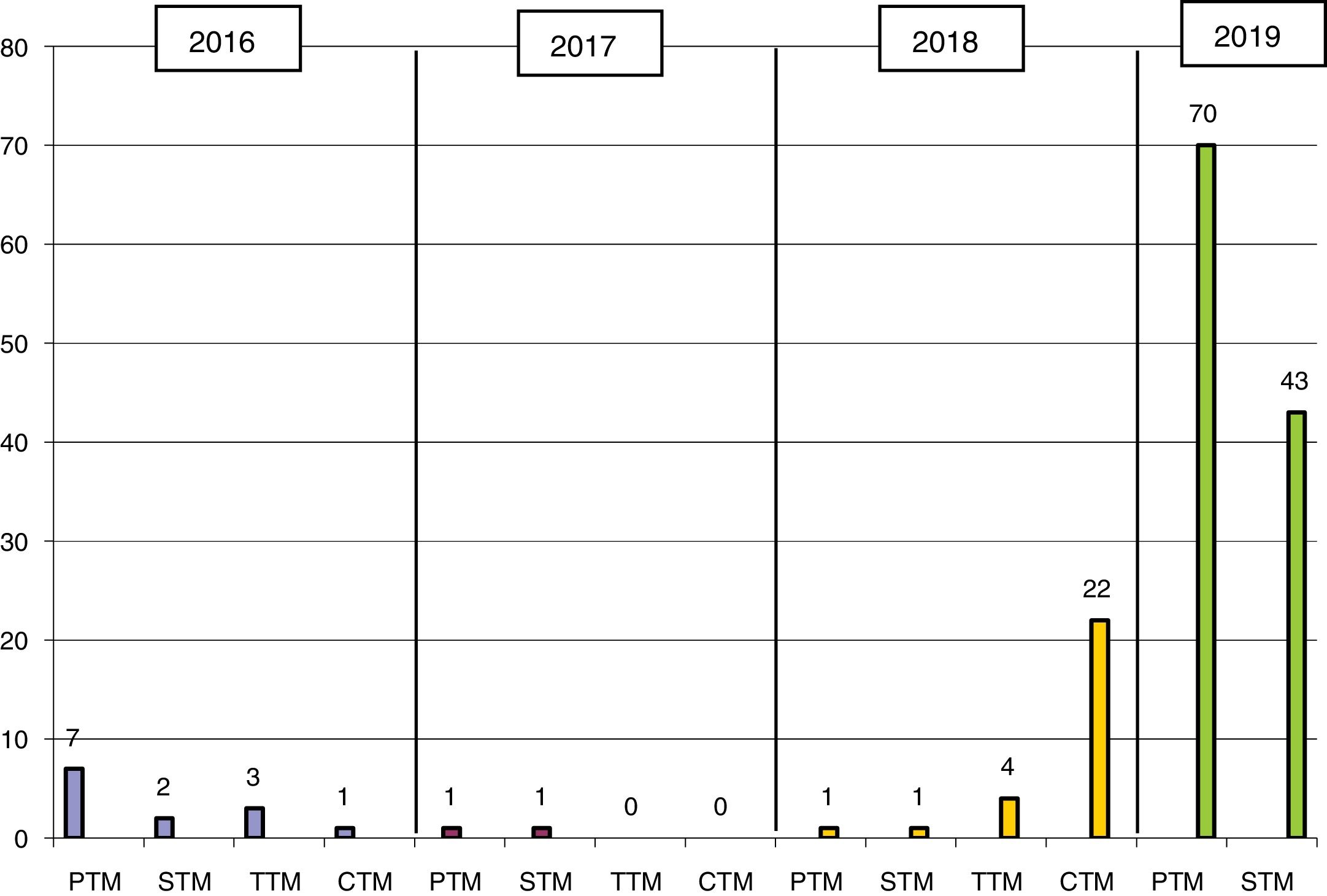
ResultadosEl primer aislamiento de KPN-KPC fue en 2016 en el HGMC, estableciéndose un brote que afectó a 13 pacientes (11 del HGMC y 2 del HGT), y que fue controlado con medidas estándar (fig. 1). El primer aislamiento del HGT procedía de un paciente que había estado ingresado en el HGMC el mes anterior; el segundo aislamiento fue 6 meses después en la misma planta de hospitalización. En 2018 ingresa en el HGT un paciente que había sido hospitalizado al mismo tiempo y en la misma planta que los 2 pacientes del 2016. En agosto de 2018 se establece un segundo brote, afectando a 28 pacientes (26 del HGT y 2 del HGMC). En 2019 vuelve a diseminarse ampliamente por ambos hospitales (fig. 1).
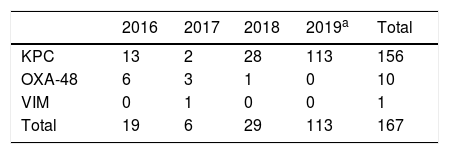
En el periodo de estudio se aislaron 156 cepas de KPN-KPC-3, que suponen el 90% de todas las EPC y el 93% de las KPN productoras de carbapenemasa en el mismo periodo (tabla 1). La secuenciación completa demostró la presencia del gen blaKPC-3. Todas las cepas estudiadas presentaron un único perfil por PFGE perteneciente al ST512 (una de ellas detectada previamente en otro hospital de la provincia).
Aislados de Klebsiella pneumoniae productores de carbapenemasas durante el brote
| 2016 | 2017 | 2018 | 2019a | Total | |
|---|---|---|---|---|---|
| KPC | 13 | 2 | 28 | 113 | 156 |
| OXA-48 | 6 | 3 | 1 | 0 | 10 |
| VIM | 0 | 1 | 0 | 0 | 1 |
| Total | 19 | 6 | 29 | 113 | 167 |
KPN: K. pneumoniae.
La mediana de edad de los pacientes fue 83 años (45-98) y el 58% eran mujeres. El 65% de las muestras fueron exudados rectales, el 24% orinas. Hubo 3 bacteriemias. Un 84% tuvo origen nosocomial y el resto estaba relacionado con la asistencia sanitaria (el 31% procedía de residencias). El 90% había recibido algún antibiótico en los 6 meses anteriores, el 72% estaba instrumentalizado y el 92% tenía alguna enfermedad de base. Hubo un 31% de exitus al mes del aislamiento.
Las cepas presentaron resistencia extensa a antibióticos, siendo sensibles únicamente a colistina (excepto 2 cepas, detección del gen mrc negativa), tigeciclina (24%) y/o gentamicina (62%). El 25% de los pacientes se infectaron (3 pacientes previamente colonizados desarrollaron una infección); el 38% recibió un aminoglucósido, el 31% un carbapenémico y el 27% colistina o tigeciclina. El 62% recibió terapia combinada.
El pico característico de la KPC (fig. 2) se detectó en el 90% de las cepas portadoras del gen blaKPC-3 y permitió su identificación directa en todos los hemocultivos estudiados, con sensibilidad del 90%, especificidad y VPP del 100% y VPN del 75% (tabla 2). La correlación con las pruebas fenotípicas testadas fue del 100%. Se observó una baja adherencia del personal implicado a la correcta higiene de manos. No detectamos ningún reservorio ambiental.
Describimos la diseminación clonal de una cepa de KPN ST512, extremadamente resistente, productora de KPC-3 entre los distintos niveles asistenciales de un área sanitaria de Castilla-La Mancha (España). Este clon se asocia frecuentemente a multirresistencia y es considerado de alto riesgo epidemiológico por su amplia diseminación mundial9. Se detectó por primera vez en España en 2012 en un hospital de Córdoba, donde causó un brote con 67 pacientes infectados10; el caso índice provenía de un hospital de Italia11, donde este clon había sido descrito previamente12. Tras la aparición del brote en nuestra área, la incidencia acumulada de EPC ha pasado del 0,01 en 2017 al 0,24 en 2018; sin embargo, no es hasta el año 2019 que se produce la rápida diseminación de la cepa por el continuo acceso de los pacientes, ancianos pluripatológicos, entre la planta de medicina interna de ambos hospitales y las distintas instituciones de crónicos, estableciéndose una endemia por este clon en nuestra área sanitaria.
Los factores que contribuyen en la diseminación de estos aislados son múltiples. Dada la falta de localización de un reservorio y la elevada colonización rectal, que indica la contaminación cruzada entre pacientes y personal sanitario, se planteó cribar las manos del personal. Finalmente, se desistió ante la baja adherencia al protocolo de higiene de manos, que puede oscilar entre el 5-89%13, y la escasez de medidas de actuación ante un resultado positivo.
La visualización del pico característico en el espectro de MALDI-TOF nos ha permitido la detección de KPN-KPC durante la identificación rutinaria de los cultivos, aunque con una eficacia diagnóstica inferior a la encontrada en otros estudios14,15, lo que lo convierte en el método fenotípico más rápido y económico para detectar KPC. Dado que no detectamos directamente la carbapenemasa, la sensibilidad de la detección dependerá de la prevalencia de clones que portan el plásmido en una determinada área. La falta del pico podría deberse a la ausencia de expresión de la proteína o a la subjetividad en visualizar el pico, lo que constituye una limitación. El siguiente paso sería la búsqueda automática del pico mediante el software del aparato14.
En conclusión, detectamos la diseminación clonal de una cepa de KPN-ST512 productora de KPC-3 en 3 hospitales de Ciudad Real. Hasta la fecha, las medidas implementadas no han resultado eficaces para controlar el brote, pasando a una situación de endemia. Además, evidenciamos la eficacia de MALDI-TOF en la detección precoz de estas cepas sin un gasto adicional, con la consecuente implicación clínica.
Conflicto de interesesNinguno.
Agradecemos al Dr. Jesús Oteo su asesoramiento.
La presente investigación no ha recibido ayudas específicas provenientes de agencias del sector público, sector comercial o entidades sin ánimo de lucro.