Actinomyces y géneros relacionados son bacilos grampositivos; estos patógenos oportunistas han sido involucrados, principalmente, en infecciones endógenas. Dado lo complejo que resulta abordar la identificación de estos microorganismos en la mayoría de los laboratorios clínicos, existe un desconocimiento acerca de su real significado clínico. En este trabajo se estudió una colección de 166 aislados correspondientes a 13 especies diferentes de Actinomyces/Actinotignum, recuperados de muestras clínicas de pacientes atendidos en un hospital universitario. La identificación se realizó por estudios moleculares y por espectrometría de masas MALDI-TOF MS. Mediante MALDI-TOF MS se identificó al 91,57% de los aislados (152/166) a nivel de especie, con un score ≥ 1,7; un 3,61% (6/166) solo se pudo identificar a nivel de género, con un score ≥ 1,5. Esta metodología no produjo resultados de identificación confiable en el 4,82% (8/166) de los aislados. Estas 13 especies de Actinomyces/Actinotignum se hallaron en diversos tipos de muestras: partes blandas (n = 47), orina (n = 35), abscesos de cabeza/cuello (n = 19), abscesos genitales (n = 11), sangre (n = 10), abscesos de mama (n = 8), osteoarticulares (n = 6), líquido ascítico/abdominal (n = 3), abscesos abdominales (n = 5), esputos/BAL (n = 4), abscesos cerebrales (n = 3), otros (n = 15). El análisis estadístico mostró una frecuencia diferencial elevada (> 2) para la asociación localización/especie en los siguientes casos: orina/A. schaalii/sanguinis; abscesos cerebrales/A. europaeus; muestras osteoarticulares/A. urogenitalis; abscesos abdominales/A. turicensis; muestras respiratorias/A. naeslundii/viscosus. Consideramos que esta información contribuirá a una mejor comprensión de la relevancia clínica y epidemiológica de estas especies; al mismo tiempo, muestra que el papel patogénico de Actinomyces spp. podrá ser develado en la medida que estos microorganismos puedan ser reconocidos gracias al cultivo prolongado y los avances de la tecnología de identificación, facilitada por MALDI-TOF MS.
Actinomyces and related genera are grampositive bacilli, opportunistic pathogens, which have been mainly involved in endogenous infections. However, due to the complexity in identifying them for most clinical laboratories, there is scant knowledge about their real clinical significance. In this work, 166 isolates of 13 different species of Actinomyces/Actinotignum species recovered from clinical samples of patients treated in a university hospital were studied. The identification was performed by MALDI-TOF MS and molecular identification. MALDI-TOF MS identified 91.57% of the isolates (152/166) at the species level using a score ≥ 1.7 and 3.61% (6/166) of the isolates were identified only at the gender level with a score ≥ 1.5. MALDI-TOF MS did not yield reliable identification results for 4.82% (8/166) of the isolates. Actinomyces/Actinotignum species were isolated from: soft tissue (n: 47), urine samples (n: 35), head / neck abscesses (n: 19), genital abscesses (n: 11), blood samples (n: 10), breast abscesses (n: 8), osteoarticular samples (n: 6), abdominal/ascitic fluids (n: 3), abdominal abscesses (n: 5), sputum/BAL (n: 4), brain abscesses (n: 3), and others (n: 15). The results obtained from the statistical analysis showed a high differential frequency (> 2) for the location/species association: urine/A. schaalii/sanguinis; brain abscesses/A. europaeus; osteoarticular samples/A. urogenitalis; abdominal abscesses/ A. turicensis; respiratory samples/A. naeslundii/viscosus. This information provides a greater understanding of the clinical and epidemiological relevance of these species. The pathogenic role of Actinomyces spp. will be increasingly revealed as these microorganisms could be recognized thanks to prolonged culture and the advances in identification technology facilitated by MALDI-TOF MS.
Actinomyces y géneros relacionados son bacilos grampositivos; estos patógenos oportunistas han sido implicados en actinomicosis humanas y pueden ocasionar otras infecciones endógenas, tanto en el paciente inmunocomprometido como en el inmunocompetente31,33. La frecuencia de infecciones en las que especies de Actinomyces están involucradas es poco conocida; esto obedece a que los microbiólogos clínicos pueden no estar familiarizados con su reconocimiento. Asimismo, dadas sus características microscópicas y culturales corineformes, estas especies pueden confundirse con otros bacilos grampositivos difteromorfos y, además, ser considerados organismos contaminantes. Esta problemática se explica por la compleja identificación fenotípica que hace falta para abordar la identificación a nivel de especie. En ese sentido, la espectrometría de masas ha facilitado el hallazgo de estas especies, lo cual puede permitir clarificar su rol en nichos ecológicos y en infecciones humanas14,28. El objetivo de este trabajo fue evaluar si existe alguna relación estadísticamente significativa entre las distintas especies de Actinomyces/Actinotignum aisladas de muestras clínicas y las localizaciones infecciosas de donde fueron recuperadas.
Materiales y métodosSe estudiaron 166 aislados de especies de Actinomyces/Actinotignum provenientes de especímenes clínicos de pacientes atendidos en un hospital universitario en el período 2012-2019. Las muestras fueron cultivadas siguiendo lo indicado en los manuales de procedimiento de nuestra institución. Todos los aislados fueron recuperados de los medios de cultivo en aerobiosis: agar sangre Columbia o agar chocolate, incubados en atmósfera con 5% de CO2 a 35°C por tiempo prolongado, de cinco a siete días. Luego, estos aislados fueron conservados a -70°C en caldo cerebro corazón (BHI) (Britania, Argentina), suplementado con glicerol al 20% hasta el momento de su estudio. Posteriormente fueron subcultivados en agar sangre ovina al 5% (bioMérieux, Marcy-l’Étoile, Francia) e incubados por 48-72 h en atmósfera con un 5% de CO2 a 35°C.
Identificación por espectrometría de masasLa identificación por espectrometría de masas, matrix-assisted laser desorption ionization-time of flight mass spectrometry (MALDI-TOF MS) (Bruker, Becton Dickinson), se llevó a cabo con el espectrómetro Microflex MALDI-TOF MS y empleando el software FlexControl (Bruker Daltonics, Bremen, Alemania). Para ello, se depositó una colonia del aislado seguido de la extracción con fórmico 70% (técnica «on spot») y se dejó secar a temperatura ambiente. Las muestras fueron fijadas con 1 μl de ácido α-ciano-4-hidroxicinámico hasta su cristalización a temperatura ambiente. Cuando dicha extracción no condujo a una identificación confiable, se siguió un protocolo de extracción con etanol 70% y ácido fórmico 70%, y luego de centrifugar 2 min a 13.000 rpm, se agregó acetonitrilo. Se tomó 1 μl de sobrenadante como inóculo sobre la placa MALDI-TOF. Los positrones fueron extruidos a una aceleración de 20 kV en modo lineal. Cada espectro fue la suma de los iones obtenidos a partir de 240 disparos de láser realizados de forma automática en las diferentes regiones del mismo pocillo. Los espectros se analizaron en un rango de m/z de 2.000 a 20.000. Los aislados se identificaron mediante el software MALDI Biotyper 3.1. Este software comparó el espectro de masas de los aislados probados con el espectro incluido en la base de datos de referencia MBT-8468 MSP (que contiene 158 y nueve perfiles de espectros principales de especies de Actinomyces y Actinotignum, respectivamente) (Bruker Daltonics, Alemania). Todas las especies de Actinomyces/Actinotignum incluidas en este trabajo se encuentran depositadas en dicha base de datos. A partir de estudios realizados anteriormente por nuestro grupo de trabajo, y por otros autores, se consideró un score ≥ 1,5 y < 1,7 para la identificación a nivel de género y ≥ 1,7 para la identificación a nivel de especie. Scores < 1,5 fueron considerados como no identificados1,5. Se utilizó el criterio de validez de interpretación entre la primera especie y la siguiente distinta de los diez primeros resultados con una divergencia del 10%26.
Identificación molecularPara aquellos aislados en los que no se pudo arribar a la identificación por espectrometría de masas MALDI-TOF MS a nivel de especie (score < 1,7) o género (score < 1,5), o frente a un porcentaje de divergencia entre especies < 10%, se realizó la identificación molecular mediante ampliación y secuenciación del gen que codifica la subunidad 16S del ARNr34. Los criterios utilizados para arribar a la identificación de especie fueron definidos utilizando como punto de corte un alto grado de identidad, es decir, ≥ 99% al comparar la identidad del producto de secuenciación obtenido (1,3-1,5 kb) con las especies depositadas en el GenBank7. Sin embargo, dada la escasa diversidad de pares de bases del citado gen que se describe entre ciertas especies estrechamente relacionadas, como Actinomyces naeslundii, Actinomyces viscosus y Actinomyces oris, o entre Actinotignum schaalii y Actinotignum sanguinis, por ejemplo, se consideró correcta la identificación a nivel de grupo cuando las secuencias de genes coincidían con alguna de las especies del grupo o dupla23. Es oportuno destacar que las especies A. naeslundii y A. viscosus comprenden un grupo heterogéneo que incluye A. naeslundii sensu stricto, A. oris, Actinomyces johnsonii y A. viscosus8.
Análisis estadísticoPara determinar la dependencia entre las variables localización del sitio de infección y especies bacterianas encontradas, se realizó la prueba χ2 de Pearson con los valores p simulados por prueba de Monte Carlo. El software utilizado fue RStudio, versión 1.2.5033, 2009-2019 RStudio, Inc. (R versión 3.6.2)24. Cuando las variables resultaron dependientes, posteriormente se calcularon los residuos de Pearson –a los que se denominó frecuencias diferenciales (FD)–; estos miden el alejamiento de cada una de las frecuencias particulares respecto del valor teórico de frecuencia homogénea (misma frecuencia en todos los sitios para cada especie de bacteria). Las unidades de los residuos de Pearson están en desvíos estándar, con lo que un residuo mayor que 2 (frecuencia diferencialmente elevada), o menor que -2 (frecuencia diferencialmente baja), representa un alejamiento de la independencia (homogeneidad de frecuencias), con un nivel de significación del 95%.
ResultadosEntre los 166 aislados de Actinomyces/Actinotignum, se identificaron en total 13 especies (o duplas), de las cuales 12 ya habían sido descriptas en infecciones humanas; solo Actinomyces canis no ha sido reportada hasta el momento en asociación con este tipo de infecciones. Esta última especie fue identificada por MALDI-TOF MS y confirmada por amplificación y secuenciación del gen que codifica la subunidad 16S del ARNr.
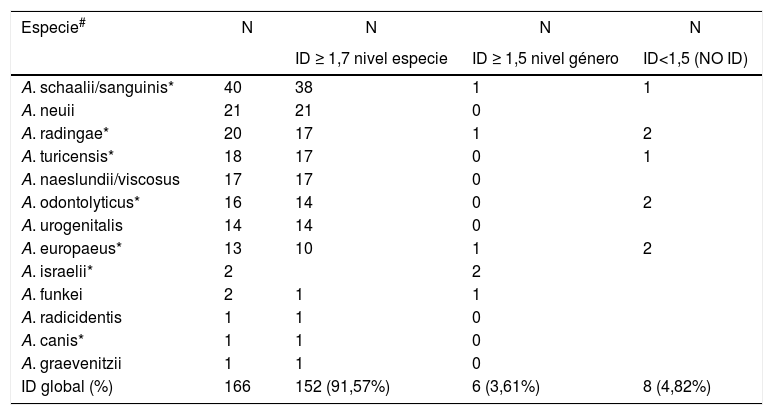
Mediante la técnica de MALDI-TOF MS se identificó el 91,57% de los aislados (152/166), a nivel de especie utilizando un score ≥ 1,7; el 3,61% (6/166) de los aislados se identificaron solo a nivel de género utilizando un score ≥ 1,5 (tabla 1). Dicho método no produjo resultados de identificación confiable en el 4,82% (8/166) de los aislados (dos Actinomyces radingae, dos Actinomyces odontolyticus, dos Actinomyceseuropaeus, un Actinomyces turicensis y un Actinotignum schaalii/sanguinis) (tabla 1). El porcentaje de identificación correcta a nivel de especie utilizando el score establecido por el fabricante (≥ 2,0) fue del 50% (83/166); con el score aquí propuesto fue del 91,57% (152/166)1,5 (datos no mostrados). Los aislados que no fueron identificados por el método de extracción directa on spot tampoco brindaron identificación correcta utilizando el método de extracción fórmico-etanólica.
Identificación de especies de Actinomyces/Actinotignum por MALDI-TOF MS
| Especie# | N | N | N | N |
|---|---|---|---|---|
| ID ≥ 1,7 nivel especie | ID ≥ 1,5 nivel género | ID<1,5 (NO ID) | ||
| A. schaalii/sanguinis* | 40 | 38 | 1 | 1 |
| A. neuii | 21 | 21 | 0 | |
| A. radingae* | 20 | 17 | 1 | 2 |
| A. turicensis* | 18 | 17 | 0 | 1 |
| A. naeslundii/viscosus | 17 | 17 | 0 | |
| A. odontolyticus* | 16 | 14 | 0 | 2 |
| A. urogenitalis | 14 | 14 | 0 | |
| A. europaeus* | 13 | 10 | 1 | 2 |
| A. israelii* | 2 | 2 | ||
| A. funkei | 2 | 1 | 1 | |
| A. radicidentis | 1 | 1 | 0 | |
| A. canis* | 1 | 1 | 0 | |
| A. graevenitzii | 1 | 1 | 0 | |
| ID global (%) | 166 | 152 (91,57%) | 6 (3,61%) | 8 (4,82%) |
# Con la excepción de Actinotignum schaalii/sanguinis, el resto de las especies pertenece al género Actinomyces.
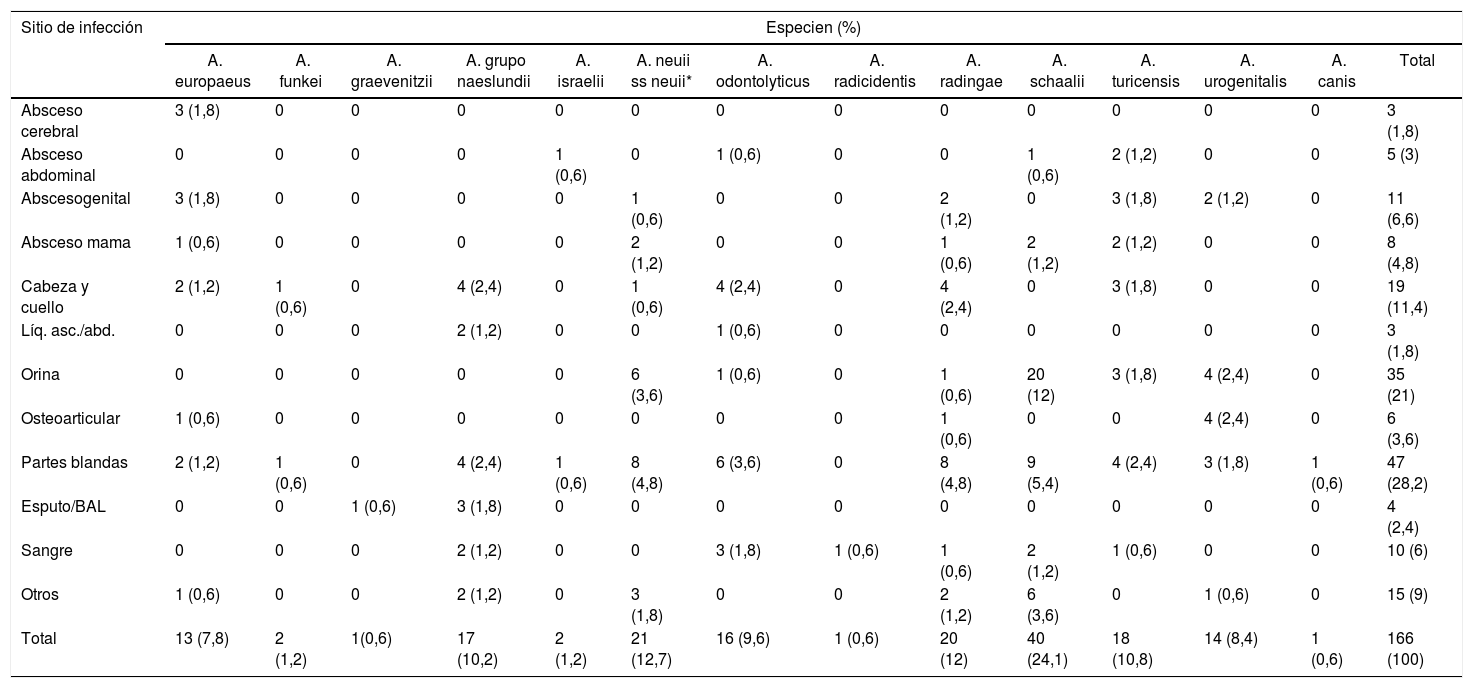
Las especies de Actinomyces/Actinotignum halladas y la fuente de origen del sitio primario de infección se detallan en la tabla 2. El número de aislados en cultivo monomicrobiano fue de 90 (54,2%), y en cultivo polimicrobiano 36 (21,7%).
Distribución de las especies de Actinomyces/Actinotignum por sitio de infección
| Sitio de infección | Especien (%) | |||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| A. europaeus | A. funkei | A. graevenitzii | A. grupo naeslundii | A. israelii | A. neuii ss neuii* | A. odontolyticus | A. radicidentis | A. radingae | A. schaalii | A. turicensis | A. urogenitalis | A. canis | Total | |
| Absceso cerebral | 3 (1,8) | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 (1,8) |
| Absceso abdominal | 0 | 0 | 0 | 0 | 1 (0,6) | 0 | 1 (0,6) | 0 | 0 | 1 (0,6) | 2 (1,2) | 0 | 0 | 5 (3) |
| Abscesogenital | 3 (1,8) | 0 | 0 | 0 | 0 | 1 (0,6) | 0 | 0 | 2 (1,2) | 0 | 3 (1,8) | 2 (1,2) | 0 | 11 (6,6) |
| Absceso mama | 1 (0,6) | 0 | 0 | 0 | 0 | 2 (1,2) | 0 | 0 | 1 (0,6) | 2 (1,2) | 2 (1,2) | 0 | 0 | 8 (4,8) |
| Cabeza y cuello | 2 (1,2) | 1 (0,6) | 0 | 4 (2,4) | 0 | 1 (0,6) | 4 (2,4) | 0 | 4 (2,4) | 0 | 3 (1,8) | 0 | 0 | 19 (11,4) |
| Líq. asc./abd. | 0 | 0 | 0 | 2 (1,2) | 0 | 0 | 1 (0,6) | 0 | 0 | 0 | 0 | 0 | 0 | 3 (1,8) |
| Orina | 0 | 0 | 0 | 0 | 0 | 6 (3,6) | 1 (0,6) | 0 | 1 (0,6) | 20 (12) | 3 (1,8) | 4 (2,4) | 0 | 35 (21) |
| Osteoarticular | 1 (0,6) | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 (0,6) | 0 | 0 | 4 (2,4) | 0 | 6 (3,6) |
| Partes blandas | 2 (1,2) | 1 (0,6) | 0 | 4 (2,4) | 1 (0,6) | 8 (4,8) | 6 (3,6) | 0 | 8 (4,8) | 9 (5,4) | 4 (2,4) | 3 (1,8) | 1 (0,6) | 47 (28,2) |
| Esputo/BAL | 0 | 0 | 1 (0,6) | 3 (1,8) | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 4 (2,4) |
| Sangre | 0 | 0 | 0 | 2 (1,2) | 0 | 0 | 3 (1,8) | 1 (0,6) | 1 (0,6) | 2 (1,2) | 1 (0,6) | 0 | 0 | 10 (6) |
| Otros | 1 (0,6) | 0 | 0 | 2 (1,2) | 0 | 3 (1,8) | 0 | 0 | 2 (1,2) | 6 (3,6) | 0 | 1 (0,6) | 0 | 15 (9) |
| Total | 13 (7,8) | 2 (1,2) | 1(0,6) | 17 (10,2) | 2 (1,2) | 21 (12,7) | 16 (9,6) | 1 (0,6) | 20 (12) | 40 (24,1) | 18 (10,8) | 14 (8,4) | 1 (0,6) | 166 (100) |
# Con la excepción de Actinotignum schaalii/sanguinis, el resto de las especies pertenece al género Actinomyces.
Prueba χ2 de Pearson, con valor p simulado (basado en 2.000 repeticiones): χ2 = 295,6, valor p = 0.01299.
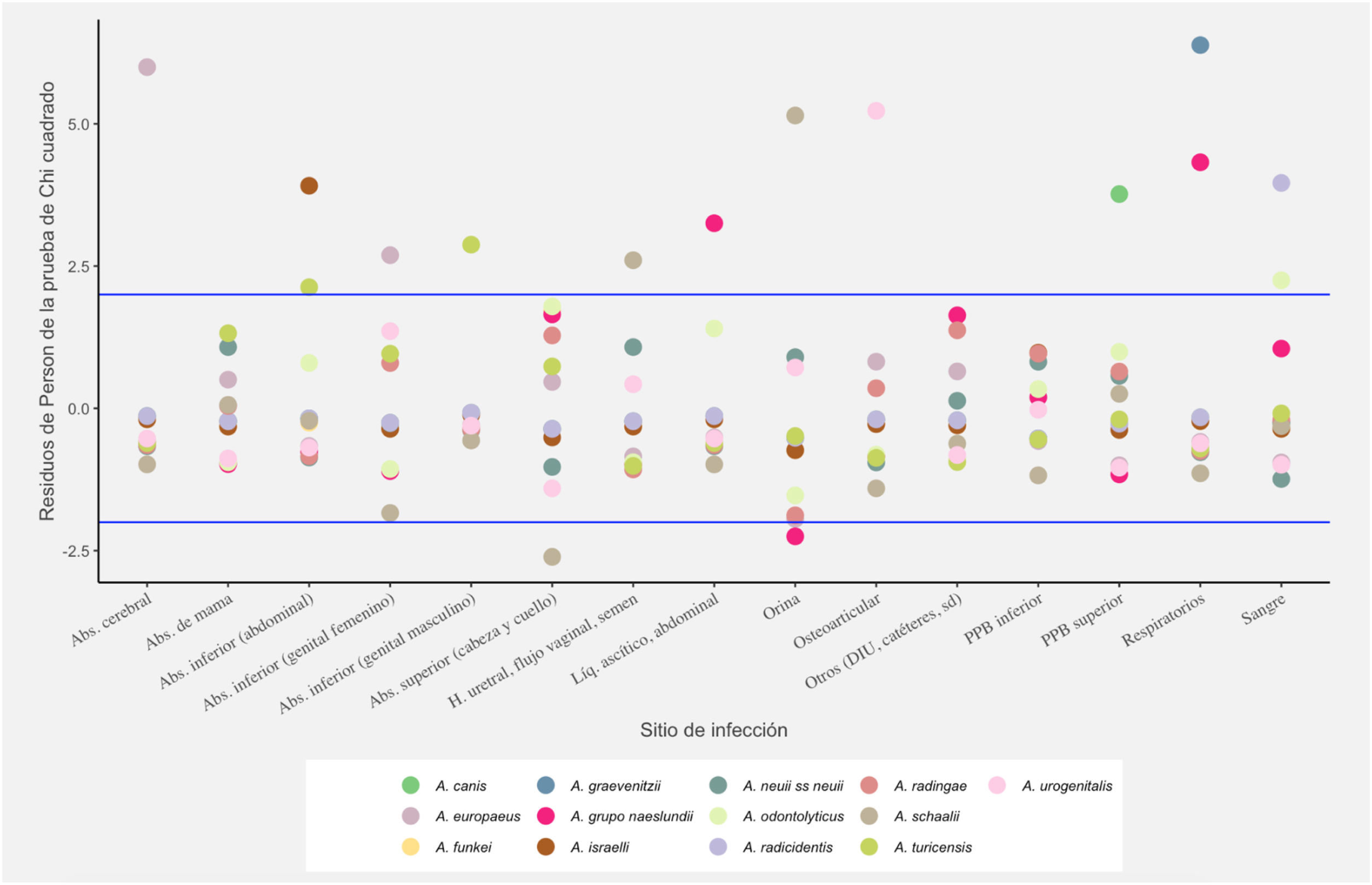
El análisis estadístico mostró una FD elevada (> 2) para las siguientes asociaciones localización/especie: orina/A. schaalii/sanguinis; abscesos cerebrales/A. europaeus; muestras osteoarticulares/A. urogenitalis; abscesos abdominales/A. turicensis y muestras respiratorias/A. naeslundii/viscosus. Tanto en abscesos de cabeza-cuello como de partes blandas y en abscesos de mama, no hubo una especie con frecuencia diferencial elevada. A. europaeus mostró una frecuencia diferencial elevada, pero no significativa, de identificación no confiable, al igual que A. odontolyticus y A. radingae. En el eje «x» de la fig. 1, se muestran los distintos sitios de infección y en el eje «y», el valor de los residuos Pearson (FD); los valores de corte en 2 y -2 se muestran en líneas azules. Las especies se encuentran codificadas por círculos de distintos colores, los cuales se muestran en la leyenda de la fig. 1.
DiscusiónEste trabajo aborda la identificación de una extensa colección de bacilos grampositivos del género Actinomyces y géneros relacionados, como Actinotignum, y documenta el nicho que diferentes especies ocupan y su frecuencia en ciertas infecciones humanas en Argentina.
En otras publicaciones realizadas por nuestro grupo de trabajo, en colaboración con el Hospital Necker-Enfants maladies, de París, el porcentaje de identificación de 71 aislados de Actinomyces y géneros relacionados fue del 92,96 y 95,77%, a nivel de especie y género, respectivamente. Similares resultados se obtuvieron en este trabajo, en el cual, sobre un total de 166 aislados, se identificaron correctamente el 91,57% de los aislados de Actinomyces/Actinotignum a nivel de especie y el 95,18% a nivel de género. Ferrand et al. mostraron menor porcentaje de identificación correcto, 58,2 y 85,4% a nivel especie y género, respectivamente; y Fong et al., sobre 77 aislados de Actinomyces spp., informaron un 57,1 y un 88,3% a nivel de especie y género, respectivamente10,11. Estas diferencias pueden explicarse con el hecho de que estos autores utilizaron los scores sugeridos por el fabricante, en lugar de los scores más bajos sugeridos en la literatura1,5.
Las especies de Actinomyces y géneros relacionados son parte de la microbiota normal de mucosas; por tal motivo, la mayoría de las infecciones en las que se involucran son endógenas (monomicrobianas o polimicrobianas), en donde ocurre disrupción de mucosas del hospedero6,33.
En un trabajo publicado por Könönen y Wade (2015) se detalla la distribución de las especies de Actinomyces y géneros relacionados en nichos específicos del cuerpo humano. Conocer esta distribución puede contribuir a afrontar el desafío que supone determinar la significancia clínica del aislamiento de estos microorganismos17.
En este trabajo, la mayoría de las infecciones por Actinotignum schaalii estuvieron relacionadas con infección del tracto urinario (ITU) y correspondieron a infecciones monomicrobianas. Si bien las infecciones urinarias por esta especie son poco frecuentes (0,1-0,2% de la prevalencia de la ITU en nuestro hospital), consideramos importante el hallazgo de este microorganismo, especialmente en pacientes añosos o con patologías urinarias de base. Además, en las ITU por A. schaalii, se debe considerar la posibilidad de falla terapéutica con tratamientos empíricos con quinolonas o trimetoprima-sulfametoxazol, dado que a menudo este microorganismo presenta resistencia a ambos antimicrobianos. A. schaalii fue descripto en 1997 y numerosas publicaciones, principalmente en países europeos, hacen referencia a su rol en la infección urinaria, cistitis o pielonefritis, con o sin bacteriemia, lo que sugiere, además, su rol invasivo3,4,18,19,25. La mayoría de los pacientes incluidos en este trabajo presentaron algún factor urinario predisponente. Si bien la mayoría de los pacientes eran añosos, un aislamiento correspondió a un paciente de 17 años con pielonefritis, con falla renal y hemodiálisis como factor predisponente3.
En los tres casos de abscesos cerebrales, la especie aislada fue A. europaeus; esto ocurrió en pacientes con focos otogénicos como otitis media crónica (OMC) y colesteatoma y, en todos los casos, el cultivo fue polimicrobiano, asociado con bacterias anaerobias, tales como Prevotella intermedia, Prevotella bergensis, Bacteroides thetaiotaomicron y Slackia exigua. En este trabajo se observó una FD elevada para la asociación absceso cerebral/A. europaeus. No existen hasta el momento publicaciones que describan el hallazgo de dicha especie en esta localización infecciosa. En 1997, Funke et al. describen la especie A. europaeus aislada de muestras de abscesos, pero ninguno en localización cerebral: abscesos perianales, suprapúbicos y de mama12. En este trabajo, solo en una paciente se aisló este microorganismo en absceso de mama en una mastitis no puerperal sin factor predisponente, por lo que la FD no mostró asociación entre A. europaeus y absceso de mama. Sin embargo, esta localización fue descripta por varios autores21,30,35.
A. urogenitalis fue originalmente descripto en muestras genitourinarias, de allí su nombre22. En este trabajo se incluyeron casos de pacientes con sedimento urinario patológico (> 5 leucocitos/cpo) y síntomas de disuria, con aislamiento monomicrobiano y un recuento significativo (> 10.000 UFC/mL) de A. urogenitalis en cuatro muestras de orina. Sin embargo, lo que resultó de particular interés fue el hallazgo de esta especie en infecciones de piel y partes blandas (PPB) y hueso, en algunos casos, en pacientes diabéticos en cultivo polimicrobiano, y también en pacientes con prótesis osteoarticular. La FD fue elevada para esta asociación. Excepto por nuestro grupo de trabajo que ha documentado la sensibilidad de aislados de A. urogenitalis y una inusual resistencia a clindamicina en aislamientos de muestras de PPB y hueso, hasta el momento no hay otras publicaciones que hagan referencia a dicha localización en relación con esta especie2.
En coincidencia con lo que documenta la literatura, A. turicensis también fue una de las especies prevalentes en este estudio9,27,32. Además de la localización urogenital, esta especie se presentó en casos de abscesos abdominales. La localización pélvico-abdominal suele ser una de las más frecuentes en los casos de actinomicosis clásica, además de la localización cervicofacial y torácica. Sin embargo, ni en relación con esta especie ni con ninguna otra de las halladas en este estudio se logró conocer si estos casos correspondían a este tipo de presentación clásica. La importancia del aislamiento de una especie de Actinomyces puede ser incierta, en ausencia del hallazgo anatomopatológico de la actinomicosis; sin embargo, otros autores han demostrado una asociación entre sitio de aislamiento e infección para estas especies6.
El grupo A. naeslundii/viscosus es un grupo heterogéneo cuyas especies muestran más del 99% de similitud en el gen que codifica la subunidad 16S del ARNr y, por lo tanto, no pueden diferenciarse por comparación de estas secuencias; sí, en cambio, comparando secuencias parciales de los genes housekeepingatpA y metG15. Si bien los factores de virulencia de los miembros de Actinomyces son poco conocidos, especies como A. oris y A. naeslundii juegan un importante rol en la formación de la placa dental (biofilm), a través de sus fimbrias, las que se unen a las proteínas ricas en prolina de la saliva adsorbidas a la superficie dental13,20. En nuestro trabajo, los aislados de A. naeslundii/viscosus de cabeza y cuello estuvieron relacionados con foco odontógeno. A diferencia de otros autores que afirman que el 25% de las infecciones odontogénicas pueden estar representadas por actinomicosis orocervicofacial29, nosotros no pudimos establecer que se trataran de actinomicosis orales clásicas. Algunos autores han identificado a A. naeslundii como causa de infección periprotésica de cadera36. En nuestro caso, la FD resultó significativa en muestras respiratorias (esputos y BAL), en pacientes inmunodeprimidos y en aislamiento polimicrobiano.
Asimismo, este estudio mostró un solo caso de infección por Actinomyces canis, cuyo foco correspondió a PPB, generado por mordedura de perro en una paciente de sexo femenino con infección por HIV bajo tratamiento antirretroviral. La infección fue de tipo polimicrobiana, acompañada por Candida spp. y bacterias anaerobias. Esta especie fue descripta por Hoyles et al. en relación con patología dentaria canina16. Fong et al. mencionan un solo caso de aislamiento de Actinomyces en humano de un conjunto de 77 aislados, sin mencionar el rol patogénico de esta reciente especie en humanos11.
ConclusiónLa espectrometría de masas MALDI-TOF MS es una herramienta rápida, poco laboriosa y confiable para la identificación de especies de Actinomyces/Actinotignum.
En este trabajo, el elevado porcentaje de aislados de especies de Actinomyces en forma monomicrobiana sugiere su relevancia clínica. Los datos aquí presentados mostraron algunos sitios más frecuentes en términos estadísticos para ciertas especies: A. schaalii/A. sanguinis en orina; A. europaeus en abscesos cerebrales, A. turicensis en abscesos abdominales, A. urogenitalis en muestras osteoarticulares y A. naeslundii/viscosus en muestras respiratorias. El papel patogénico de Actinomyces spp. podrá determinarse con mayor claridad en la medida que estos microorganismos puedan ser reconocidos gracias al cultivo prolongado y a los avances de la tecnología de identificación facilitada por MALDI-TOF MS.
FinanciaciónEl presente trabajo ha sido financiado con fondos del proyecto UBACYT 2018, modalidad I: código 20020170100109BA.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.