Escherichia coli diarreogénica abarca un grupo heterogéneo de patotipos o variantes patogénicas que comparten características fenotípicas con marcadas diferencias en genes de virulencia, sitios de colonización, patogenia, presentación clínica y epidemiología de la infección. Los patotipos más estudiados son E.coli productora de toxina Shiga (STEC), E.coli enterotoxigénica (ETEC), E.coli enteropatogénica (EPEC), E.coli enteroagregativa (EAEC) y E.coli enteroinvasiva (EIEC). El objetivo del estudio fue caracterizar los aislamientos de E.coli diarreogénica provenientes de población pediátrica ambulatoria con diarrea, atendida en dos hospitales públicos de Buenos Aires, Argentina. Los patotipos de E.coli diarreogénica se investigaron mediante la amplificación de fragmentos de genes de virulencia característicos: intimina (eae), toxina termolábil (lt), toxinas termoestables (stp,sth), antígeno plasmídico de invasiónH (ipaH), activador transcripcional R (aggR) y toxinas Shiga (stx1, stx2). La subtipificación molecular de aislamientos se realizó mediante PFGE (XbaI). E.coli diarreogénica fue detectada en el 14% (84/601) de los casos. El patotipo EAEC fue prevalente, mientras que ETEC, STEC, EPEC y EIEC fueron hallados en menor proporción. Los aislamientos de EAEC presentaron un alto grado de diversidad genética. Todos los patotipos fueron hallados en niños menores de 5años, mientras que solamente EAEC, EIEC y ETEC fueron detectados en población de mayor edad. Futuros estudios que incluyan la caracterización de aislamientos a partir de un mayor número de genes y población de otras áreas geográficas serán necesarios para determinar la relevancia de E.coli diarreogénica en Argentina.
Diarrheagenic Escherichia coli comprises a heterogeneous group of pathotypes or pathogenic variants that share phenotypic characteristics with marked differences in virulence genes, colonization sites, pathogenesis, clinical presentation, and epidemiology of infection. The most studied pathotypes are Shiga toxin-producing E.coli (STEC), enterotoxigenic E.coli (ETEC), enteropathogenic E.coli (EPEC), enteroaggregative E.coli (EAEC), and enteroinvasive E.coli (EIEC). The objective of the study was to characterize the isolates of diarrheagenic E.coli from an outpatient pediatric population with diarrhea attended in two public hospitals from Buenos Aires, Argentina. Diarrheagenic E.coli pathotypes were investigated by amplifying characteristic virulence gene fragments: intimin (eae), heat-labile toxin (lt), heat-stable toxins (stp, sth), invasion plasmid antigen H (ipaH), transcriptional activator R (aggR) and Shiga toxins (stx1, stx2). Molecular subtyping of isolates was performed using PFGE (XbaI). Diarrheagenic E.coli was detected in 14% (84/601) of cases. The EAEC pathotype was prevalent, while ETEC, STEC, EPEC and EIEC were found in a lower proportion. EAEC isolates exhibited a high degree of genetic diversity. All pathotypes were found in children under 5years of age, while only EAEC, EIEC and ETEC were detected in the older population. Future studies that include the characterization of isolates from a greater number of genes and populations from other geographical areas will be necessary to determine the relevance of diarrheagenic E.coli in Argentina.
Escherichia coli es una bacteria gram negativa que forma parte de la microbiota del tracto digestivo del humano y de animales de sangre caliente. Los patotipos o variantes patogénicas más estudiados incluyen E.coli enteropatogénica (EPEC), E.coli enterotoxigénica (ETEC), E.coli enteroagregativa (EAEC), E.coli productora de toxina Shiga (STEC) y E.coli enteroinvasiva (EIEC).
Los patotipos de E.coli asociados al tracto intestinal presentan marcadas diferencias en la fisiopatogenia, los factores de virulencia y la epidemiología de la infección. La adherencia es un paso crucial para la patogenia bacteriana. En EPEC, la unión íntima a los enterocitos produce una lesión característica denominada A/E (del inglés attaching and effacing) que conduce a la disminución de la superficie absortiva intestinal asociada al cuadro diarreico. La patogenia de ETEC comienza con la adherencia a los enterocitos mediante sus factores de colonización seguido de la producción de enterotoxinas (termolábil y/o termoestable) que conducen a una diarrea secretoria. El patotipo EAEC presenta una gran diversidad de factores de virulencia. Esta variante patogénica suele formar microcolonias que subsisten en el intestino por largos períodos dando cuadros de diarrea aguda y persistente. Por el contrario, el principal factor de virulencia de STEC es la toxina Shiga, cuyo mecanismo de acción es la inhibición de la síntesis proteica celular. Dicha toxina interviene en la destrucción de la mucosa colónica dando cuadros de diarrea, colitis hemorrágica y síndrome urémico hemolítico. Por último, EIEC presenta un mecanismo de patogenia asociado a la invasión intestinal. La bacteria ingresa por las célulasM de la mucosa colónica, luego invade a los enterocitos, se multiplica en el citoplasma y produce cuadros de diarrea disentérica3,5,8,14,16,17. En las últimas décadas se han incorporado nuevos patotipos, como E.coli de adherencia difusa (DAEC) y E.coli adherente invasiva (AIEC). Sin embargo, el alcance epidemiológico de estas variantes patogénicas no está completamente esclarecido8,24.
La frecuencia de infección con E.coli diarreogénica (DEC) en la población con diarrea ha presentado amplias variaciones. En países de Latinoamérica como Chile, México, Bolivia y Brasil reportaron una frecuencia de infección entre el 10% y el 25%, mientras que Uruguay, Perú, Venezuela y Paraguay informaron valores más elevados5,6,11,12,19,22,33,35.
En Argentina, la mayoría de los estudios han sido realizados en STEC, un patógeno de gran relevancia clínica y epidemiológica para la salud pública. Sin embargo, los otros patotipos DEC no se investigan de manera sistemática en los pacientes pediátricos con diarrea. Por lo tanto, el objetivo del estudio fue caracterizar los aislamientos de E.coli diarreogénica provenientes de población pediátrica ambulatoria con diarrea, atendida en dos hospitales públicos de Buenos Aires, Argentina.
Materiales y métodosEl diseño del estudio fue observacional, descriptivo, prospectivo y de corte transversal. La población accesible estuvo constituida por pacientes menores de 15años que se atendieron por un cuadro gastrointestinal en el Hospital de Niños Debilio Blanco Villegas de Tandil y en el Hospital de Niños Sor María Ludovica de La Plata, provincia de Buenos Aires, Argentina. La selección se realizó aplicando un procedimiento de colección por casos consecutivos durante 2017 y 2018. La población con antecedentes de enfermedad intestinal inflamatoria, inmunosuprimidos, con enfermedades oncohematológicas o con diarrea crónica fueron excluidos del estudio. El protocolo de trabajo fue aprobado por el Comité de Bioética de la Investigación de la Facultad de Ciencias Médicas de la Universidad Nacional de La Plata. Los investigadores condujeron el estudio de acuerdo con las normas de buenas prácticas clínicas, la declaración de Helsinki con todas sus enmiendas y de conformidad con la normativa vigente aplicable. La confidencialidad de la investigación fue observada mediante la asignación de un código alfanumérico interno que permitió la disociación permanente de la información personal de los pacientes.
Los patotipos de E.coli diarreogénica (DEC) se investigaron mediante la amplificación de genes de virulencia característicos con dos PCR múltiples20. La mPCR1 amplificó los codificantes de intimina (eae), la toxina termolábil LT (lt), las toxinas termoestables STP (stp) y STH (sth) y 16S. La mPCR2 amplificó los codificantes de antígeno plasmídico de invasión H (ipaH), el activador transcripcional R (aggR), las toxinas Shiga 1 (stx1) y Shiga 2 (stx2) y 16S7,20. La detección molecular fue realizada aplicando una estrategia diagnóstica en etapas. La primera etapa consistió en la realización de las dos PCR múltiples con ADN proveniente del cultivo confluente. En caso de que se hubiera observado amplificación de algún gen, la segunda etapa consistió en la identificación de la colonia positiva (con genes de virulencia). Para tal efecto, el ADN de 10 a 20 colonias aisladas se utilizó como molde para las PCR individuales. La colonia que presentó amplificación fue identificada por métodos fenotípicos (VITEK®2, bioMérieux) para confirmar la especie bacteriana20. Los patotipos fueron identificados según los genes de virulencia hallados: EPEC (presencia de eae, ausencia de stx1 y stx2), ETEC (presencia de lt, stp y/o sth), EIEC (presencia de ipaH), STEC (presencia de stx1 y/o stx2, presencia o ausencia de eae) y EAEC (presencia de aggR)20.
La recolección de las heces, el cultivo, la extracción de ADN y la amplificación molecular se llevaron a cabo según metodología previamente descrita20. Brevemente, la materia fecal fue sembrada en agar MacConkey e incubada a 37°C por 24h. Las colonias características se resuspendieron en buffer TE con Tritón X-100, se lisaron a 100°C y se centrifugaron a 10.000rpm20. El ADN se conservó a −20°C. Las concentraciones en la mezcla de reacción fueron: buffer 1X y dNTP, 0,1mM; Taq pol, 0,03U/μl y ADN, 1-5μl. La concentración de MgCl2 fue 1,5mM para mPCR1 y 0,75mM en la mPCR2. Las concentraciones de primers fueron 0,2pmol/μl (SK1, SK2, STh-f, STh-r, STp-f y STp-r); 0,25pmol/μl (LT-R, LT-F, IpaH8 e IpaH15); 1pmol/μl (aggRks1 y aggRkas2); 1,6pmol/μl (stx1-F y stx1-R); 0,4pmol/μl (stx2-F y stx2-R) y 0,125pmol/μl (16S-f y 16S-r)20 (Anexo, tabla S1). El protocolo de ciclado de mPCR1 consistió en 94°C 5min, 30 ciclos de 94°C 30 s, 56°C 1min y 72°C 1min. El protocolo de mPCR2 fue 94°C 5min, 30 ciclos de 94°C 30 s, 57°C 1min y 72°C 30s. La extensión final para ambos protocolos fue 72°C 2min20. En cada amplificación se utilizaron cepas control: E.coli 2348/69 (eae), E.coli KNH-172 (lt, stp), E.coli O126-53 (sth), E.coli C-481 (ipaH), E.coli 17-2 (aggR), E.coli EDL933 (stx1, stx2) y E.coli ATCC25922. Los productos de amplificación fueron detectados en geles de agarosa y los perfiles de bandas fueron documentados con UVP Doc-It® LS, LifeScience Software.
La subtipificación molecular de aislamientos se realizó mediante PFGE según metodología descrita23. Brevemente, el ADN contenido en bloques de agarosa fue digerido con 30U de la enzima de restricción XbaI (Thermo Scientific) a 37°C por 3h. Los perfiles moleculares obtenidos por XbaI-PFGE fueron documentados utilizando el sistema de adquisición de imágenes MaestroGen slider® imager (Maestrogen Inc., Nevada, EE.UU.). Las imágenes en formato TIFF fueron analizadas con el software BioNumerics versión 6.6 (Applied Maths, Sint-Martens-Latem, Bélgica). La relación entre los perfiles fue estimada mediante la proporción de bandas compartidas aplicando el coeficiente de similitud de Dice y generando dendrogramas basados en el método unweighted pair group method with arithmetic mean (UPGMA). Las cepas fueron agrupadas en clústeres cuando mostraron idéntico patrón XbaI-PFGE (100% de similitud).
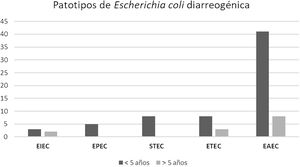
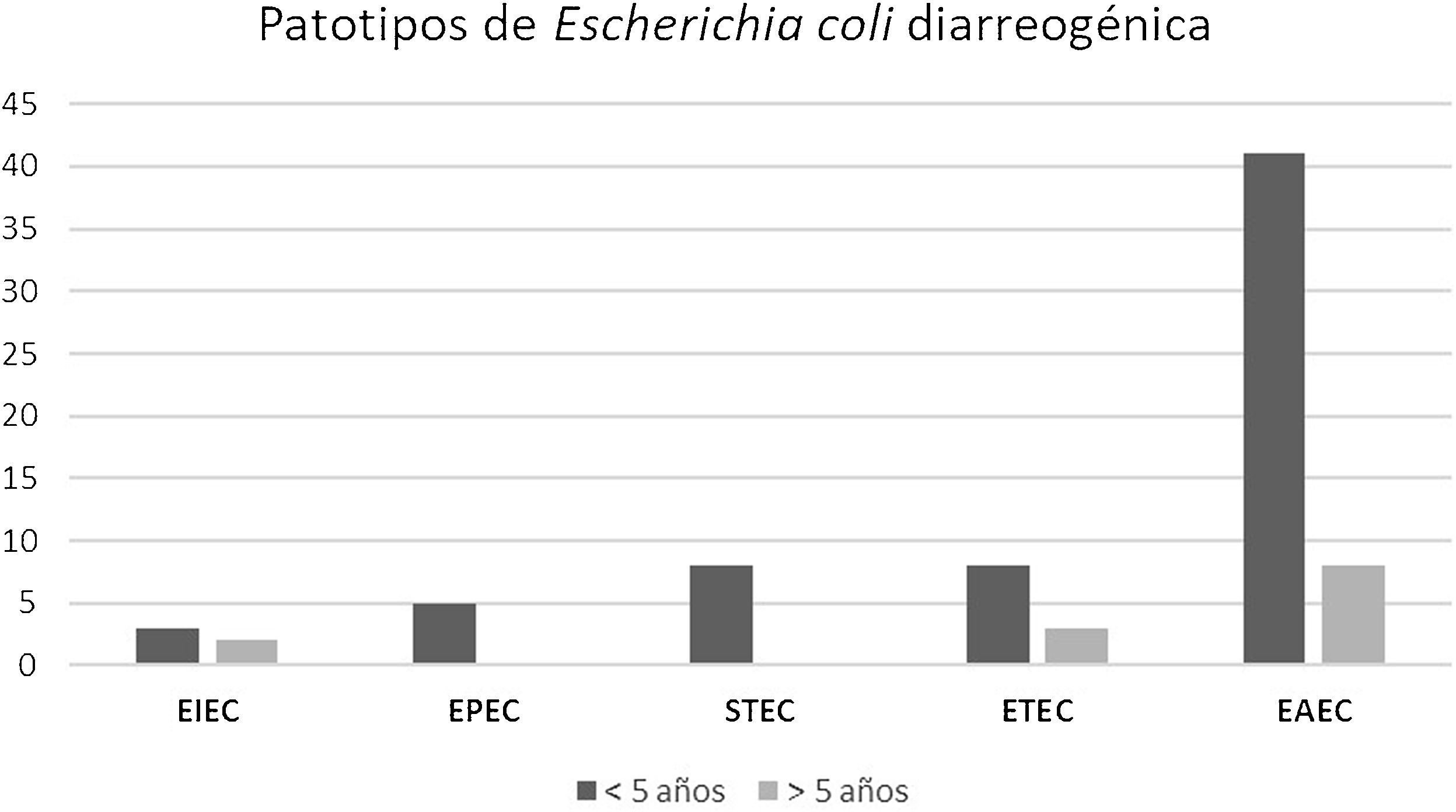
ResultadosEl grupo de estudio incluyó 601 pacientes con diarrea. La población menor de 5años representó el 76% de los casos. E.coli diarreogénica fue detectada en el 14% (84/601) (tabla 1). Las frecuencias de infección con EAEC, ETEC, STEC, EPEC y EIEC fueron del 8,7%, del 2,3%, del 1,3%, del 0,8% y del 0,8%, respectivamente. El patotipo hallado con mayor frecuencia fue EAEC. Las relaciones genéticas entre aislamientos de EAEC se muestran en la figura 1. Los genes de las toxinas halladas fueron stx2, lt, stp y sth. Todos los aislamientos STEC presentaron los genes eae y stx2 (100%, 8/8). En ETEC, el gen lt fue prevalente (85,7%, 12/14). La distribución de genes fue lt (9/14), lt+stp (3/14), stp (1/14) y sth (1/14) (figs. 2 y 3). Todos los patotipos fueron hallados en pacientes menores de 5años, mientras que solamente EAEC, EIEC y ETEC fueron detectados en pacientes de mayor edad (fig. 4).
Distribución de patotipos de Escherichia coli diarreogénica en pacientes con diarrea provenientes de La Plata y Tandil entre 2017 y 2018
| Patotipo | n | % |
|---|---|---|
| EAEC (E.coli enteroagregativa) | 52 | 61,9 |
| ETEC (E.coli enterotoxigénica) | 14 | 16,8 |
| STEC (E.coli productora de toxina Shiga) | 8 | 9,5 |
| EPEC (E.coli enteropatogénica) | 5 | 5,9 |
| EIEC (E.coli enteroinvasiva) | 5 | 5,9 |
| Total | 84 | 100 |
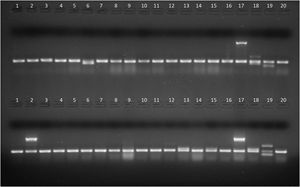
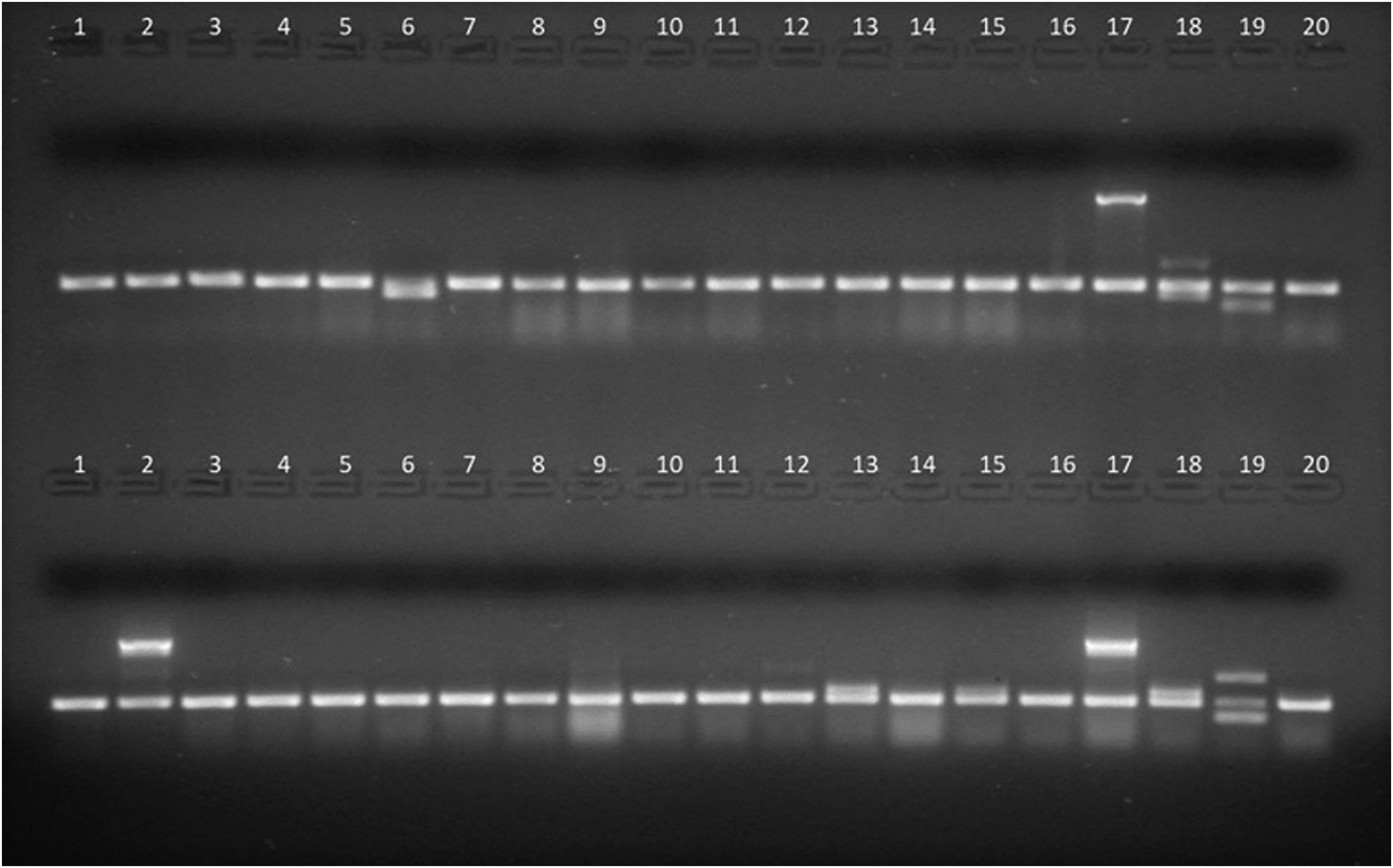
Productos de amplificación en gel de agarosa de aislamientos de E.coli intestinal (líneas 1 a 16) y cepas control (líneas 17 a 20). Arriba, PCR1. línea 6: gen stp, líneas 1 a 5 y 7 a 16: sin factores de virulencia, línea 17: gen eae (E.coli 2348/69), línea 18: genes lt y stp (E.coli KNH-172), línea 19: gen sth (E.coli O126-53), línea 20: gen 16S (E.coli ATCC25922, control de amplificación). Abajo, PCR2. Línea 2: gen ipaH; líneas 13 y 15: gen aggR; líneas 1, 3 a 12, 14 y 16: sin factores de virulencia; línea 17: gen ipaH (E.coli C-481); línea 18: gen aggR (E.coli 17-2); línea 19: genes stx1 y stx2 (E.coli EDL933); línea 20: gen 16S (E.coli ATCC 25922, control de amplificación).
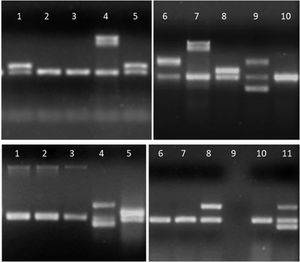
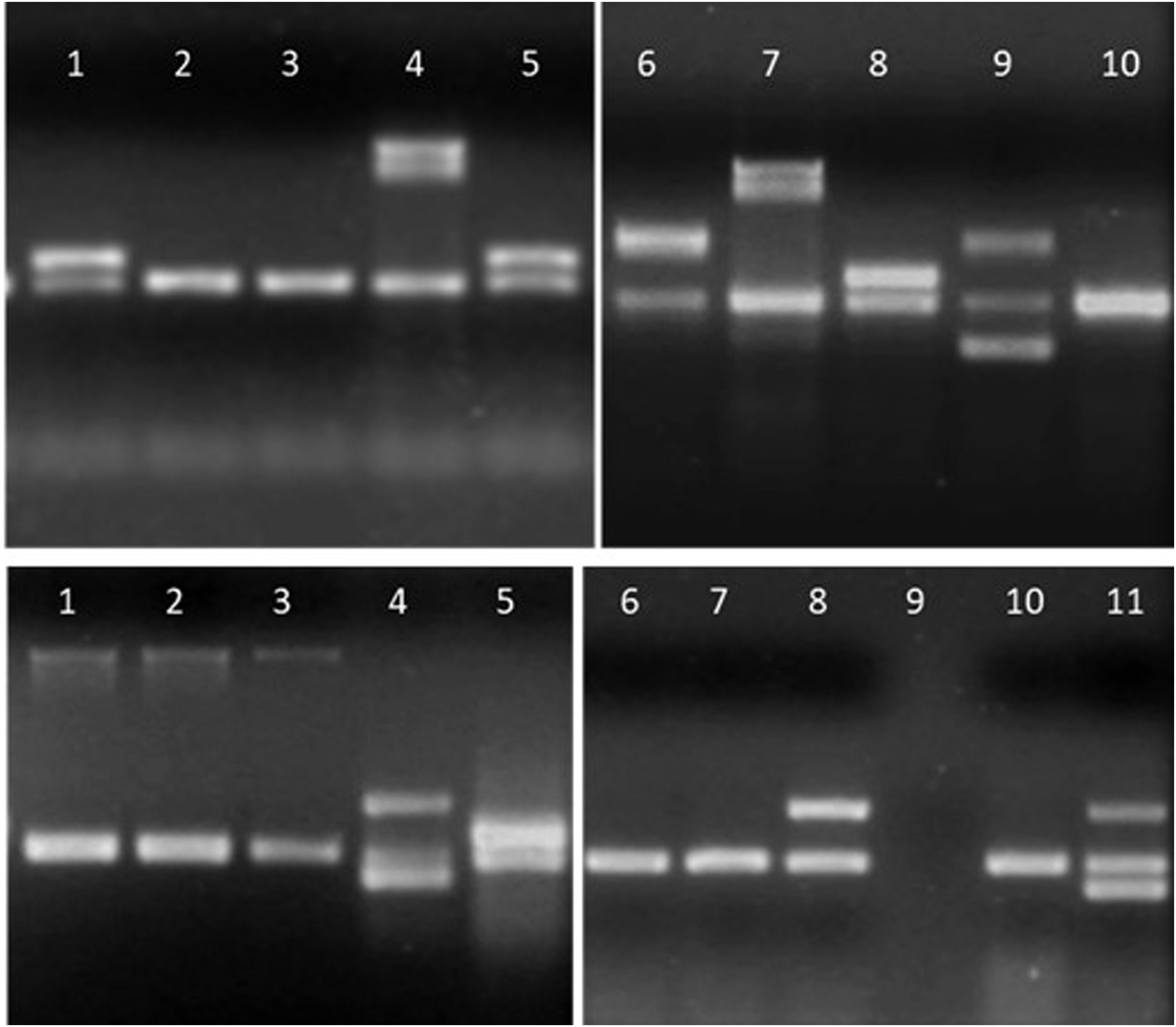
Detalle de los productos de amplificación en gel de agarosa de aislamientos de E.coli. Arriba, línea 1, 5 y 8: gen aggR; líneas 4 y 7: gen ipaH; línea 6: gen stx2; línea 9: genes stx1 y stx2; líneas 2, 3 y 10: sin factores de virulencia. Abajo, líneas 1 a 3: gen eae; líneas 4 y 11: genes lt y stp; línea 5: gen aggR; línea 8: gen lt; línea 9: sin muestra; líneas 6, 7 y 10: sin factores de virulencia.
Distribución de patotipos de Escherichia coli diarreogénica según la edad de los pacientes. EAEC (E.coli enteroagregativa), ETEC (E.coli enterotoxigénica), STEC (E.coli productora de toxina Shiga), EPEC (E.coli enteropatogénica) y EIEC (E.coli enteroinvasiva) provenientes de La Plata y Tandil, entre 2017 y 2018.
Los resultados de este estudio han permitido caracterizar los aislamientos de E.coli diarreogénica provenientes de población pediátrica ambulatoria con diarrea, atendida en dos hospitales públicos de Buenos Aires, Argentina.
La frecuencia de infección entérica por DEC fue del 14%. Este valor está en concordancia con un estudio previo de nuestro grupo20. Resultados similares también han sido reportados en Chile, Perú, Venezuela, Costa Rica y Paraguay13,25,29,35,36. Por el contrario, otros estudios realizados en Argentina, Brasil, Bolivia y México señalaron frecuencias de infección por DEC mayores a las reportadas en este estudio4,6,10,12,18. La disparidad en los valores reportados en los estudios podría deberse a diferencias en el grupo etario investigado, en el diseño del estudio, en las características sociodemográficas de los participantes, en el sesgo en la selección de las muestras fecales o en los genes de virulencia amplificados20,23.
El principal factor de virulencia de STEC son las toxinas Shiga. En este estudio, la frecuencia de infección fue del 1,3% y todos los aislamientos presentaron el gen de la toxina STX2. Dicho predominio está en concordancia con lo reportado en otros estudios realizados en el país17,23,39. Frecuencias similares fueron reportadas en niños con diarrea de otros países, como Bangladesh, México, Perú, Kenia, Tailandia y Arabia Saudita6,15,16,22,30,31. Por el contrario, un estudio retrospectivo realizado en Argentina por Oderiz et al.23 reportó una frecuencia de infección con STEC del 4,8%. Sin embargo, dichos autores solamente estudiaron las heces de niños con cuadros de diarrea sanguinolenta. Estas diferencias en las tasas de infección con STEC pueden deberse a diversos factores, entre los que se encuentran el tiempo transcurrido entre el inicio de los síntomas y la recolección de la muestra fecal, la metodología de detección bacteriana, el uso de terapia antimicrobiana antes del diagnóstico etiológico certero, y otros17,20,23.
Los aislamientos ETEC mostraron un predominio del gen de la toxina termolábil. Este resultado está en concordancia con otros estudios de Argentina3,18,20,27. La prevalencia de LT es coincidente con lo reportado en otros estudios. Por ejemplo, Isidean et al.14 señalaron el predominio de dicha toxina en más de 17.000 cepas estudiadas. Asimismo, los aislamientos ETEC con toxina LT fueron prevalentes en estudios realizados en México y en Perú. Por el contrario, las cepas productoras de ST fueron predominantes en Bangladesh, Egipto, Colombia, España y Tanzania21,26,34,37.
En este estudio, el patotipo EAEC fue prevalente, mientras que los otros patotipos fueron hallados en una menor proporción. El predominio de EAEC está en concordancia con estudios realizados en Argentina, Bolivia, Brasil e India10,12,18,20,27,28. El patotipo EAEC ha sido reconocido como un patógeno emergente, tanto por su reciente descripción como por su relevancia en cuadros gastrointestinales agudos y persistentes. La prevalencia de EAEC podría deberse a sus características patogénicas, ya que dicha bacteria es capaz de adherirse a la pared intestinal, favorecer la producción de moco, liberar enterotoxinas, estimular la respuesta inflamatoria intestinal, evadir el sistema inmune y persistir en el intestino.
En este trabajo se evaluó la diversidad genética de los aislamientos de EAEC mediante el método de electroforesis en gel de campos pulsados (PFGE). Mediante dicha técnica se hallaron 23 patrones de bandas diferentes de EAEC, lo que demostró la circulación regional de aislamientos con un alto grado de diversidad genética. El PFGE permitió detectar dos pares de aislamientos con el 100% de similitud. Ambos pares de EAEC fueron recuperados dentro del mismo mes del año (enero de 2018 y septiembre de 2017); sin embargo, no se pudo establecer el nexo epidemiológico entre los portadores de dichos aislamientos debido a la ausencia de información personal, clínica o geográfica de los pacientes. Todos los patotipos fueron hallados en pacientes menores de 5años, mientras que solamente EAEC, EIEC y ETEC fueron detectados en población de mayor edad. Diversos autores han postulado que la menor frecuencia de infección a mayor edad podría estar relacionada con la maduración de la respuesta inmune, con la persistencia intestinal o con cierta protección inmune generada por las reinfecciones en la infancia1,2,9,21,28,32,37,38.
Esta investigación demostró la presencia de cinco patotipos diferentes de E.coli diarreogénica en los pacientes pediátricos con diarrea. La caracterización molecular de los aislamientos reveló que EAEC fue el patotipo prevalente y que presentó un alto grado de diversidad genética en la población estudiada. En relación con los genes de las toxinas de E.coli diarreogénica, se observó un marcado predominio de la toxina termolábil en los aislamientos ETEC y la preponderancia absoluta de la STX2 en los aislamientos STEC. La detección de las toxinas Shiga forma parte del protocolo para el diagnóstico etiológico de síndrome urémico hemolítico, mientras que la detección de los genes de las toxinas contribuye a la vigilancia epidemiológica. Teniendo en cuenta el enfoque de Una Salud, los estudios moleculares podrían aportar información sobre los subtipos de toxinas Shiga circulantes en humanos, en animales y en el ambiente. Los resultados de este estudio constituyen un aporte relevante para la medicina pediátrica, ya que aporta información inédita sobre la infección con E.coli diarreogénica en la población atendida en dos hospitales públicos de la provincia de Buenos Aires. Sin embargo, en este estudio solamente se realizó la subtipificación molecular de EAEC debido a la baja frecuencia de infección de los otros patotipos. Además, este trabajo fue realizado en la población de áreas urbanas y podría no representar exactamente las características genotípicas de E.coli diarreogénica en otras regiones del país.
En conclusión, E.coli diarreogénica fue detectada en el 14% de los casos. El patotipo EAEC fue prevalente, mientras que ETEC, STEC, EPEC y EIEC fueron hallados en menor proporción. Los aislamientos de EAEC presentaron un alto grado de diversidad genética. Todos los patotipos fueron hallados en niños menores de 5años, mientras que, solamente EAEC, EIEC y ETEC fueron detectados en población de mayor edad. En las últimas décadas se han hecho grandes avances en el diagnóstico de los agentes productores de diarrea. Sin embargo, la investigación de los patotipos diarreogénicos de E.coli por métodos moleculares no se realiza en forma rutinaria en la mayoría de los laboratorios de diagnóstico clínico. Futuros estudios que incluyan la caracterización de aislamientos a partir de un mayor número de genes y población de otras áreas geográficas serán necesarios para determinar la relevancia de E.coli diarreogénica en Argentina.
FinanciaciónEl presente trabajo ha sido parcialmente financiado por un subsidio a la investigación clínica de la Fundación Alberto J. Roemmers.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.





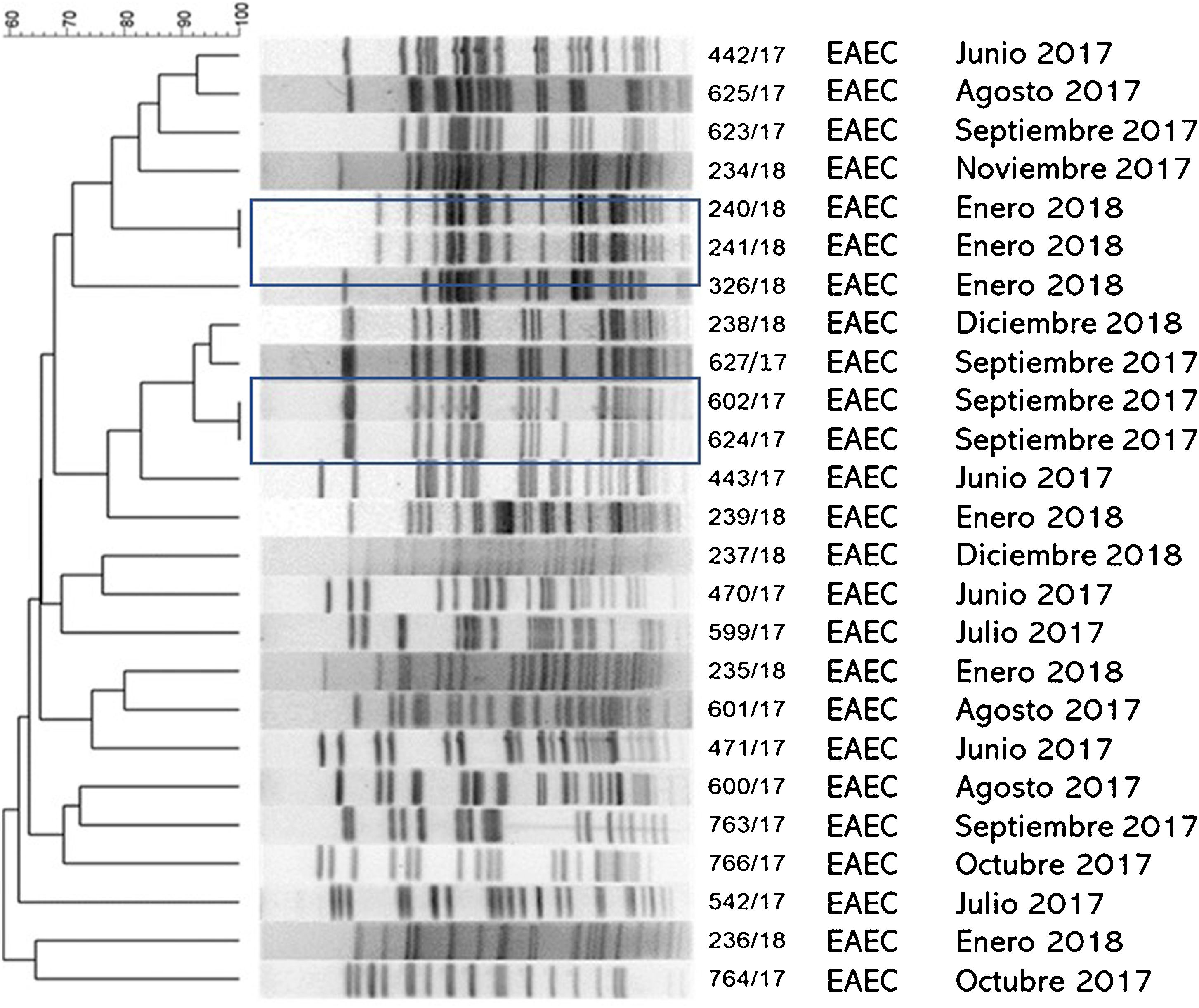

![Relación clonal de aislamientos EAEC recuperados de heces de pacientes con diarrea durante 2017-2018 en dos ciudades de la provincia de Buenos Aires, Argentina. Dice (Opt: 1,50%) (Tol 1,5%-1,5%) (H>0,0% S>0,0%) [0,0%-100.0%] PFGE-XbaI. Relación clonal de aislamientos EAEC recuperados de heces de pacientes con diarrea durante 2017-2018 en dos ciudades de la provincia de Buenos Aires, Argentina. Dice (Opt: 1,50%) (Tol 1,5%-1,5%) (H>0,0% S>0,0%) [0,0%-100.0%] PFGE-XbaI.](https://static.elsevier.es/multimedia/03257541/0000005600000001/v1_202403171031/S0325754123000536/v1_202403171031/es/main.assets/thumbnail/gr1.jpeg?xkr=ue/ImdikoIMrsJoerZ+w96p5LBcBpyJTqfwgorxm+Ow=)