El cáncer se origina por mutaciones conductoras que entregan ventajas en el crecimiento celular, por medio de la inhibición de los puntos de control y la activación exacerbada de vías de señalización involucradas en la sobrevivencia y la proliferación.
El cáncer de pulmón es la principal causa de muerte por cáncer en 89/185 países, y la medicina de precisión ha mejorado el diagnóstico y tratamiento de esta enfermedad, considerando la importancia del perfil mutacional del tumor. Los inhibidores de tirosina quinasa (TKIs) dirigidos a mutaciones conductoras en EGFR, uno de los genes más mutado en cáncer de pulmón de células no pequeñas (NSCLC), han demostrado una disminución significativa en la mortalidad al ser drogas más específicas y menos tóxicas. Los genes accionables en NSCLC son EGFR, ALK, ROS1, ERBB2, MET, MAP2K1, BRAF, KRAS, NTRK1/2/3 y RET, y combinados impactan al 64% de los pacientes. Sin embargo, el acceso a NGS (secuenciación de próxima generación por sus siglas en inglés) y a las drogas dirigidas es desigual por país y la ausencia de mutaciones en genes accionables y el desarrollo de mutaciones de resistencia a la terapia dirigida, son desafíos a nivel mundial. La incorporación de nuevos biomarcadores como PD-L1, la validación del DNA circulante en plasma, la medición de la carga mutacional del tumor, y el desarrollo de ensayos clínicos con combinación de terapias, son parte de las estrategias actuales en investigación.
Esta revisión está enfocada en entregar a lectores de lengua española el estado actual de la medicina de precisión en NSCLC
Cancer originates from driver mutations that provide growth benefits to cells through inhibition of cell cycle checkpoints and exacerbated activation of signaling pathways involved in survival and proliferation.
Lung cancer is the leading cause of cancer death in 89/185 countries, and precision medicine has now improved the diagnosis and treatment of this disease, considering the importance of the mutational profile of the tumor in the diagnosis. For example, tyrosine kinase inhibitors (TKIs) targeting driver mutations in EGFR, one of the most mutated gene in non-small cell lung cancer (NSCLC), have significantly decreased mortality and improved quality of life with more specific and less toxic drugs. The actionable genes in NSCLC are EGFR, ALK, ROS1, ERBB2, MET, MAP2K1, BRAF, KRAS, NTRK1/2/3, and RET, and combined they impact 64% of patients. However, unequal access to NGS and targeted drugs, along with the absence of actionable gene mutations and the development of therapy resistance mutations are global challenges.
The incorporation of biomarkers such as PD-L1, the validation of circulating DNA in plasma instead of re-biopsy, the measurement of the mutational burden of the tumor, and the development of clinical trials with a combination of target therapies are some of the strategies in the current research in NSCLC. This review is focused providing Spanish-language readers with the current state of precision medicine in NSCLC in developed countries and Latin America.
El cáncer, se origina por modificaciones estables en el DNA y el RNA conocidas como mutaciones, que inducen la inmortalidad celular y la expansión del clon alterado mediante la inhibición de los puntos de control y la activación exacerbada de las vías de señalización claves para la sobrevivencia y la proliferación celular1. Estos eventos que favorecen la transformación celular, podrían conducir a la formación de un tumor sólido y, eventualmente, formar crecimientos secundarios y distantes al sitio de origen, lo cual se conoce como metástasis, la etapa de no retorno de la enfermedad2.
Varios factores han sido descritos como agentes cancerígenos, siendo la radiación UV, las carnes procesadas, la obesidad y el consumo de tabaco3, los que presentan mayor evidencia hasta ahora, dentro de los cuales el consumo de tabaco, sigue siendo una de las adicciones más dañinas para la salud, ya que está asociada al desarrollo de todos los cánceres, sobre todo al cáncer de pulmón4,5. La herencia también juega un rol importante y fluctúa entre el 5 al 10% de la carcinogénesis a nivel global6.
Los agentes cancerígenos originan dos tipos de mutaciones; las germinales, que afectan los gametos y por lo tanto se heredan, y las mutaciones somáticas, que están presentes en el DNA de cualquier célula que hace mitosis y no son heredadas a la descendencia, pero se acumulan en nuestro DNA a lo largo de la vida, y son las causantes del 90-95% de la carcinogénesis global7.
2Conceptos claves en medicina de precisiónHasta el año 2002, el fenotipo histopatológico del tumor era lo que determinaba las decisiones terapéuticas, las cuales estaban limitadas sólo a la cirugía, la radioterapia y la quimioterapia citotóxica; pero ahora sabemos que el DNA y RNA de los tumores tienen un perfil mutacional distintivo en cada paciente lo cual hoy en día permite el uso de terapias dirigidas8. La implementación de estas terapias se ha desarrollado bajo el concepto de “Medicina de Precisión”, y tiene como objetivo tiene como objetivo encontrar el mejor tratamiento a través de la evaluación del perfil mutacional del tumor9.
Para la genómica del cáncer, las mutaciones conductoras son aquellas que entregan ventajas en el crecimiento y sobrevivencia, dando lugar a la expansión del clon alterado, con el consecuente inicio de un tumor10–12. Cuando las células portadoras de mutaciones conductoras muestran sensibilidad como respuesta a una terapia dirigida, se conoce como mutación accionable13,14. Por esta razón, descubrir nuevos biomarcadores y comprobar la accionabilidad de mutaciones conductoras, ayuda al desarrollo de la medicina de precisión mediante el incremento del abanico de drogas dirigidas12,15, ya que existe un porcentaje de pacientes que no portan mutaciones accionables conocidas en sus tumores, lo cual no se debe confundir con los pacientes portadores de mutaciones conductoras pero que no pueden ser atacadas farmacológicamente, como las mutaciones conductoras presentes en los genes TP53 y MYC16.
Debido a que las decisiones terapéuticas son guiadas cada vez más por la presencia de mutaciones accionables en el tumor, se deben considerar variables importantes al momento de diagnosticar mutaciones accionables, como la heterogeneidad del tumor, los tipos de biopsia (tumores primarios o metastásicos, biopsias sólidas o líquidas), el momento de la toma de muestra (antes o después de los tratamientos) y los paneles de secuenciación utilizados17.
3Clasificación clínica de las mutaciones somáticasHay que considerar que un tumor de NSCLC puede contener desde menos de cincuenta a más de quinientas mutaciones, y debido a la gran cantidad de mutaciones reportadas a partir del uso de la secuenciación de próxima generación (NGS), se ha diseñado un sistema para clasificar las mutaciones somáticas, de acuerdo con su impacto en la práctica clínica. La clasificación TIER I agrupa las mutaciones con un fuerte significado clínico, ya que cuentan con terapias aprobadas por la Agencia Federal de Administración de Alimentos y Medicamentos de los EE.UU. (FDA) y por estar incluidas en las guías clínicas; TIER II agrupa las mutaciones con el potencial de ser tratadas con drogas validadas pero en otros tumores, y TIER III y IV son para las mutaciones de significado clínico incierto y mutaciones benignas, respectivamente18. Las mutaciones accionables y conductoras pueden ser clasificadas clínicamente como mutaciones TIER I y II de acuerdo con la evidencia clínica que presenten19,20.
4Cáncer de pulmón en latinoamerica y en el mundoAl año 2020, el cancer de pulmón sigue siendo la principal causa de muerte por cáncer en todo el mundo. El NSCLC es el subtipo histológico más prevalente (85% de los casos) subdiviendose a su vez en adenocarcinomas (40%) y escamoso (25%). En general, el NSCLC es diagnosticado en etapas avanzadas, sobre todo en Latinoamérica21–23, donde el acceso a NGS y drogas dirigidas a las mutaciones reconocidas como accionables sigue siendo un desafío24,25.
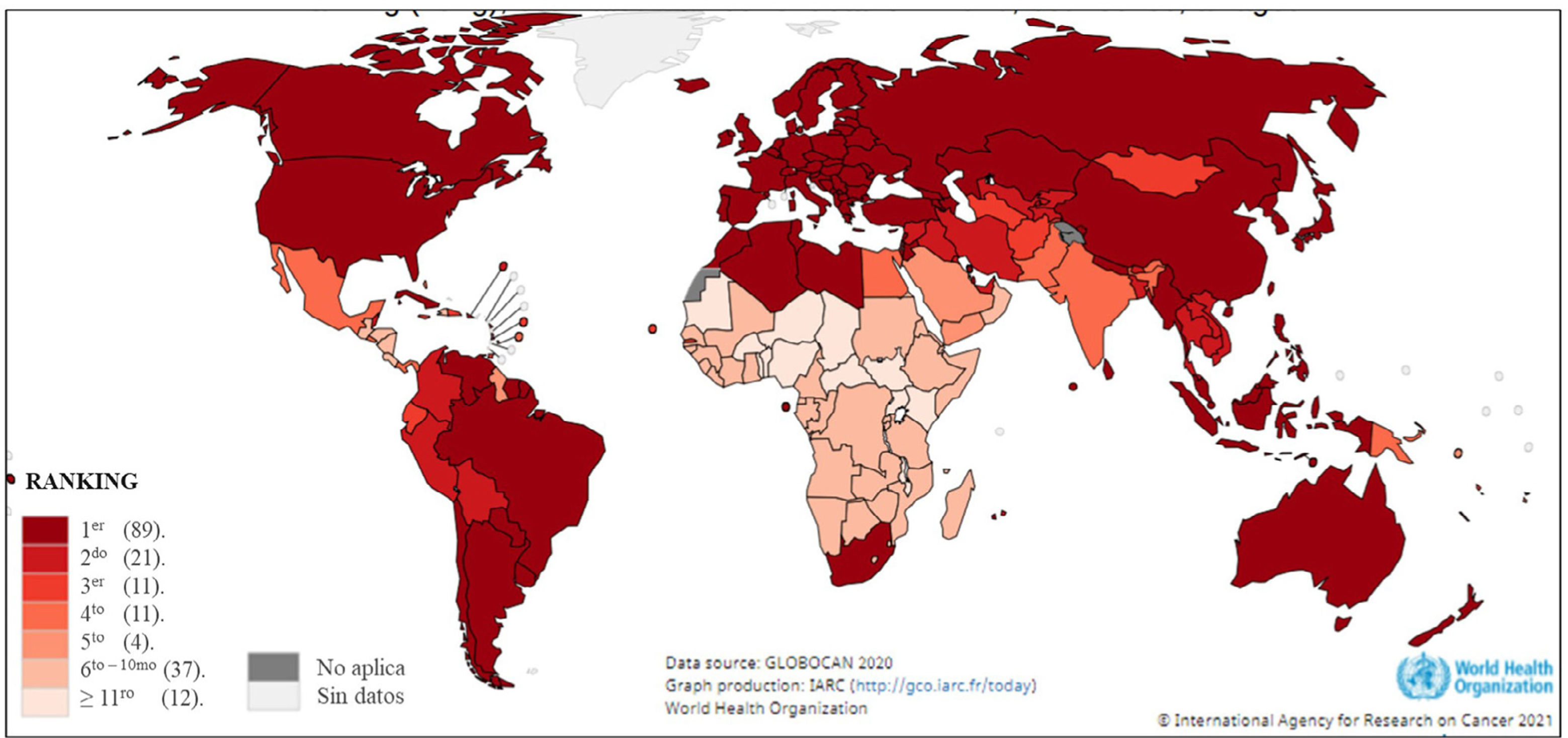
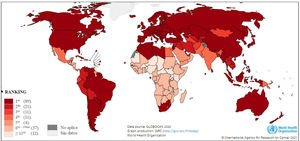
Según los datos de mortalidad del Global Cancer Observatory (Globocan), el año 2020 fallecieron un millón ochocientas personas por cáncer de pulmón a nivel mundial, y Chile presentó al cáncer de pulmón como la primera causa de muerte por cáncer, junto con otros 88 países. Sin embargo, en Latinoamérica, países como Guatemala, Honduras, Nicaragua y Costa Rica, presentan la mortalidad por cáncer de pulmón entre el sexto y décimo lugar (Fig. 1)26. Al mismo tiempo que el NSCLC ocupa el primer lugar en mortalidad, es el subtipo histológico más avanzado en la implementación de la medicina de precisión, sobre todo el adenocarcinoma.
5Genes accionables en nsclc, diagnóstico y terapia dirigidaLas mutaciones en el gen EGFR y los rearrreglos del gen ALK fueron las primeras alteraciones conductoras encontradas en NSCLC27–29 y presentaron una alta sensibilidad a las primeras drogas dirigidas, conocidas como los inhibidores de los receptores con actividad tirosina quinasa (TKIs)30.
A nivel mundial, las mutaciones más frecuentes en NSCLC ocurren en el gen EGFR, y las deleciones en el exón 19 y sustituciones en el exón 21 (como el cambio de leucina a arginina en la posición 858 (L858R)), representan aproximadamente 45% y 40% de todas las mutaciones de EGFR, evidenciando notables mejoras en los resultados clínicos, aunque pacientes con mutaciones atípicas en EGFR han mostrado respuestas heterogéneas y reducidas a los inhibidores de EGFR31,32.
Posterior a estos hallazgos, diversos esfuerzos internacionales como el atlas del genoma del cáncer (TCGA), el Consorcio Internacional del Genoma del Cáncer (ICGC) y la Sociedad Americana de Oncología Clínica (ASCO), enfocaron sus esfuerzos en incrementar la lista de genes accionables, abriendo la prescripción de terapias dirigidas, de EGFR y, ALK, a ROS1, ERBB2, MET, MAP2K1, BRAF, KRAS y RET, los cuales se encuentran alterados en aproximadamente un 64% de los pacientes con NSCLC (Tabla 1)33,34. Esto significa que existen pacientes que no poseen alteraciones conductoras en los genes reconocidos como accionables, aunque como veremos más adelante, interrogar nueve genes accionables no es la única alternativa para beneficiarse de las terapias dirigidas.
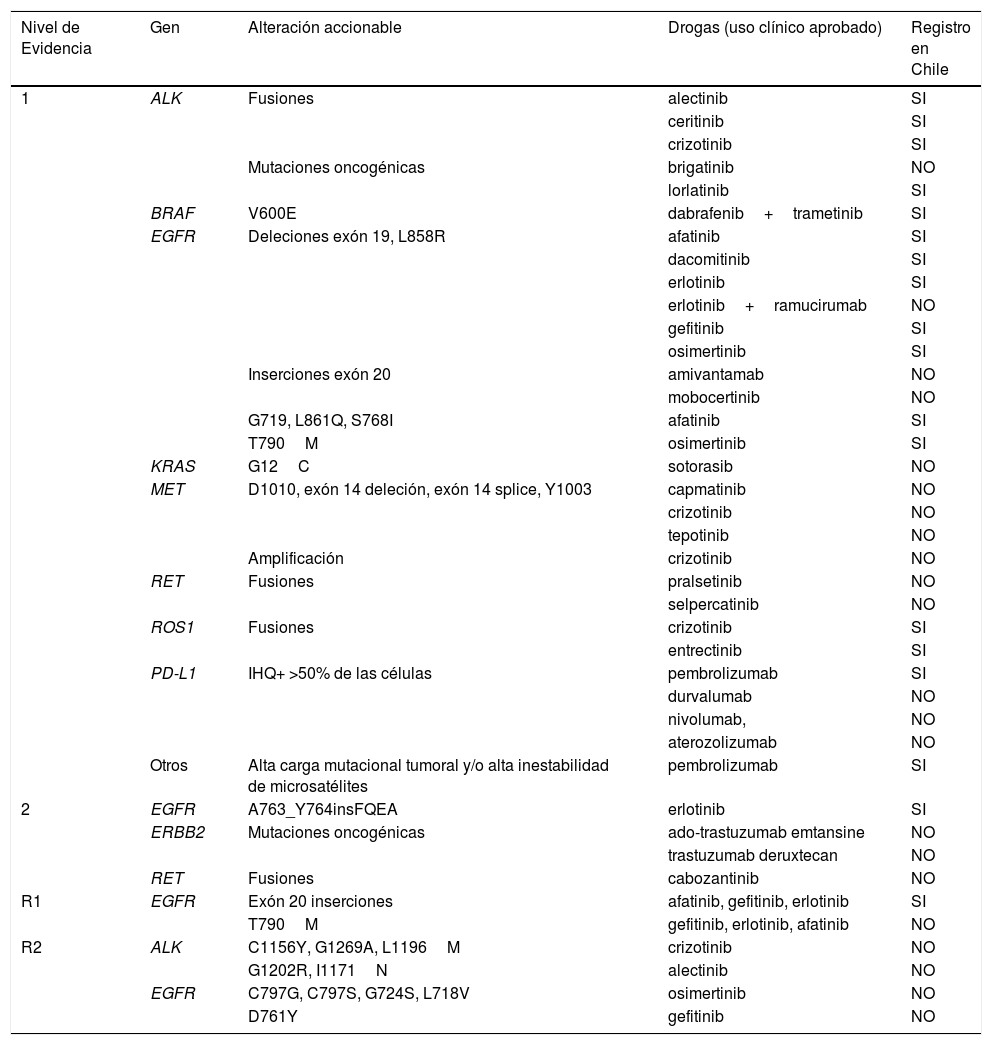
Drogas dirigidas a alteraciones accionables en NSCLC de acuerdo con los niveles de evidencia reportados en OncoKB81 y su registro de aprobación en el Instituto de Salud Pública de Chile (octubre 2021)
| Nivel de Evidencia | Gen | Alteración accionable | Drogas (uso clínico aprobado) | Registro en Chile |
|---|---|---|---|---|
| 1 | ALK | Fusiones | alectinib | SI |
| ceritinib | SI | |||
| crizotinib | SI | |||
| Mutaciones oncogénicas | brigatinib | NO | ||
| lorlatinib | SI | |||
| BRAF | V600E | dabrafenib+trametinib | SI | |
| EGFR | Deleciones exón 19, L858R | afatinib | SI | |
| dacomitinib | SI | |||
| erlotinib | SI | |||
| erlotinib+ramucirumab | NO | |||
| gefitinib | SI | |||
| osimertinib | SI | |||
| Inserciones exón 20 | amivantamab | NO | ||
| mobocertinib | NO | |||
| G719, L861Q, S768I | afatinib | SI | ||
| T790M | osimertinib | SI | ||
| KRAS | G12C | sotorasib | NO | |
| MET | D1010, exón 14 deleción, exón 14 splice, Y1003 | capmatinib | NO | |
| crizotinib | NO | |||
| tepotinib | NO | |||
| Amplificación | crizotinib | NO | ||
| RET | Fusiones | pralsetinib | NO | |
| selpercatinib | NO | |||
| ROS1 | Fusiones | crizotinib | SI | |
| entrectinib | SI | |||
| PD-L1 | IHQ+ >50% de las células | pembrolizumab | SI | |
| durvalumab | NO | |||
| nivolumab, | NO | |||
| aterozolizumab | NO | |||
| Otros | Alta carga mutacional tumoral y/o alta inestabilidad de microsatélites | pembrolizumab | SI | |
| 2 | EGFR | A763_Y764insFQEA | erlotinib | SI |
| ERBB2 | Mutaciones oncogénicas | ado-trastuzumab emtansine | NO | |
| trastuzumab deruxtecan | NO | |||
| RET | Fusiones | cabozantinib | NO | |
| R1 | EGFR | Exón 20 inserciones | afatinib, gefitinib, erlotinib | SI |
| T790M | gefitinib, erlotinib, afatinib | NO | ||
| R2 | ALK | C1156Y, G1269A, L1196M | crizotinib | NO |
| G1202R, I1171N | alectinib | NO | ||
| EGFR | C797G, C797S, G724S, L718V | osimertinib | NO | |
| D761Y | gefitinib | NO |
Niveles de evidencia OncoKB: (1) Biomarcador reconocido por la FDA que predice la respuesta a un fármaco aprobado por la FDA en esta indicación. (2) Biomarcadores de atención estándar o biomarcadores en etapa de investigación que predicen la respuesta a un medicamento aprobado por la FDA en una indicación específica. (R1) Cuidado estándar del biomarcador predictivo de resistencia una droga aprobada por la FDA, y (R2) Evidencia clínica convincente respalda el biomarcador como predictivo de resistencia a un fármaco, pero ni biomarcador ni la droga es la atención estándar. Los genes NTRK1/2/3 no fueron considerados.
Referente a los métodos diagnósticos usados para identificar mutaciones accionables, tanto la FDA como distintos comités de expertos y guías clínicas recomiendan el uso de NGS para la evaluación de los genes accionables en NSCLC35,36 (Tabla 1), al igual que la guía práctica clínica de la red nacional integral de cáncer de EE.UU. del año 2022, (NCCN). La evaluación por qPCR es aceptada, pero la principal desventaja es que permite evaluar un número restringido y específico de mutaciones por análisis37.
Una de las desventajas es el costo excesivo de las plataformas de secuenciación, paneles e insumos, lo que constituye un desafío para la mayoría de las economías latinoamericanas, agravándo la desigualdad en el acceso a la medicina de precisión25,38.
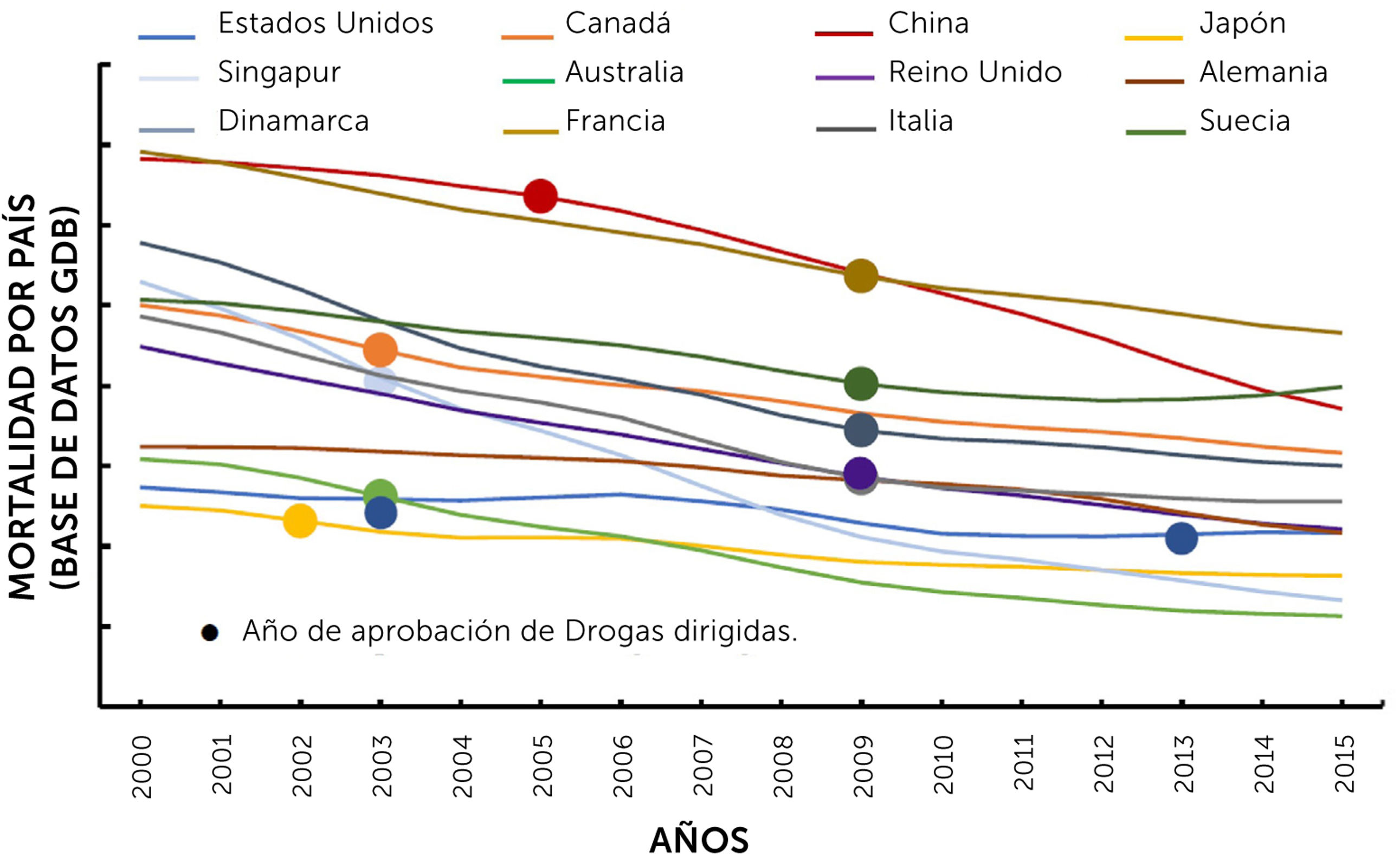
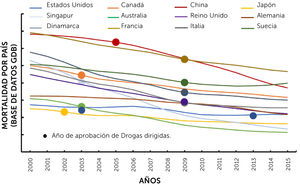
6El perfil mutacional de un tumor es importante para el pronóstico de vida de los pacientes con nsclcPaíses como China, Estados Unidos, Japón y Australia, entre otros, ya han demostrado una mejoría en la supervivencia de los pacientes con NSCLC, al comparar la tasa de mortalidad antes y después de la implementación de TKIs como se muestra en la Fig. 2 (previamente publicada por Howlader, et al). De esta manera se evidenció que el diagnóstico de mutaciones en genes accionables mejora la sobrevida de los pacientes con NSCLC.
Cambios en la mortalidad por cáncer de pulmón de células no pequeñas (NSCLC). Año de aprobación de EGFR-TKI en EE.UU., Canadá, China, Japón, Singapur, Australia, Reino Unido, Alemania, Dinamarca, Francia, Italia y Suecia, entre 2000 y 2015 de acuerdo con la base de datos Global Disease Burden (GDB, http://www.healthdata.org/gbd). Figura traducida al español de la Fig. 1B original con licencia de Elsevier (número 5177010593414)41.
Varios trabajos han destacado la importancia del perfil mutacional en el diagnóstico y terapia, como Shen Zhao et al., quienes estudiaron una cohorte de 1.166 pacientes con NSCLC avanzado (aNSCLC) en busca de la mejor terapia dirigida, y encontraron 781 pacientes con alteraciones potencialmente accionables, 440 clasificaron para terapias ya existentes; de los cuales 196 recibieron una terapia ya aprobada por la FDA y 244 pacientes fueron inscritos en ensayos clínicos. Luego del análisis de respuesta a terapias, concluyeron que el perfil mutacional del tumor es importante para la prescripción de terapias dirigidas, ya que aumenta significativamente la sobrevivencia libre de progresión (PFS) y la sobrevivencia promedio (OV) en comparación a aquellos pacientes que presentaron mutaciones conductoras sin accionabilidad y que fueron tratados con terapia convencional40. Fränce Hardtstock et al. seleccionaron 1.741 pacientes diagnosticados con NSCLC entre los años 2012 y 2015, encontrando que solo el 26,3% de los diagnósticos presentó un perfil mutacional con prescripción de terapia dirigida, y presentaron una mejor sobrevida en comparación a los pacientes sin perfil mutacional tratados con quimioterapia41.
Un estudio que incluyó a Argentina, Colombia, Chile y Uruguay, evaluó retrospectivamente la aplicación de pruebas para mutaciones en EGFR. En promedio, la frecuencia de las pruebas moleculares para EGFR fue del 66%, siendo Argentina la más alta (79%), con una población que pertenece principalmente al sector privado (87%) y Uruguay la tasa de pruebas más baja (28%), con una población que pertenece principalmente al sector público de salud (91%). Respecto a los pacientes con progresión de la enfermedad, solo el 70,97% contaron con pruebas moleculares para interrogar mutaciones de resistencia24. El acceso a las pruebas moleculares sigue siendo un desafío que afecta la elección del terapias dirigidas de primera línea en pacientes latinoamericanos con NSCLC25.
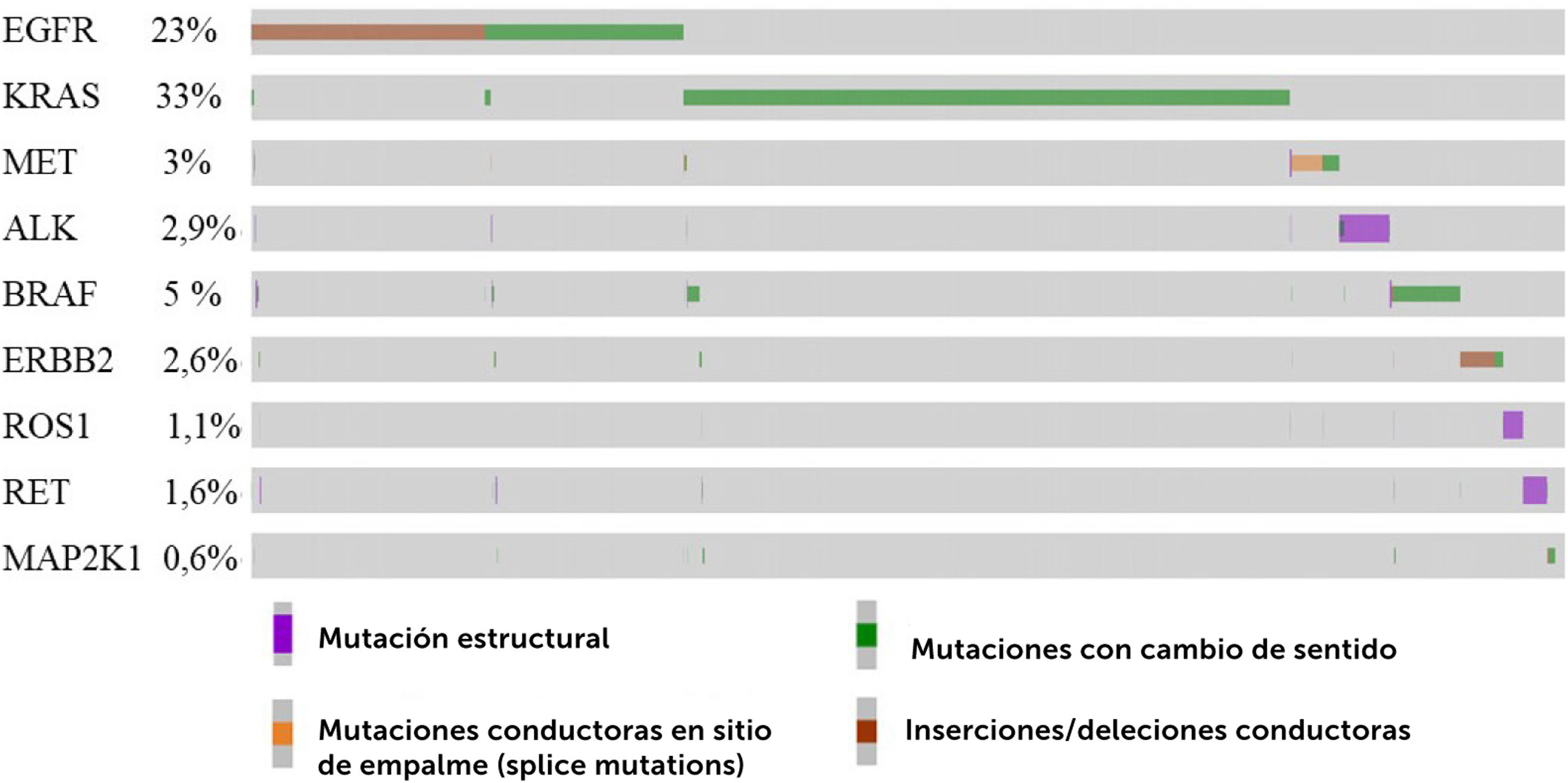
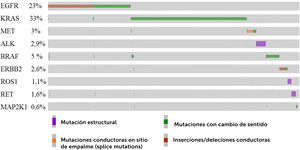
7Frecuencia de las mutaciones conductoras en genes accionablesPara conocer solamente las frecuencias de alteraciones conductoras en los nueve genes accionables, utilizamos información del proyecto GENIE, seleccionando los datos públicos de 12.382 pacientes con NSCLC, específicamente adenocarcinoma. Luego se filtraron 2.915 pacientes no perfilados para todos los genes y 2.460 mutaciones de significado incierto (VUS). Finalmente, se interrogaron los genes accionables en 9.467 pacientes representados en un oncoplot (Fig. 3). El gen más mutado fue KRAS (33%), seguido por EGFR (23%) y BRAF (5%)42,43. De acuerdo con otros estudios, EGFR es el gen más mutado en NSCLC, alcanzando un 15% en caucásicos y cerca del 50% en asiáticos, por lo que la ancestría jugaría un rol importante44,45. En Latinoamérica, los porcentajes de mutaciones en EGFR son variables. En Brasil se ha reportado un 11%21 y 22,4%46 de mutaciones en EGFR, en Chile ha alcanzado el 8,3%24 y 14%21, Perú 25%21 y un 51%47, Argentina 26%24 y 14,4%47. Por este motivo, al comparar, es importante considerar varios factores, como la ancestría, etnia o raza, el subtipo histológico del tumor, la prueba diagnóstica empleada (qPCR o NGS), el número de muestras interrogadas y la metodología para reclutar las cohortes del estudio.
Oncoplot representativo del perfil mutacional de 12.434 pacientes con NSCLC, específicamente adenocarcinoma. A partir de datos disponibles en GENIE (https://genie.cbioportal.org)82, se seleccionaron los pacientes y el análisis sólo de las mutaciones conductoras presentes en los genes accionables EGFR, KRAS, MET, ALK, BRAF, ERBB2, ROS1, RET y MAP2K1. El 77% de los pacientes presentó al menos un gen alterado. La clave de color representa el tipo de mutación encontrada. El oncoplot muestra sólo los pacientes con alteraciones42,43.
Las terapias dirigidas a mutaciones accionables aprobadas hasta ahora por la FDA en NSCLC, están disponibles en ONCOKB y están listadas en la Tabla 1. Sin embargo, a pesar de la alta eficacia inicial de las terapias dirigidas a alteraciones accionables, las células portadoras de mutaciones conductoras, desarrollarán en algún momento de la terapia mutaciones de resistencia como mecanismo de sobrevivencia y evolución tumoral. El desarrollo de mutaciones de resistencia puede afectar al mismo blanco molecular y a las vías de señalización rio abajo o paralelas al gen blanco48,49. Otro desafío importante, son los pacientes que no son portadores de ninguna alteración molecular en los genes reconocidos hasta ahora como accionables. Sin embargo, el cálculo de la carga mutacional del tumor (TMB), el cual se define como el número de mutaciones por mega base (Mb) de DNA y que necesita la secuenciación del exoma completo (o un panel de gran tamaño) para ser calculado, ha demostrado ser un buen biomarcador para el uso de inhibidores de puntos de control inmunes (ICI), en tumores sólidos con TMB alta como el NSCLC50, lo cual es independiente de los niveles de expresión de PD-L1 y de mutaciones en genes accionables51. Un bajo TMB es ≤5 mutaciones/Mb y un TMB alto es entre >20 y ≤50 Mb.
9Resistencia a tkis y evaluación de mutaciones de resistenciaLas mutaciones de resistencia son el principal desafío para la medicina de precisión, por lo que es necesario evaluar la frecuencia de las mutaciones conductoras diagnosticadas inicialmente a lo largo del tratamiento, y también, evaluar la aparición de mutaciones de resistencia reconocidas (Tabla 1). Para evitar una nueva biopsia, el cual es un diagnóstico invasivo, se ha validado y aceptado el uso de DNA tumoral circulante en plasma (ctDNA) para evaluar la respuesta a terapias dirigidas52,53.
La primera y segunda línea de TKIs dirigidos a pacientes con alteraciones en EGFR, fueron gefitinib, erlotinib y afatinib, pero luego de estos tratamientos emergió la resistencia a TKIs a través del desarrollo de mutaciones entre los 9 a 14 meses de tratamiento54,55, siendo la mutación EGFR T790M, el 50% de los casos de resistencia a TKIs. Luego se aprobó osimertinib, un EGFR-TKI de tercera generación como opción terapéutica para pacientes con mutaciones de resistencia en EGFR56. Otra de las mutaciones de resistencia a EGFR-TKIs bien estudiada, es la amplificación del gen MET, tanto para TKIs de primera como de segunda generación57. Esta alteración se ha observado también como el mecanismo más común de resistencia afectando a un 15% de los ctDNA de pacientes con NSCLC (NCT02296125). De acuerdo con estos hallazgos, estudios preclínicos han demostrado que el uso concomitante de inhibidores de MET, tales como crizotinib, en combinación con osimertinib tienen el potencial de mejorar la sensibilidad de pacientes con mutaciones en EGFR resistentes a osimertinib y con MET amplificado, las cuales fueron detectadas usando el ctDNA58.
A pesar de que estudios evidencian que las mutaciones en EGFR y KRAS son excluyentes en NSCLC59, un estudio basado en la evaluación de mutaciones de resistencia demostró que un 48,5% de muestras de plasma fueron positivas para mutaciones en el codón 12 de KRAS después de la resistencia a TKI y el 39,4%, tenían una co-mutación de KRAS y EGFR60. También se han descrito mutaciones de resistencia en KRAS en pacientes positivos para la mutación activante que elimina al exón 14 (METex14) luego de la terapia con crizotinib, y también se han repostado casos positivos para BRAF V600E (rio abajo de MET y KRAS) con mutación de resistencia en el codón 12 de KRAS61,62.
Comprender los mecanismos de resistencia se ha convertido en una tarea urgente junto con la evaluación de combinación de drogas, tanto para los casos de resistencia y como para los casos que presenten co-ocurrencia de mutaciones accionables en NSCLC63.
10Inmunoterapia en pacientes con nsclcLos inhibidores de PD-1 y de PD-L1 son un grupo de fármacos anticancerosos inhibidores de los puntos de control inmunológico para pacientes cuyos tumores expresan PD-L1 en al menos el 50% de las células64.
Cabe destacar que existe una creciente contribución de estudios clínicos que consideran la terapia de PD-L1 en combinación con otras terapias65. El ensayo clínico KEYNOTE-189 comenzó el año 2015 evaluando la combinación de anti-PD-L1 con quimioterapia, específicamente pembrolizumab más pemetrexed-platino mostraron una OV (22 v/s 10 meses) y PFS (9 v/s 4,9) de casi el doble en comparación grupo tratado con placebo más pemetrexed-platino. Hasta ahora, el uso de ICIs en pacientes sin mutaciones accionables conocidas, muestran una mejora significativa en el pronóstico de vida de los pacientes con NSCLC66,67.
Hasta ahora, la expresión de PD-L1 no se ha asociado a co-ocurrencia de mutaciones en los principales RTK como EGFR, los reordenamientos de ALK o mutaciones de KRAS68, Sin embargo, los pacientes que presentaron la amplificación del gen MET (METamp) con un bajo número de copias del gen o gene copy-number (GCN, por sus siglas en inglés) MET (METamp GCN ≤10) y tratados con ICI, mostraron una mediana de sobrevida de 19 v/s 8 meses con quimioterapia, y los pacientes con niveles de amplificación altos (GCN ≥10), la mediana de sobrevida fue de 36 meses con ICI en comparación a 4 meses con quimioterapia69.
11Avances de la medicina de precisión en el mundo y en latinoaméricaLa oncología de precisión ofrece tecnologías terapéuticas para pacientes diagnosticados de NSCLC que mejoran significativamente el pronóstico de vida, pero debido al problema de acceso a pruebas moleculares y a las drogas dirigidas, los pacientes de todo el mundo no han sido beneficiados por igual69.
De acuerdo con la Federación Europea de Industrias Farmacéuticas (EFPIA), casi el 90% de los nuevos medicamentos que se han lanzado al mercado en los últimos años han sido consumidos exclusivamente por EE.UU., Europa Occidental y Japón (>60% solo en los EE.UU.), quedando muy atrás al resto del mundo70,71.
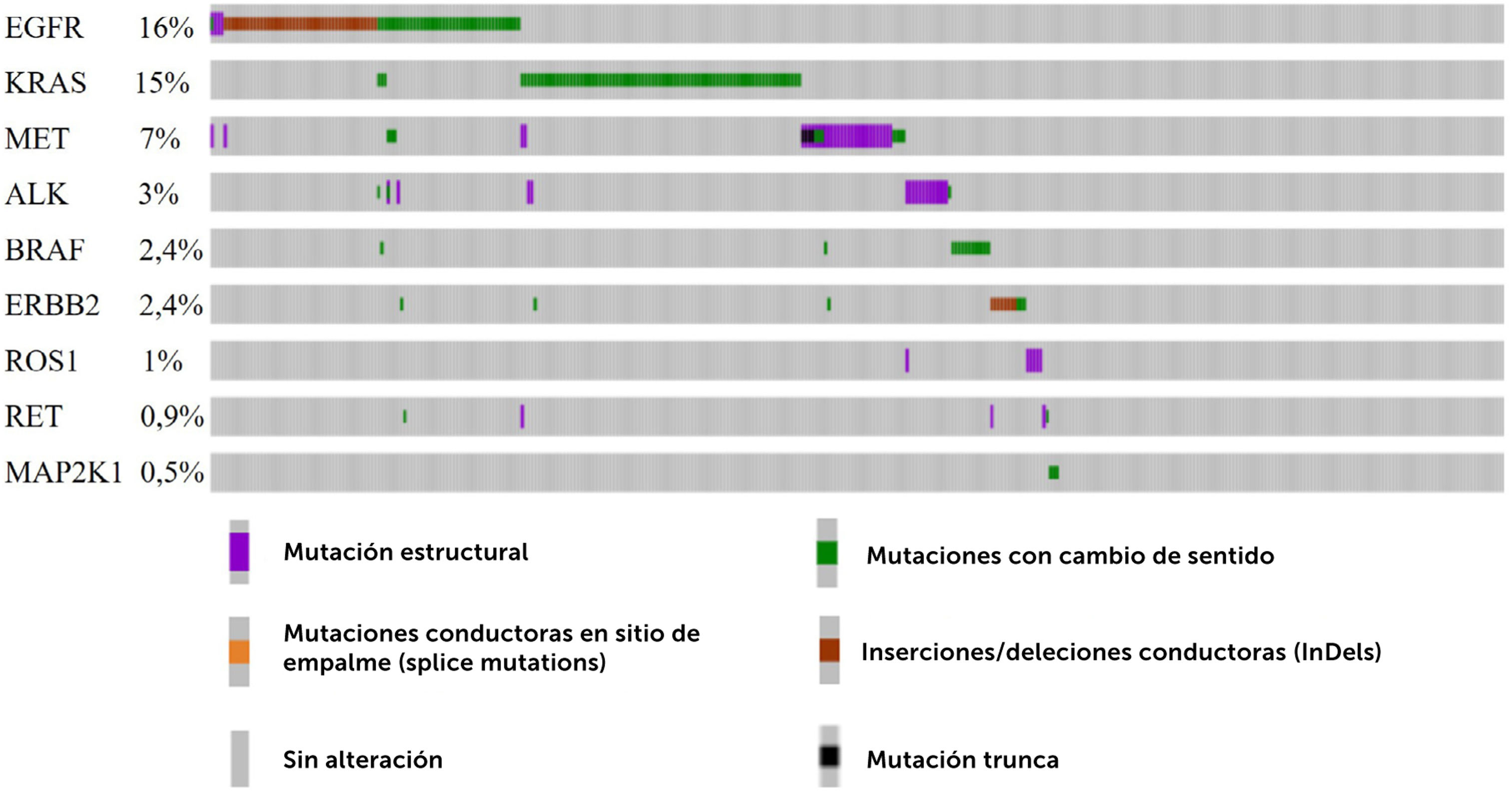
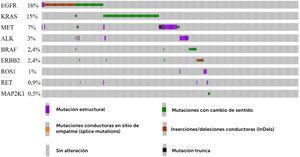
En Chile, se han aprobado algunas drogas dirigidas para su uso en NSCLC y adenocarcinoma de pulmón (Tabla 1), pero no han sido implementadas dentro de un plan de salud universal. Según registros de 579 pacientes chilenos secuenciados por NGS (Fig. 4), los genes más mutados en NSCLC (adenocarcinomas) son EGFR (16%), KRAS (15%) y MET (7%). Casi todas las drogas dirigidas contra EGFR están aprobadas para su uso en Chile, pero no existen drogas disponibles contra alteraciones en KRAS ni MET (Tabla 1). La primera droga dirigida a pacientes positivos para KRAS G12S es sotorasib, la cual ha producido un beneficio clínico duradero en los pacientes del ensayo clínico de fase 2 (NCT03600883) y en mayo 2021 fue aprobada por la FDA. La aprobación de sotorasib se basó en los resultados de CodeBreaK 100, un ensayo global, multicéntrico y multicohorte, que reclutó a 427 pacientes con tumores sólidos avanzados con la mutación KRAS G12C, incluidos 250 pacientes con NSCLC72,73, y actualmente, sotorasib está aprobada para su uso en EE.UU., Canadá y en el Reino Unido.
Oncoplot representativo del perfil mutacional de 579 pacientes chilenos diagnosticados con NSCLC, específicamente adenocarcinoma de pulmón. A partir de los datos disponible en NIRVANA (NCT03220230)21. Se seleccionaron los pacientes de Chile (columnas) y el análisis de la mutaciones conduc-toras en los genes EGFR, KRAS, MET, ALK, BRAF, ERBB2, ROS1, RET y MAP2K1. El 45% de los pacientes presenta al menos un gen alterado. La clave de color representa el tipo de mutación encontrada y las barras grises indican ausencia de mutaciones en los genes interrogados.
El tercer gen más mutado en Chile es MET (7%), el cual es el único gen que está afectado por sobre el promedio de los datos de GENIE (3%). Esto se podría explicar mediante la ancestría, la cual ha demostrado ser importante en la frecuencia de mutaciones en EGFR y posiblemente podría explicar la frecuencia de alteraciones en MET44,45. Sin embargo, debido a la falta de información sobre alteraciones en MET en Latinoamérica, no fue posible analizar estos resultados con mayor profundidad. Otra posible explicación es que ambos oncoplots no sean directamente comparables, ya que la cohorte de GENIE agrupa varios estudios que utilizan distintas metodologías diagnósticas basadas en el estudio del DNA principalmente, y los pacientes de Chile fueron evaluados con un ensayo NGS (OFA) que utiliza tanto el DNA como el RNA del tumor, por lo que puede haber diferencias importantes en la sensibilidad técnica de los ensayos74. Actualmente, las mutaciones más frecuentes en MET (METamp y METex14), cuentan con varias drogas dirigidas aprobadas por la FDA como crizotinib75, tepotinib76, capmatinib77 y savolitinib78.
En Chile, las drogas contra MET y KRAS no están disponibles para su uso en la práctica clínica, lo cual resulta perentorio de evaluar ya que KRAS y MET cuentan con un gran porcentaje de mutaciones que no coexisten con otras mutaciones accionables con opciones terapéuticas (NCT03220230)21.
Crizotinib es un multi TKI que ha mostrado un aumento significativo de la PFS en comparación a quimioterapia, cuando se usa en pacientes con mutaciones en ALK, ROS1 y MET79. En Chile, crizotinib está aprobado sólo para pacientes con alteraciones en ALK (Tabla 1). Es importante evaluar y aprobar nuevas pruebas diagnósticas y drogas dirigidas a los genes accionables más mutados en pacientes chilenos con NSCLC.
12DiscusiónEl perfil mutacional de los genes accionables junto con los niveles de expresión de PD-L1 y la TMB son hasta ahora las mejores opciones diagnósticas para prescribir terapias dirigidas en NSCLC39,41,80. De la información tabulada en los oncoplots gracias a la gran cantidad de datos almacenados en bases de datos públicas, podemos rescatar dos conceptos importantes. Lo más común son los genes mutados en una frecuencia menor al 2%, y lo menos común, son los genes mutados en un porcentaje superior al 20%. Otro aspecto importante, es que la mayoría de los perfiles mutacionales (columnas) poseen solo una mutación conductora, lo que se evidencia al observar la figura completa, donde las mutaciones (colores) forman una especie de escalera que desciende hacia la derecha, siendo muy pocos pacientes los que expresan más de una mutación en los genes interrogados. Sin embargo, existen pacientes que no presentaron ninguna mutación conductora. En esos casos, si se quiere encontrar la alteración conductora predictiva de accionabilidad, es importante utilizar paneles con una mayor cantidad de genes en NGS y para los casos que no posean mutaciones conductoras, medir los niveles de PD-L1 o calcular la TMB son de gran ayuda para encontrar la mejor alternativa terapéutica37.
Dado que la principal causa de muerte por cáncer en Chile y América del Sur es el cáncer de pulmón y dada la dificultad que genera encontrar datos de las frecuencias de mutaciones en pacientes latinoamericanos, sería importante contar con una base de datos local y pública que agrupe datos moleculares de tumores, junto con el fortalecimiento de la red oncológica y almacenamiento de datos en los servicios asistenciales.
Declaración de conflicto de interésR.A. ha recibido honorarios de parte de Roche por concepto de asesorías, conferencias y actividades educativas, y subvenciones para proyectos de investigación científica.