El estudio de la sensibilidad a antimicrobianos de las diferentes bacterias aisladas en muestras biológicas tiene 2 objetivos fundamentales: guiar al clínico en la elección del mejor tratamiento individual, y monitorizar la evolución de la resistencia bacteriana con objeto de revisar el espectro del antimicrobiano y poder actualizar los tratamientos empíricos1. Este estudio se realiza mediante el antibiograma, que mide la sensibilidad de una bacteria frente a diferentes antimicrobianos in vitro y a partir de estos resultados predice la eficacia in vivo. Con un antibiograma se pueden obtener resultados cualitativos que indican si la bacteria es sensible o resistente a un antibiótico, o cuantitativos que determinan la concentración mínima (CMI) de antimicrobiano que inhibe el crecimiento bacteriano (en μg/ ml o en mg/l). La interpretación de los resultados del antibiograma (sensible, intermedio o resistente) se realiza en función de los valores establecidos por diferentes comités, como el Clinical and Laboratory Standards Institute en Estados Unidos2, el European Committee on Antimicrobial Susceptibility Testing en Europa3 y la Mesa Española de Normalización de la Sensibilidad y Resistencia a los Antimicrobianos4. Estos comités determinan y establecen puntos de corte basados en propiedades microbiológicas, farmacocinéticas y de eficacia clínica, para definir la sensibilidad (éxito terapéutico) o resistencia de las diferentes especies bacterianas a cada antimicrobiano5,6. En este capítulo se desarrollan aspectos relacionados con las técnicas microbiológicas del antibiograma, y se describen algunos ejemplos de fenotipos de resistencia característicos de algunas especies. En un próximo capítulo se profundizará más acerca de los fenotipos de resistencia, básicos para entender la lectura interpretada del antibiograma.
Puntos clave
El estudio de la sensibilidad in vitro de las bacterias a los antimicrobianos se realiza mediante métodos fenotípicos (técnicas de dilución y de difusión), bioquímicos y genéticos7.
Los métodos fenotípicos (antibiograma) son los más utilizados. Consisten en enfrentar un inóculo bacteriano estandarizado a una única o a diferentes concentraciones de antibiótico. La interpretación de los resultados obtenidos permite clasificar a los microorganismos en categorías clínicas: sensibles, intermedios o resistentes. Hay que tener en cuenta que no siempre un valor de CMI más bajo indica mayor actividad de este antimicrobiano, ya que las CMI que definen la sensibilidad o resistencia son diferentes para cada especie bacteriana y cada antimicrobiano. Si un microorganismo es sensible indica que con las dosis habituales se espera una evolución favorable de la infección, siempre que se alcancen valores adecuados en el lugar de la infección, lo que en ocasiones no es posible (p. ej., en el sistema nervioso central). Por el contrario, si el microorganismo es intermedio o resistente, es probable que la evolución sea desfavorable. La interpretación de la sensibilidad predice mejor el fracaso (cuando es resistente) que el éxito de un tratamiento.
Entre los métodos fenotípicos, las técnicas de dilución determinan la CMI utilizando un medio líquido (dilución en caldo) o un medio sólido (dilución en agar) para disolver las diferentes concentraciones del antimicrobiano. El medio estandarizado para la realización del antibiograma es el medio Mueller-Hinton, al que se le añade sangre u otros suplementos para bacterias que no crecen en él. La CMI es la dilución más baja de antimicrobiano en la que no se observa crecimiento bacteriano. La dilución en caldo suele realizarse en micrométodo (microdilución), en paneles multipocillos, y es el sistema mayoritariamente adoptado por los sistemas automáticos comerciales para determinar la sensibilidad a los antimicrobianos. En estos sistemas, la lectura de los valores de CMI y la interpretación de resultados se realizan de forma automática.
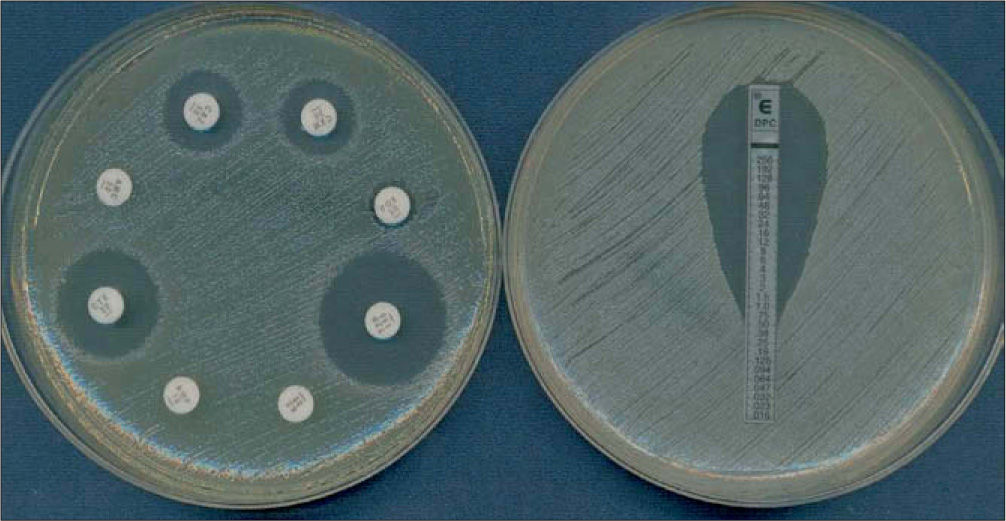
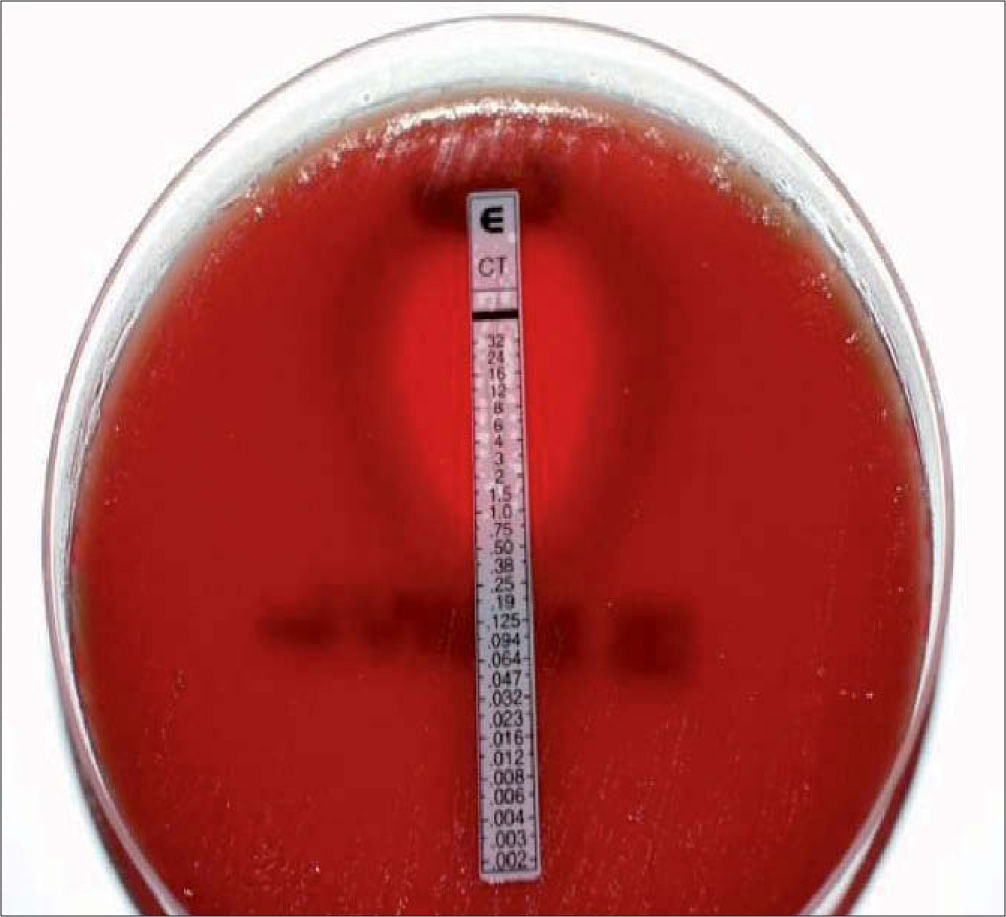
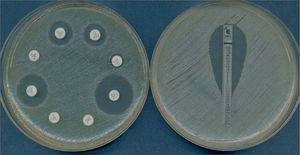
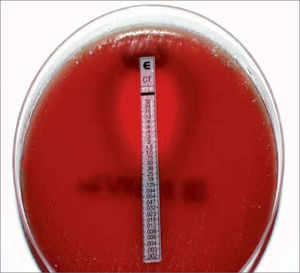
Las técnicas de difusión emplean discos de papel impregnados con una solución estandarizada de antibiótico que se disponen sobre la superficie de un medio sólido previamente inoculado en su superficie con una suspensión bacteriana. Tras un período de incubación de 18 h, el diámetro del halo formado está en relación con el grado de sensibilidad del microorganismo. La carga del disco está ajustada para que los halos de inhibición permitan diferenciar los microorganismos sensibles de los resistentes y pueda establecerse una correlación con los valores de CMI: halos pequeños se relacionan con valores altos de CMI (resistentes) y halos grandes con CMI bajas (sensibles) (fig. 1A). Otra técnica de difusión es el E-test, que además permite la determinación directa del valor de la CMI (fig. 1B). Utiliza tiras de plástico impregnadas con un antibiótico en concentraciones decrecientes. Al contacto de la tira con el agar, el antibiótico difunde e impide el crecimiento del microorganismo. Después de la incubación se observa una zona de inhibición en forma de elipse: el valor de la CMI es el punto de intersección de la elipse con la tira y está indicado en la escala impresa sobre la superficie de la tira. Esta técnica puede utilizarse directamente sobre muestras clínicas para obtener resultados preliminares en menos de 24 h, que siempre deben confirmarse mediante pruebas de sensibilidad estandarizadas con bacterias en cultivo puro8 (fig. 2).
Antibiograma mediante E-test directo de una muestra de LCR de un paciente con meningitis por Streptococcus pneumoniae. El medio utilizado contiene sangre de carnero necesaria para el crecimiento de S. pneumoniae. Se observa una elipse de inhibición con la tira de cefotaxima. El punto donde la elipse corta con la tira es el valor de CMI, en este caso 0,75 mg/l.
Los métodos bioquímicos consisten en la determinación del mecanismo por el cual una bacteria es resistente a un antimicrobiano. Los más utilizados son la detección de β-lactamasa con discos impregnados con una cefalosporina cromogénica que cambia de color cuando se hidroliza (método que se utiliza para la detección rápida de la resistencia a ampicilina en Haemophilus spp., Neisseria spp. y Moraxella spp.), o la detección de la PBP2a responsable de la resistencia a cloxacilina en Staphylococcus aureus, por una técnica de aglutinación con látex. Finalmente, los métodos genéticos detectan genes de resistencia, generalmente mediante técnicas de PCR, como en el caso del gen mecA que codifica la producción de la PBP2a.
Del antibiograma a la práctica clínicaComo ya se ha indicado, el estudio de la sensibilidad a antimicrobianos de las diferentes bacterias aisladas en muestras biológicas tiene interés individual y epidemiológico. La realización rutinaria del antibiograma proporciona los patrones de resistencias locales y regionales que deben actualizarse periódicamente, ya que éstos pueden cambiar sustancialmente en cortos períodos9. Por otra parte, el antibiograma también puede ayudar a diferenciar entre microorganismos verdaderamente patógenos o contaminantes (aislamientos sucesivos de estafilococo coagulasa negativo en hemocultivos con diferente sensibilidad indicaría contaminación cutánea) y en la evaluación inicial ante la sospecha de un brote nosocomial (aislamiento en distintos pacientes de Klebsiella pneumoniae productora de β-lactamasa de espectro extendido [BLEE]).
Lectura interpretada del antibiogramaEl análisis de los resultados de sensibilidad es un aspecto esencial para una adecuada información del antibiograma y tiene una gran trascendencia clínica10. En este sentido, la lectura interpretada del antibiograma analiza los fenotipos de sensibilidad y permite deducir posibles mecanismos de resistencia. Además, este proceso permite inferir la sensibilidad de antibióticos no estudiados en el antibiograma y la corrección, en su caso, de falsas sensibilidades observadas in vitro, como ocurre en el caso del antibiograma de una enterobacteria con una BLEE, en el que no siempre aparecen como resistentes todas las cefalosporinas, si bien, en la práctica debe evitarse su uso. Asimismo, favorece la adecuación del tratamiento, el control de las políticas de antimicrobianos, la detección de nuevos mecanismos de resistencia y el conocimiento de su epidemiología11. Un requisito esencial para poder realizar una adecuada lectura interpretada es conocer la identidad del microorganismo estudiado, tanto el género como la especie, ya que sin ella el resultado puede llevar a errores en la utilización de los antimicrobianos. Así, una cepa de S. aureus con CMI de cloxacilina de 1 mg/l es sensible a cloxacilina y a todos los β-lactámicos, mientras que si se trata de un estafilococo coagulasa negativa la CMI de 1 mg/l indica resistencia a cloxacilina. Otro ejemplo es que una CMI de ampicilina de 8 mg/l frente a una enterobacteria indica sensibilidad, pero frente a un estafilococo esta misma CMI indica resistencia, ya que un estafilococo es ya resistente a ampicilina con una CMI de 0,5 mg/l. De esta manera, CMI más bajas no siempre indican mayor actividad y, además, son variables dependiendo del microorganismo y del antibiótico, como se ha indicado anteriormente. También hay otros casos de microorganismos que, aunque pertenecen al mismo género, presentan mecanismos de resistencia diferentes, como es el caso de Proteus vulgaris que es siempre resistente a ampicilina, mientras que Proteus mirabilis es generalmente sensible; o el de Citrobacter freundii que siempre es resistente a ampiclina, amoxicilina-clavulánico y a cefalosporinas de primera y segunda generación, mientras que Citrobacter koseri es siempre resistente a ampicilina pero no a amoxicilina-clavulánico.
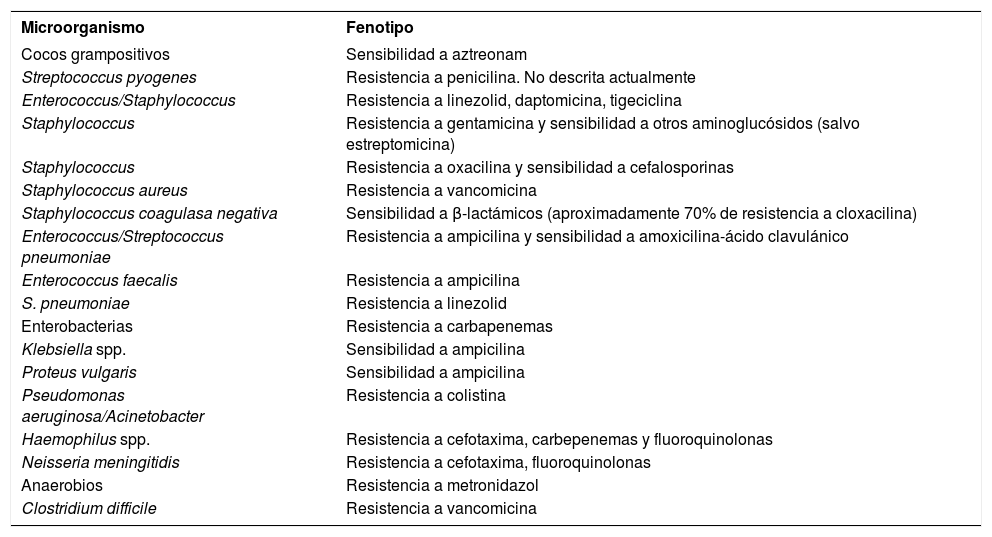
Otro requisito para poder realizar correctamente la lectura interpretada del antibiograma es conocer el fenotipo de sensibilidad de un microorganismo, ya que hay bacterias que siempre son resistentes a determinados antibióticos y otras que siempre son sensibles, y la desviación de estos patrones indica si el patrón del antibiograma corresponde a un fenotipo habitual, raro o imposible (tabla 1)12,13. Los fenotipos habituales son los aislamientos con mecanismos de resistencia cuya presencia es epidemiológicamente normal en el medio donde se realiza el antibiograma. Un ejemplo de ello son la resistencia a penicilina y sensibilidad a cloxacilina en un aislado de S. aureus. Los fenotipos raros son los que presentan resistencias poco habituales, bien porque han sido recientemente caracterizadas o porque son muy poco frecuentes en nuestro medio. Un ejemplo de los primeros es la resistencia a imipenem en Enterobacter cloacae14, y de los segundos las cepas de enterococo resistentes a la vancomicina15. Finalmente, los fenotipos imposibles no responden a mecanismos de resistencia conocidos y, por tanto, es necesaria su comprobación. Estos fenotipos imposibles, en muchas ocasiones, representan un error en la identificación del microorganismo o bien problemas técnicos en la realización del antibiograma, pero también hay que tener en cuenta que la repetición de estos fenotipos en bacterias correctamente identificadas puede suponer un nuevo mecanismo de resistencia, tal es el caso de la resistencia a linezolid en enterococos16.
Fenotipos de resistencia raros o imposibles
| Microorganismo | Fenotipo |
|---|---|
| Cocos grampositivos | Sensibilidad a aztreonam |
| Streptococcus pyogenes | Resistencia a penicilina. No descrita actualmente |
| Enterococcus/Staphylococcus | Resistencia a linezolid, daptomicina, tigeciclina |
| Staphylococcus | Resistencia a gentamicina y sensibilidad a otros aminoglucósidos (salvo estreptomicina) |
| Staphylococcus | Resistencia a oxacilina y sensibilidad a cefalosporinas |
| Staphylococcus aureus | Resistencia a vancomicina |
| Staphylococcus coagulasa negativa | Sensibilidad a β-lactámicos (aproximadamente 70% de resistencia a cloxacilina) |
| Enterococcus/Streptococcus pneumoniae | Resistencia a ampicilina y sensibilidad a amoxicilina-ácido clavulánico |
| Enterococcus faecalis | Resistencia a ampicilina |
| S. pneumoniae | Resistencia a linezolid |
| Enterobacterias | Resistencia a carbapenemas |
| Klebsiella spp. | Sensibilidad a ampicilina |
| Proteus vulgaris | Sensibilidad a ampicilina |
| Pseudomonas aeruginosa/Acinetobacter | Resistencia a colistina |
| Haemophilus spp. | Resistencia a cefotaxima, carbepenemas y fluoroquinolonas |
| Neisseria meningitidis | Resistencia a cefotaxima, fluoroquinolonas |
| Anaerobios | Resistencia a metronidazol |
| Clostridium difficile | Resistencia a vancomicina |
Cefotaxima es equivalente en cuanto a actividad antibacteriana a ceftriaxona y ampicilina a amoxicilina.
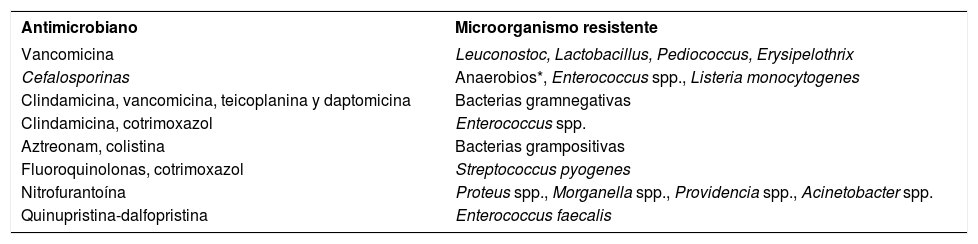
En la tabla 1 se indican algunos ejemplos de fenotipos habituales, raros e imposibles, y en la tabla 2 algunos casos de bacterias intrínsecamente resistentes a ciertos antibióticos. En un próximo capítulo, en el que se analizarán con mayor detalle los fenotipos de sensibilidad más comunes, se expondrán algunos ejemplos de lectura interpretada del antibiograma.
Microorganismos intrínsecamente resistentes a ciertos antibióticos
| Antimicrobiano | Microorganismo resistente |
|---|---|
| Vancomicina | Leuconostoc, Lactobacillus, Pediococcus, Erysipelothrix |
| Cefalosporinas | Anaerobios*, Enterococcus spp., Listeria monocytogenes |
| Clindamicina, vancomicina, teicoplanina y daptomicina | Bacterias gramnegativas |
| Clindamicina, cotrimoxazol | Enterococcus spp. |
| Aztreonam, colistina | Bacterias grampositivas |
| Fluoroquinolonas, cotrimoxazol | Streptococcus pyogenes |
| Nitrofurantoína | Proteus spp., Morganella spp., Providencia spp., Acinetobacter spp. |
| Quinupristina-dalfopristina | Enterococcus faecalis |