Las infecciones genitales femeninas suponen el 20-30% de las consultas ginecológicas; eso es así por la morbilidad que ocasionan y por su importante incidencia en nuestro medio: el 75% de las mujeres experimentan un episodio de vulvovaginitis en toda su vida, que será recidivante hasta en el 50% de la población femenina1,2 y al menos el 40% de las mujeres asistidas en las Unidades de Infecciones de Transmisión Sexual presentan clínica con signos y síntomas de cervicitis3. El objetivo del estudio fue analizar la epidemiología de los microorganismos encontrados en la población general. Se realizó un estudio transversal, descriptivo de carácter retrospectivo, en el que se incluyeron los resultados emitidos por el Laboratorio de Microbiología, tras el procesamiento de todas las muestras genitales de mujeres con diagnóstico presuntivo de un episodio de infección, entre el 1 de enero de 2016 y 31 de diciembre de 2018. Se evaluaron los resultados obtenidos tras el procesamiento de estas, mediante cultivo y/o técnicas moleculares (PCR o hibridación), de agentes productores de infección genital, sin incluir el estudio de infección por Treponema pallidum. Todas las muestras fueron procesadas siguiendo los protocolos previamente descritos2,4–6. Se recogieron las variables tipo de muestra, prueba de laboratorio, resultados y datos demográficos anonimizados, para su posterior evaluación. Para las muestras diferentes de una misma paciente con identificación del mismo microorganismo en un periodo de 30 días o menos se consideró un solo episodio infeccioso. Se realizó un análisis estadístico descriptivo, calculando frecuencias absolutas y relativas para las variables cualitativas, así como medidas de tendencia central para las cuantitativas. Los datos se analizaron con el software IBM SPSS Statistics 19.
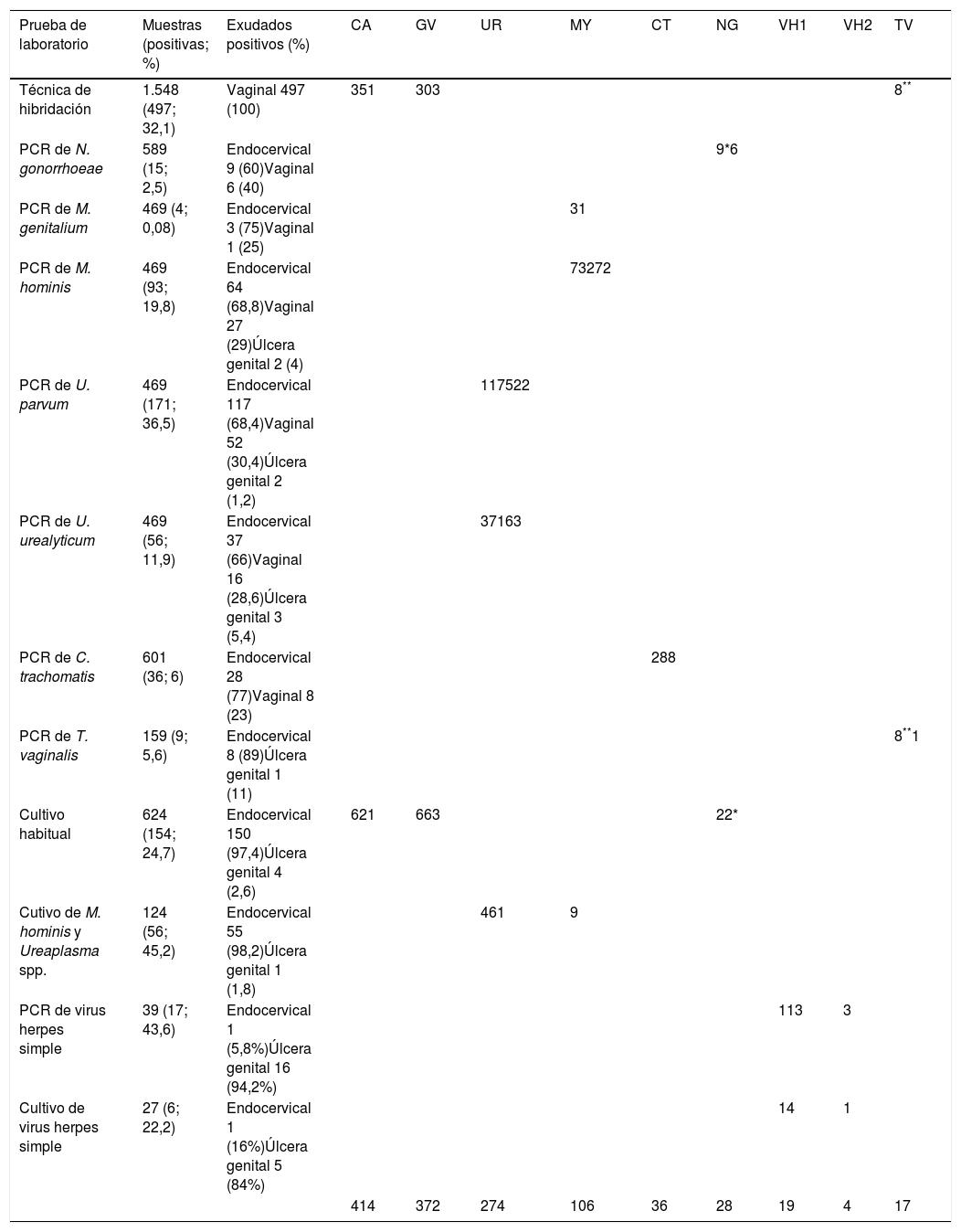
Durante los tres años estudiados se realizaron 5.587 pruebas de laboratorio (tabla 1) de mujeres con diagnóstico de sospecha de infección del tracto genital (1.722 episodios), con una edad media de 32,88 años (rango entre 16 años y 95). Del total de las muestras se obtuvo un resultado positivo para algún microorganismo patógeno en 1.114 (19,9%), 497 (44,6% de positivos) mediante prueba de hibridación y 154 (13,8%) mediante cultivo habitual en medio artificial para recuperar microorganismos que crecen formado colonias. Mediante hibridación los microorganismos detectados con más frecuencia fueron Candida spp. (351 episodios; 70,6%) y Gardnerella vaginalis (G. vaginalis) (303; 61%). En cuanto al cultivo habitual, los microorganismos aislados con más frecuencia fueron esta (69 episodios; 44,8% de los cultivos positivos) y Candida spp. (63; 40,9%). Otros microorganismos detectados en menor frecuencia fueron Candida glabrata (C. glabrata) 16 (25,4%) y Neisseria gonorrhoeae 22 (14,3%) episodios. Por último, los virus herpes simplex se investigaron mediante cultivo, antes de 2018, o mediante PCR, durante 2018, cuando se solicitaron, detectándose en el 34,8% de las muestras analizadas; el 82,6% correspondieron a virus herpes simplex tipo 1, mientras que el resto al tipo 2. Al comparar la edad de las pacientes según los diferentes microorganismos detectados, se encontró una mayor edad en el grupo con Trichomonas vaginalis y una menor edad en el grupo con presencia de virus herpes simplex tipo 1 y Chlamydia trachomatis, con medias de 36,6+12,2, 23,9+5,7 y 25,0+7,4 años, respectivamente, si bien esta fue mayor en pacientes con C. glabrata (40 años). El resto de microorganismos ocupan una posición intermedia en las edades de los pacientes. Estos resultados evidencian dos aspectos: por un lado la necesidad de estudios protocolizados amplios, que no se limiten a la investigación molecular; y por otro, que la detección de microorganismos, con G. vaginalis y Candida spp. encabezando la mayor presencia seguidas por Ureaplasma spp. y Mycoplasma hominis, sigue la misma línea que otros estudios individuales publicados.
Infección genital femenina. Distribución de las pruebas empleadas, muestras estudiadas en el laboratorio de microbiología y microorganismos detectados
| Prueba de laboratorio | Muestras (positivas; %) | Exudados positivos (%) | CA | GV | UR | MY | CT | NG | VH1 | VH2 | TV |
|---|---|---|---|---|---|---|---|---|---|---|---|
| Técnica de hibridación | 1.548 (497; 32,1) | Vaginal 497 (100) | 351 | 303 | 8** | ||||||
| PCR de N. gonorrhoeae | 589 (15; 2,5) | Endocervical 9 (60)Vaginal 6 (40) | 9*6 | ||||||||
| PCR de M. genitalium | 469 (4; 0,08) | Endocervical 3 (75)Vaginal 1 (25) | 31 | ||||||||
| PCR de M. hominis | 469 (93; 19,8) | Endocervical 64 (68,8)Vaginal 27 (29)Úlcera genital 2 (4) | 73272 | ||||||||
| PCR de U. parvum | 469 (171; 36,5) | Endocervical 117 (68,4)Vaginal 52 (30,4)Úlcera genital 2 (1,2) | 117522 | ||||||||
| PCR de U. urealyticum | 469 (56; 11,9) | Endocervical 37 (66)Vaginal 16 (28,6)Úlcera genital 3 (5,4) | 37163 | ||||||||
| PCR de C. trachomatis | 601 (36; 6) | Endocervical 28 (77)Vaginal 8 (23) | 288 | ||||||||
| PCR de T. vaginalis | 159 (9; 5,6) | Endocervical 8 (89)Úlcera genital 1 (11) | 8**1 | ||||||||
| Cultivo habitual | 624 (154; 24,7) | Endocervical 150 (97,4)Úlcera genital 4 (2,6) | 621 | 663 | 22* | ||||||
| Cutivo de M. hominis y Ureaplasma spp. | 124 (56; 45,2) | Endocervical 55 (98,2)Úlcera genital 1 (1,8) | 461 | 9 | |||||||
| PCR de virus herpes simple | 39 (17; 43,6) | Endocervical 1 (5,8%)Úlcera genital 16 (94,2%) | 113 | 3 | |||||||
| Cultivo de virus herpes simple | 27 (6; 22,2) | Endocervical 1 (16%)Úlcera genital 5 (84%) | 14 | 1 | |||||||
| 414 | 372 | 274 | 106 | 36 | 28 | 19 | 4 | 17 |
CA: Candida spp.; CT: Chlamydia trachomatis; GV: Gardnerella vaginalis; MY: Mycoplasma spp.; NG: Neisseria gonorrhoeae; TV: Trichomonas vaginalis; UR: Ureaplasma spp.; VH: virus herpes simple.
Este fue un estudio no intervencionista, con ninguna investigación adicional a los procedimientos rutinarios. El material biológico se utilizó solo para el diagnóstico estándar de infecciones del tracto genital, siguiendo las prescripciones de los médicos. No se realizó muestreo adicional ni modificación del protocolo diagnóstico de rutina. Se realizaron los análisis de datos utilizando una base de datos anónima. La entidad que concedió el permiso para acceder y utilizar los datos fue la Unidad de Gestión Clínica de Microbiología Clínica del Hospital Virgen de las Nieves de Granada, España.