La diabetes mellitus tipo 2 (DM2) se define genéticamente como una enfermedad compleja originada por la interacción del medio ambiente y los genes, que se encuentran ubicados en diferentes regiones del genoma humano. El gen calpaína 10 (CAPN10), localizado en 2q37.3, constituye un gen de susceptibilidad para esta enfermedad. La asociación alélica y haplotípica observada de los polimorfismos de nucleótido simple (SNP) 19, 43 y 63 en poblaciones amerindias y algunas europeas confirmaría estos resultados.
En esta investigación se postuló que los SNP 19, 43 y 63 del gen CAPN10 estaban asociados a DM2 en la población peruana. Se analizaron 45 pacientes diabéticos y 58 familiares no diabéticos. Además de los factores de riesgo como la obesidad, el índice de masa corporal (IMC), la hipertrigliceridemia (HTG), la hipertensión arterial (HTA) y la hipercolesterolemia (HC), se identificaron -mediante reacción en cadena de la polimerasa (PCR)los SNP 19, 43 y 63 del CAPN10. Se analizaron los resultados utilizando las pruebas de equilibrio de Hardy-Weinberg y el análisis de regresión logística condicional.
Se observó asociación del SNP19 a DM2 (x2=8,31; p=0,01) e IMC, HTG e HC en familias diabéticas peruanas. El aporte de la investigación radica en haber detectado estas asociaciones en la población diabética peruana.
Type 2 diabetes mellitus (T2DM) is genetically defined as a complex disease caused by the interaction of environment and genes, which are located in different regions from the human genome. Calpain 10 (CAPN10) is a susceptibility gene for the disease, located in 2q37.3. The allelic and haplotypic association observed of SNP's 19, 43 and 63 in Amerindian and some European populations would confirm these results.
In this research, it has been postulated that SNP's 19, 43 and 63 of CAPN10 gene were associated with T2DM in Peruvian population. It has been analyzed 45 diabetic patients and 58 non-diabetic relatives. Among other risk factor for obesity, such body mass index (BMI), hypertriglyceridemia (HTG), arterial hypertension (HTA), and hypercholesterolemia (HC), it has been identified by polymerase chain reaction (PCR) SNP's 19, 43 and 63 of CAPN10. The Hardy-Weinberg test and conditional regression logistic were used to analyze the results.
The results showed association of SNP19 with T2DM (x2=8,31; p=0,01) and BMI, HTG, HC in Peruvian diabetic families. The main contribution of this research is having detected these associations in the diabetic Peruvian population.
calpaína 10;
diabetes mellitus tipo 2;
hipercolesterolemia;
hipertensión arterial;
hipertrigliceridemia;
equilibrio de Hardy-Weinberg;
índice de masa corporal;
reacción en cadena de la polimerasa;
polimorfismo de nucleótido simple.
La diabetes mellitus tipo 2 (DM2) es la forma más frecuente de diabetes (corresponde aproximadamente al 90% del total de casos), y se caracteriza por resistencia a la insulina y deficiencia relativa en la acción y en la secreción de dicha hormona1.
Al igual que otras enfermedades complejas, la tasa de prevalencia de DM2 depende del grupo étnico en estudio y también de los genes candidatos de esta enfermedad. Seclen et al.2 realizaron estudios en población peruana sobre prevalencia de la DM2, que resultó ser mayor en la población urbana de la costa (7,6%) comparado con la población urbana de la sierra (1,3%). Los estudios en población peruana confirman lo observado en otras investigaciones, relacionando la prevalencia de DM2 y el componente genético de la población: 20% en mexicano-americanos, 15% en personas de origen africano y 11,9% en poblaciones de origen caucásico3.
Los estudios genéticos de asociación han permitido encontrar aproximadamente 21 genes candidatos de DM24, si bien sólo tres o cuatro de ellos han presentado resultados reproducibles en diferentes poblaciones4,5.
De los estudios llevados a cabo hasta la fecha, uno de los más interesantes fue realizado por Hanis6, quien encontró evidencias de ligamiento y, en consecuencia, la presencia de un gen de susceptibilidad en el cromosoma 2, que fue observado en población mexicana.
Posteriormente, Horikawa et al.7 delimitaron aún más la región y encontraron que, de los tres genes candidatos, sólo calpaína 10 (CAPN10) presentó asociación entre las frecuencias haplotípicas de los polimorfismos de nucleótido simple (SNP) 19, 43 y 63 y la DM2 en un 20-30% de la población pima y mexicano-americana, y en un 3% en población finlandesa y alemana. Esto llevó a postular el CAPN10 como un gen de susceptibilidad de DM2.
Se ha comprobado, a nivel celular, que CAPN10 interviene en la regulación de varias funciones celulares, incluyendo señales reguladas por calcio, proliferación, diferenciación y apoptosis8, expresándose en los tejidos fetales y adultos9. Se conoce que una de las funciones más importantes del CAPN10 es facilitar la translocación del transportador de glucosa 4 que responde a insulina (GLUT4), e interviene en la liberación de insulina actuando sobre la glucosa en tejidos periféricos. El gen CAPN10 interviene en un proceso esencial para mantener la homeostasis de la glucosa10, corroborando su función y su relación con la etiología de la DM2.
Asimismo, otros antecedentes demuestran que los mexicanoamericanos son más propensos a la obesidad, la DM2, las dislipidemias y otras complicaciones metabólicas3, y que esta población, al igual que la peruana, está caracterizada por un incremento de la adiposidad y la distribución centralizada de la grasa corporal2.
Considerando la cercanía filogenética entre las poblaciones pima, mexicano-americana11 y peruana, así como los antecedentes que relacionan la DM2 y los factores de riesgo, se postula al CAPN10 como un gen de susceptibilidad de DM2 en la población peruana.
Por lo anteriormente expuesto, se propuso analizar 45 personas diabéticas y 58 familiares no diabéticos que presentaban factores de riesgo asociados a esta enfermedad. Además, se analizaron las 45 personas diabéticas frente a 123 individuos control sin antecedentes de DM2 u otro factor de riesgo.
Material y métodoPoblación en estudioSe obtuvo la participación de 45 diabéticos y 58 familiares no diabéticos con más de un familiar afectado y diagnosticado de DM2, provenientes del Hospital Arzobispo Loayza y el Hospital Edgardo Rebagliati Martins, además de 123 individuos control (personas sin relación de parentesco con los afectados y sus familiares y, a su vez, sin antecedentes familiares de DM2 u otros factores de riesgo). Los pacientes, familiares y controles llenaron una ficha de captación de datos y el consentimiento informado aprobado por los comités de ética de la Universidad de San Martín de Porres (USMP) y los respectivos hospitales. Los participantes en esta investigación fueron mayoritariamente mestizos (amerindios y caucásicos).
Al mismo tiempo, se analizaron el índice de masa corporal (IMC) (≥25kg/m2 según criterios de la OMS)12, la obesidad abdominal (definida como un valor del perímetro abdominal >102cm en el hombre y >88cm en la mujer)3, la hipertensión arterial (HTA) (definida como presión arterial ≥140/90mmHg según criterios de la OMS)13, la hipercolesterolemia (HC) (definida como colesterol total >200mg/dL) y la hipertrigliceridemia (HTG) (definida como triglicéridos >150mg/dL)14.
Análisis molecularA partir de una muestra de ADN, se realizó la amplificación de los SNP 19 (intrón 6), 43 (intrón 3) y 63 (intrón 13) del gen CAPN10 utilizando los protocolos de amplificación descritos por Evans et al.15 y que fueron los siguientes:
SNP-19 (CAPN10-g.7920 indel 32bp). Se realizó la mezcla de reacción en cadena de la polimerasa (PCR) en un volumen de 10 μL que contenía buffer de PCR 1X, 200 μimol de cada dNTP, 1,5mmol de MgCl2, 5% de dimetilsulfóxido, 250nmol de cada primer, 0,25 UI de AmpliTaq Gold® (Applied Biosystems) y 40ng de ADN genómico. Las condiciones del protocolo de PCR fueron 94°C durante 12 minutos, 35 ciclos de 94°C durante 30 segundos, 60°C durante 30 segundos y 72°C durante 30 segundos, y 72°C durante 10 minutos. Los productos de PCR se separaron en un gel de agarosa al 2%. El alelo 1 mostró dos repeticiones de una secuencia de 32 pb y el amplificado tenía un tamaño de 155 pb, y el alelo 2 presentó tres repeticiones que sumaban 187 pb.
SNP-43 (CAPN10-g.4852G/A). Se realizó el protocolo de PCR en un volumen total de 10μL que contenía buffer de PCR 1X, 200 μimol de cada dNTP, 1,5mol de MgCl2, 0,25 UI de AmpliTaq Gold® (Applied Biosystems) y 40ng de ADN genómico. Las condiciones de PCR fueron 96°C durante 12 minutos, 35 ciclos de 94°C durante 30 segundos, 57°C durante 30 segundos y 72°C durante 30 segundos y 72°C durante 10 minutos. Los productos de PCR fueron separados en un gel de agarosa al 2%. El alelo 1(G) fue de 134 pb y el alelo 2(A) de 152 pb.
SNP-63 (CAPN10-g.16378 C/T). Las condiciones de PCR fueron similares a las del SNP-19, excepto en que la temperatura de alineamiento fue de 62°C. Los productos de PCR se digirieron con 2 UI de la enzima de restricción Hhal (NEB) en buffer plus NE4 1X y albúmina de suero bovino al 1X, a 37°C durante 2 horas. Luego, estos productos de digestión fueron separados en gel de agarosa al 2%. El alelo 1(C) fue de 162 pb y el alelo 2(T) de 192 pb.
Este análisis fue obtenido en el Centro de Genética y Biología Molecular del Instituto de Investigación de la Facultad de Medicina Humana de la USMP.
Análisis estadísticoSe realizó la prueba de equilibrio de Hardy-Weinberg (H-W), validada con una ji al cuadrado (x2) de Pearson y de razón de verosimilitud, para determinar si las poblaciones eran genéticamente homogéneas en estos marcadores. Asimismo, se realizó el análisis de frecuencias alélicas y de regresión logística condicional entre los diabéticos y sus familiares, y entre los diabéticos y los controles. En ambos casos se llevó a cabo con el programa STATA 8.0 (StataCorp., 2003).
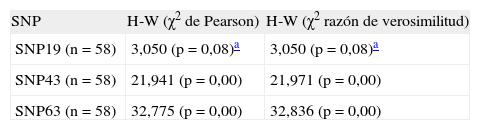
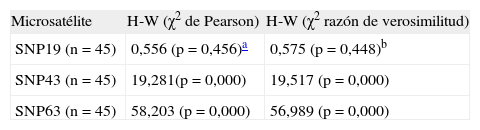
ResultadosLas tablas 1 y 2 presentan los resultados de la prueba de equilibrio de Hardy-Weinberg (H-W) en los individuos diabéticos y no diabéticos (familiares de primer grado del individuo afectado) para los SNP 19, 43 y 63, observándose que solamente el SNP19 se encontraba en equilibrio H-W tanto en los individuos diabéticos como en los familiares no diabéticos. Estos resultados permitieron demostrar que los diabéticos y sus familiares eran genéticamente homogéneos para el SNP19, y se continuó con los posteriores análisis sólo para este SNP.
Equilibrio de Hardy-Weinberg (H-W) en familiares no diabéticos, utilizando los polimorfismos de nucleótido simple (SNP) 19, 43 y 63 del gen calpaína 10 (CAPN10)
Equilibrio de Hardy-Weinberg (H-W) en diabéticos, utilizando los polimorfismos de nucleótido simple (SNP) 19, 43 y 63 del gen calpaína 10 (CAPN10)
| Microsatélite | H-W (χ2 de Pearson) | H-W (χ2 razón de verosimilitud) |
| SNP19 (n=45) | 0,556 (p=0,456)a | 0,575 (p=0,448)b |
| SNP43 (n=45) | 19,281(p=0,000) | 19,517 (p=0,000) |
| SNP63 (n=45) | 58,203 (p=0,000) | 56,989 (p=0,000) |
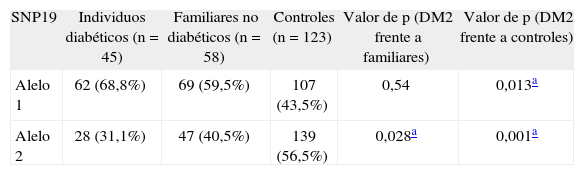
La tabla 3 presenta la frecuencia alélica del SNP19 en individuos diabéticos frente a familiares no diabéticos, y en diabéticos frente a controles. Al comparar las frecuencias alélicas de los diabéticos frente a las de los familiares no diabéticos, se demostró una diferencia estadísticamente significativa en el alelo 2 (p=0,028), pero no en el alelo 1. Además, se observa que el análisis de frecuencias alélicas de diabéticos frente a controles presenta una diferencia estadísticamente significativa en el alelo 1 (p=0,013) y en el alelo 2 (p=0,001). Esto indicaría que el SNP19 del CAPN10 estaría asociado a DM2 y que el problema observado en el alelo 1 al comparar diabéticos frente a familiares no diabéticos se debería al tamaño de la muestra; esto fue corroborado al comparar con el grupo control poblacional.
Análisis de frecuencias alélicas del SNP19 en diabéticos, familiares no diabéticos y controles de la población peruana
Posteriormente se realizó el análisis de regresión logística entre 45 individuos diabéticos y 58 familiares no diabéticos, observándose una asociación entre el SNP19 y la DM2 (x2=8,31; p=0,01). Además, se comparó a los 45 diabéticos con los 123 individuos control, analizándose la población control peruana con la prueba de equilibrio de H-W para el SNP19 (x2=1,176; p=0,758; x2=1,545; p=0,672). Los resultados de homogeneidad genética del SNP19 permitieron continuar con el análisis de regresión logística, observándose la asociación significativa entre el SNP19 y la DM2 (x2=17,99; p=0,001).
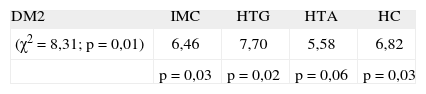
La tabla 4 presenta la asociación del SNP19 con los factores de riesgo asociados a DM2. El análisis de regresión logística permitió analizar el SNP19 asociado a obesidad, HTG, HTA e HC, encontrándose una asociación del IMC (x2=6,46; p=0,03), la HTG (X2=7,70; p=0,02) y la HC (x2=6,82; p=0,03) con el SNP19.
Asociación entre SNP19 del gen calpaína 10 (CAPN10) y factores de riesgo en pacientes diabéticos (n=45) y familiares no diabéticos (n=58)
| DM2 | IMC | HTG | HTA | HC |
| (χ2=8,31; p=0,01) | 6,46 | 7,70 | 5,58 | 6,82 |
| p=0,03 | p=0,02 | p=0,06 | p=0,03 |
p <0,05 (significativo).
HC: hipercolesterolemia; HTA: hipertensión arterial; HTG: hipertrigliceridemia; IMC: índice de masa corporal.
Se considera importante la prueba de H-W porque permite realizar una adecuada estratificación de la población16 y determinar si ésta es genéticamente homogénea. Los resultados del H-W del SNP19 permitieron continuar con los estudios de asociación entre este marcador y la DM2.
Un aporte de esta investigación ha sido determinar que el SNP19 está asociado a la DM2 en la población peruana (un hecho observado tanto para el alelo 1 como para el alelo 2), indicando que este marcador intrónico se encuentra en el gen CAPN10, el cual está asociado a la enfermedad pero que ninguno de los dos alelos puede ser discriminado como alelo de susceptibilidad o de protección (tabla 3). Publicaciones recientes señalan que las frecuencias alélicas de los SNP pueden variar drásticamente entre diferentes grupos étnicos. Spielman et al.17 sostienen que la expresión de genes individuales varía en diferentes grupos étnicos y que el cambio en la frecuencia de estos genes modificaría la expresión génica de los mismos. Incluso, se ha estimado que altos niveles del índice de fijación (FST) están relacionados con una mejor adaptación y conservación de este marcador, y bajos niveles de FST con una selección neutral o negativa. En consecuencia, los SNP mejor adaptados se encuentran más frecuentemente en regiones codificantes que en no codificantes18. Es probable que altos valores de FST tengan una selección positiva y una mayor conservación de las frecuencias alélicas de un marcador molecular asociado a una enfermedad, y que las variaciones de estas frecuencias se relacionen con la prevalencia o la progresión de la enfermedad. En cambio, el marcador SNP19, al encontrarse en una región intrónica (no codificante), tendría una presión de selección neutral o negativa en la población peruana y, a su vez, esto podría explicar que no se observen diferencias entre los alelos 1 y 2 de este marcador.
El análisis de regresión logística presenta la asociación entre la DM2 y el SNP19 en población peruana (tabla 4). Antecedentes similares han sido observados entre este marcador y la DM2 en población mexicano-americana6. Otros autores, en cambio, han encontrado asociación entre el SNP43 y la DM2 en población europea19, y del SNP44 en población turca20. Sin embargo, los SNP 19, 43, 44 y 63 no estarían asociados a esta enfermedad en población finlandesa21. Estos resultados aparentemente contradictorios se relacionan con el origen étnico de las poblaciones estudiadas, el tamaño de la muestra y la posición del marcador en el gen a estudiar18.
Otros investigadores han relacionado el gen CAPN10 con la función microvascular22, el metabolismo de los adipocitos, la función del adrenorreceptor beta y la liberación de glucosa23,24. En algunas poblaciones, los SNP 43 y 44 están asociados a obesidad y a obesidad abdominal en diabéticos25, pero en otras poblaciones no26. Asimismo, el genotipo 1/1 del SNP19 estaría asociado a una disminución de la actividad lipolítica, y el genotipo 2/2 a un IMC elevado27. Estos antecedentes sustentan la asociación de los SNP dependiendo del origen étnico de la población. Esto indicaría que no todos los SNP localizados en CAPN10 estarían asociados a la DM2, y que para algunas poblaciones este gen sería importante en la etiología de la enfermedad y para otras no7,26. Los resultados (recogidos en la tabla 4) que asocian el IMC y la HC al SNP19 se sustentan en lo observado en población japonesa, pero la asociación del SNP19 con la HTA no ha sido demostrada en otra población; por tanto, sería necesario realizar un análisis con una mayor población para confirmar estos resultados.
Los antecedentes anteriormente expuestos relacionan el gen CAPN10 con la DM2 y el IMC, la HTA y la HC; esto, a su vez, indica que este gen es compartido en vías metabólicas comunes que involucran a estas enfermedades.
En resumen, el SNP19 estaría asociado a la DM2 y a los factores de riesgo IMC, HTA y HC en población diabética peruana. Estos resultados se explicarían por el origen multifactorial de esta enfermedad y por las diferencias genéticas entre los grupos étnicos. La asociación observada en distintas poblaciones y en la peruana sustentaría la idea del CAPN10 como un gen candidato de DM24,5,28. Es probable que las variaciones en los intrones de CAPN10 (que modifican la expresión del ARNm en el músculo esquelético29) sustentan la idea de una variación similar del SNP19 asociando así esta variación con la expresión diferencial del CAPN10 en los pacientes diabéticos estudiados.
AgradecimientosEste proyecto ha sido financiado por la Facultad de Medicina Humana de la Universidad de San Martín de Porres y el Consejo Nacional de Ciencia y Tecnología (CONCYTEC), PROCYT-N.° 155 (2006–2007). ¿
Declaración de potenciales conflictos de interesesM. Paredes, F. Lizaraso, R. Lissón, E. Giovanna Rodríguez, J. Calderón y J. Huapaya declaran que no existen conflictos de intereses en relación con el contenido del presente artículo.