Justificación: La endocarditis infecciosa (EI) es una enfermedad grave, de diagnóstico complejo y mortalidad elevada. La microbiología constituye el pilar fundamental del diagnóstico etiológico. En ocasiones, la microbiología clásica no permite identificar el microorganismo causal. Las técnicas de amplificación de ácidos nucleicos (TAAN) de secuenciación directa de tejido valvular de bacterias y hongos (PCR universal) pueden optimizar el diagnóstico etiológico.
Objetivo: Comprobar en nuestro centro la utilidad de la secuenciación directa de bacterias y hongos a partir de tejido valvular en el diagnóstico etiológico de la EI, comparando con un grupo control.
Método: De enero a junio de 2017 se recogieron muestras de válvulas de pacientes con EI (grupo estudio) y pacientes sin EI intervenidos por disfunción valvular (grupo control).
Se realizó el cultivo microbiológico habitual y las TAAN.
La extracción de ADN se realizó con el kit Virus/Pathogen Mini (QiaSymphony®, QIAGEN®). La PCR universal se realizó con los primers PSL y P13P (gen 16S rRNA de bacterias) y 18S-1 y 18S-2 (gen 18S rRNA de hongos). Se amplificó el gen de la β-globina humana (primers β-gloF y β-gloR) para detectar inhibiciones y fallos en la extracción. Los amplicones se purificaron (QIAquickPCR Purification kit QIAGEN®) y se realizó la PCR de secuenciación empleando los mismos primers que en la amplificación. El producto se precipitó (Agencourt®CleanSEQ®) y secuenció en el equipo GenomeLabTM Genetic Analysis System. Las secuencias obtenidas se cotejaron en la web de GenBank a través del software BLAST.
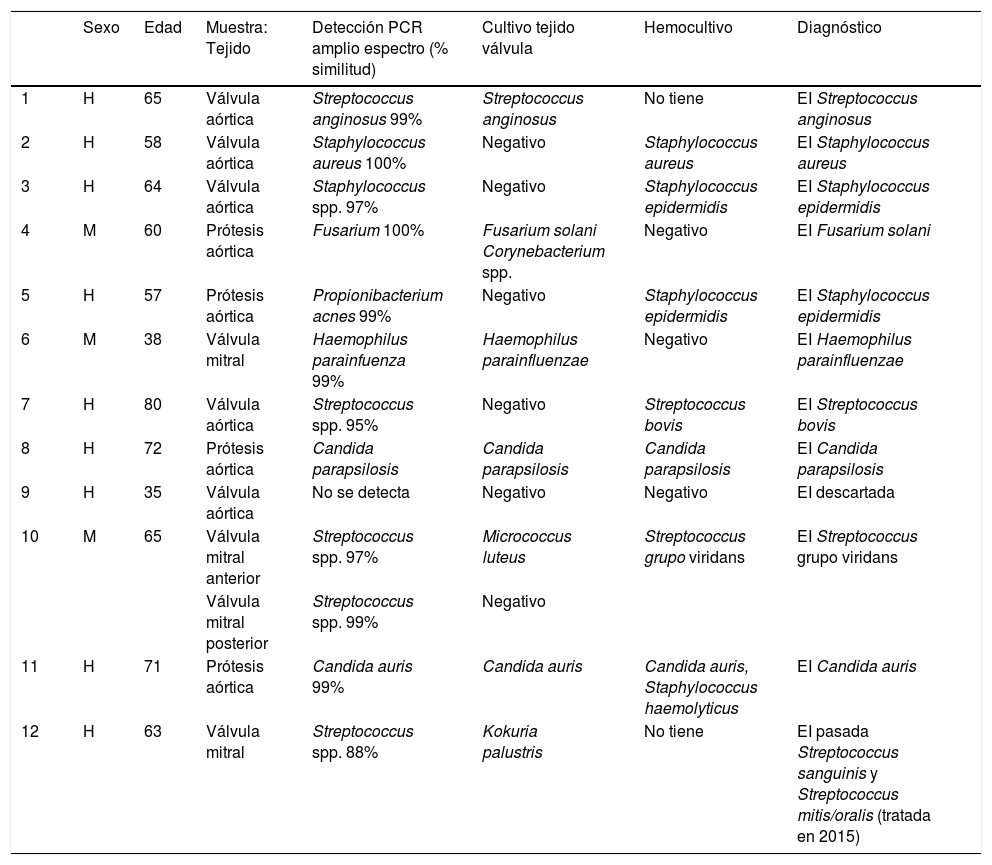
Resultados: Se recogieron 11 muestras de pacientes con EI sospechada o confirmada y un paciente con EI pasada (tabla 1).
Grupo de estudio
| Sexo | Edad | Muestra: Tejido | Detección PCR amplio espectro (% similitud) | Cultivo tejido válvula | Hemocultivo | Diagnóstico | |
|---|---|---|---|---|---|---|---|
| 1 | H | 65 | Válvula aórtica | Streptococcus anginosus 99% | Streptococcus anginosus | No tiene | EI Streptococcus anginosus |
| 2 | H | 58 | Válvula aórtica | Staphylococcus aureus 100% | Negativo | Staphylococcus aureus | EI Staphylococcus aureus |
| 3 | H | 64 | Válvula aórtica | Staphylococcus spp. 97% | Negativo | Staphylococcus epidermidis | EI Staphylococcus epidermidis |
| 4 | M | 60 | Prótesis aórtica | Fusarium 100% | Fusarium solani Corynebacterium spp. | Negativo | EI Fusarium solani |
| 5 | H | 57 | Prótesis aórtica | Propionibacterium acnes 99% | Negativo | Staphylococcus epidermidis | EI Staphylococcus epidermidis |
| 6 | M | 38 | Válvula mitral | Haemophilus parainfuenza 99% | Haemophilus parainfluenzae | Negativo | EI Haemophilus parainfluenzae |
| 7 | H | 80 | Válvula aórtica | Streptococcus spp. 95% | Negativo | Streptococcus bovis | EI Streptococcus bovis |
| 8 | H | 72 | Prótesis aórtica | Candida parapsilosis | Candida parapsilosis | Candida parapsilosis | EI Candida parapsilosis |
| 9 | H | 35 | Válvula aórtica | No se detecta | Negativo | Negativo | EI descartada |
| 10 | M | 65 | Válvula mitral anterior | Streptococcus spp. 97% | Micrococcus luteus | Streptococcus grupo viridans | EI Streptococcus grupo viridans |
| Válvula mitral posterior | Streptococcus spp. 99% | Negativo | |||||
| 11 | H | 71 | Prótesis aórtica | Candida auris 99% | Candida auris | Candida auris, Staphylococcus haemolyticus | EI Candida auris |
| 12 | H | 63 | Válvula mitral | Streptococcus spp. 88% | Kokuria palustris | No tiene | EI pasada Streptococcus sanguinis y Streptococcus mitis/oralis (tratada en 2015) |
En 11 de los 12 casos hubo concordancia con el diagnóstico microbiológico clásico. En el caso 12 (EI pasada) el cultivo de válvula se consideró contaminado, pero se detectó ADN de Streptococcus spp. (aunque con porcentaje bajo de similitud). En los casos 1, 4 y 6 (hemocultivo negativo o no recogido) las TAAN coincidieron con el cultivo del tejido, afianzando el diagnóstico etiológico. En 5 muestras se detectó ADN de Streptococcus spp., pero solo en una se llegó a la identificación de especie.
En el grupo control se recogieron 17 muestras. En 5 no amplificó el gen de la β-globina, lo que invalidó el resultado de las TAAN. En las otras 12 el resultado fue negativo. Respecto al cultivo, fue positivo en un caso (Staphylococcus epidermidis, considerado contaminación).
Conclusiones: Nuestra experiencia, todavía muy preliminar, concuerda con lo descrito en la literatura:
- •
La secuenciación directa de tejido valvular presenta una elevada concordancia con el cultivo microbiológico clásico. Nos permite el diagnóstico etiológico cuando no se disponen de hemocultivos o son negativos.
- •
La PCR del gen 16S es insuficiente para diferenciar Streptococcus spp. a nivel de especie.
- •
En un caso hemos podido detectar ADN bacteriano tras años de un episodio de EI.