La obesidad común o no sindrómica es un rasgo poligénico complejo condicionado por polimorfismos bialélicos o de un sola base denominados single nucleotide polimorphisms (SNP) que presentan un efecto aditivo y que actúan sinérgicamente. La mayor parte de los estudios de asociación genotipo-fenotipo obeso incluyen índice de masa corporal (IMC) o índice cintura talla (ICT), siendo escasos los que introducen un amplio perfil antropométrico.
ObjetivoComprobar si una puntuación de riesgo genético (PRG) desarrollada a partir de 10SNP se encuentra asociada al fenotipo de obesidad evaluado a partir de medidas antropométricas indicativas de exceso ponderal, adiposidad y distribución de la grasa.
Material y métodosUna serie de 438 escolares españoles (de 6 a 16años) fueron evaluados antropométricamente (peso, talla, perímetro de la cintura, pliegues adiposos subcutáneos, IMC, ICT, porcentaje de grasa corporal [%GC]). Se genotiparon 10SNP a partir de muestras de saliva que generaron una puntuación de riesgo genético de obesidad y establecieron asociación genotipo-fenotipo.
ResultadosLos escolares categorizados como obesos mediante IMC, ICT y %GC presentaron puntuaciones más altas de riesgo poligénico que sus pares no obesos. La prevalencia de exceso de peso y adiposidad fue más elevada en los sujetos con una PRG por encima de la mediana. Así mismo, entre los 11 y los 16años todas las variables antropométricas presentaron promedios superiores.
ConclusionesLa PRG estimada a partir de los 10SNP puede ser un instrumento diagnóstico del riesgo potencial de obesidad en escolares españoles, con utilidad desde una perspectiva preventiva.
Common or non-syndromic obesity is a complex polygenic trait conditioned by biallelic or single-base polymorphisms called SNPs (Single-Nucleotide Polymorphisms) that present an additive effect and act synergistically. Most genotype-obese phenotype association studies include body mass index (BMI) or waist-to-height ratio (WtHR), and very few introduce a broad anthropometric profile.
ObjectiveTo verify whether a genetic risk score (GRS) developed from 10SNPs is associated with the obesity phenotype assessed from anthropometric measures indicative of excess weight, adiposity and fat distribution.
Material and methodsA series of 438 Spanish schoolchildren (6 to 16years old) were evaluated anthropometrically (weight, height, waist circumference, skinfold thickness, BMI, WtHR, body fat percentage [%BF]). Ten SNPs were genotyped from saliva samples, generating a GRS for obesity, establishing genotype-phenotype association.
ResultsSchoolchildren categorised as obese by BMI, ICT and %BF had higher GRS than their non-obese peers. The prevalence of overweight and adiposity was higher in subjects with a GRS above the median. Similarly, between 11 and 16years of age, all anthropometric variables presented higher averages.
ConclusionsGRS estimated from the 10SNPs can be a diagnostic tool for the potential risk of obesity in Spanish schoolchildren and could be useful from the preventive perspective.
Como han demostrado los estudios de asociación de genoma completo conocidos como Genome-Wide Association Studies (GWAS), la obesidad común o no sindrómica es un rasgo poligénico condicionado por polimorfismos bialélicos o de un sola base denominados single nucleotide polimorphisms (SNP) que presentan un efecto aditivo y que actúan sinérgicamente1. La revisión de Buniello et al.2 sobre un total de 60 estudios GWAS mostró que ya se han identificado cientos de variantes asociadas al fenotipo obeso en población general. La contribución de cada una de ellas es más o menos modesta y su expresión depende en gran medida del ambiente, que actúa sobre todo a través de los hábitos alimentarios y el ejercicio3.
La mayor parte de los estudios de barrido genómico se han efectuado a partir de muestras de población adulta, aunque en los últimos años ciertas investigaciones se han centrado en la edad pediátrica y juvenil. Se puede mencionar como ejemplo el metaanálisis publicado por Bradfield et al.4, que analiza la asociación entre SNP e índice de masa corporal (IMC) a partir de 30 estudios previos. Dichos antecedentes se llevaron a cabo con muestras en edades comprendidas entre los 2 y los 18años y con una amplia diversidad por lo que se refiere a su origen étnico y poblacional, ya que integraban series con ancestría europea, africana, sudamericana y asiática.
Cabe recordar que el primer SNP asociado a obesidad, localizado en el gen FTO, fue dado a conocer en el año 20075. Pronto se identificaron otras variantes situadas en otros genes o en sus proximidades, como los TMEM18, INSIG2, GNPDA2, CLOCK, FAIM2, FAM120AOS y OLFM4, implicados en el desarrollo de exceso ponderal a través de distintos mecanismos como la regulación del apetito y la saciedad, el aumento del gasto energético o el metabolismo de las grasas, entre otros6. Dado el efecto sumatorio que puede suponer el hecho de poseer un genotipo homocigoto o heterocigoto para el alelo de riesgo en cada uno de los SNP, surgió el concepto de puntuación de riesgo genético (PRG). Esta puntuación es una herramienta que permite evaluar la predisposición genética a cualquier rasgo, característica o enfermedad compleja, sometida a una heredabilidad de tipo aditivo7. La capacidad predictiva de la PRG está recomendada especialmente en los casos donde el tamaño muestral es heterogéneo o relativamente pequeño8.
Conocer con mayor profundidad las interacciones genotipo-condición nutricional en la edad pediátrica y en la juvenil es un aspecto primordial para una mayor comprensión de la obesidad, así como para un abordaje integral e individualizado de la misma, desde la prevención hasta el tratamiento en edades tempranas. En esta línea, el objetivo del presente estudio es conocer si la PRG basada en una batería de 10SNP (previamente asociados a obesidad en la literatura científica) predice el perfil antropométrico en una muestra de escolares españoles.
Material y métodosSe trata de un estudio transversal descriptivo y analítico en una muestra de 438 escolares madrileños de entre 6 a 16años de edad (60,05% varones y 39,95% mujeres). Los datos se recopilaron entre 2019 y 2021 en centros escolares y polideportivos de la Comunidad de Madrid, España.
Los datos empleados en el estudio son anónimos y se encuentran desagregados de información que pueda identificar al sujeto. Fue requisito obtener el consentimiento informado de los padres o tutores y el asentimiento de los participantes, respetando los principios bioéticos de la Declaración de Helsinki en su versión más actualizada9, además de ser aprobado por el Comité de Ética de la Universidad Autónoma de Madrid (CEI-91-1699).
Cada sujeto fue evaluado antropométricamente y se calculó su PRG de obesidad mediante el genotipado de 10SNP a partir de muestras de saliva.
AntropometríaSiguiendo los protocolos del Programa Internacional de Biología (IBP)10 y de la Sociedad Internacional para el Avance de la Cineantropometría (ISAK)11 se midieron la estatura (cm), el peso (kg), el perímetro de umbilical de la cintura (cm) y los pliegues adiposos del bíceps, tríceps, subescapular y suprailíaco (mm). Se utilizaron antropómetro GPM (1mm de precisión), balanza digital SECA (precisión 100g), cinta antropométrica Holtain y adipómetro Holtain (presión constante y 0,2mm de precisión), siempre calibrados antes del proceso de medición. A partir de estas dimensiones directas, se calculó el IMC (peso kg/talla m2) y el índice de cintura-talla (ICT: perímetro cintura/talla). Se estimó el porcentaje de grasa corporal (%GC) mediante la expresión de Siri, previo cálculo de la densidad mediante las fórmulas de Brook o Durnin y Ramahan, todas descritas por Marrodán y González Montero de Espinosa12. El diagnostico nutricional se efectuó, tomando como puntos de corte para el perímetro de la cintura, pliegues adiposos, ICT y %GC las referencias percentilares de Marrodán et al.13-16. Por lo que respecta al IMC, se siguió el criterio de Cole y Lobstein17.
GenotipadoSe analizaron un total de 10 SNP previamente identificados como asociados a la obesidad común en población infanto-juvenil, en un total de 8 genes: rs6548238 (gen TMEM18), rs7566605 (gen INSIG2), rs10938397 (gen GNPDA2), rs801260 (gen CLOCK), rs944990 (gen FAM120AOS), rs7138803 (gen FAIM2), rs12429545 (gen OLFM4) y tres SNP del gen FTO (rs1558902, rs17817449 y rs9939609). El ADN se extrajo en muestras de saliva utilizando el kit Speedtools Tissue DNA Extraction de BioTools B&M S.A. Posteriormente, el genotipado se llevó a cabo en los laboratorios del Nodo de la Universidad de Santiago de Compostela del Centro Nacional de Genotipado (CeGen), que forma parte de la Plataforma en Red de Recursos Biomoleculares y Bioinformáticos del Instituto de Salud CarlosIII. Se empleó la tecnología iPLEX® Gold para la plataforma MassArray de Agena Bioscience Inc.18. Las frecuencias genotípicas y del alelo de riesgo para cada uno de los SNP se pueden consultar en el material suplementario, tabla S.1.
Procedimiento estadísticoDe acuerdo a la metodología de Jääskeläinen et al.19, se clasificó el genotipo de cada SNP adjudicando 2puntos si el individuo era homocigoto para el alelo de riesgo, 1punto si era heterocigoto y 0puntos si era homocigoto para el alelo alterno. La PRG total podía situarse por tanto en un rango ente 0 y 20 (ningún alelo de riesgo en el primer caso, homocigoto de riesgo para los 10SNP en el segundo). Siguiendo el criterio recomendado por Zhao et al.20, en función de la mediana y los cuartiles se clasificó a los sujetos en niveles de menor (PRG ≤Q1) a mayor (PRG >Q3) riesgo genético. Para establecer las comparaciones entre grupos, comprobada la normalidad de las distribuciones, se emplearon pruebas paramétricas (t de Student, ANOVA) o no paramétricas (U de Man Whitney, Kruskal-Wallis). Para el análisis de prevalencias, la muestra se estratificó por grupos de edad (6-10 y 11-18años). El contraste de proporciones se efectuó mediante una prueba de chi cuadrado (χ2). Los resultados se consideraron estadísticamente significativos si p ≤ 0,05. Los datos fueron analizados con el paquete estadístico IBM SPSS 24.
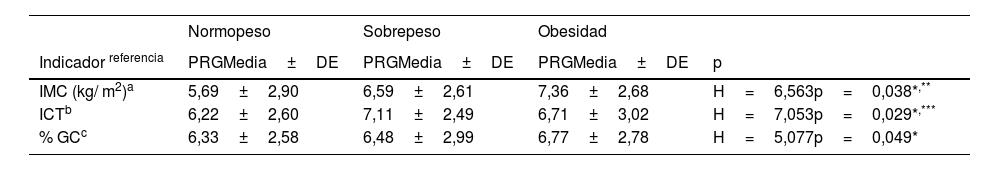
ResultadosEl 31,10% de los individuos de la muestra presentaron exceso ponderal según el IMC (22,50% sobrepeso y 7,60% obesidad). El 47,10% de los participantes tenían un %GC elevado (17,60% entre el percentil 90-97 y 29,50% >percentil 97) y el 43,40% mostraron obesidad abdominal de acuerdo a los puntos de corte del ICT. Como se refleja en la tabla 1, para las medidas directas (perímetro de la cintura y pliegues adiposos) no se observaron diferencias entre la PRG promedio obtenida por los escolares clasificados por encima del percentil90 frente al resto. Sin embargo, los sujetos con sobrepeso y obesidad según el IMC, el %GC y el ICT obtuvieron PRG significativamente más altas que los que mostraban una condición de normopeso. El perfil antropométrico de los participantes puede consultarse en el material suplementario, tabla S.2.
Comparación de la puntuación de riesgo genético (PRG) en función de la condición nutricional de los escolares, evaluada según distintos indicadores antropométricos (n=438)
| Normopeso | Sobrepeso | Obesidad | ||
|---|---|---|---|---|
| Indicador referencia | PRGMedia±DE | PRGMedia±DE | PRGMedia±DE | p |
| IMC (kg/ m2)a | 5,69±2,90 | 6,59±2,61 | 7,36±2,68 | H=6,563p=0,038*,** |
| ICTb | 6,22±2,60 | 7,11±2,49 | 6,71±3,02 | H=7,053p=0,029*,*** |
| % GCc | 6,33±2,58 | 6,48±2,99 | 6,77±2,78 | H=5,077p=0,049* |
| <p90 | ≥ p90 | ||
|---|---|---|---|
| Perímetro umbilical (cm)d | 6,45±2,65 | 6,61±3,24 | H=0,352p=0,657 |
| Pliegue tricipital (mm)e | 6,41±2,67 | 6,75±2,83 | H=1,165p=0,280 |
| Pliegue bicipital (mm)e | 6,38±2,72 | 6,89±2,70 | H=2,784p=0,085 |
| Pliegue subescapular (mm)e | 6,42±2,71 | 6,88±2,71 | H=1,961p=0,161 |
| Pliegue suprailíaco (mm)e | 6,39±2,67 | 6,89±2,93 | H=2,512p=0,113 |
DE: desviación estándar; ICT: índice de cintura-talla; IMC: índice de masa corporal; PRG: puntuación de riesgo genético; %CG: porcentaje de grasa corporal.
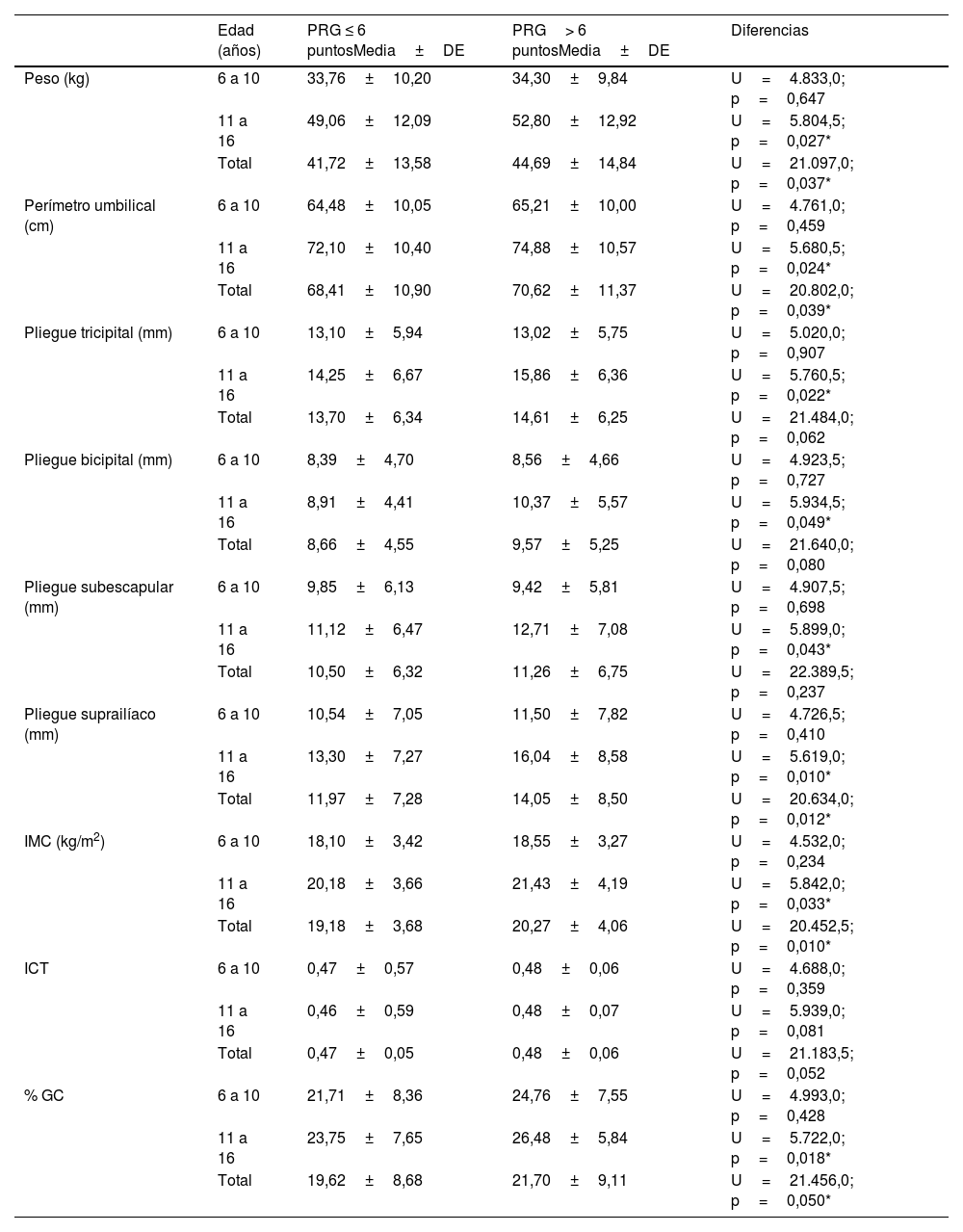
Por otra parte, en la tabla 2 se comparan los promedios para las variables antropométricas directas y derivadas en función de la PRG (por encima o por debajo de 6puntos, valor de la mediana). Para todos los parámetros analizados, los promedios más altos correspondieron a la categoría de mayor riesgo genético. Como puede observarse, los sujetos con PRG >6 puntos presentaron mayor peso, perímetro umbilical de la cintura, grosor de los pliegues adiposos, IMC y %GC entre los 11 y los 16años (p<0,05).
Comparación de promedios de las variables antropométricas relativas a exceso de peso y adiposidad entre grupos establecidos en función de la edad y la puntuación de riesgo genético (PRG)
| Edad (años) | PRG ≤ 6 puntosMedia±DE | PRG> 6 puntosMedia±DE | Diferencias | |
|---|---|---|---|---|
| Peso (kg) | 6 a 10 | 33,76±10,20 | 34,30±9,84 | U=4.833,0; p=0,647 |
| 11 a 16 | 49,06±12,09 | 52,80±12,92 | U=5.804,5; p=0,027* | |
| Total | 41,72±13,58 | 44,69±14,84 | U=21.097,0; p=0,037* | |
| Perímetro umbilical (cm) | 6 a 10 | 64,48±10,05 | 65,21±10,00 | U=4.761,0; p=0,459 |
| 11 a 16 | 72,10±10,40 | 74,88±10,57 | U=5.680,5; p=0,024* | |
| Total | 68,41±10,90 | 70,62±11,37 | U=20.802,0; p=0,039* | |
| Pliegue tricipital (mm) | 6 a 10 | 13,10±5,94 | 13,02±5,75 | U=5.020,0; p=0,907 |
| 11 a 16 | 14,25±6,67 | 15,86±6,36 | U=5.760,5; p=0,022* | |
| Total | 13,70±6,34 | 14,61±6,25 | U=21.484,0; p=0,062 | |
| Pliegue bicipital (mm) | 6 a 10 | 8,39±4,70 | 8,56±4,66 | U=4.923,5; p=0,727 |
| 11 a 16 | 8,91±4,41 | 10,37±5,57 | U=5.934,5; p=0,049* | |
| Total | 8,66±4,55 | 9,57±5,25 | U=21.640,0; p=0,080 | |
| Pliegue subescapular (mm) | 6 a 10 | 9,85±6,13 | 9,42±5,81 | U=4.907,5; p=0,698 |
| 11 a 16 | 11,12±6,47 | 12,71±7,08 | U=5.899,0; p=0,043* | |
| Total | 10,50±6,32 | 11,26±6,75 | U=22.389,5; p=0,237 | |
| Pliegue suprailíaco (mm) | 6 a 10 | 10,54±7,05 | 11,50±7,82 | U=4.726,5; p=0,410 |
| 11 a 16 | 13,30±7,27 | 16,04±8,58 | U=5.619,0; p=0,010* | |
| Total | 11,97±7,28 | 14,05±8,50 | U=20.634,0; p=0,012* | |
| IMC (kg/m2) | 6 a 10 | 18,10±3,42 | 18,55±3,27 | U=4.532,0; p=0,234 |
| 11 a 16 | 20,18±3,66 | 21,43±4,19 | U=5.842,0; p=0,033* | |
| Total | 19,18±3,68 | 20,27±4,06 | U=20.452,5; p=0,010* | |
| ICT | 6 a 10 | 0,47±0,57 | 0,48±0,06 | U=4.688,0; p=0,359 |
| 11 a 16 | 0,46±0,59 | 0,48±0,07 | U=5.939,0; p=0,081 | |
| Total | 0,47±0,05 | 0,48±0,06 | U=21.183,5; p=0,052 | |
| % GC | 6 a 10 | 21,71±8,36 | 24,76±7,55 | U=4.993,0; p=0,428 |
| 11 a 16 | 23,75±7,65 | 26,48±5,84 | U=5.722,0; p=0,018* | |
| Total | 19,62±8,68 | 21,70±9,11 | U=21.456,0; p=0,050* |
IMC: índice de masa corporal; ICT: índice de cintura-talla; %GC: porcentaje de grasa corporal.
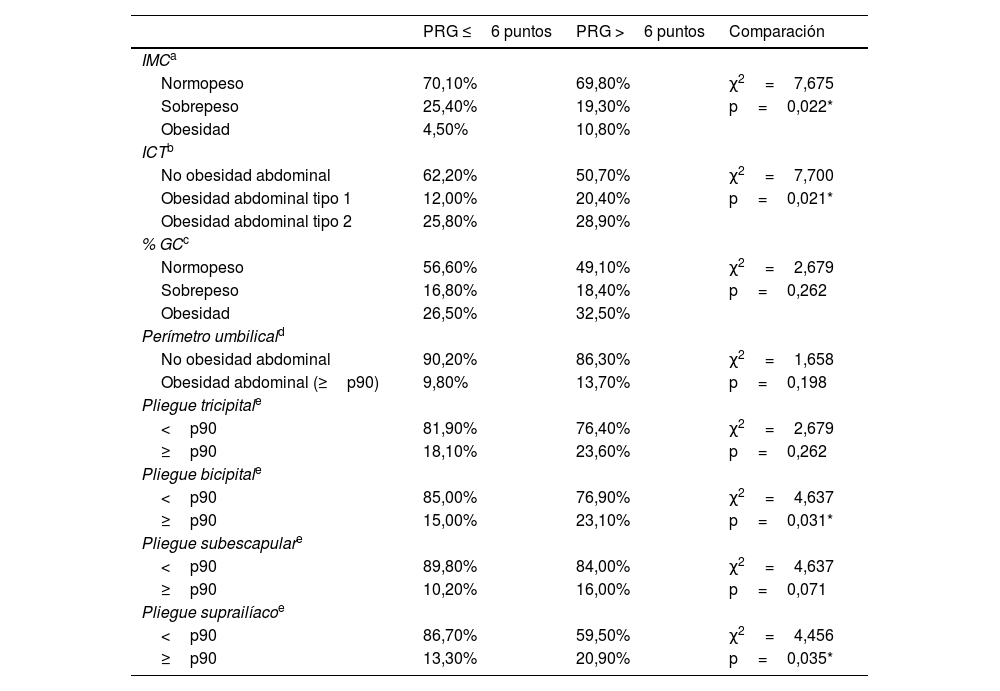
La tabla 3 reúne la comparación de las prevalencias de normopeso, sobrepeso y obesidad según el IMC, el ICT y el %GC entre escolares con menor y mayor predisposición genética a la obesidad en función de la mediana de la PRG (6puntos). La misma tabla incluye la comparación de prevalencias de obesidad abdominal según la medida del perímetro umbilical de cintura y de los pliegues tricipital, bicipital, subescapular y suprailíaco. El porcentaje de sobrepeso y obesidad categorizado por IMC e ICT es significativamente mayor entre los sujetos con mayor predisposición genética a la obesidad. Los valores percentilares de los pliegues bicipital y suprailíaco también son significativamente mayores en los sujetos con puntuaciones más altas de predisposición genética a la obesidad.
Comparación de prevalencias de exceso de peso y adiposidad en función de la PRG comparando entre grupos de riesgo genético de obesidad establecidos en función de la mediana de la puntuación
| PRG ≤6 puntos | PRG >6 puntos | Comparación | |
|---|---|---|---|
| IMCa | |||
| Normopeso | 70,10% | 69,80% | χ2=7,675 |
| Sobrepeso | 25,40% | 19,30% | p=0,022* |
| Obesidad | 4,50% | 10,80% | |
| ICTb | |||
| No obesidad abdominal | 62,20% | 50,70% | χ2=7,700 |
| Obesidad abdominal tipo 1 | 12,00% | 20,40% | p=0,021* |
| Obesidad abdominal tipo 2 | 25,80% | 28,90% | |
| % GCc | |||
| Normopeso | 56,60% | 49,10% | χ2=2,679 |
| Sobrepeso | 16,80% | 18,40% | p=0,262 |
| Obesidad | 26,50% | 32,50% | |
| Perímetro umbilicald | |||
| No obesidad abdominal | 90,20% | 86,30% | χ2=1,658 |
| Obesidad abdominal (≥p90) | 9,80% | 13,70% | p=0,198 |
| Pliegue tricipitale | |||
| <p90 | 81,90% | 76,40% | χ2=2,679 |
| ≥p90 | 18,10% | 23,60% | p=0,262 |
| Pliegue bicipitale | |||
| <p90 | 85,00% | 76,90% | χ2=4,637 |
| ≥p90 | 15,00% | 23,10% | p=0,031* |
| Pliegue subescapulare | |||
| <p90 | 89,80% | 84,00% | χ2=4,637 |
| ≥p90 | 10,20% | 16,00% | p=0,071 |
| Pliegue suprailíacoe | |||
| <p90 | 86,70% | 59,50% | χ2=4,456 |
| ≥p90 | 13,30% | 20,90% | p=0,035* |
ICT: índice de cintura-talla; IMC: índice de masa corporal.
A día de hoy está plenamente establecido que la predisposición genética a la obesidad común, como otras características complejas, se fundamenta en el efecto aditivo de una multiplicidad de SNP. Por este motivo, la utilización de la PRG no ponderada, método empleado en el presente estudio, se considera un buen procedimiento de análisis. Esta herramienta es particularmente útil en los estudios de asociación entre perfil genético y fenotipo antropométrico de obesidad, incluso con muestras relativamente pequeñas21.
Los 10 SNP analizados se escogieron por haberse encontrado asociados aisladamente a obesidad medida por IMC en población infantil o adolescente en la literatura científica consultada y habían sido analizados en un estudio previo publicado por los autores sobre una pequeña muestra22. Se trata, en su mayoría, de marcadores que se ubican dentro o cerca de genes que tienes que ver con procesos metabólicos; de ellos, sin duda son los SNP del gen FTO los que más se han citado en relación con el exceso ponderal, tanto en niños como en adultos. Desde el primer artículo de Frayling et al.5, diversos autores han relacionado este polimorfismo con la capacidad de ingesta, con alteraciones del apetito, con la regulación energética, con la acumulación de triglicéridos en tejido adiposo o con el tamaño de los adipocitos blancos, especialmente con las variantes del SNP rs993960923. También se seleccionaron SNP del gen TMEM18 que codifica para la proteína transmembrana18 que regula la carga de insulina, el INSIG2 que interviene en el metabolismo del colesterol, el GNPDA2 que codifica para la glucosamina-6-fosfato desaminasa2, isoenzima que interviene en el metabolismo de la glucosa y el gen CLOCK que regula los ritmos circadianos24. Además se incluyeron en la batería de SNP alguno del gen OLFM4, relacionado con la inhibición de la apoptosis celular en procesos inflamatorios, o de los FAM120AOS y FAIM2 que codifican para una proteína de membrana que se expresa en el hipocampo cerebral y regula la apoptosis neuronal24.
Cabe señalar que, si bien hay abundante bibliografía sobre la relación entre PRG evaluada a partir de SNP y fenotipo de obesidad, esta es más numerosa en población adulta, y por lo que se refiere a las variables antropométricas, la mayor parte de las publicaciones incluyen el IMC, el perímetro de la cintura y el ICT, pero aún son escasas las que incorporan un perfil antropométrico amplio, incluyendo la medida de la adiposidad directa o relativa. En este sentido, en el presente estudio la PRG se ha asociado al fenotipo obeso a través de diversos indicadores antropométricos que incorporan no solo el IMC e indicadores de distribución de la grasa, sino el valor de los pliegues adiposos y el %GC. Los resultados han puesto de manifiesto que la PRG se asocia no solo al exceso ponderal, sino la adiposidad, tanto general, como focalizada, es decir, a la obesidad abdominal y a la medida de los pliegues. Esta situación se hace particularmente evidente al desagregar la muestra por grupos de edad, ya que entre los 11 y los 16años se magnifican las diferencias antropométricas para las medidas directas y la composición corporal en función de la PRG.
De las evidencias obtenidas podemos extraer que la variabilidad de la condición nutricional asociada a la predisposición genética, pese a que se va forjando durante la infancia (donde la tendencia a exceso ponderal ya es mayor en los que presentan mayor riesgo genético), expone su máxima influencia en esta etapa de rápido crecimiento y desarrollo puberal. Este hecho queda reflejado especialmente en estudios longitudinales, en los que se observa que la tendencia a desarrollar exceso ponderal en individuos con alta PRG alcanza su máximo exponente en la adolescencia, pese a que empiecen a encontrarse diferencias en edades anteriores25.
En relación con lo expuesto, y en apoyo a lo aquí constatado, cabe señalar que ciertos estudios recientes, efectuados sobre cohortes de adolescentes europeos, apuntan que el riesgo poligénico de obesidad se expresa preferentemente en la fase de la ontogenia que se corresponde con la edad adolescente. Ello sucede por la mediación de distintas vías biológicas o factores hormonales que actúan en la acumulación de grasa corporal, en el control de apetito y en la ingesta energética26. En contraposición, algunas investigaciones ponen de relieve que el riesgo genético condiciona tempranamente el fenotipo obeso. Este es el caso del trabajo efectuado por Monnereau et al.27 sobre una muestra cercana a 4.000 escolares holandeses en la que se asoció una PRG construida a partir de 91SNP (asociados a obesidad en adultos) a indicadores antropométricos de exceso ponderal y obesidad abdominal en sujetos de 6años de edad. En misma línea, Belsky et al.28 reportaron que, si bien la PRG no se asociaba al peso al nacimiento, los niños con puntuación elevada ganaban peso y grasa rápidamente durante la fase de aceleración del crecimiento que tiene lugar en la temprana infancia.
Como se indicó anteriormente, son relativamente pocos los estudios destinados a valorar el perfil de riesgo genético para el desarrollo de obesidad con sujetos en proceso de crecimiento. Hay que citar el trabajo de Seyednasrollah et al.29 en una muestra de 1.142 adolescentes finlandeses en el que se construyó una PRG a partir de 97SNP que lograba predecir exceso ponderal. También el trabajo surgido a partir del estudio HELENA (Healthy Lifestyle in Europe by Nutrition in Adolescence), que enroló a 1.069 adolescentes y en el que la PRG asociada a la obesidad estaba conformada por un total de 21SNP30.
El presente trabajo presenta limitaciones. A la vista de los resultados obtenidos, se hace necesario incrementar el efectivo muestral para asegurar la potencia de las asociaciones observadas. Sin embargo, también tiene algunas fortalezas. En los dos estudios referidos en el párrafo anterior27,30 la PRG se elaboró con un mayor número de SNP que al menos duplica el actual. Por otra parte, la PRG aquí desarrollada se asocia no solo al IMC, sino también a otros indicadores antropométricos que miden la cantidad y la distribución del tejido adiposo, por lo que aportan nueva información. En conclusión, puede afirmarse que la PGR estimada a partir de los 10SNP considerados constituye un instrumento diagnóstico del potencial riesgo de obesidad en escolares españoles. De ahí su utilidad como un elemento más a tener en cuenta para la evaluación de la condición nutricional con un enfoque preventivo.
FinanciaciónProject PR41/17_21008 Banco de Santander.
Conflicto de interesesLos autores declaran que no tienen conflictos de interés relacionados con esta publicación.