El receptor de melanocortina 4 (MC4R) participa en el control del apetito a nivel del sistema nervioso central, a través de la vía de la leptina-melanocortina. Se ha reportado asociación entre diferentes polimorfismos del gen MC4R y la obesidad; sin embargo, existen escasos estudios del polimorfismo de nucleótido simple (SNP) rs483145 de este gen.
ObjetivoInvestigar su prevalencia y asociación con marcadores de adiposidad en adultos chilenos.
MétodosLa prevalencia del SNP rs483145, del gen MC4R, fue determinada en 259 participantes del estudio Genes, Ambiente, Diabetes y Obesidad (GENADIO) mediante reacción en cadena de la polimerasa (PCR) en tiempo real. La asociación del alelo de riesgo de MC4R (A) con marcadores de adiposidad (peso corporal, índice de masa corporal, porcentaje de masa grasa, perímetro de cadera, perímetro de cintura e índice cintura/cadera), se realizó mediante análisis de regresión lineal y fue ajustada por variables de confusión (sociodemográficas y de actividad física) mediante 3 modelos estadísticos.
ResultadosSe determinó que la prevalencia del alelo de riesgo (A) del SNP rs483145 del gen MC4R es del 24,5% en la población adulta chilena incluida en este estudio, sin encontrar asociación con ninguno de los marcadores de adiposidad estudiados, tanto en modelos ajustados como sin ajustar.
ConclusiónLa presencia del alelo de riesgo del SNP rs483145 del gen MC4R no se asocia con marcadores de adiposidad en la población adulta chilena estudiada. Nuevos estudios que incluyan una muestra más numerosa serán necesarios para confirmar estos resultados.
The melanocortin receptor 4 (MC4R) participates in the control of appetite at the level of the central nervous system, through the leptin-melanocortin pathway. An association between different polymorphisms of the MC4R gene and obesity has been reported. However, there are few studies of the rs483145 single nucleotide polymorphism (SNP) of this gene.
ObjectiveTo investigate its prevalence and association with adiposity markers in Chilean adults.
MethodsThe prevalence of SNP rs483145, of the MC4R gene, was determined in 259 participants of the GENADIO study (genes, environment, diabetes and obesity) by means of real-time polymerase chain reaction (PCR). The association between the risk allele of MC4R (A) and adiposity markers (body weight, body mass index, fat mass percentage, hip circumference, waist circumference, waist-to-hip ratio) was performed by linear regression analysis and adjusted for confusion variables (socio-demographic and physic activity) using three statistical models.
ResultsIt was determined that the prevalence of the risk allele (A) of the SNP rs483145 of the MC4R gene is 24.5% in the Chilean adult population included in this study, without finding an association with any of the adiposity markers studied, both in adjusted and unadjusted models.
ConclusionThe presence of the risk allele of SNP rs483145 of the MC4R gene is not associated with adiposity markers in the Chilean adult population studied. New studies with a bigger sample size will be necessary to confirm these results.
Durante los últimos años, la obesidad se ha convertido en uno de los problemas de salud pública más importantes a nivel mundial, siendo responsable de 2,6 millones de muertes anuales1,2. Inicialmente, se reconocía solo como un factor de riesgo de cardiopatía isquémica; pero luego se asoció con enfermedades como diabetes mellitus de tipo 2, hipertensión arterial y cáncer3. Actualmente un 34,4% de la población chilena es obesa, y un 74,2% de la población adulta presenta exceso de peso4.
El desarrollo de la obesidad es multifactorial, y conjuga factores modificables como la alimentación y la actividad física, con factores no modificables como edad, sexo y factores genéticos5,6. Actualmente, se describen más de 700 marcadores genéticos asociados con la obesidad común, destacando los polimorfismos de nucleótido simple (SNP) asociados al gen FTO, por presentar la mayor fuerza de asociación, y los SNP del receptor de melanocortina 4 (MC4R), por haber sido uno de los primeros identificados7,8.
El MC4R es un receptor acoplado la proteína G relacionado con el control del apetito por la vía hipotalámica de la leptina-melanocortina9. La activación del MC4R es producida por su unión a moléculas derivadas de la proopiomelanocortina (POMC), como las formas α y β de la hormona estimulante de melanocitos (αMSH y βMSH)10. La proteína Agouti (Agouti-related peptide [AGRP]) es otro péptido de unión a MC4R, que antagoniza la señal anorexigénica de los derivados de POMC11. Debido a esto, se ha investigado el potencial uso de algunos agonistas sintéticos del MC4R como fármacos para el tratamiento de la obesidad, especialmente en la obesidad sindrómica de origen monogénica12. Aunque los primeros agonistas mostraron efectos cardiovasculares adversos13, posteriores estudios clínicos de fase 3 para setmelanotide mostraron una reducción significativa del apetito y del peso en pacientes con deficiencias en POMC o el receptor de leptina (LEPR), abriendo paso para su uso clínico14,15.
La eliminación de MC4R mediante modificación genética en ratones provoca obesidad asociada a hiperfagia y falta de saciedad, mientras que en humanos las mutaciones con pérdida de función están asociadas a obesidad monogénica severa de inicio temprano16,17. Estas mutaciones inactivantes de MC4R provocan aumentos de hasta 7kg de peso corporal en homocigotos, pero presentan una prevalencia menor al 0,1% en poblaciones caucásicas18. Por otro lado, el MC4R presenta otras variaciones genéticas de alta prevalencia del tipo SNP, algunas de las cuales están asociadas con aumento modesto en el riesgo de obesidad15. Los SNP de MC4R se distribuyen a lo largo de todo el gen, incluyendo a rs483145 y rs11872992 ubicados en la región promotora, a rs2229616 (V103I) y rs52820871 (I251L) presentes en el único exón, y a rs17782313 y rs12970134 ubicados a 188 y 150kb del extremo 3’ del gen (locus distal), respectivamente19. Estos últimos SNP han sido los más estudiados y presentan asociación con riesgo aumentado de obesidad en asiáticos, europeos y americanos, alcanzando una variación de hasta 0,3kg/m2 en el índice de masa corporal (IMC) por cada copia del alelo de riesgo20,21. Por el contrario, para el SNP rs483145 existe solo un estudio de asociación realizado en una población de indios pima (originarios de Arizona, EE. UU.), donde se evidenció un aumento de 0,58kg/m2 en IMC por cada copia del alelo de riesgo y una frecuencia del alelo de riesgo mayor al 80%22. Debido a la ausencia de estudios que asocien la prevalencia del SNP rs483145 de MC4R con obesidad en población adulta chilena, el objetivo de este trabajo fue determinar su prevalencia en esta población chilena e investigar su posible asociación con marcadores de adiposidad.
Materiales y métodosSe realizó un estudio descriptivo transversal que incluyó a 259 individuos con datos disponibles para el genotipo del SNP rs483145 en el gen MC4R, pertenecientes al estudio Genes, Ambiente, Diabetes y Obesidad (GENADIO), realizado en Chile entre los años 2009-2011. El proyecto GENADIO estudió una población total de 475 individuos de ascendencia mapuche o europea (249 y 226 individuos, respectivamente) de las regiones del Biobío y Los Ríos, sin historial de enfermedad metabólica o cardiovascular, y que al momento de la evaluación no estuvieran bajo prescripción de medicamentos23. La muestra fue calculada en base a los datos del CENSO 2002, en el cual la población indígena chilena constituía el 4,6% de la población. Para la selección de participantes de ascendencia mapuche o europea y el descarte de individuos de ancestría mestiza, se incluyeron solo aquellos cuyos apellidos paterno y materno fuesen de origen mapuche o europeo, respectivamente. Además, para la selección de mapuches, se incluyeron solo aquellos cuyo grupo sanguíneo fuese 0. El reclutamiento de los participantes se realizó mediante invitaciones abiertas a la comunidad o por intermedio de organizaciones comunitarias como centros de atención de salud primaria, centros educacionales o clubes sociales. El estudio contó con la aprobación de los comités de ética de la Universidad de Chile, Universidad de Concepción y Universidad de Glasgow, que se adhieren a la Declaración de Helsinki sobre estudios en humanos. Todos los participantes firmaron su consentimiento informado previo a la recolección de datos.
Determinación de variantes alélicas del gen MC4RPara la genotipificación del SNP rs483145 del gen MC4R, se obtuvo ADN genómico de leucocitos periféricos mediante el kit QIAamp® DNA blood midi kit (QIAGEN, Ltd. UK). La discriminación alélica se realizó mediante la reacción en cadena de la polimerasa (PCR) de tiempo real en termociclador ABI 7900-HT usando sondas TaqMan® (Applied Biosystems, Warrington, UK). Todos los análisis de las muestras fueron realizados en duplicados, con el 98% de éxito en la determinación del genotipo.
Marcadores de adiposidadLa evaluación antropométrica fue realizada por un profesional capacitado utilizando protocolos estandarizados24. El peso corporal y la talla fueron medidos con una balanza electrónica (Tanita TBF 300A, EE. UU.) y tallímetro (SECA Modelo A800, EE. UU.) con una precisión de 100g y 1mm, respectivamente. El estado nutricional fue clasificado en base a los puntos de corte sugeridos por la Organización Mundial de la Salud (OMS): bajo peso: <18,5kg/m2; normo peso: 18,5-24,9kg/m2; sobrepeso: 25,0-29,9kg/m2 y obesidad: ≥30,0kg/m2. El perímetro de cintura (PC) fue medido con una cinta métrica no distendible (SECA Modelo 201, EE. UU.). Los valores utilizados para definir la obesidad central fueron los siguientes: PC≥94cm y ≥80cm en varones y mujeres, respectivamente25. El perímetro de cadera fue medido alrededor del punto más ancho alrededor de la región de la cadera, aproximadamente al nivel de la sínfisis púbica. Para la medición de grasa abdominal se utilizó el índice de cintura/cadera, el cual fue obtenido tras la división entre estas 2 variables. La composición corporal se determinó mediante la medición de 4 pliegues cutáneos (bicipital, subescapular, suprailíaco y tricipital) a través de un caliper Harpenden (Cranlea & Company, UK), con una precisión de 0,2mm23,26. Se aplicó la ecuación de Durnin y Womersley para estimar el porcentaje de masa grasa corporal26.
Variables sociodemográficas y actividad físicaLos datos sociodemográficos (edad, sexo, zona de residencia, etnia y nivel educacional) fueron recolectados mediante encuestas validadas19. Los niveles de actividad física (AF) y el tiempo sedentario fueron estimados por acelerometría de movimiento (ActiGraph GTM1, EE. UU.). La intensidad de la AF y el gasto energético se determinaron mediante el algoritmo de Freedson27.
Análisis estadísticoLos datos de caracterización de la población estudiada son presentados como promedio y desviación estándar (DE) para variables continuas, y como porcentaje para variables categóricas. Las diferencias entre genotipo fueron determinadas con análisis de regresión para variables continuas y con el test χ2 para variables categóricas. Para investigar la asociación entre el polimorfismo rs483145 del gen MC4R y los marcadores de obesidad (peso corporal, IMC, PC, perímetro de cadera, índice cintura/cadera y porcentaje de masa grasa), se realizó un análisis de regresión lineal. También se realizó análisis de interacción entre MC4R y sexo, así como también entre MC4R y etnia; para determinar si la asociación entre el MC4R y marcadores de adiposidad es diferente para hombres y mujeres o para población mapuche y no mapuche. Debido a que no se encontró evidencia de una interacción significativa (p-interacción sexo p=0,369 y etnia p=0,560), no fue necesario estratificar los análisis de asociación por sexo o etnia.
El genotipo del SNP rs483145 del gen MC4R fue codificado siguiendo un modelo genético aditivo, siendo: 0=TT homocigoto para el alelo protector; 1=AT heterocigoto para el alelo de riesgo; 2=AA homocigoto para el alelo de riesgo.
Todos los análisis fueron ajustados por variables de confusión mediante la utilización de 3 modelos estadísticos: Modelo 0: sin ajustar; Modelo 1: ajustado por edad, sexo, etnia, nivel educacional y zona de residencia (urbano/rural); y Modelo 2: ajustado por el modelo 1, pero también por actividad física. Para todos los análisis se utilizó el programa STATA® SE v14. El nivel de significación fue definido como p<0,05.
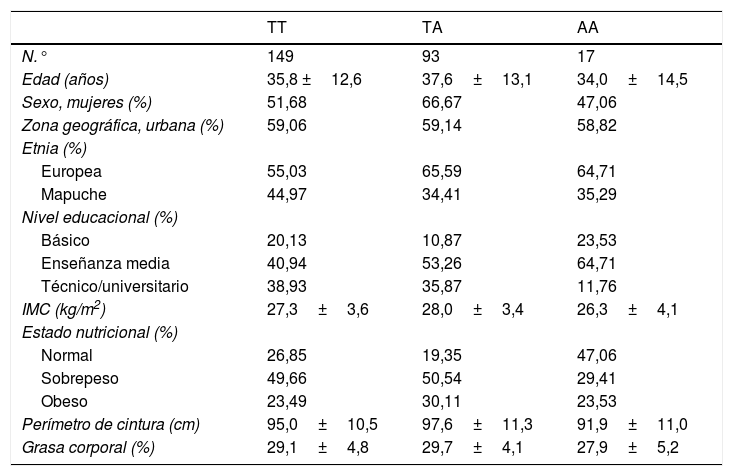
ResultadosLas características generales de la población estudiada según genotipo se encuentran en la tabla 1. Los 3 genotipos presentaron un promedio de edad similar, correspondiente a 35,8 años para TT; 37,6 años para TA y 34,0 años para AA. El genotipo TA presentó un 66,6% de mujeres, mientras que el genotipo TT presentó un 51,6% y el AA un 47,0%. Se observó una menor pertenencia a la etnia mapuche entre los individuos con genotipos de riesgo (34,4% para TA y 35,2% para AA) vs. los portadores de genotipo protector TT, (44,9%). No se observaron diferencias apreciables en otras variables sociodemográficas, ni en los parámetros de actividad física entre grupos. En la tabla 2 se presentan las frecuencias alélicas del SNP rs483145, las cuales se distribuyen de acuerdo con el equilibrio de Hardy Weinberg (χ2=0,616) y corresponden a 0,754 para el alelo protector (T) y 0,245 para el alelo de riesgo (A).
Características de la población según el genotipo del gen MC4R (rs483145)
| TT | TA | AA | |
|---|---|---|---|
| N.° | 149 | 93 | 17 |
| Edad (años) | 35,8 ±12,6 | 37,6±13,1 | 34,0±14,5 |
| Sexo, mujeres (%) | 51,68 | 66,67 | 47,06 |
| Zona geográfica, urbana (%) | 59,06 | 59,14 | 58,82 |
| Etnia (%) | |||
| Europea | 55,03 | 65,59 | 64,71 |
| Mapuche | 44,97 | 34,41 | 35,29 |
| Nivel educacional (%) | |||
| Básico | 20,13 | 10,87 | 23,53 |
| Enseñanza media | 40,94 | 53,26 | 64,71 |
| Técnico/universitario | 38,93 | 35,87 | 11,76 |
| IMC (kg/m2) | 27,3±3,6 | 28,0±3,4 | 26,3±4,1 |
| Estado nutricional (%) | |||
| Normal | 26,85 | 19,35 | 47,06 |
| Sobrepeso | 49,66 | 50,54 | 29,41 |
| Obeso | 23,49 | 30,11 | 23,53 |
| Perímetro de cintura (cm) | 95,0±10,5 | 97,6±11,3 | 91,9±11,0 |
| Grasa corporal (%) | 29,1±4,8 | 29,7±4,1 | 27,9±5,2 |
Datos presentados como promedio y desviación estándar (DE) para variables continuas y como porcentaje (%) para variables categóricas.
AF: actividad física; IMC: índice de masa corporal.
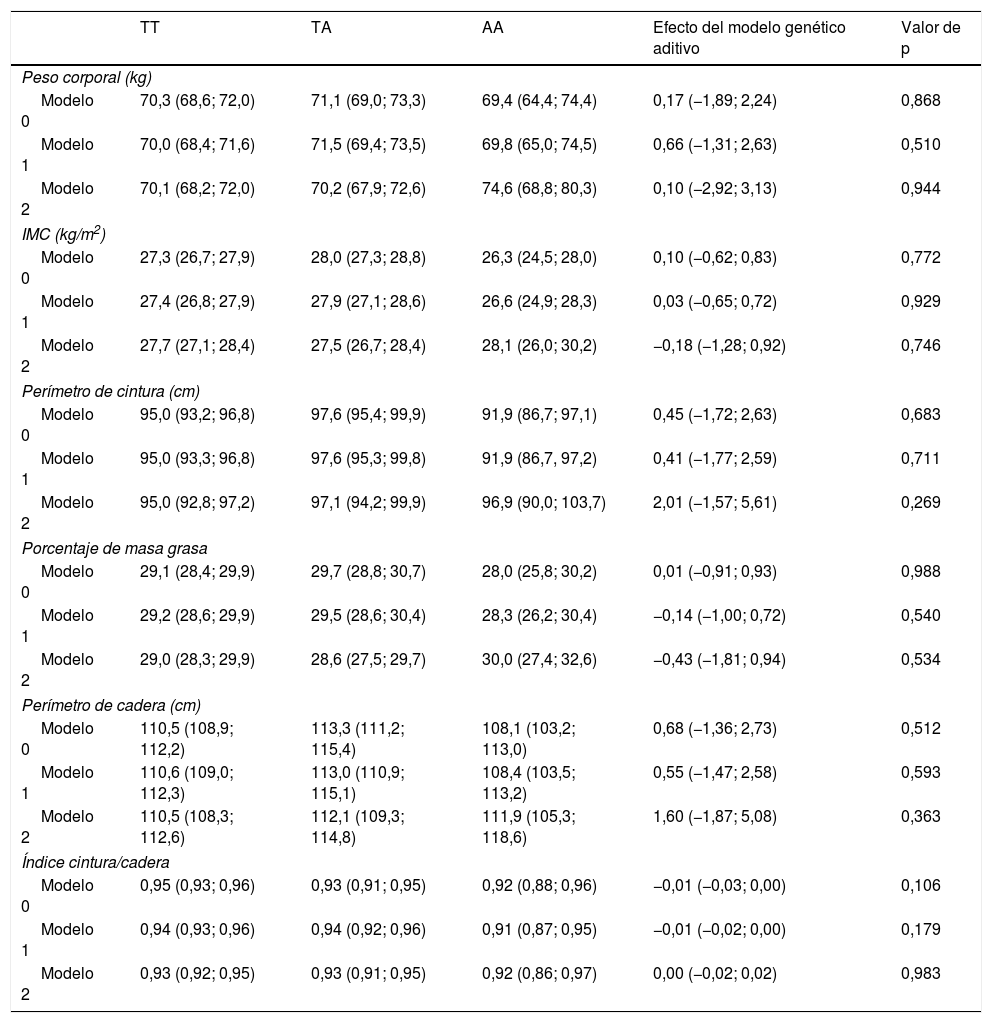
Los resultados de la asociación entre el SNP rs483145 del gen MC4R y marcadores de adiposidad se presentan en la tabla 3. No se identificaron asociaciones significativas entre el genotipo de riesgo y los marcadores de adiposidad estudiados (peso corporal, IMC, perímetro de cintura, porcentaje de masa grasa, perímetro de cadera e índice cintura/cadera). Para el peso corporal se observó en el modelo no ajustado (modelo 0) un valor de 70,3kg para el genotipo protector TT, de 71,1kg para el genotipo TA; y de 69,4kg para el genotipo AA. Para el resto de los marcadores de adiposidad, exceptuando el índice cintura/cadera, se observaron variaciones similares de aumento para el genotipo TA y de disminución para el genotipo AA, en los modelos 0 y 1; pero sin significación estadística.
Asociación entre el genotipo del gen de MC4R (rs483145) con marcadores de obesidad
| TT | TA | AA | Efecto del modelo genético aditivo | Valor de p | |
|---|---|---|---|---|---|
| Peso corporal (kg) | |||||
| Modelo 0 | 70,3 (68,6; 72,0) | 71,1 (69,0; 73,3) | 69,4 (64,4; 74,4) | 0,17 (−1,89; 2,24) | 0,868 |
| Modelo 1 | 70,0 (68,4; 71,6) | 71,5 (69,4; 73,5) | 69,8 (65,0; 74,5) | 0,66 (−1,31; 2,63) | 0,510 |
| Modelo 2 | 70,1 (68,2; 72,0) | 70,2 (67,9; 72,6) | 74,6 (68,8; 80,3) | 0,10 (−2,92; 3,13) | 0,944 |
| IMC (kg/m2) | |||||
| Modelo 0 | 27,3 (26,7; 27,9) | 28,0 (27,3; 28,8) | 26,3 (24,5; 28,0) | 0,10 (−0,62; 0,83) | 0,772 |
| Modelo 1 | 27,4 (26,8; 27,9) | 27,9 (27,1; 28,6) | 26,6 (24,9; 28,3) | 0,03 (−0,65; 0,72) | 0,929 |
| Modelo 2 | 27,7 (27,1; 28,4) | 27,5 (26,7; 28,4) | 28,1 (26,0; 30,2) | −0,18 (−1,28; 0,92) | 0,746 |
| Perímetro de cintura (cm) | |||||
| Modelo 0 | 95,0 (93,2; 96,8) | 97,6 (95,4; 99,9) | 91,9 (86,7; 97,1) | 0,45 (−1,72; 2,63) | 0,683 |
| Modelo 1 | 95,0 (93,3; 96,8) | 97,6 (95,3; 99,8) | 91,9 (86,7, 97,2) | 0,41 (−1,77; 2,59) | 0,711 |
| Modelo 2 | 95,0 (92,8; 97,2) | 97,1 (94,2; 99,9) | 96,9 (90,0; 103,7) | 2,01 (−1,57; 5,61) | 0,269 |
| Porcentaje de masa grasa | |||||
| Modelo 0 | 29,1 (28,4; 29,9) | 29,7 (28,8; 30,7) | 28,0 (25,8; 30,2) | 0,01 (−0,91; 0,93) | 0,988 |
| Modelo 1 | 29,2 (28,6; 29,9) | 29,5 (28,6; 30,4) | 28,3 (26,2; 30,4) | −0,14 (−1,00; 0,72) | 0,540 |
| Modelo 2 | 29,0 (28,3; 29,9) | 28,6 (27,5; 29,7) | 30,0 (27,4; 32,6) | −0,43 (−1,81; 0,94) | 0,534 |
| Perímetro de cadera (cm) | |||||
| Modelo 0 | 110,5 (108,9; 112,2) | 113,3 (111,2; 115,4) | 108,1 (103,2; 113,0) | 0,68 (−1,36; 2,73) | 0,512 |
| Modelo 1 | 110,6 (109,0; 112,3) | 113,0 (110,9; 115,1) | 108,4 (103,5; 113,2) | 0,55 (−1,47; 2,58) | 0,593 |
| Modelo 2 | 110,5 (108,3; 112,6) | 112,1 (109,3; 114,8) | 111,9 (105,3; 118,6) | 1,60 (−1,87; 5,08) | 0,363 |
| Índice cintura/cadera | |||||
| Modelo 0 | 0,95 (0,93; 0,96) | 0,93 (0,91; 0,95) | 0,92 (0,88; 0,96) | −0,01 (−0,03; 0,00) | 0,106 |
| Modelo 1 | 0,94 (0,93; 0,96) | 0,94 (0,92; 0,96) | 0,91 (0,87; 0,95) | −0,01 (−0,02; 0,00) | 0,179 |
| Modelo 2 | 0,93 (0,92; 0,95) | 0,93 (0,91; 0,95) | 0,92 (0,86; 0,97) | 0,00 (−0,02; 0,02) | 0,983 |
Datos presentados como promedio e IC 95% según genotipo. El modelo genético aditivo indica el promedio de incremento en la variable de adiposidad por cada copia adicional de la variante de riesgo (A). Este efecto aditivo y su respectivo IC 95% fue determinado mediante regresión lineal. Los análisis fueron ajustados por: Modelo 0: sin ajustar; Modelo 1: ajustado por edad, sexo, etnia, nivel educacional, ingreso económico, nivel socioeconómico, y zona de residencia (urbano/rural); Modelo 2: ajustado por el modelo 1, pero también por actividad física.
IC 95%: intervalo de confianza del 95%; IMC: índice de masa corporal.
El presente estudio evidencia ausencia de asociación significativa entre el SNP rs483145 de MC4R y marcadores de obesidad en la población adulta chilena. La frecuencia del alelo de riesgo (A), se estimó en el 24,5%; valor similar al reportado en bases de datos europeas como 1.000 genomas, pero considerablemente menor a la de indios americanos Pima, donde presentó una frecuencia del 80%22. Por otro lado, el SNP rs17782313 de MC4R, que es más prevalente en población caucásica (30%), presenta una frecuencia de solo el 3% en los indios pima, revelando una fuerte dependencia del componente étnico en la variación genética de MC4R. En contraste con nuestros resultados, el alelo de riesgo de rs483145 en indios pima se asoció con un aumento de 0,58kg/m2 en el IMC por cada copia extra del alelo de riesgo, aunque no se asoció con alteraciones en otros marcadores de adiposidad, como porcentaje de masa grasa y gasto energético22. Es posible que la discrepancia en la asociación del SNP rs483145 con IMC en chilenos vs. indios pima, pueda estar relacionada con características étnicas particulares o con interacciones específicas con componentes ambientales que difieren en estas dos poblaciones, tal como ha sido demostrado en estudios longitudinales donde la interacción del perfil genético con ambientes afecta el grado de obesidad presentada28,29. Sin embargo, no podemos descartar que la falta de asociación se deba a un bajo poder estadístico, ya que en el estudio realizado en indios pima se incluyó a 11.268 participantes en comparación a los 259 incluidos en el presente estudio.
Estudios previos han abordado la relación entre MC4R con obesidad en población chilena, pero a través del análisis del SNP rs17782313, un polimorfismo ubicado en el locus distal, a 317kb del SNP aquí estudiado23. El SNP rs17782313 presenta una prevalencia del alelo de riesgo cercano al 30% en niños y adultos chilenos, sin presentar una mayor frecuencia en los individuos con obesidad30–32. Interesantemente, estos estudios también revelaron que, tanto en niños como en adultos que presentaban obesidad, el alelo de riesgo de rs17782313 se correlacionó con conductas alimentarias alteradas, como niveles de saciedad disminuidos y mayor disfrute de las comidas, pero no se logró establecer asociación alguna con IMC30. En conjunto, los estudios de distintas variantes genéticas de MC4R realizados en población chilena, coinciden en evidenciar una alta prevalencia de variantes de riesgo cercana al 30% en diversas regiones del gen, sin asociación a obesidad, posiblemente debido al reducido tamaño muestral (<500). En europeos, por otro lado, la presencia del alelo de riesgo de rs17782313 se acompañó de un aumento de 0,2kg/m2 en el IMC, de 0,66kg en el peso corporal y un aumento del 14% en la probabilidad de ser obeso8. Para el caso de rs12970134, otro SNP del locus distal, estudios de asociación genómica (Genomic Wide Association [GWAS]) en población asiática y europea, revelaron un aumento de 1cm en la circunferencia de cintura por cada copia del alelo de riesgo8.
Para los SNP que se asocian a cambios en la secuencia proteica de MC4R, como rs2229616 (V103I) y rs52820871 (I251L), estudios de metaanálisis, que incluyen más de 10.000 participantes, revelaron que los alelos menores reducen el riesgo de obesidad en un 20 y 50% y se presentan con una frecuencia entre 2 y 7% en distintas poblaciones, incluyendo americanos, asiáticos y europeos3,8. Para ambos SNP, se ha reportado una disminución cercana a 0,8kg/m2 en IMC por cada copia del alelo de riesgo, efecto asociado a una respuesta disminuida de MC4R a la señal orexigénica mediada por AGRP19. Estudios en europeos también revelaron alteraciones del comportamiento alimentario asociado a variantes y polimorfismos no-sinónimos de MC4R, como preferencia de consumo de grasa, menor consumo de sacarosa y menor respuesta a saciedad33,34. Los distintos estudios revisados revelan que la asociación de obesidad con polimorfismos en MC4R y los loci cercanos, se deben a cambios en el eje hipotalámico de control del apetito, asociados con la respuesta a derivados de POMC o a AGRP, pero también con la regulación del consumo de macronutrientes a nivel de la amígdala, y con la homeostasis térmica, asociada al consumo de oxígeno en tejido adiposo9,34,35. A pesar que la variante genética analizada en este estudio no se localiza en la secuencia codificante y, por lo tanto, no involucra cambios directos en la función proteica de MC4R, su ubicación hacia el extremo 5’ del gen la convierten en un potencial elemento cis-regulatorio que podría influir sobre los niveles de expresión de MC4R. De ser esto probado, por ejemplo, a través de estudios de eQTL, es posible postular que los portadores del SNP rs483145 de MC4R, podrían beneficiarse del tratamiento con agonistas del MC4R que pudieran aumentar la señalización disminuida por efecto de una eventual menor expresión del receptor. Recientemente, la administración de alimentos y fármacos de los EE. UU. (FDA) aprobó el setmelanotide para el tratamiento de obesidad severa en pacientes con déficit en la síntesis de POMC o en el receptor de Leptina; constituyendo el primer agonista de MC4R aprobado para su uso farmacológico.
Las limitaciones de este estudio incluyen el tamaño muestral reducido y la selección de una población con un promedio de edad inferior a los 40 años sin historial de enfermedades metabólicas. El reducido tamaño muestral de este estudio impidió realizar análisis comparativos entre varones y mujeres (112 y 147); y entre individuos con ascendencia mapuche y europea (153 y 106), sin embargo, se pudo observar una menor prevalencia del alelo de riesgo en la población mapuche (35 vs. 65%). Cabe mencionar que, en estudios previamente publicados en la misma cohorte, identificamos asociaciones positivas entre variantes genéticas de FTO, TCF7 y SLC16A11 con marcadores de obesidad36–38. Lo anterior sugiere que, en la eventualidad de existir una asociación subrepticia entre el SNP rs483145 de MC4R con obesidad, esta tendría una fuerza de asociación significativamente menor que las reportadas para FTO, TCF7 y SLC16A11. Por otro lado, dado que para otros SNP de este gen se han encontrado diferencias en las conductas alimentarias, resultaría interesante realizar estudios similares para rs48314516.
ConclusionesNuestros resultados y los estudios reportados revelan la importancia del componente étnico en la prevalencia de los distintos SNP de MC4R, así como en su relación con marcadores de obesidad. Las diferentes asociaciones que presentan otras variantes genéticas de MC4R con obesidad (predisposición vs. protección) agregan mayor complejidad a la relación entre MC4R y obesidad, revelando la necesidad de estudiar la prevalencia de los diversos polimorfismos de MC4R en las distintas poblaciones y su asociación con obesidad, conductas alimentarias y metabolismo. Particularmente, la variante genética rs483145 no se encontró asociada a obesidad en población chilena, contrariamente a lo reportado en indios pima22. El presente estudio aporta nueva información para comprender la relación entre los polimorfismos en el gen MC4R y obesidad, lo cual podría considerarse al diseñar programas de prevención primaria y estrategias terapéuticas personalizadas en población chilena.
Conflicto de interesesLos autores declaran no tener conflicto de intereses.
FinanciaciónLa presente investigación no ha recibido ayudas específicas provenientes de agencias del sector público, sector comercial o entidades sin ánimo de lucro.