Los métodos más frecuentemente utilizados en Microbiología Clínica para la determinación de la sensibilidad de las bacterias a los antibióticos se basan en un estudio fenotípico, observando el crecimiento bacteriano de la cepa incubada en presencia del antibiótico a estudiar. Estos métodos requieren normalmente un tiempo de unas 24h para la obtención de resultados. El objetivo de este trabajo es revisar el fundamento y los resultados de las principales técnicas instrumentales que proporcionan un antibiograma rápido. De manera pormenorizada se exponen datos relativos a técnicas moleculares, citometría de flujo, quimioluminiscencia, espectrometría de masas, métodos comerciales utilizados en el trabajo de rutina, métodos colorimétricos, nefelometría, microarrays, microfluidos y métodos de lisis bacteriana
The most widely used antibiotic susceptibility testing methods in Clinical Microbiology are based on the phenotypic detection of antibiotic resistance by measuring bacterial growth in the presence of the antibiotic being tested. These conventional methods take typically 24hours to obtain results. A review is presented here of recently developed techniques for the rapid determination of antibiotic susceptibility. Data obtained with different methods such as molecular techniques, flow cytometry, chemiluminescence, mass spectrometry, commercial methods used in routine work, colorimetric methods, nephelometry, microarrays, microfluids, and methods based on cell disruption and sequencing, are analyzed and discussed in detail.
En los laboratorios de Microbiología Clínica, la identificación microbiana y la determinación de la sensibilidad de los microorganismos a los antibióticos son tareas con un gran impacto en el manejo del paciente infectado. Los métodos utilizados para la determinación de la sensibilidad se basan en un estudio fenotípico, observando el crecimiento bacteriano de la cepa incubada en presencia del antibiótico a estudiar. Estos métodos incluyen dilución en agar, macrodilución y microdilución en caldo, tiras con un gradiente de antibiótico y difusión disco-placa. La microdilución en caldo es la técnica utilizada por los diversos sistemas automatizados comerciales como el MicroScan Walkaway (Siemens, Nueva York, EE.UU.), Phoenix (BD Diagnostics, Sparks, MD, EE.UU.) o VITEK (bioMérieux, Marcy l’Etoile, Francia)1. Para la interpretación de los resultados de sensibilidad obtenidos mediante las técnicas anteriormente citadas se siguen las normas del antibiograma publicadas por diversos organismos, como el Clinical and Laboratory Standards Institute (CLSI)2 y el European Committee on Antimicrobial Susceptibility Testing (EUCAST)3. En el trabajo de rutina, todas estas técnicas se aplican a partir de las colonias crecidas en las placas de aislamiento; los métodos Phoenix y VITEK, que son los más rápidos, requieren un tiempo medio mínimo de 9h para obtener los resultados4. Dado que las infecciones graves requieren la administración de antibióticos con la mayor prontitud posible5, el objetivo de este trabajo es revisar las diferentes técnicas instrumentales aplicadas en Microbiología Clínica para la obtención de un antibiograma rápido, tanto a partir de las colonias, como directamente a partir de diferentes muestras.
Para llevar a cabo la determinación de la sensibilidad de forma rápida se han utilizado diversas técnicas instrumentales, como son técnicas moleculares (incluyendo la secuenciación del genoma completo), citometría de flujo, quimioluminiscencia, espectrometría de masas, métodos comerciales utilizados en la rutina de trabajo, métodos colorimétricos, nefelometría, microarrays, microfluidos o métodos de lisis bacteriana. El objetivo de esta revisión es exponer el fundamento de cada una de estas técnicas y los principales resultados obtenidos.
Técnicas molecularesLas técnicas moleculares permiten la detección de material genético, tanto ácido desoxirribonucleico (ADN) como ácido ribonucleico (ARN). De entre todas las técnicas moleculares utilizadas, la reacción en cadena de la polimerasa (PCR) es la que ha adquirido un mayor valor diagnóstico, permitiendo la detección de agentes infecciosos, además de caracterizar sus genotipos de virulencia y de resistencia6. El primer paso de la PCR consiste en una extracción del material genético, seguido de la desnaturalización térmica del ADN que se va a usar como molde, el anillamiento de cebadores sintéticos y la extensión catalizada por la ADN polimerasa de los oliogonucleótidos anillados que actúan como cebadores. Este proceso de 3 pasos se repite un número determinado de veces (de 25 a 35), duplicándose cada vez el número de moléculas de producto. Esta amplificación exponencial tiene como resultado un gran número de copias de la secuencia de ADN, lo que le confiere una elevada sensibilidad7.
Los importantes avances en el conocimiento de las bases genéticas de la resistencia a antibióticos han permitido que distintas PCR puedan detectar, en unas horas, la presencia de genes de resistencia a una gran variedad de antibióticos para un gran número de especies bacterianas8. Además, la PCR a tiempo real ha sido utilizada para monitorizar el crecimiento bacteriano mediante la detección de los genes de resistencia cuando las bacterias han sido incubadas en presencia del antibiótico a ensayar9,10.
Se han comercializado numerosos ensayos basados en técnicas moleculares para la detección de genes de resistencia a partir del frasco de hemocultivo crecido. Para la detección de Staphylococcus aureus y su resistencia a meticilina se puede utilizar el sistema GeneXpert MRSA/SA (Cepheid, Sunnyvale, CA, EE.UU.) o el Staph ID/R Blood Culture Panel (Great Basin Diagnostics, Salt Lake City, UT, EE.UU.). Estos sistemas requieren una hora para realizar las determinaciones y presentan una sensibilidad y especificidad muy cercanas al 100%11–13. El sistema GeneOhm Staph SR (Becton Dicksinson, Maryland, EE.UU.) es otro sistema que permite la detección de S.aureus y su resistencia a meticilina, también con una sensibilidad y especificidad muy cercanas al 100%. Sin embargo, este sistema requiere 2h14,15. En la detección de bacterias grampositivas, el sistema Verigene BC-GP Blood Culture Nucleic Acid Test (Nanosphere, Northbrook, IL, EE.UU) es capaz de detectar estafilococos, estreptococos, enterococos, micrococos y Listeriaspp., así como la resistencia a meticilina en estafilococos y a vancomicina en enterococos. Este sistema requiere 3h para proporcionar un resultado y muestra una sensibilidad y una especificidad muy cercanas al 100%16. En la detección de bacterias (grampositivas y gramnegativas) y levaduras, el sistema FilmArray Blood Culture Identification panel (BioFire Diagnostics, Salt Lake City, UT, EE.UU.) realiza una PCR múltiple que permite detectar, en una hora, 15 géneros bacterianos, 11 especies bacterianas y 5 especies de levaduras, y además los genes de resistencia mecA, vanA/B y blaKPC. La sensibilidad va del 83 al 100% y la especificidad es >99%, dependiendo del patógeno estudiado17. Hyplex BloodScreen PCR ELISA (Amplex BioSystems, Giessen, Alemania) es un sistema que detecta varias especies bacterianas grampositivas y gramnegativas, además de la presencia del gen mecA. Esta metodología incluye una PCR en la que se amplifica el gen que codifica para el ARNr 16S; seguidamente, el producto de amplificación de la PCR es detectado mediante un ensayo enzimático del tipo del Enzyme-Linked Immunosorbent Assay (ELISA). Este sistema requiere un tiempo de 6h y presenta una sensibilidad del 100% y una especificidad del 92,5 al 100% para la detección de bacterias gramnegativas, una sensibilidad del 96,6 al 100% y una especificidad del 100% para bacterias grampositivas, y una sensibilidad y especificidad del 100% para la detección de S.aureus resistente a meticilina18.
Otra técnica molecular disponible para realizar un antibiograma rápido es la secuenciación del genoma completo. Los avances en la secuenciación del ADN han posibilitado secuenciar un genoma bacteriano completo en horas. Una vez obtenida esta gran cantidad de datos, los programas bioinformáticos se encargan de su procesamiento y análisis. De este modo, esta técnica puede utilizarse para el estudio de determinantes genéticos de resistencias a antibióticos19,20. Además, Zankari et al.21 utilizan la secuenciación del genoma completo para caracterizar los perfiles de resistencia de 200 aislados bacterianos pertenecientes a 4 especies bacterianas. Cuando comparan los resultados obtenidos mediante secuenciación con los obtenidos mediante métodos fenotípicos obtienen un 99,74% de concordancia, demostrando así que la sensibilidad obtenida mediante secuenciación puede correlacionar bien con la sensibilidad obtenida mediante métodos fenotípicos.
Citometría de flujoLa citometría de flujo es un proceso que permite que las partículas (generalmente células) pasen en fila dentro de un flujo a través del aparato con una intensidad de 500-4.000 partículas/s. Cuando esto sucede, es posible realizar la medición simultánea de múltiples características de una sola célula, de tal forma que es posible caracterizar, separar y cuantificar las diferentes subpoblaciones celulares que se engloban en un conjunto22. A principios de la década de los ochenta aparecen los primeros estudios en los que se demuestra la aplicación de la citometría de flujo fluorescente en la determinación de la sensibilidad de bacterias23,24. Posteriormente se han utilizado otros fluorocromos con el fin de obtener información sobre diversos parámetros bacterianos, como el potencial de membrana, el tamaño celular, la cantidad de ADN o la actividad enzimática25. Ramani y Chaturvedi26 determinan la sensibilidad de distintas especies de levaduras a anfotericinaB y fluconazol. Al cabo de unas horas de incubación de las levaduras en presencia de los antifúngicos se añade yoduro de propidio, que solo tiñe los microorganismos con alteraciones en la membrana y, por lo tanto, con deterioro celular. La CMI por citometría de flujo se define como la mínima concentración de antifúngico que proporciona un incremento del 50% de la señal de fluorescencia comparada con la señal obtenida a partir de la disolución control (levadura incubada sin la presencia del antifúngico). Los resultados obtenidos de sensibilidad mediante citometría de flujo son rápidos (6h como máximo, dependiendo de la cepa a estudiar), reproducibles y estadísticamente comparables con los obtenidos por el método de macrodilución en caldo, que es el método de referencia indicado por el CLSI2. Shrestha et al.27 logran diferenciar, en 4h, entre cepas de S.aureus resistentes y sensibles a meticilina mediante histogramas en los que se representa la luz dispersa en un ángulo de 90 grados (SSC) frente a la señal de fluorescencia. Broeren et al.28 calculan la CMI mediante el citómetro de flujo Sysmex UF-1000i de Escherichia coli, Pseudomonas aeruginosa y S.aureus frente a amoxicilina, gentamicina y piperacilina. Definen la CMI por citometría de flujo como la mínima concentración de antibiótico que proporciona una reducción del 80% del recuento bacteriano a los 240min de incubación comparado con el recuento del control sin antibiótico al mismo tiempo de incubación. Los resultados obtenidos mediante citometría de flujo concuerdan en un 100% con los obtenidos mediante los sistemas comerciales VITEK2, E-test y macrodilución en caldo. En micobacterias, Pina-Vaz et al.29 determinan la sensibilidad de Mycobacterium tuberculosis a estreptomicina, isoniazida, rifampicina y etambutol. Después de 3días de incubación en presencia del antituberculoso en el sistema BACTEC MGIT960 (Becton Dickinson, Sparks, MD, EE.UU.), los microorganismos se tiñen con el fluorocromo SYTO16, que solo penetra en las células con alteración de la membrana celular y, comparando la intensidad de la señal de fluorescencia obtenida a partir de estos microorganismos con la obtenida a partir microorganismos incubados durante el mismo tiempo sin la presencia del antituberculoso, se puede diferenciar entre cepas sensibles, intermedias o resistentes, obteniendo unos resultados que muestran una excelente concordancia con los obtenidos mediante una incubación de unas 3semanas en el sistema BACTEC MGIT960. Kirk et al.30 determinan la sensibilidad de M.tuberculosis estudiando la habilidad de la micobacteria para hidrolizar, mediante esterasas, el sustrato diacetato de fluoresceína que pasa a fluoresceína, compuesto que emite fluorescencia cuando se excita con luz de longitud de onda adecuada. Si la micobacteria es sensible al antituberculoso, la capacidad hidrolítica de la micobactería disminuye y, por lo tanto, la señal de fluorescencia detectada también disminuye. Comparando la señal de fluorescencia y la luz dispersa a 90 grados detectada mediante citometría de flujo obtenidas a partir de micobacterias sin contacto con el antituberculoso con las señales obtenidas a partir de micobacterias con una incubación de tan solo 24h con isoniazida, etambutol y rifampicina, obtienen concordancias del 95, del 92 y del 83%, respectivamente, con respecto al antibiograma obtenido mediante el método de las proporciones.
Cohen y Sahar31 diseñan un procedimiento, mediante citometría de flujo, para la detección bacteriana y el estudio de la sensibilidad a amikacina a partir de 43 muestras clínicas y obtienen, en menos de 2h, una sensibilidad del 74% y una especificidad del 88% para la detección de bacterias y una concordancia del 92% en la sensibilidad a amikacina. Por otra parte, Gauthier et al.32 realizan un antibiograma mediante citometría de flujo a partir de muestras de orina. Para ello, usan 2 fluorocromos, el yoduro de propidio y el bis-(1,3-dibutilbarbiturato) trimetilnaoxonol o DiBAC4(3); este último penetra en las células que presentan la membrana despolarizada, y ambos aumentan la señal cuando la célula no es viable. Analizan 114 muestras de orina y realizan las mediciones de la señal fluorescente tras una incubación de 2h en la presencia de los diferentes antibióticos. De este modo obtienen un 2% de discrepancias con respecto a la sensibilidad determinada mediante microdilución en caldo.
QuimioluminiscenciaLa quimioluminiscencia es un proceso de relajación radiante que tiene lugar cuando en una reacción química se genera una especie excitada electrónicamente. La bioluminiscencia es una forma de quimioluminiscencia que aparece como consecuencia de una reacción química que tiene lugar en los organismos vivos, como por ejemplo luciérnagas (insectos de la familia Lampyridae). En esta reacción química, la sustancia luminiscente luciferina es oxidada, en presencia de adenosín trifosfato (ATP), por la acción catalítica de la enzima luciferasa. El sistema bioluminiscente de las luciérnagas tiene muchas aplicaciones analíticas, dado que la enzima luciferasa posee una gran especificidad por determinados sustratos y, además, la cantidad de luz producida es directamente proporcional a la cantidad de ATP de la reacción33. Los trabajos realizados sobre la determinación de la sensibilidad bacteriana mediante bioluminiscencia se fundamentan en la medida de los niveles de ATP bacteriano de origen intracelular, extracelular o total. Para calcular la CMI mediante bioluminiscencia se compara la señal de ATP obtenida a partir de microorganismos incubados sin la presencia de antibiótico (grupo control) con la señal obtenida a partir de microorganismos incubados en presencia de antibiótico a diferentes tiempos, formulando así diferentes criterios de sensibilidad34,35. Con respecto al grupo control, si se mide el ATP intracelular las cepas sensibles al antibiótico estudiado van a proporcionar una disminución de la señal de bioluminiscencia36; si se mide el ATP extracelular las cepas sensibles van a producir un aumento de la señal37, y si se mide el ATP total la cepas sensibles van a proporcionar una disminución de la señal de ATP34,35. En cambio, si el microorganismo es resistente al antibiótico las medidas del grupo control serán prácticamente iguales que las obtenidas a partir de las cepas a estudiar. Para determinar la sensibilidad en bacterias se requieren unas 4h y se obtienen unas CMI muy similares a las obtenidas a partir de la dilución en medio sólido34,35,37. En el caso de micobacterias, en un tiempo mínimo de 5días se obtiene una concordancia prácticamente del 100 % con respecto al método de las proporciones y a los métodos radiométricos33,38,39. En la literatura se encuentran escasos trabajos de determinación de la sensibilidad en levaduras mediante bioluminiscencia. En estos se apunta que con una incubación de aproximadamente 5h se obtiene un acuerdo del 100% con respecto a la CMI obtenida mediante macrodilución40. También se ha demostrado que, mediante bioluminiscencia, es posible determinar la sensibilidad a antibióticos monitorizando la actividad de la enzima adenilato quinasa. Para ello se añade adenosín difosfato (ADP) a los cultivos bacterianos incubados con y sin antibiótico durante 6h; el ADP se transforma en ATP por acción de la enzima adenilato quinasa, y finalmente este se mide mediante bioluminiscencia. Si la cepa es sensible al antibiótico, la actividad de la enzima se verá disminuida y la señal obtenida a partir del ATP será menor que la obtenida a partir de las cepas incubadas sin antibiótico; si la cepa es resistente, la señal obtenida a partir del ATP será muy similar, tanto en las cepas incubadas con antibiótico como en las que no41.
Ivancic et al.42 realizan un antibiograma directamente a partir de muestras de orina mediante bioluminiscencia en el que ensayan 85 muestras monobacterianas y estudian los antibióticos ampicilina, cefalotina, ciprofloxacino, gentamicina, nitrofurantoína, trimetropima-sulfametoxazol y vancomicina. Para la realización del antibiograma primero es preciso eliminar el ATP presente en la orina de origen no bacteriano y después realizar la extracción del ATP bacteriano intracelular. Para estudiar la sensibilidad comparan la señal de ATP procedente de una alícuota de orina con cultivo positivo sin antibiótico con la señal de ATP procedente de otra alícuota de la misma orina que ha estado incubada durante 2h con antibiótico. De esta manera logran un 91% de concordancia con la CMI obtenida a partir de la colonia mediante dilución en caldo.
A diferencia de la bioluminiscencia, la quimioluminiscencia solo requiere la adición de menadiona al cultivo del microorganismo, pudiendo prescindir de la rotura celular. La membrana bacteriana es permeable a esta molécula, y en el interior del microorganismo la menadiona es reducida, generándose diversos compuestos que difunden al medio extracelular, donde se autooxidan y emiten fotones de luz43,44. A finales de la década de los ochenta aparecen diversas publicaciones en las que se apunta que la quimioluminiscencia es una herramienta útil para el estudio de la viabilidad de células y microorganismos45,46. La viabilidad de los microorganismos se predice comparando la señal quimioluminiscente de cepas incubadas con antibióticos con la de cepas incubadas sin antibióticos a diferentes tiempos, proponiendo así diferentes criterios de sensibilidad47–50. Lógicamente, si la cepa es sensible al antibiótico estudiado, se detectará una señal mucho menor que la obtenida a partir del cultivo sin antibiótico. A partir del año 2000 aparecen numerosos trabajos en los que se realiza un antibiograma rápido mediante quimioluminiscencia. Para bacterias, con una incubación de unas 4h se obtiene desde un 88% hasta un 100% de acuerdo entre la CMI obtenida mediante quimioluminiscencia y la obtenida mediante macrodilución o microdilución en caldo47,48,50. Mediante quimioluminiscencia también es posible, con un tiempo de 8h, detectar cepas de S.aureus intermedias y heterorresistentes a vancomicina49. De igual forma, con esta metodología se ha observado que linezolid disminuye, a las 4h de incubación, la intensidad de la señal de quimioluminiscencia por igual en cepas sensibles a linezolid que presentan diferentes sensibilidades a vancomicina51. Finalmente, en micobacterias se ha observado que se necesitan como mínimo 4días para poder llevar a cabo la determinación de la sensibilidad52.
Espectrometría de masasLa espectrometría de masas (MALDI-TOF) es una herramienta basada en espectrometría de masas que permite obtener en unos minutos la identificación de microorganismos tales como bacterias (incluyendo micobacterias), levaduras y hongos filamentosos. La denominación «MALDI» proviene de Matrix-Assisted Laser Desorption/Ionization, y «TOF» alude al analizador de iones que se acopla al MALDI, que es del tipo de tiempo de vuelo (time of flight)53. Para realizar el análisis mediante MALDI-TOF es necesario que las proteínas de los microorganismos se ionicen. Para ello, las colonias de microorganismos se depositan sobre la placa portamuestras, y a continuación sobre esa muestra se deposita una disolución matriz. A continuación, la placa es introducida en la cámara de alto vacío, donde la superficie cristalina de la muestra es expuesta a disparos de un láser de longitud de onda en la zona ultravioleta del espectro, con lo que la matriz absorbe la energía del láser produciéndose la sublimación del analito y de la matriz. Ya en fase gaseosa, la estabilización de la matriz tiene lugar mediante la liberación de protones que, en parte, son captados por las proteínas de las bacterias, generándose fragmentos de proteínas con carga positiva. Mediante un electrodo se genera un campo eléctrico que acelera los iones formados desde las proximidades de la muestra hacia el analizador de masas. De esta forma, los iones entran en un tubo (de 1 a 4m de longitud) con la misma energía cinética y siguiendo una trayectoria lineal. Así, el tiempo que tardan los iones en recorrer el tubo es proporcional a la relación masa/carga (m/z) de los mismos. En última instancia está el detector de los iones previamente separados. Si la carga (z) es igual a uno, como es habitual en la ionización blanda de la técnica MALDI-TOF, el espectro de masas es la representación de la intensidad frente a la masa de los iones formados54,55. El sistema MALDI-TOF realiza la identificación rápida de microorganismos mediante el método de la «huella peptídica». En este método, las proteínas del microorganismo a identificar son hidrolizadas en pequeños péptidos y se obtiene el correspondiente espectro de masas del hidrolizado, que se conoce con el nombre de huella peptídica. Esta huella, que es única para cada microorganismo, es comparada con las huellas de microorganismos conocidos presentes en la base de datos, de tal forma que la huella problema se puede asociar con la huella más semejante y obtener así la identificación del microorganismo56,57.
En referencia al antibiograma rápido, el sistema MALDI-TOF permite predecir en unas horas si las bacterias poseen enzimas que degradan los antibióticos. Para ello, los microorganismos previamente aislados en las placas de cultivo deben ser incubados durante un tiempo con el antibiótico. Posteriormente, con el análisis realizado mediante el sistema MALDI-TOF se observa, en caso de que el microorganismo posea la enzima responsable de la degradación del antibiótico, la desaparición del pico correspondiente al antibiótico y la aparición de nuevos picos que corresponden a los metabolitos resultantes de la rotura del antibiótico. En caso de que la bacteria no hidrolice el antibiótico, se observa únicamente el pico correspondiente al antibiótico. Mediante este procedimiento ha sido posible detectar diferentes carbapenemasas58–61. Esta misma metodología también ha sido aplicada directamente a partir de frascos de hemocultivo crecidos preparados artificialmente con cepas previamente caracterizadas62. También es posible predecir la resistencia a cloranfenicol y clindamicina mediante la detección de la metilación del ARNr16S llevada a cabo por metiltransferasas63. Por otra parte, se ha observado que tanto las cepas de S.aureus sensibles a meticilina como las resistentes proporcionan unos picos de identificación específicos64. De esta forma, a partir de una base de datos, normalmente elaborada en el propio laboratorio, es posible diferenciar entre cepas de S.aureus resistentes y sensibles a meticilina65. De la misma manera, el sistema MALDI-TOF también permite discriminar entre cepas de enterococos resistentes y sensibles a vancomicina66. Finalmente, es posible estudiar la sensibilidad mediante la incubación de microorganismos con antibiótico en un medio con isótopos marcados. Si el microorganismo es resistente, este incorporará el isótopo que posteriormente podrá ser detectado mediante el sistema MALDI-TOF67.
Métodos comercialesTambién se han empleado diferentes métodos comerciales utilizados en la rutina de trabajo del laboratorio de Microbiología Clínica para llevar a cabo un antibiograma directamente a partir de diferentes muestras clínicas. Las tiras comerciales con un gradiente de antibiótico se han utilizado para realizar un antibiograma a partir de muestras respiratorias. Para ello las muestras se siembran en placas de agar Mueller Hinton y seguidamente se depositan las tiras con antibiótico. Las placas son incubadas durante 24h y, una vez crecidas las colonias, la CMI puede ser calculada. Para expresar los resultados del antibiograma directo, estos se clasifican de acuerdo a la Food and Drug Administration (FDA)68 como agreements, minor errors, major errors y very major errors. Tomando como gold standard los resultados de sensibilidad obtenidos mediante microdilución en caldo, Boyer et al.69 obtienen un 88,9% de agreements, un 1,5% de very major errors y un 9,6% de major errors, y Bouza et al.70 obtienen un 96,44% de agreements, un 1,98% de major errors y un 1,56% de minor errors.
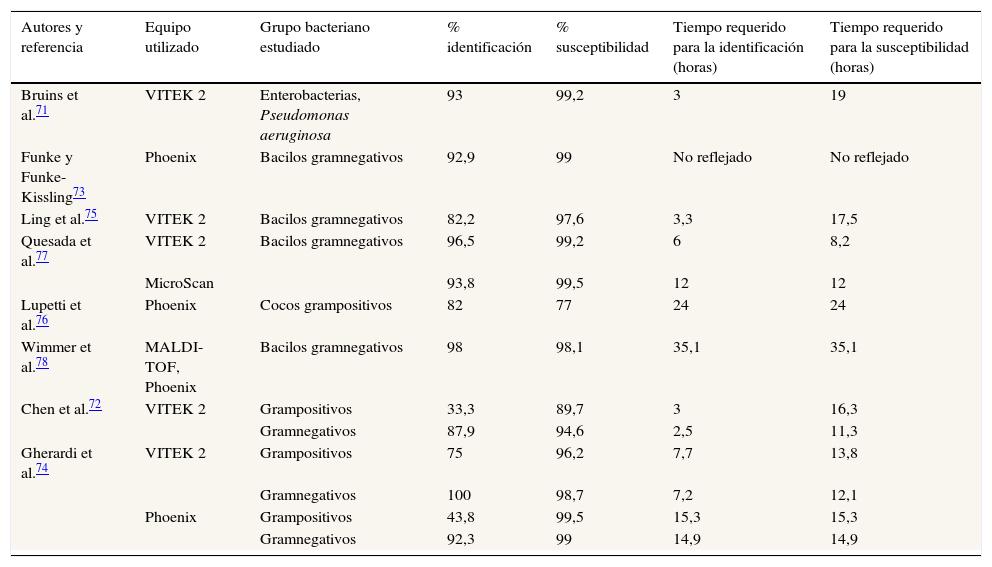
Los métodos de microdilución en caldo estandarizados permiten llevar a cabo la identificación bacteriana y la obtención del antibiograma a partir del frasco de hemocultivo crecido. Los resultados obtenidos se muestran en la tabla 1. Para la identificación bacteriana se requieren 3h, con pobres resultados en bacterias grampositivas y aceptables en gramnegativas; sin embargo, para obtener el antibiograma se precisan 14h, obteniendo buenos resultados en bacterias tanto grampositivas como gramnegativas71–78.
Resultados de la identificación directa y de la determinación de la susceptibilidad a partir de frasco de hemocultivo crecido mediante sistemas comerciales
| Autores y referencia | Equipo utilizado | Grupo bacteriano estudiado | % identificación | % susceptibilidad | Tiempo requerido para la identificación (horas) | Tiempo requerido para la susceptibilidad (horas) |
|---|---|---|---|---|---|---|
| Bruins et al.71 | VITEK 2 | Enterobacterias, Pseudomonas aeruginosa | 93 | 99,2 | 3 | 19 |
| Funke y Funke-Kissling73 | Phoenix | Bacilos gramnegativos | 92,9 | 99 | No reflejado | No reflejado |
| Ling et al.75 | VITEK 2 | Bacilos gramnegativos | 82,2 | 97,6 | 3,3 | 17,5 |
| Quesada et al.77 | VITEK 2 | Bacilos gramnegativos | 96,5 | 99,2 | 6 | 8,2 |
| MicroScan | 93,8 | 99,5 | 12 | 12 | ||
| Lupetti et al.76 | Phoenix | Cocos grampositivos | 82 | 77 | 24 | 24 |
| Wimmer et al.78 | MALDI-TOF, Phoenix | Bacilos gramnegativos | 98 | 98,1 | 35,1 | 35,1 |
| Chen et al.72 | VITEK 2 | Grampositivos | 33,3 | 89,7 | 3 | 16,3 |
| Gramnegativos | 87,9 | 94,6 | 2,5 | 11,3 | ||
| Gherardi et al.74 | VITEK 2 | Grampositivos | 75 | 96,2 | 7,7 | 13,8 |
| Gramnegativos | 100 | 98,7 | 7,2 | 12,1 | ||
| Phoenix | Grampositivos | 43,8 | 99,5 | 15,3 | 15,3 | |
| Gramnegativos | 92,3 | 99 | 14,9 | 14,9 |
Finalmente, las pruebas inmunocromatográficas han sido aplicadas a partir del frasco de hemocultivo crecido. El test BinaxNOW PBP2a (Alere, Waltham, MA, EE.UU.), utilizado para la determinación de sensibilidad de S.aureus a meticilina, requiere unos 30min y muestra una sensibilidad del 95,4% y una especificidad del 100%79. El test Keypath MRSA/MSSA Blood Culture Test (MicroPhage, Longmont, CO, EE.UU.) permite la detección de S.aureus y del gen mecA. Requiere unas 5h y presenta una sensibilidad y una especificidad del 84 al 100%16.
Métodos colorimétricosLas diferentes metodologías basadas en procedimientos colorimétricos requieren unas 6h para poder informar la sensibilidad de las bacterias a los antibióticos y han mostrado muy buenos resultados. La resazurina es un compuesto que, cuando se añade a un cultivo bacteriano en medio líquido resistente al antibiótico con el que es incubado, es reducida por la actividad metabólica de la bacteria. En cambio, si la bacteria es sensible al antibiótico se detiene su metabolismo, con lo que la resazurina permanece en su forma oxidada. Dado que la forma reducida de resazurina es estable y de color azul, y fotométricamente distinguible de la forma oxidada, que es de color rojo, la viabilidad celular puede ser monitorizada mediante espectrofotometría, colorimetría o fluorometría. Utilizando este compuesto se ha determinado la sensibilidad de S.aureus a meticilina y vancomicina80,81. Asimismo ha permitido estudiar el carácter bactericida o bacteriostático de diferentes antibióticos frente S.aureus82. Otro método colorimétrico consiste en la medición de la actividad de la enzima bacteriana nitrato reductasa. Cuando se incuba la bacteria en presencia de iones NO3– y antibiótico, si la bacteria es resistente al antibiótico se van a generar iones NO2– que, con los reactivos de Gries, originan un compuesto de color rojo. Con este método se ha determinado la sensibilidad de S.aureus a meticilina y vancomicina80,81. Del mismo modo, la resazurina y el ensayo de la nitrato reductasa ha sido aplicado a micobacterias. Dixit et al.83 estudian la sensibilidad de M.tuberculosis a rifampicina, isoniazida, estreptomicina y etambutol, y obtienen, en un tiempo de 8 a 14días, una sensibilidad y una especificidad que van del 83,6 al 100% respecto al método de las proporciones.
NefelometríaLa nefelometría es otra técnica analítica basada en la dispersión de luz que permite realizar un antibiograma rápido. El sistema UroQuick o HBandL™ (Alifax, Padua, Italia), diseñado para realizar un cribado de muestras de orina, detecta la presencia de microorganismos en un tubo que contiene un medio de enriquecimiento dado que el crecimiento de estos causa una desviación de la luz medible mediante los correspondientes detectores. Las señales obtenidas son procesadas por un software que monitoriza las curvas de crecimiento. De este modo, este sistema es capaz de informar la concentración microbiana de la muestra84. Este equipo ha sido aplicado con éxito para realizar la detección fenotípica de diversos mecanismos de resistencia de cepas caracterizadas genéticamente o fenotípicamente, requiriendo 24h para bacterias grampositivas y 8h para bacterias gramnegativas85.
La nefelometría también ha sido aplicada para la realización de un antibiograma directo a partir de muestra de orina. Ilki et al.84 centrifugan los tubos del sistema UroQuick o HBandL™ que contienen las orinas incubadas que han sido marcados como positivos. A partir del sedimento obtenido se realiza la identificación y la determinación de la sensibilidad mediante el sistema VITEK2. Tomando como gold standard de identificación la obtenida a partir de la colonia mediante el sistema VITEK2, y como gold standard de sensibilidad la obtenida mediante difusión en disco-placa, obtienen, con un tiempo de 11h, una concordancia >90%. Por otra parte, Roveta et al.86 introducen, de forma manual en el sistema UroQuick o HBandL™, una alícuota de la orina con cultivo positivo positiva junto con el antibiótico en los diferentes tubos que contienen un caldo de enriquecimiento. En 5h predicen la sensibilidad del microorganismo presente en la orina con un acuerdo >90% con respecto a los resultados de sensibilidad obtenidos mediante difusión en disco-placa.
MicroarraysLa metodología de los microarrays consiste en el uso de un sustrato sólido que contiene un gran número de moléculas de ácido nucleico de una sola cadena ensambladas. A estos fragmentos de ácido nucleico se les denomina normalmente sondas, y suelen ir marcadas mediante diversos métodos (enzimáticos, fluorocromos, etc.). Los ácidos nucleicos de las muestras a analizar se incuban en contacto con las sondas, permitiendo así la hibridación a las sondas complementarias. Finalmente, mediante las herramientas informáticas es posible determinar en las muestras la secuencia de los ácidos nucleicos, detectar pequeñas variaciones en la secuencia de los genes o estudiar la expresión de genes87. De este modo, para el estudio de la sensibilidad, los microarrays permiten la detección de un gran número de genes de resistencia en un solo ensayo, caso contrario de lo que ocurre en las PCR. En bacterias gramnegativas es posible detectar, en unas horas, un gran número de genes que codifican para diferentes β-lactamasas88-90.
MicrofluidosLos avances en nanotecnología han posibilitado la miniaturización de los diferentes ensayos usados para la detección de las resistencias a los antibióticos desarrollando plataformas o chips que utilizan volúmenes muy pequeños de muestra y de reactivos, y que además pueden albergar múltiples ensayos como cultivo bacteriano, hibridación y amplificación de ácidos nucleicos y rotura celular, entre otros. De este modo, los métodos de detección varían ampliamente, dependiendo del ensayo realizado8. Tang et al.91 desarrollan una plataforma que incorpora un sensor de pH mediante el cual, midiendo los cambios de pH que ocurren como consecuencia de la acumulación de productos metabólicos, es posible detectar el crecimiento bacteriano en 2h. Choi et al.92 utilizan un sistema de microfluidos con cámaras de cultivo que contienen agarosa. Este compuesto es capaz de inmovilizar las bacterias individualmente, y mediante microscopia se monitoriza el crecimiento bacteriano. Esta metodología permite obtener la CMI en 3-4h. Finalmente, Mach et al.93 realizan un antibiograma directamente a partir de muestras de orina utilizando un sistema de microfluidos que incorpora un sensor electroquímico para la cuantificación del ARN ribosómico 16S. Este grupo obtiene, en 3h y media, un 94% de concordancia con la sensibilidad obtenida mediante microdilución en caldo.
Métodos de lisis bacterianaOtra metodología para la determinación de la sensibilidad consiste en la detección de la lisis bacteriana. Para ello, la bacteria es incubada con la presencia del antibiótico a la concentración deseada; seguidamente la bacteria se inmoviliza en un microgel de agarosa y es expuesta a una solución de lisis que produce la liberación del ADN8. Posteriormente, la preparación es incubada con el fluorocromo SYBR Gold y mediante observación al microscopio de fluorescencia es posible estudiar la integridad del ADN. Esta metodología requiere menos de 2h y ha sido aplicada tanto a bacterias gramnegativas como grampositivas94–96.
ConclusiónA modo de conclusión podemos señalar que las metodologías descritas en este trabajo permiten acortar el tiempo necesario para obtener la sensibilidad de las bacterias a los antibióticos respecto a los métodos habituales. Para la interpretación del antibiograma es indispensable conocer la especie bacteriana que se está estudiando. En este sentido, la identificación rápida proporcionada por el sistema MALDI-TOF permite la implementación de las metodologías que realizan un antibiograma rápido. Sin embargo, para determinar si estas técnicas proporcionan una sensibilidad y una especificidad aceptables para la práctica clínica, es necesario ampliar el número de cepas bacterianas estudiadas. Además, en los ensayos en los que se detecta la presencia de ciertos determinantes de resistencia, como las técnicas moleculares y el sistema MALDI-TOF, es importante establecer la concordancia que existe entre la sensibilidad obtenida mediante métodos fenotípicos y la presencia de los determinantes de resistencia. Dados los importantes beneficios de proporcionar un resultado rápido de sensibilidad (aumento de la supervivencia, reducción de gastos y retraso de la selección de cepas bacterianas resistentes)28,97, el antibiograma en Microbiología Clínica, lejos de estar definitivamente establecido, es un campo de continuo avance.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.