El objetivo de este estudio fue caracterizar microbiológicamente 154 aislados de Listeria monocytogenes procedentes de casos humanos ocurridos en Andalucía en el período 2005-2009.
Material y métodosLa serotipificación se realizó mediante antisueros comerciales frente a antígenos somáticos de L. monocytogenes y por PCR multiplex de acuerdo con el método descrito por Doumith et al. (2004). La sensibilidad antibiótica fue estudiada por Epsilon test e interpretada por criterios CLSI. La relación clonal mediante PFGE de los fragmentos de restricción obtenidos con la enzima ApaI se realizó según protocolo PulseNet y el software Bionumerics. La identificación de clones epidémicos ECI, ECII y ECIII se estableció mediante un protocolo de PCR multiplex descrito por Chen y Knabel (2007).
ResultadosLos 154 aislados fueron agrupados en 4 serotipos: 4b [n=94 (61%)], 1/2b [n=30 (19%)], 1/2a [n=27 (18%)] y 1/2c [n=3 (2%)], siendo el 100% sensibles a ampicilina y cotrimoxazol. Se identificaron 62 pulsotipos diferentes. Treinta y siete aislados (24%) mostraron pulsotipos únicos, y los aislados restantes (76%) fueron asignados a 25 grupos PFGE (60% en grupos de más de 2 aislados). Los marcadores EC se encontraron en 62 (40,3%) aislados. El marcador de ECI estuvo presente en 43 (46,2%) de los aislados serotipo 4b, ECII en 10 (10,7%) aislados serotipo 4b, y ECIII en 9 (33,3%) aislados serotipo 1/2a.
DiscusiónUna gran proporción de los aislados de L. monocytogenes pertenecientes a casos de listeriosis humana de nuestro estudio se agrupan clonalmente mediante técnicas de subtipado molecular, lo que sugiere la presencia de clones circulantes que podrían estar relacionados con brotes internacionales de origen alimentario o corresponder a cepas mas virulentas.
The aim of this study was to perform a retrospective study by genotyping 154 isolates from human listeriosis cases occurred in the region of Andalusia (southern Spain) in the period 2005-2009.
Material and methodsSerotyping was performed for 1 and 4 somatic antigens using commercial Listeria antisera, and by multiplex-PCR serogrouping according to the method described by Doumith et al. (2004). The antimicrobial susceptibility was performed by Epsilon test and interpreted by CLSI criteria. PFGE was performed according to the PulseNet protocol with the ApaI enzyme. The similarity of PFGE profiles was evaluated using the Bionumerics software. The multiplex PCR protocol described by Chen and Knabel (2007) was used for the identification of isolates belonging to L. monocytogenes ECI, ECII, and ECIII epidemic clones.
ResultsThe 154 isolates were grouped into four serotypes: 4b [94 (61%)] strains, 1/2b [30 (19%)] strains, 1/2a [27 (18%)] strains, and 1/2c [3 (2%)] strains, with 100% of susceptibility to ampicillin and cotrimoxazole. A further sixty-two ApaI distinct pulsotypes were recognized. Thirty-seven isolates (24%) showed unique ApaI pulsotypes, and the remaining 117 strains (76%) were assigned to 25 ApaI clusters (60% in clusters of more than two isolates). The EC markers were found in 62 (40.3%) of the L. monocytogenes isolates tested. The ECI marker was present in 43 (46.2%) 4b serotype isolates, ECII in 10 (10.7%) 4b serotype isolates, and ECIII in 9 (33,3%) 1/2a serotype isolates.
DiscussionA large proportion of the human listeriosis cases under investigation could be grouped into molecular subtype clusters, and our cases could be related to international food-borne outbreaks.
La listeriosis es una enfermedad invasiva producida por una bacteria intracelular, Listeria monocytogenes. Además, es una zoonosis que en humanos afecta a grupos de riesgo bien definidos: mayores de 65 años, inmunodeprimidos, gestantes y neonatos. Este microorganismo posee la habilidad excepcional de cruzar barreras biológicas, causando generalmente aborto, septicemia o infección del sistema nervioso central, y su mortalidad es cercana al 30%1,2.
La importancia de la listeriosis no está suficientemente reconocida, ya que se trata de una enfermedad relativamente poco común. De hecho, está considerada una enfermedad rara, y como tal está incluida en ORPHANET, el Consorcio Europeo para Enfermedades y Medicamentos Huérfanos.
A nivel europeo, de los 1.454 casos confirmados de listeriosis durante el año 2008 (últimos datos suministrados por el European Centre for Disease Prevention and Control [ECDC]), la mayoría (56%) correspondían a personas mayores de 65 años, con una incidencia de 0,94 casos/100.000, seguido de los niños menores de 5 años, con 0,35 casos/100.000 habitantes3. En nuestro país no existen datos de incidencia de la infecciones por L. monocytogenes, pues no se considera una enfermedad de declaración obligatoria. Desde 1999 la listeriosis se incluyó dentro de las enfermedades de declaración obligatoria en Andalucía, declarándose 50 nuevos casos en el año 2010, con una tasa de 0,60 casos/100.000 habitantes e incrementándose respecto a los 0,54 casos/100.000 habitantes del periodo 2003-20094, lo que supone el doble que el declarado en la población europea.
En Europa, la mayoría de los casos de listeriosis no están vinculados a una fuente común, y por tanto se definen como esporádicos. Sin embargo, debido a las características epidemiológicas y clínicas de la enfermedad, los sistemas tradicionales de vigilancia no son capaces de detectar casos asociados, sobre todo cuando un número limitado de casos están dispersos en una amplia área geográfica.
La caracterización molecular de los aislados de origen humano de L. monocytogenes debería ser una parte esencial de la investigación epidemiológica. La combinación de métodos fenotípicos y genotípicos se ha demostrado altamente eficaz en la investigación de brotes de la enfermedad5,6. La subtipificación a nivel molecular permite caracterizar L. monocytogenes más allá de la especie y subespecie, ayudando a diferenciar los brotes de enfermedad de los casos esporádicos y proporcionando una mejor comprensión de la relación entre los aislados y sus patrones de diseminación.
Varios estudios han demostrado que, en general, los brotes de listeriosis están causados por un pequeño número de cepas estrechamente relacionadas, lo que sugiere que estas cepas pueden pertenecer a grupos clonales bien definidos7-9. Así, se han descrito clones epidémicos que incluyen cepas que están genéticamente relacionadas e implicadas en uno o más brotes a lo ancho del mundo. Actualmente existen métodos moleculares para la detección de los clones epidémicos mas prevalentes: ECI, ECII y ECIII10, y estos podrían ser empleados de forma rutinaria en la caracterización de aislados procedentes de casos humanos11.
El objetivo de este trabajo fue caracterizar microbiológicamente, tanto desde el punto de vista fenotípico como genotípico, 154 cepas de L. monocytogenes aisladas de casos humanos ocurridos en Andalucía en el periodo 2005-2009.
Material y métodosAislados bacterianosEl estudio incluyó a 154 aislados procedentes de casos de listeriosis humana. Ciento treinta y nueve se obtuvieron de los laboratorios de microbiología de 15 hospitales de Andalucía entre 2005 y 2009, en el marco del proyecto LISAND (colaboración de la Sociedad Andaluza de Microbiología y Parasitología y de la Sociedad Andaluza de Enfermedades Infecciosas para estudiar los casos humanos de listeriosis en Andalucía). Adicionalmente, se han incluido 15 aislados esporádicos humanos de L. monocytogenes que habían sido identificados por los mismos laboratorios desde 1993 hasta 2004. Todos los aislados se caracterizaron fenotípicamente según procedimientos recomendados12. Noventa y ocho (64%) cepas habían sido aisladas de sangre, 46 cepas de líquido cefalorraquídeo (30%), 5 (3%) de líquido ascítico y 5 (3%) de otras procedencias (líquidos articular, pleural, peritoneal y biliar). El estudio no incluye aislados procedentes de casos neonatales. Cada aislado en este estudio representaba un caso de listeriosis humana. Ningún caso había sido asignado como perteneciente a brotes epidémicos.
SerotipadoEl serotipado de los antígenos somáticos 1 y 4 de L. monocytogenes se realizó con antisueros comerciales para Listeria (BBL-Difco), de acuerdo con las instrucciones del fabricante.
Complementariamente se realizó el estudio de serogrupos por PCR multiplex, de acuerdo con el método descrito por Doumith et al.13. Se utilizaron 5 parejas de cebadores para amplificar los genes Lmo0737, Lmo1118, ORF 2819, ORF 2110 y prs. Los fragmentos amplificados se sometieron a electroforesis en gel de agarosa al 2%.
Estudio de sensibilidad antibióticaEl estudio de la actividad de ampicilina y cotrimoxazol se realizó en agar Mueller-Hinton 5% sangre de caballo, mediante Epsilon-test (E-test. BioMerieux. Francia) según las instrucciones del fabricante, empleando una cepa de Streptococcus pneumoniae ATCC 49619 como control de calidad. Los valores de CMI fueron interpretados según recomendaciones CLSI14.
Electroforesis en campo pulsadoEl estudio de las cepas mediante electroforesis en campo pulsado (PFGE) se realizó según el protocolo de PulseNet15, con pequeñas modificaciones16, empleando como enzima de restricción ApaI y como estándar de referencia ADN de Salmonella enterica serotipo Braenderup H9812 digerido con XbaI.
La similitud de los perfiles de PFGE se evaluó utilizando el software Bionumerics version 5.1 (Applied Maths, Sint-Martens-Latem, Bélgica). El agrupamiento se realizó mediante la aplicación del coeficiente de correlación de Dice y el método UPGMA (Unweighted Pair Group Method Using Arithmetic Average), con un 1,5% de tolerancia según lo recomendado por Martin et al.17. El índice de discriminación de Simpson, expresado como porcentaje, se determinó según lo descrito por Hunter y Gaston18. Un pulsotipo se definió para un porcentaje de similitud de bandas de ≥99% y de ≥90% para cepas muy relacionadas19.
Multiplex PCR para la detección de clones epidémicosEn la identificación de los aislados pertenecientes a clones epidémicos ECI, ECII y ECIII, se utilizó el protocolo descrito por Chen y Knabel10, con pequeñas modificaciones (se realizaron las amplificaciones por separado), empleándose una escalera de ADN 1kpb (Promega) como marcador de peso molecular.
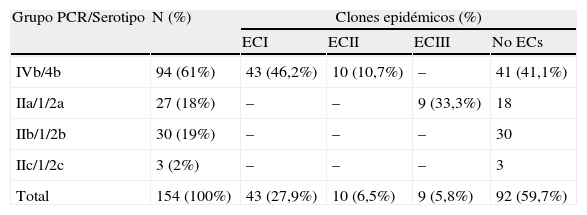
ResultadosSerotipadoDe los 154 aislados clínicos, 94 (61%) pertenecían al serogrupo 4 y 60 (39%) al serogrupo 1.
El agrupamiento mediante PCR multiplex permitió asignar los aislados a 4 grupos: IIa: 27 cepas (18%); IIb: 30 cepas (19%); IIc: 3 cepas (2%), y IVb: 94 cepas (61%) (tabla 1).
Características microbiológicas de los aislados de Listeria monocytogenes
| Grupo PCR/Serotipo | N (%) | Clones epidémicos (%) | |||
| ECI | ECII | ECIII | No ECs | ||
| IVb/4b | 94 (61%) | 43 (46,2%) | 10 (10,7%) | – | 41 (41,1%) |
| IIa/1/2a | 27 (18%) | – | – | 9 (33,3%) | 18 |
| IIb/1/2b | 30 (19%) | – | – | – | 30 |
| IIc/1/2c | 3 (2%) | – | – | – | 3 |
| Total | 154 (100%) | 43 (27,9%) | 10 (6,5%) | 9 (5,8%) | 92 (59,7%) |
En función de la combinación de los 2 métodos, los aislados se agruparon en 4 serotipos: 4b: 94 cepas (61%); 1/2b: 30 cepas (19%); 1/2a: 27 cepas (18%), y 1/2c: 3 cepas (2%).
Sensibilidad antibióticaEl 100% de las cepas fueron sensibles a ampicilina y cotrimoxazol según criterios CLSI. Los valores de CMI modal para ampicilina fueron CMI50: 0,25mg/l y CMI90: 0,75mg/l (rango: 0,064-1mg/l) y para cotrimoxazol CMI50: 0,016mg/l y CMI90: 0,125mg/l (rango: 0,002-0,5mg/l).
Estratificando según el serotipo y el clon epidémico, los más prevalentes fueron:
• Serotipo 4b (94 cepas): ampicilina CMI50: 0,25mg/l; CMI90: 0,5mg/l (rango: 0,094-1mg/l) y cotrimoxazol CMI50: 0,016mg/l; CMI90: 0,032mg/l (rango: 0,002-0,094mg/l).
• Clon epidémico ECI (43 cepas): ampicilina CMI50: 0,25mg/l; CMI90: 0,5mg/l (rango: 0,125-1mg/l) y cotrimoxazol CMI50: 0,016mg/l; CMI90: 0,047mg/l (rango: 0,004-0,094mg/l).
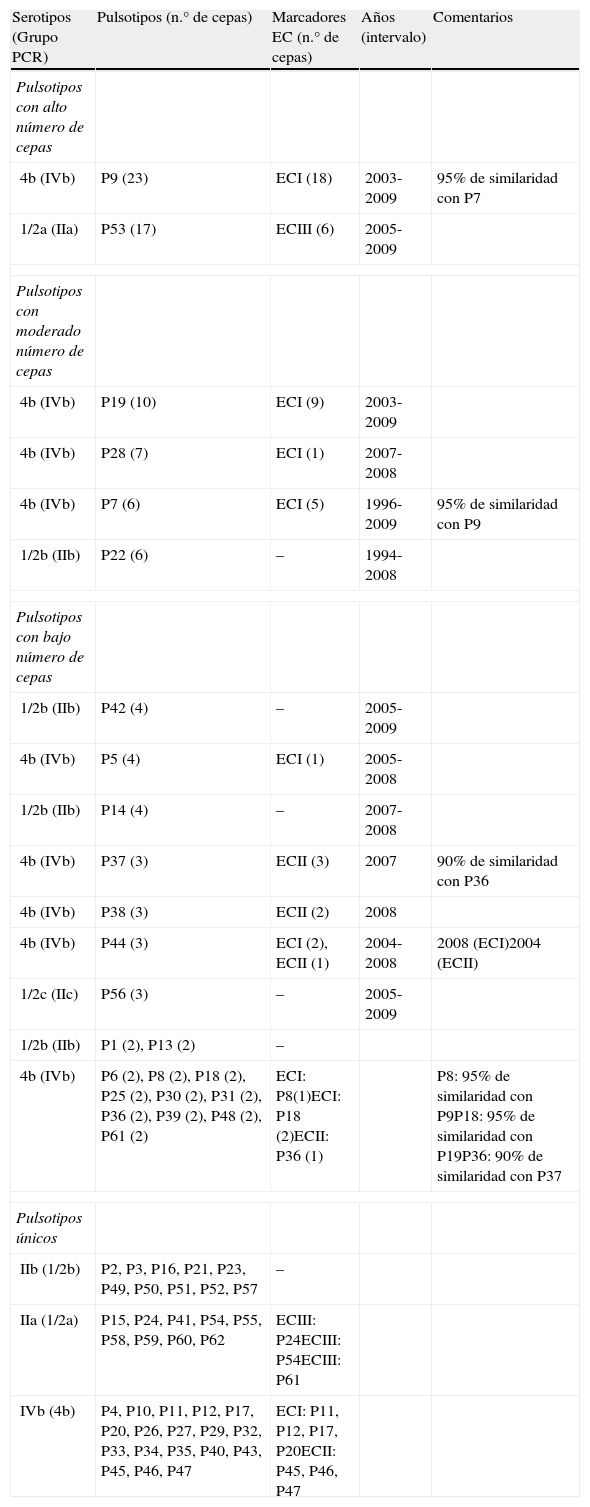
Caracterización molecular y agrupamiento por electroforesis en campo pulsadoEntre los 154 aislados se diferenciaron 62 pulsotipos distintos. Treinta y siete aislados (24%) mostraron pulsotipos únicos, y las restantes 117 cepas (76%) fueron asignadas a 25 grupos ApaI, el 60% en grupos de más de 2 cepas (tabla 2).
Distribución de pulsotipos y marcadores EC en los aislados de Listeria monocytogenes
| Serotipos (Grupo PCR) | Pulsotipos (n.° de cepas) | Marcadores EC (n.° de cepas) | Años (intervalo) | Comentarios |
| Pulsotipos con alto número de cepas | ||||
| 4b (IVb) | P9 (23) | ECI (18) | 2003-2009 | 95% de similaridad con P7 |
| 1/2a (IIa) | P53 (17) | ECIII (6) | 2005-2009 | |
| Pulsotipos con moderado número de cepas | ||||
| 4b (IVb) | P19 (10) | ECI (9) | 2003-2009 | |
| 4b (IVb) | P28 (7) | ECI (1) | 2007-2008 | |
| 4b (IVb) | P7 (6) | ECI (5) | 1996-2009 | 95% de similaridad con P9 |
| 1/2b (IIb) | P22 (6) | – | 1994-2008 | |
| Pulsotipos con bajo número de cepas | ||||
| 1/2b (IIb) | P42 (4) | – | 2005-2009 | |
| 4b (IVb) | P5 (4) | ECI (1) | 2005-2008 | |
| 1/2b (IIb) | P14 (4) | – | 2007-2008 | |
| 4b (IVb) | P37 (3) | ECII (3) | 2007 | 90% de similaridad con P36 |
| 4b (IVb) | P38 (3) | ECII (2) | 2008 | |
| 4b (IVb) | P44 (3) | ECI (2), ECII (1) | 2004-2008 | 2008 (ECI)2004 (ECII) |
| 1/2c (IIc) | P56 (3) | – | 2005-2009 | |
| 1/2b (IIb) | P1 (2), P13 (2) | – | ||
| 4b (IVb) | P6 (2), P8 (2), P18 (2), P25 (2), P30 (2), P31 (2), P36 (2), P39 (2), P48 (2), P61 (2) | ECI: P8(1)ECI: P18 (2)ECII: P36 (1) | P8: 95% de similaridad con P9P18: 95% de similaridad con P19P36: 90% de similaridad con P37 | |
| Pulsotipos únicos | ||||
| IIb (1/2b) | P2, P3, P16, P21, P23, P49, P50, P51, P52, P57 | – | ||
| IIa (1/2a) | P15, P24, P41, P54, P55, P58, P59, P60, P62 | ECIII: P24ECIII: P54ECIII: P61 | ||
| IVb (4b) | P4, P10, P11, P12, P17, P20, P26, P27, P29, P32, P33, P34, P35, P40, P43, P45, P46, P47 | ECI: P11, P12, P17, P20ECII: P45, P46, P47 | ||
El 45% (n=69) de las cepas se agrupaban en 6 pulsotipos (fig. 1). El pulsotipo más frecuente fue el P9, con 23 cepas del serogrupo 4b, aisladas en el período 2003-2009; había además 2 pulsotipos estrechamente relacionados, P7 y P8, con 6 y 2 cepas, respectivamente, por lo que el número de cepas asciende a 31. El siguiente pulsotipo en frecuencia fue P53, con 17 cepas del serogrupo 1/2a, aisladas entre 2005 y 2009. El pulsotipo P19 incluía 10 cepas del serogrupo 4b aisladas entre 2003-2009, presentaba una estrecha relación con el pulsotipo P18 y elevaba la agrupación a 12 cepas. Seguía en frecuencia el pulsotipo P28, con 7 cepas del serogrupo 4b aisladas en el período 2007-2008. Por último, el pulsotipo P22 incluía 6 cepas del serogrupo 1/2b aisladas en el período 1994-2008.
El 31% de los pulsotipos (19/62) incluían un bajo número de cepas (entre 2 y 4). El 25% de estas cepas (12/48) pertenecían al serogrupo 1/2b, y el 69% (33/48), al serogrupo 4b. El serogrupo 1/2c incluyó 3 cepas (aisladas entre 2005 y 2009) en un único pulsotipo (P56).
Los pulsotipos únicos representaron el 24% de las cepas y el 60% de la pulsotipos: el 49% del serogrupo 4b, el 27% del serogrupo 1/2b y el 24% del serogrupo 1/2a.
Investigación de clones epidémicosLos marcadores EC se encontraron en 62 (40,3%) de las aislados estudiados (tabla 1). El marcador ECI estaba presente en 43 (46,2%) de los aislados serotipo 4b, ECII en 10 (10,7%) de los aislados serotipo 4b, y ECIII en 9 (33,3%) aislados del serotipo 1/2a.
El marcador ECI se detectó en 18 de las 23 cepas del grupo P9, y en 6 de las 8 cepas de los pulsotipos estrechamente relacionados P7 y P8. También en 8 de las 10 cepas que componían el pulsotipo P19, y en las 3 cepas de los pulsotipos estrechamente relacionados P18 y P20. Las 8 restantes (21,6%) con este marcador fueron aisladas de casos esporádicos (tabla 2).
El marcador ECII se detectó en 6 cepas de los pulsotipos P36, P38 y P39 (83% de similitud), mientras que las otras 4 cepas con este marcador fueron de casos esporádicos (tabla 2).
El marcador ECIII fue encontrado en 7 de 17 cepas del pulsotipo P53, y las 2 restantes correspondieron a casos esporádicos (tabla 2).
DiscusiónLa distribución de serotipos en nuestro estudio es similar a la descrita en otros países europeos20, con un predominio del serotipo 4b. Los posibles mecanismos por los que existe una mayor prevalencia del serotipo 4b, así como de 1/2a y 1/2b, en la enfermedad humana siguen siendo poco conocidos21.
Al igual que en otros estudios europeos, no se constataron problemas de resistencia antibiótica a ampicilina y cotrimoxazol en las cepas estudiadas22.
Durante el período de 4 años que abarca este estudio, ciertos pulsotipos de L. monocytogenes se mantuvieron estables en el tiempo y se distribuyeron ampliamente, concretamente 5 pulsotipos representaron el 39,9% de las cepas estudiadas. De ellos, 2 grandes grupos de los serotipos 1/2a (P53) y 4b (P9) representaron el 26% de los aislados, distribuyéndose en un período prolongado de tiempo (2003-2009 y 2005-2009, respectivamente), sin que exista constancia de que los casos fueran asignados por las autoridades sanitarias a brotes epidémicos de la enfermedad.
Estos resultados demuestran la importancia del subtipado molecular de rutina de los aislados clínicos de L. monocytogenes, ya que podría alertar sobre posibles brotes de enfermedad. Este aspecto ha sido puesto de manifiesto por los ECDC23,24. Sin embargo, aunque el uso de técnicas de PFGE para la tipificación molecular de aislados ha demostrado gran utilidad en la caracterización de casos esporádicos o epidémicos, los criterios de Tenover25 no son generalmente aplicables en estudios a largo plazo y solo aislados con patrones indistinguibles deberían ser incluidos en los pulsotipos19. Por ello, en nuestro estudio se requirió una similitud del 99% en el patrón PFGE. Además, el uso de la enzima de restricción ApaI, que proporciona un mayor número de bandas, permite un mayor poder discriminatorio, como se ha descrito previamente26.
Por otro lado, la mayoría de los brotes de listeriosis descritos en la literatura han involucrado a un pequeño grupo de cepas genéticamente muy relacionadas. Debido a los problemas potenciales de los estudios de PFGE en estudios a largo plazo, reconocer grupos clonales puede cambiar el enfoque de los estudios epidemiológicos. En L. monocytogenes se han reconocido 3 grupos principales de clones epidémicos designados como ECI, ECII y ECIII7-9. En nuestro estudio se ha encontrado una alta prevalencia (40,5%) de estos marcadores. El marcador ECI, relacionado con brotes de listeriosis en Nueva Escocia (1981), Massachusetts (1983), Los Ángeles (1985), Suiza (1983-87), Dinamarca (1985-87) y Francia (1992)27, se ha encontrado en el 28% de nuestras cepas, sobre todo en pulsotipos con amplio número de cepas. El primer aislado con este marcador aparece en el año 1996, distribuyéndose de manera uniforme a lo largo de nuestro período de estudio.
La distribución del marcador ECII, vinculado a 2 brotes en Estados Unidos en 1998-1999 y 200227, parece ser más heterogéneo y aparece en pulsotipos de bajo número de aislados o en pulsotipos de casos aparentemente esporádicos. La heterogeneidad del grupo ECII ha sido demostrada por otros investigadores en Europa28.
En nuestro estudio, el marcador ECIII (este grupo clonal, a diferencia de los anteriores, solo incluye cepas del serotipo 1/2a), vinculado a un brote multiestatal de listeriosis asociada al consumo de embutidos de pavo en Estados Unidos en el año 200029, está asociado con un solo pulsotipo (P53), incluyendo aislados del periodo 2005-2008. Esto concuerda con el estudio de Mammina et al.11 realizado en Italia, donde las 3 cepas con marcadores ECIII descritas estaban agrupadas en un único pulsotipo.
Es importante señalar que la presencia de marcadores ECII y ECIII ha sido poco comunicada en casos humanos en Europa. Solo un estudio realizado en Italia en 2009 evidenció la existencia de estos marcadores en aislados clínicos de L. monocytogenes11.
En consecuencia, un alto porcentaje de los casos de listeriosis humana en Andalucía podrían estar relacionados con brotes epidémicos ocurridos a nivel internacional y/o la presencia de clones más virulentos.
Este es el primer gran estudio de epidemiología molecular de casos humanos de listeriosis en España, y más concretamente en Andalucía, y sería conveniente disponer de datos similares de otras zonas de España para comprobar la distribución de serotipos, pulsotipos y marcadores EC. Por tanto, nuestro trabajo podría ser útil en la reinterpretación de la epidemiología de la listeriosis en el sur de España, así como contribuir a la adopción de medidas de prevención de la enfermedad en el futuro.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
María Dolores López-Prieto (Hospital de Jerez, Cádiz), José Porras (Hospital Universitario Carlos Haya, Málaga), Marina de Cueto (Hospital Universitario Virgen Macarena, Sevilla), Concepción Carazo (Complejo Hospitalario de Jaén, Jaén), Ana Isabel Aller (Hospital Universitario de Valme, Sevilla), Alberto de la Iglesia (Hospital Infanta Elena, Huelva), José Antonio Pérez (Hospital Universitario San Cecilio, Granada), Manuel Causse (Hospital Universitario Reina Sofía, Córdoba), María Victoria García (Hospital Universitario Virgen de la Victoria, Málaga), Consuelo Miranda (Hospital Universitario Virgen de las Nieves, Granada), José María Saavedra (Hospital Juan Ramón Jiménez, Huelva), Rocío Tejero (Hospital Infanta Margarita, Cabra, Córdoba), Francisco Franco (Hospital de Riotinto, Huelva), Ana García-Tapia (Hospital Universitario Puerta del Mar, Cádiz), Manuel Rodríguez-Iglesias (Hospital Universitario Puerto Real, Cádiz), Miguel Martínez-Lirola (Complejo Hospitalario Torrecárdenas, Almería), Inmaculada López-Rodríguez (Hospital Costal del Sol, Marbella, Málaga), María del Carmen Domínguez (Hospital de Osuna, Sevilla), María Jesús Pérez-Santos (Hospital de la Serranía, Ronda, Málaga), María Teresa Cabezas (Hospital de Poniente, Almería), Carmen Serrano (Hospital San Juan de Dios, Bormujos, Sevilla).
Los componentes del grupo LISAND-Microbiología se relacionan en el anexo 1.