Evaluamos tres procedimientos de identificación rápida de microorganismos a partir de hemocultivos positivos.
MétodosAplicamos dos métodos basados en la extracción directa desde el frasco de hemocultivo: Sepsityper® (Bruker Daltonics) (ST) y un método casero con saponina (MCS), y un tercer método basado en un subcultivo con incubación corta (SIC). Se comparan las identificaciones por espectrometría de masas Matrix-Assisted Laser Desorption Ionization-Time of Flight (EM-MALDI-TOF) aplicando los criterios de interpretación del fabricante y los puntos de corte corregidos (PCC).
ResultadosAplicando los criterios del fabricante se identificaron a nivel de especie el 65,8%, el 45,8% y el 57,4% con ST, MCS y SIC, respectivamente. Aplicando los PCC, estos resultados fueron del 92,3%, del 80,6%, y del 85,2%, respectivamente. La identificación con ST fue significativamente mejor que el MCS. ST y SIC no mostraron diferencias significativas, excepto en levaduras.
ConclusionesST y SIC obtienen buenas tasas de identificación y pueden integrarse fácilmente en cualquier laboratorio.
Three procedures for rapid identification of microorganisms in positive blood cultures were evaluated.
MethodsWe performed two methods based on direct extraction from a blood culture: Sepsityper® (Bruker Daltonics) (ST) and a non-commercial saponin method (MCS), and another method consisting of a short incubation subculture (SIC). Identification values obtained by spectrometry Matrix-Assisted Laser Desorption Ionization-Time of Flight (EM MALDI-TOF) were compared by applying the manufacturer's interpretation criteria and corrected cut-off points.
ResultsAccording to the manufacturer, 65.8%, 45.8% and 57.4% of microorganisms were identified at the species level by using ST, MCS and SIC, respectively. When applying corrected cut-off points, the values increased to 92.3%, 80.6% and 85.2%, respectively. ST offered significantly better results than MCS, and no significant differences were found between ST and SIC, except for with respect to yeast.
ConclusionsBetter identification rates were obtained by using ST and SIC, which are easily applicable in any laboratory.
Instaurar una terapia antibiótica empírica efectiva supone mejorar el pronóstico de los pacientes y reducir el tiempo de hospitalización y los costes sanitarios asociados1.
Köck et al.2 encontraron que los clínicos cambiaban los tratamientos empíricos al 20% de los pacientes después de conocer la identificación de la especie, frente al 8% cuando solo tenían los resultados de la microscopía. En base a datos como estos, se han desarrollado nuevos métodos basados en identificaciones mediante espectrometría de masas tipo Matrix-Assisted Laser Desorption Ionization-Time of Flight (MALDI-TOF) a partir de botellas de hemocultivo positivas, tanto de forma directa utilizando técnicas de extracción comerciales o caseras3-10, como indirecta a partir de subcultivos de incubación corta11-14. En cuanto a la interpretación de las puntuaciones obtenidas por MALDI-TOF, Kohlmann et al.11 publicaron unos puntos de corte corregidos (PCC) basados en si los microorganismos eran grampositivos o no.
El primer objetivo de este estudio fue valorar la eficacia de tres procedimientos para la identificación rápida de microorganismos a partir de hemocultivo positivo y su aplicabilidad en la rutina del laboratorio. El segundo objetivo fue comparar las puntuaciones obtenidas por MALDI-TOF aplicando los criterios de interpretación del fabricante y los propuestos por Kohlmann et al., para evaluar cómo afectan en la identificación final de los microorganismos.
Material y métodosSe presenta un estudio observacional comparativo realizado entre 2014 y 2016 que evalúa tres métodos para la identificación rápida de patógenos a partir de hemocultivos positivos.
Se consideró una botella por paciente y episodio de bacteriemia. Se empleó el sistema BacT/ALERT (bioMérieux, Marcy l’Etoile, Francia). Los frascos positivos se procesaron siguiendo los protocolos habituales. Tras 24h de incubación, los microorganismos aislados fueron identificados por MALDI-TOF (Bruker Daltonics, Bremen, Alemania) y por pruebas bioquímicas automatizadas (MicroScan WalkAway, Beckman Coulter, EE.UU.). En el caso de Streptococcus pneumoniae y estreptococos del grupo viridans, las identificaciones se confirmaron mediante sensibilidad a la optoquina y solubilidad en bilis. Se excluyeron los hemocultivos positivos en los que la tinción de Gram revelaba una etiología polimicrobiana.
De la primera botella positiva de cada paciente estudiado se obtuvieron tres alícuotas para evaluar los procedimientos siguientes:
- 1)
Sepsityper® Kit (ST) (Bruker Daltonics, Bremen, Alemania). Se procesa 1ml del hemocultivo según las especificaciones del fabricante y se realiza la extracción etanol/fórmico. Posteriormente, se deposita 1μl del sobrenadante en una placa metálica.
- 2)
Método casero con saponina (MCS). Basado en la modificación del protocolo de Martiny et al.5. Se extrae 1ml del frasco de hemocultivo, se añaden 500μl de una solución de saponina al 5% y tras agitación con vórtex (10s), se deja reposar 5min a temperatura ambiente. Se realizan dos lavados con agua miliQ. Posteriormente se procede a la extracción etanol/ácido fórmico y se deposita 1μl del sobrenadante en una placa metálica.
- 3)
Subcultivo de incubación corta (SIC). Se inoculan 10 gotas (≈500μl) del hemocultivo en un medio de agar chocolate (Becton Dickinson, Franklin Lakes, NJ, EE.UU.) y se incuba en estufa a 37°C, 5% CO2 durante 3,5h. Tras este tiempo, con un asa estéril, se realizan varios arrastres sobre la zona de inoculación y se añade a la placa metálica. A continuación, se realiza una extracción directa en placa con ácido fórmico ≥96%.
Sobre los inóculos de la placa, se añadió 1μl de solución matriz (ácido α-ciano-4-hidroxicinámico) y se dejó secar a temperatura ambiente. A continuación, se introdujo la placa en el sistema MALDI-TOF. Se utilizó el calibrador Bacterial Test Standard, y para el análisis de cada muestra, el espectrómetro de masas Microflex LT y el software Biotyper 3.1 (Bruker Daltonics, Bremen, Alemania).
Los criterios de interpretación del fabricante establecen los valores ≥2,3 como alta probabilidad de identificación, entre 2 y 2,2 como probable identificación a nivel de especie, entre 1,7 y 1,9 como probable identificación a nivel de género, y los valores <1,7 se interpretan como poco fiables. Los PCC publicados por Kohlmann et al.11 consideran identificaciones correctas a nivel de género y especie aquellas con una puntuación ≥1,5 para microorganismos grampositivos y ≥1,7 para el resto de microorganismos. Además, como criterio adicional, las tres primeras opciones de identificación deben ser idénticas o, en su defecto, que las dos primeras sean idénticas con una diferencia mayor de 0,3 con la tercera. Puntuaciones diferentes a lo anterior se consideran poco fiables. Las identificaciones directas propuestas por MALDI-TOF que no se correspondieron con las definitivas del subcultivo tradicional se consideraron incorrectas.
Análisis estadísticoSe empleó la prueba t de Student con una distribución binomial para la comparación entre los métodos utilizados. Se analizaron el total de identificaciones correctas y poco fiables/incorrectas para cada método y también se realizó un análisis por subgrupos (microorganismos grampositivos, gramnegativos y levaduras).
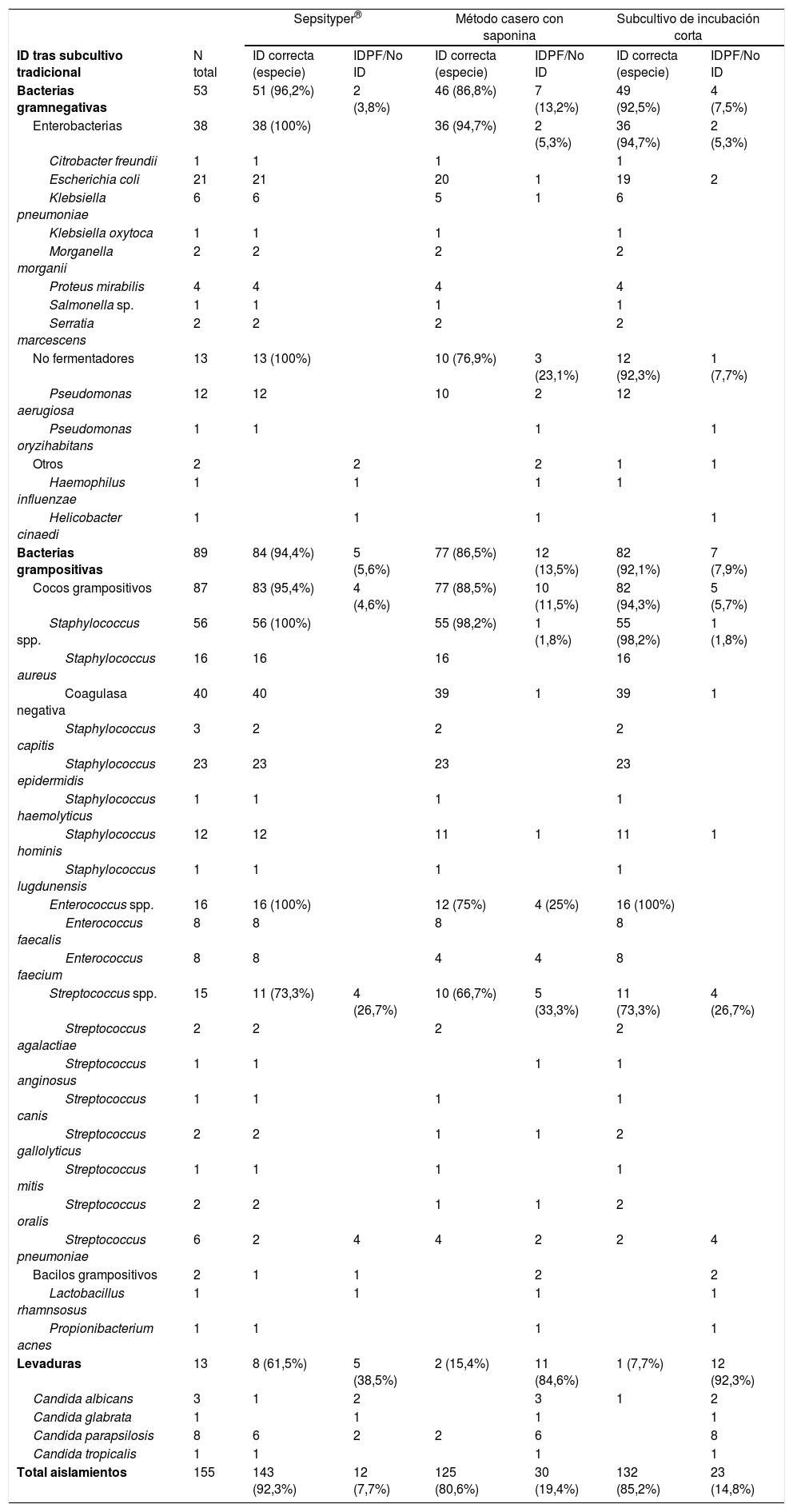
ResultadosSe analizaron 155 frascos de hemocultivo (90 aerobios y 65 anaerobios): 89 (57,4%) aislamientos de microorganismos grampositivos, 53 (34,2%) gramnegativos y 13 (8,4%) levaduras. Se identificaron 33 especies diferentes. Los resultados globales por microorganismo y método, aplicando los PCC11, se presentan en la tabla 1. En la tabla 2 se indican los resultados de cada método, según el criterio de interpretación.
Resultados de identificación por MALDI-TOF desglosados por microorganismo y métodos empleados. Los resultados se interpretaron con los criterios de Kohlmann
| Sepsityper® | Método casero con saponina | Subcultivo de incubación corta | |||||
| ID tras subcultivo tradicional | N total | ID correcta (especie) | IDPF/No ID | ID correcta (especie) | IDPF/No ID | ID correcta (especie) | IDPF/No ID |
| Bacterias gramnegativas | 53 | 51 (96,2%) | 2 (3,8%) | 46 (86,8%) | 7 (13,2%) | 49 (92,5%) | 4 (7,5%) |
| Enterobacterias | 38 | 38 (100%) | 36 (94,7%) | 2 (5,3%) | 36 (94,7%) | 2 (5,3%) | |
| Citrobacter freundii | 1 | 1 | 1 | 1 | |||
| Escherichia coli | 21 | 21 | 20 | 1 | 19 | 2 | |
| Klebsiella pneumoniae | 6 | 6 | 5 | 1 | 6 | ||
| Klebsiella oxytoca | 1 | 1 | 1 | 1 | |||
| Morganella morganii | 2 | 2 | 2 | 2 | |||
| Proteus mirabilis | 4 | 4 | 4 | 4 | |||
| Salmonella sp. | 1 | 1 | 1 | 1 | |||
| Serratia marcescens | 2 | 2 | 2 | 2 | |||
| No fermentadores | 13 | 13 (100%) | 10 (76,9%) | 3 (23,1%) | 12 (92,3%) | 1 (7,7%) | |
| Pseudomonas aerugiosa | 12 | 12 | 10 | 2 | 12 | ||
| Pseudomonas oryzihabitans | 1 | 1 | 1 | 1 | |||
| Otros | 2 | 2 | 2 | 1 | 1 | ||
| Haemophilus influenzae | 1 | 1 | 1 | 1 | |||
| Helicobacter cinaedi | 1 | 1 | 1 | 1 | |||
| Bacterias grampositivas | 89 | 84 (94,4%) | 5 (5,6%) | 77 (86,5%) | 12 (13,5%) | 82 (92,1%) | 7 (7,9%) |
| Cocos grampositivos | 87 | 83 (95,4%) | 4 (4,6%) | 77 (88,5%) | 10 (11,5%) | 82 (94,3%) | 5 (5,7%) |
| Staphylococcus spp. | 56 | 56 (100%) | 55 (98,2%) | 1 (1,8%) | 55 (98,2%) | 1 (1,8%) | |
| Staphylococcus aureus | 16 | 16 | 16 | 16 | |||
| Coagulasa negativa | 40 | 40 | 39 | 1 | 39 | 1 | |
| Staphylococcus capitis | 3 | 2 | 2 | 2 | |||
| Staphylococcus epidermidis | 23 | 23 | 23 | 23 | |||
| Staphylococcus haemolyticus | 1 | 1 | 1 | 1 | |||
| Staphylococcus hominis | 12 | 12 | 11 | 1 | 11 | 1 | |
| Staphylococcus lugdunensis | 1 | 1 | 1 | 1 | |||
| Enterococcus spp. | 16 | 16 (100%) | 12 (75%) | 4 (25%) | 16 (100%) | ||
| Enterococcus faecalis | 8 | 8 | 8 | 8 | |||
| Enterococcus faecium | 8 | 8 | 4 | 4 | 8 | ||
| Streptococcus spp. | 15 | 11 (73,3%) | 4 (26,7%) | 10 (66,7%) | 5 (33,3%) | 11 (73,3%) | 4 (26,7%) |
| Streptococcus agalactiae | 2 | 2 | 2 | 2 | |||
| Streptococcus anginosus | 1 | 1 | 1 | 1 | |||
| Streptococcus canis | 1 | 1 | 1 | 1 | |||
| Streptococcus gallolyticus | 2 | 2 | 1 | 1 | 2 | ||
| Streptococcus mitis | 1 | 1 | 1 | 1 | |||
| Streptococcus oralis | 2 | 2 | 1 | 1 | 2 | ||
| Streptococcus pneumoniae | 6 | 2 | 4 | 4 | 2 | 2 | 4 |
| Bacilos grampositivos | 2 | 1 | 1 | 2 | 2 | ||
| Lactobacillus rhamnsosus | 1 | 1 | 1 | 1 | |||
| Propionibacterium acnes | 1 | 1 | 1 | 1 | |||
| Levaduras | 13 | 8 (61,5%) | 5 (38,5%) | 2 (15,4%) | 11 (84,6%) | 1 (7,7%) | 12 (92,3%) |
| Candida albicans | 3 | 1 | 2 | 3 | 1 | 2 | |
| Candida glabrata | 1 | 1 | 1 | 1 | |||
| Candida parapsilosis | 8 | 6 | 2 | 2 | 6 | 8 | |
| Candida tropicalis | 1 | 1 | 1 | 1 | |||
| Total aislamientos | 155 | 143 (92,3%) | 12 (7,7%) | 125 (80,6%) | 30 (19,4%) | 132 (85,2%) | 23 (14,8%) |
ID: identificación; IDPF/No ID: identificación poco fiable/no identificación.
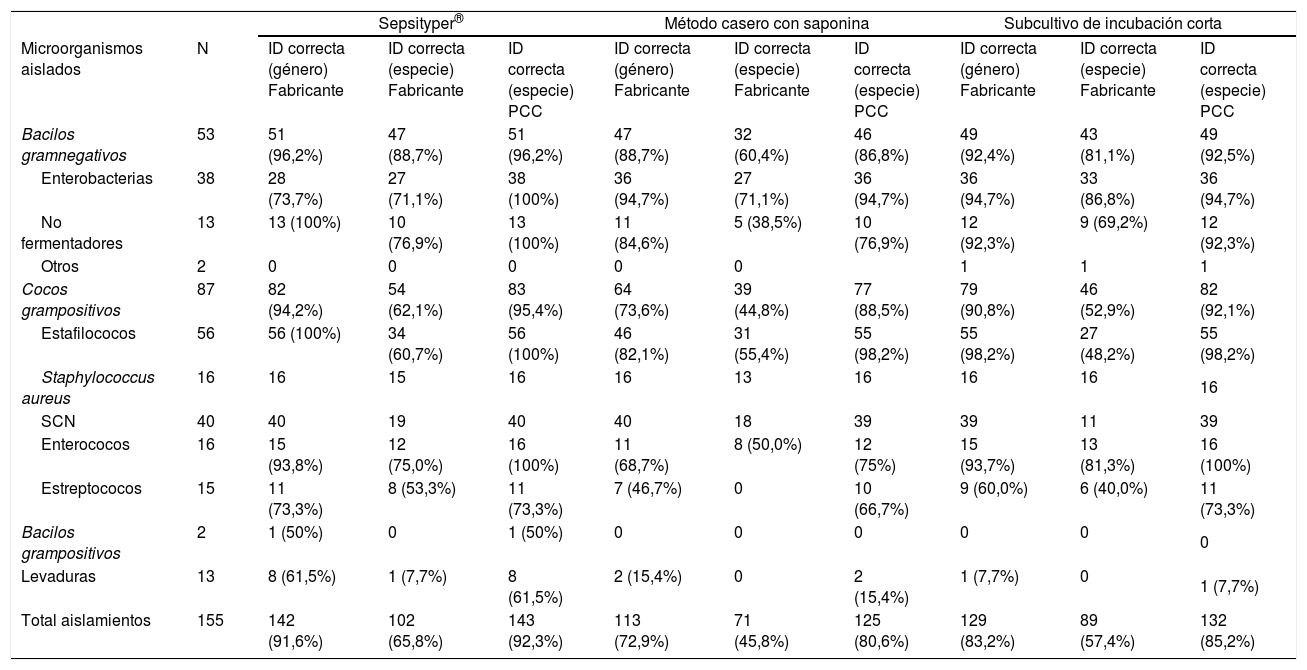
Resultados obtenidos por Sepsityper®, el método casero con saponina y el subcultivo de incubación corta, en función del criterio de interpretación aplicado a las puntuaciones obtenidas mediante EM MALDI-TOF
| Sepsityper® | Método casero con saponina | Subcultivo de incubación corta | ||||||||
| Microorganismos aislados | N | ID correcta (género) Fabricante | ID correcta (especie) Fabricante | ID correcta (especie) PCC | ID correcta (género) Fabricante | ID correcta (especie) Fabricante | ID correcta (especie) PCC | ID correcta (género) Fabricante | ID correcta (especie) Fabricante | ID correcta (especie) PCC |
| Bacilos gramnegativos | 53 | 51 (96,2%) | 47 (88,7%) | 51 (96,2%) | 47 (88,7%) | 32 (60,4%) | 46 (86,8%) | 49 (92,4%) | 43 (81,1%) | 49 (92,5%) |
| Enterobacterias | 38 | 28 (73,7%) | 27 (71,1%) | 38 (100%) | 36 (94,7%) | 27 (71,1%) | 36 (94,7%) | 36 (94,7%) | 33 (86,8%) | 36 (94,7%) |
| No fermentadores | 13 | 13 (100%) | 10 (76,9%) | 13 (100%) | 11 (84,6%) | 5 (38,5%) | 10 (76,9%) | 12 (92,3%) | 9 (69,2%) | 12 (92,3%) |
| Otros | 2 | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 1 | |
| Cocos grampositivos | 87 | 82 (94,2%) | 54 (62,1%) | 83 (95,4%) | 64 (73,6%) | 39 (44,8%) | 77 (88,5%) | 79 (90,8%) | 46 (52,9%) | 82 (92,1%) |
| Estafilococos | 56 | 56 (100%) | 34 (60,7%) | 56 (100%) | 46 (82,1%) | 31 (55,4%) | 55 (98,2%) | 55 (98,2%) | 27 (48,2%) | 55 (98,2%) |
| Staphylococcus aureus | 16 | 16 | 15 | 16 | 16 | 13 | 16 | 16 | 16 | 16 |
| SCN | 40 | 40 | 19 | 40 | 40 | 18 | 39 | 39 | 11 | 39 |
| Enterococos | 16 | 15 (93,8%) | 12 (75,0%) | 16 (100%) | 11 (68,7%) | 8 (50,0%) | 12 (75%) | 15 (93,7%) | 13 (81,3%) | 16 (100%) |
| Estreptococos | 15 | 11 (73,3%) | 8 (53,3%) | 11 (73,3%) | 7 (46,7%) | 0 | 10 (66,7%) | 9 (60,0%) | 6 (40,0%) | 11 (73,3%) |
| Bacilos grampositivos | 2 | 1 (50%) | 0 | 1 (50%) | 0 | 0 | 0 | 0 | 0 | 0 |
| Levaduras | 13 | 8 (61,5%) | 1 (7,7%) | 8 (61,5%) | 2 (15,4%) | 0 | 2 (15,4%) | 1 (7,7%) | 0 | 1 (7,7%) |
| Total aislamientos | 155 | 142 (91,6%) | 102 (65,8%) | 143 (92,3%) | 113 (72,9%) | 71 (45,8%) | 125 (80,6%) | 129 (83,2%) | 89 (57,4%) | 132 (85,2%) |
ID: identificación; PCC: puntos de corte corregidos.
Todas las identificaciones obtenidas siguiendo los tres métodos estudiados coincidieron con la identificación del subcultivo tradicional, excepto en dos cepas identificadas inicialmente como estreptococos del grupo viridans, que, tras pruebas manuales, se identificaron finalmente como Streptococcus pneumoniae.
El ST obtuvo resultados significativamente mejores (p<0,05) respecto al MCS en la identificación de los microorganismos. El ST y el SIC no mostraron diferencias significativas ni para gramnegativos (p=0,094) ni para grampositivos (p=0,234); solo en el análisis de levaduras se obtuvo una p<0,05 a favor de ST. Por último, se encontraron diferencias significativas (p<0,05) tanto en la identificación de gramnegativos como en la de grampositivos tras comparar el MCS y el SIC, obteniéndose los mejores resultados con este último.
DiscusiónAnalizamos la eficacia de tres métodos para la identificación rápida de microorganismos a partir de hemocultivos positivos. Los métodos con mejores resultados a nivel de especie, aplicando los criterios del fabricante, fueron el ST (65,8%) y el SIC (57,4%). Solo presentaban diferencias significativas en la identificación de levaduras, siendo mejor cuando se utilizaba el método comercial. El MCS obtuvo resultados significativamente inferiores (45,8%) comparados con los demás métodos.
Aplicando los PCC11, los porcentajes de identificación correcta a nivel de especie se incrementaron: ST (92,3%), SIC (85,2%) y MCS (80,6%), lo que confirma que los puntos de corte del fabricante se podrían modificar sin perder especificidad12,15.
Los resultados para ST son similares a los ya publicados, en los que las identificaciones correctas a nivel de especie, aplicando criterios del fabricante, oscilan entre el 59,5 y el 82,8%3-6. Con respecto al SIC, nuestros resultados son similares a los existentes en la literatura11,13,14. En cuanto al MCS, las modificaciones del protocolo de Martiny et al.5 permiten obtener mejores resultados cuando se aplican los PCC11.
Para la identificación de bacterias gramnegativas a nivel de especie aplicando PCC11, tanto el ST como el SIC mostraron resultados excelentes (96,2 y 92,5%, respectivamente). El MCS, aunque fue estadísticamente inferior (p<0,05) para la identificación global de bacterias gramnegativas, en el caso de enterobacterias resultó comparable con los demás métodos.
También se obtuvieron resultados óptimos para bacterias grampositivas aplicando PCC11 con ST (94,4%) y con SIC (92,1%), siendo superiores las identificaciones de estafilococos y enterococos en comparación con las de estreptococos. Es relativamente habitual no poder discriminar entre Streptococcus pneumoniae y estreptococos del grupo viridans debido a la similitud entre sus espectros proteicos. Hay que destacar que todas las cepas de Staphylococcus aureus fueron identificadas correctamente por los tres métodos y que ningún estafilococo coagulasa negativa fue identificado erróneamente como S.aureus.
Los porcentajes de identificación correcta de las levaduras fueron inferiores al resto, siendo el ST (61,5% aplicando PCC11) estadísticamente superior a los otros métodos.
En cuanto a la aplicabilidad de estas técnicas en el laboratorio, el ST es rápido (20min) pero conlleva un tiempo superior de procesamiento técnico y mayor coste económico. Sin embargo, con el SIC el resultado se obtiene más tarde (3,5h) pero necesita menor procesamiento técnico y es más económico, por lo que puede integrarse fácilmente en la rutina del laboratorio.
Por tanto, como ST y SIC tienen tasas de identificación comparables, a excepción de las levaduras, ambas técnicas podrían combinarse según la hora de positivización de los hemocultivos y la logística del laboratorio, de tal forma que se mantendría una información clínica continua con un uso sostenible de los recursos.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.