Introducción
Staphylococcus aureus es un patógeno responsable de una gran variedad de cuadros clínicos graves, como infecciones óseas, neumonía, septicemia y endocarditis. El tratamiento de elección para estas infecciones lo constituyen los antibióticos betalactámicos, asociados en ocasiones a aminoglucósidos1. Así mismo, los estafilococos coagulasa- negativos (ECN) continúan siendo una causa importante de bacteriemias en pacientes hospitalizados2,3. Desde la introducción de la meticilina en la práctica clínica en 1961, la incidencia de S. aureus resistentes a meticilina (SARM) ha aumentado de forma gradual y las infecciones nosocomiales causadas por este agente se han convertido en un grave problema en todo el mundo1,4. La resistencia a meticilina presenta incluso mayor prevalencia en ECN, sobre todo en las infecciones hospitalarias3. En los pacientes con infecciones por S. aureus, la identificación precoz de los SARM tiene importantes implicaciones para el tratamiento y manejo de los mismos al ser los glucopéptidos los fármacos de elección para estas infecciones1.
Las bacteriemias por S. aureus a menudo implican una situación médica comprometida; sin embargo, el significado clínico de los ECN aislados en hemocultivos en ocasiones no está bien definido ya que pueden ser contaminantes5. Por consiguiente, cuando se observan en la tinción de Gram de botellas de hemocultivos cocos grampositivos en racimos, se plantean dos interrogantes: diferenciar si el microorganismo es S. aureus o ECN y si el microorganismo es sensible o no a la meticilina6. En el laboratorio clínico, S. aureus se identifica por sus características de crecimiento y la detección de la actividad catalasa y coagulasa. Las pruebas convencionales de sensibilidad, por dilución en agar, difusión en disco y métodos de cribado en medio sólido detectan de forma fiable la resistencia a meticilina u oxacilina siempre que se realicen de acuerdo con las normas del National Committee for Clinical Laboratory Standards (NCCLS)7,8. Sin embargo, estas pruebas convencionales de sensibilidad son lentas, en ocasiones la expresión fenotípica de la resistencia a meticilina in vitro es heterogénea y, a veces, difícil de inducir, por lo que se pueden producir falsos negativos9. Además, los sistemas comerciales rápidos disponibles para la detección de la coagulasa pueden mostrar un resultado falso negativo o ininterpretable con algunas cepas de S. aureus10, al igual que las pruebas rápidas de detección de resistencia a meticilina, especialmente en el caso de los ECN11. Si el microorganismo está en cultivo puro, la identificación por las técnicas convencionales requiere entre 18-24 h y las pruebas de sensibilidad de 6 a 10 h adicionales, por lo que una disminución de estos tiempos tendría una gran relevancia clínica.
El gen nucA de S. aureus, que codifica una termonucleasa extracelular, tiene secuencias específicas de especie que han permitido el diseño de cebadores para la identificación de S. aureus12,13. Por otro lado, el principal determinante de resistencia a meticilina en estafilococos, el gen mecA, está presente tanto en S. aureus como en ECN14,15. Ambos genes se han utilizado para la rápida identificación de S. aureus resistente a meticilina a partir de subcultivos en medio sólido16 y también directamente en botellas de hemocultivos utilizando la técnica de reacción en cadena de la polimerasa (PCR) en tiempo real17.
En este trabajo se ha evaluado la utilidad de la técnica de PCR en tiempo real para identificar S. aureus y detectar la resistencia a meticilina tanto en S. aureus como en ECN, directamente en botellas Bactec de hemocultivos positivos. Para ello se han utilizado secuencias específicas de los genes nucA y mecA y la tecnología LightCycler®.
Material y métodos
Aislamientos clínicos
Se estudiaron 33 cepas de S. aureus sensibles a meticilina y 18 cepas de SARM, aisladas de diferentes pacientes, empleadas para la validación de la técnica molecular, y 171 microorganismos aislados de los hemocultivos positivos empleados en el estudio.
Identificación bacteriana y pruebas de sensibilidad
La caracterización fenotípica de los aislados se realizó mediante tinción de Gram, actividad de la catalasa y reacción de la coagulasa, determinada esta última por una prueba de aglutinación de látex con el kit Pastorex Staph Plus (Sanofi Diagnostic Pasteur, Marnes la Coquette, Francia). La identificación y pruebas de sensibilidad se hicieron con el sistema semiautomatizado Walk-Away MicroScan® (Dade-Behring), y mediante difusión en agar con un disco de 1 μg de oxacilina por el método Kirby-Bauer, de acuerdo con las normas del NCCLS8.
Hemocultivos
Durante los meses de junio y julio de 2002 se analizaron 171 botellas de hemocultivos positivos (Bactec 9240 Roche Diagnostics, Mannheim, Alemania), enviadas al Servicio de Microbiología de los Hospitales Universitarios Virgen del Rocío de Sevilla obtenidas de 74 pacientes y que correspondían a 83 episodios febriles. Los hemocultivos se procesaron siguiendo los procedimientos habituales que incluyen tinción de Gram e inoculación en diferentes medios sólidos. En 131 botellas se observaron cocos grampositivos en racimos en la tinción de Gram, e inmediatamente se alicuotó 1 ml de su contenido, conservándose a 70 °C para su posterior utilización en las técnicas moleculares. En los restantes 40 hemocultivos positivos se visualizaron en la tinción de Gram: 13 bacilos gramnegativos, cuatro bacilos grampositivos, 20 cocos grampositivos en cadenas, dos diplococos grampositivos y un diplococo gramnegativo.
Extracción del ADN
La extracción del ADN se realizó a partir de una alícuota de 100 μl de la sangre congelada y se llevó a cabo en el sistema automático MagNA Pure LC18 utilizando el kit MagNA Pure LC Total DNA Isolation (Roche Molecular Biochemicals). Una vez extraído, el ADN se resuspendió en un volumen final de 100 μl, utilizándose 2 μl en la reacción de PCR.
La especificidad de la nueva técnica molecular se determinó con ADN extraído por el método rápido de ebullición16 a partir de las 33 cepas de S. aureus sensibles a meticilina (SASM) y las 18 cepas de SARM.
Amplificación por PCR
Los cebadores y las sondas de hibridación, diseñadas para la amplificación y detección de un fragmento de 408 pb del gen mecA16 y de un fragmento de 279 pb del gen nucA específico de S. aureus12,13 fueron elaboradas por TIB MolBiol (Berlín, Alemania). La secuencia de nucleótidos y su posición se muestran en la tabla 1. Además, fue incluida una pareja de cebadores para la β-globina humana (KM38-GH20) como control interno de la amplificación del ADN y descartar la presencia de inhibidores de la reacción.
La mezcla de reacción para la amplificación del gen nucA contenía 2 μl de 10x LightCycler® DNA Master Hybridation Probes (Roche Molecular Biochemicals), 4 mM MgCl2 (concentración final), 0,5 μM de cada uno de los cebadores nucA-F y nucA-R, 0,2 μM de cada una de las sondas de hibridación nuc-FLU y nuc-LCR, y 2 ml de ADN, en un volumen final de 20 μl. Las condiciones de reacción fueron una primera desnaturalización a 95 °C durante 10 s, seguido de un protocolo de amplificación de 40 ciclos que consistían en calentamiento a 20 °C/s hasta 95 °C, enfriamiento a 20 °C/s hasta 55 °C, manteniendo esta temperatura durante 10 s, y calentamiento a 20 °C/s hasta 72 °C manteniéndola 15 s. La emisión de fluorescencia se midió en cada ciclo al final de la fase de desnaturalización utilizando el canal de lectura F2. Esto fue seguido de un programa de fusión de 50 °C a 80 °C a 0,1 °C/s con monitorización continua de la emisión de fluorescencia.
Para la detección del gen mecA se usó una sonda TaqMan marcada con un marcador de fluorescencia16. El volumen final de reacción fue de 20 μl, que contenía 2 μl de la mezcla 10x LightCycler® DNA Master Hybridation Probes (Roche Molecular Biochemicals), 4 mM MgCl2 (concentración final), 0,5 μM de cada uno de los cebadores mecF y mecR, 0,1 μM de la sonda de hibridación mec-HP-2, y 2 μl de ADN. Las condiciones de reacción fueron una primera desnaturalización a 95 °C durante 10 s, seguido de un protocolo de amplificación de 40 ciclos que consistían en calentamiento a 20 °C/s hasta 95 °C, enfriamiento a 20 °C/s hasta 50 °C, manteniendo esta temperatura durante 10 s, y calentamiento a 20 °C/s hasta 72 °C manteniéndola 15 s. La emisión de fluorescencia se midió en cada ciclo al final de la fase de elongación utilizando el canal de lectura F1.
Resultados
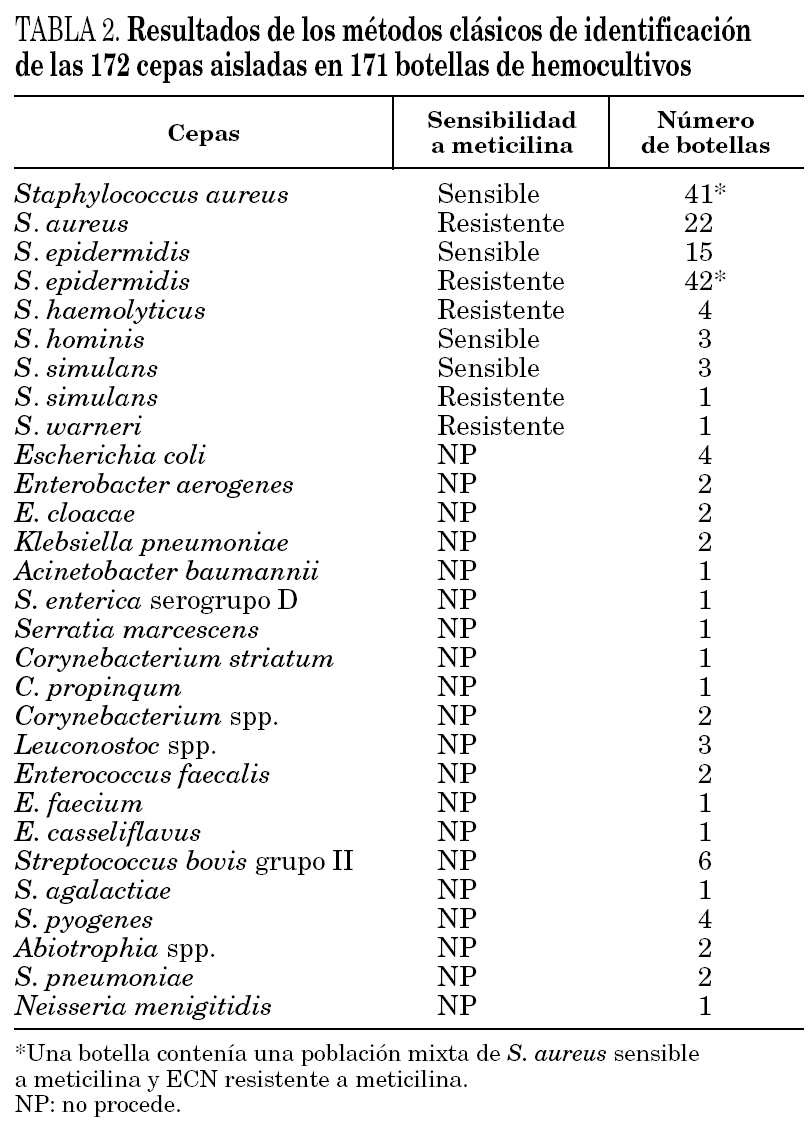
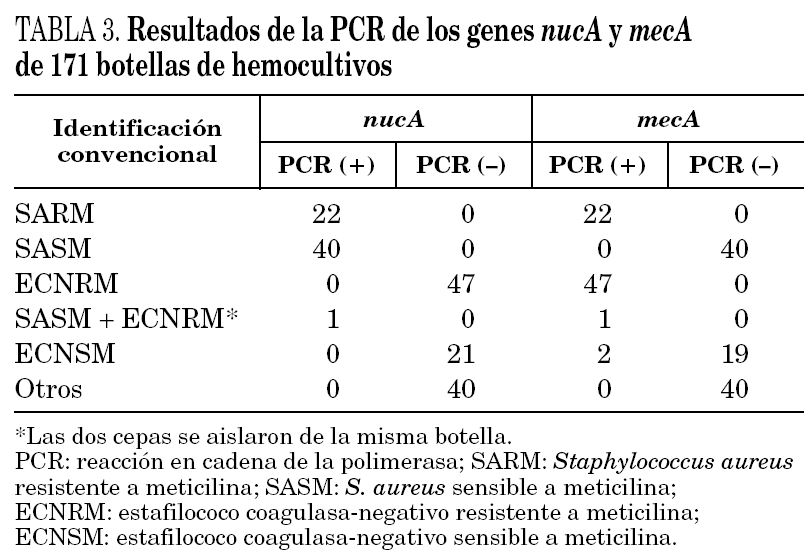
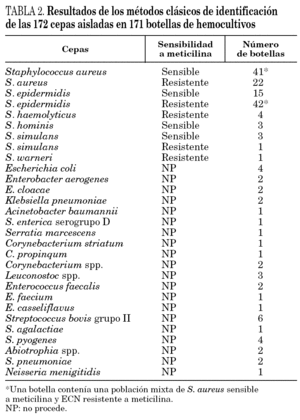
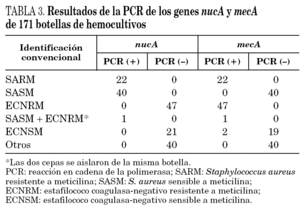
En primer lugar se evaluó la técnica de PCR en tiempo real con los genes nucA y mecA en 51 cepas de S. aureus aisladas a partir de muestras clínicas. La sensibilidad y especificidad fueron ambas del 100%, detectándose el gen nucA en todas las cepas estudiadas y el gen mecA en los 18 SARM. Posteriormente, se realizó el estudio con las 171 botellas de hemocultivos positivos. La identificación con los métodos convencionales de las cepas aisladas en las botellas de hemocultivos se presenta en la tabla 2. En todas las muestras la amplificación del gen de la β-globina fue positiva, indicando la ausencia de inhibidores en la reacción de amplificación. En la tabla 3 se resumen los resultados obtenidos en los 171 hemocultivos con la PCR en tiempo real y los genes nucA y mecA. Las 22 botellas de hemocultivos de las que se aislaron SARM fueron correctamente identificadas, siendo la PCR positiva para ambos genes. Las 41 botellas que contenían S. aureus sensible a meticilina fueron positivas para el gen nucA y negativas para el gen mecA. Las 48 botellas que contenían ECN resistentes a meticilina fueron correctamente identificadas por PCR, con resultados negativos para el gen nucA y positivos para el mecA. Sin embargo, en una botella con un cultivo mixto de SASM y ECN resistente a meticilina se detectó amplificación de ambos genes. Por lo tanto, en hemocultivos con cultivos mixtos de Staphylococcus spp. la interpretación de las técnicas de amplificación puede ser confusa.
En todas las botellas con ECN sensibles a meticilina la PCR del gen nucA fue negativa y la amplificación del gen mecA fue negativa en todas excepto en dos de ellas. Después de realizar la prueba de la coagulasa en tubo y el método del disco difusión se confirmó que ambas cepas eran ECN sensibles a meticilina y se consideraron como falsos positivos de la técnica de PCR. No se detectó amplificación de los genes estudiados en ninguno de los hemocultivos en los que se aislaron especies diferentes a Staphylococcus spp. (tabla 3).
La sensibilidad y la especificidad de la PCR en tiempo real del gen nucA fue del 100%, identificando todos los S. aureus correctamente. En la evaluación de la detección de la resistencia a meticilina mediante PCR del gen mecA en botellas que contenían S. aureus y ECN, la prueba convencional de sensibilidad, con el disco de oxacilina de 1 mg de acuerdo con las normas del NCCLS8, se consideró como el método de referencia. La sensibilidad y la especificidad de la técnica de PCR en tiempo real fueron del 100 y 97,5%, respectivamente.
Discusión
La identificación de S. aureus basada exclusivamente en los métodos convencionales fenotípicos puede producir errores diagnósticos. Las técnicas moleculares basadas en la detección de ácidos nucleicos podrían ser más sensibles, especialmente en la detección de bacteriemias en pacientes con tratamiento antimicrobiano19, en los que los hemocultivos podrían ser negativos.
La detección del gen nucA se ha utilizado con fines diagnósticos para la identificación de S. aureus por varios autores13,20,21, y nuestros resultados confirman su utilidad. Cuando se aplicó directamente en botellas de hemocultivos positivos, fue igualmente eficaz, no obteniéndose ningún resultado positivo cuando se aplicaron a botellas de hemocultivos que contenían microorganismos distintos de S. aureus.
En otros estudios se ha utilizado la amplificación del gen sa442 para la identificación de S. aureus, a partir de botellas de hemocultivos con resultados idénticos a los nuestros19, o con una sensibilidad ligeramente inferior17. Hay que tener presente que la utilización de diferentes medios de hemocultivos, con distinta composición, podría tener como consecuencia la presencia de sustancias inhibidoras de la PCR. En nuestro caso la sensibilidad fue del 100%, y puede deberse, a diferencia de los dos trabajos antes mencionados, a la utilización de un sistema de extracción automático que posee en principio una mayor reproducibilidad de la extracción del ADN.
Algunos autores consideran como técnica de referencia para la determinación de la resistencia a meticilina la detección del gen mecA10, ya que muchas cepas de estafilococos expresan el gen mecA de forma heterogénea, y sólo unas pocas colonias de una población bacteriana pueden ser PBP2a (proteína producto de la expresión del gen mecA y que determina la resistencia a meticilina) positivas, siendo esta heterogeneidad más común en cepas de ECN que en cepas de S. aureus. Esto hace que la detección fenotípica de la resistencia a la meticilina sea en ocasiones problemática, especialmente en ECN. El método de difusión en disco y el sistema Walk Away mostraron resultados equivalentes para la identificación de las cepas que poseían el gen mecA, y sólo 2 de 69 cepas de ECN (2,9%) no fueron reconocidas como tal. Si se considera la detección del gen mecA como el método de referencia, estas dos cepas deberían ser consideradas resistentes, siendo falsos negativos del método fenotípico. A pesar de que se han descrito otros mecanismos de resistencia a meticilina, está demostrado que la concentración inhibitoria mínima (CIM) de oxacilina para los ECN que no poseen el gen mecA es pocas veces mayor o igual a 4 μg/ml22,23. Al igual que Grisold et al24 en el estudio de aislamientos clínicos, hemos utilizado un sistema automático de extracción de ADN, MagNA Pure, que puede superar los problemas de inhibición encontrados por Shrestha et al19 en la prueba del gen mecA cuando la aplicaban directamente a botellas de hemocultivos positivos.
En nuestro estudio, ninguna de las botellas que contenían microorganismos diferentes a estafilococos obtuvieron resultados positivos, demostrándose la elevada especificidad de nuestra técnica de PCR. En todas las cepas de SASM se obtuvieron resultados positivos para el gen nucA y resultados negativos para el gen mecA. La PCR identificó una cepa de SASM como un SARM debido a la presencia en la misma botella de una cepa de ECN mecA positivo. El estudio que describimos hubiera identificado correctamente que la botella contenía una cepa de S. aureus pero la hubiera clasificado de forma inadecuada como resistente. Por lo tanto, creemos que el subcultivo de rutina de los hemocultivos positivos en medios de cultivo selectivos y no selectivos deber seguir haciéndose para identificar los cultivos mixtos. Una limitación de este estudio es que las botellas que contengan Micrococcus spp. deberían resultar negativas para el gen nucA, y podrían ser identificados como ECN. Este hecho tiene poca relevancia desde el punto de vista clínico porque estas especies son consideradas en la mayoría de las ocasiones contaminantes de los hemocultivos. En estos casos habría que esperar al resultado del subcultivo para su correcta identificación, y así determinar su significado clínico en función no sólo del resultado de la PCR para los genes nucA y mecA, sino también del número de botellas positivas y del contexto clínico del paciente.
Esta es la primera vez que dos pruebas de PCR en tiempo real usando los genes nucA y mecA combinadas con un protocolo automático de extracción de ADN con el sistema MagNA Pure LC se han aplicado directamente en botellas de hemocultivos positivos para la identificación y detección de Staphylococcus spp. resistentes a meticilina. La información proporcionada por estas pruebas podría ayudar en la práctica clínica en la rápida identificación de las bacteriemias por S. aureus y ECN detectando simultáneamente la resistencia a meticilina, y así modificar si fuera necesario la terapia antimicrobiana 24-36 h antes, tiempo mínimo necesario para obtener resultados con las pruebas convencionales de identificación y sensibilidad.
En conclusión, creemos que este protocolo de extracción de ADN combinado con dos pruebas de PCR en tiempo real aplicado directamente a hemocultivos positivos es un método rápido y seguro que podría ser introducido en la rutina del laboratorio clínico, ya que su duración es de 3 h una vez realizada la tinción de Gram.
Agradecimientos
Este trabajo ha sido en parte financiado por la Red Española de Investigación en Patología Infecciosa (REIPI-C03/14).