Los virus transmitidos por artrópodos (arbovirus) y los transmitidos por roedores (robovirus) o por otros animales se engloban en el epígrafe «virus transmitidos por vector» (VTV). En nuestro entorno son 3 los principales VTV autóctonos que causan enfermedad: los virus Toscana, West Nile y de la coriomeningitis linfocitaria; además, se diagnostican enfermedades por VTV importados (virus dengue, chikungunya) que actualmente suponen un riesgo de asentamiento por la circulación de vectores competentes de transmisión en nuestro territorio, como es el mosquito Aedes albopictus. La Sociedad Española de Enfermedades Infecciosas y Microbiología Clínica se ha hecho eco de la emergencia de las enfermedades por VTV y ha redactado un procedimiento sobre diagnóstico microbiológico de arbovirosis y robovirosis emergentes que supone una actualización sobre los VTV con mayor sospecha diagnóstica en nuestro entorno y los métodos de detección disponibles para el diagnóstico de las enfermedades que producen.
Vector borne viruses (VBV) include viruses transmitted by arthropods, rodents and other animals. In Spain the three main autochthonous VBVs causing human diseases are: Toscana, West Nile and Lymphocytic Choriomeningitis viruses. There are also other imported viruses that are potential threats to our public health, due to the presence of competent transmission vectors (dengue and chikungunya viruses in areas infested with Aedes albopictus), or due to the potential person-to-person transmission (Lassa and other viruses causing haemorrhagic fever). The Spanish Society for Infectious Diseases and Clinical Microbiology has responded to the emergence of VBVs by publishing a special issue of Microbiological Proceedings focused on the diagnosis of those emerging vector borne viruses of major concern in our country.
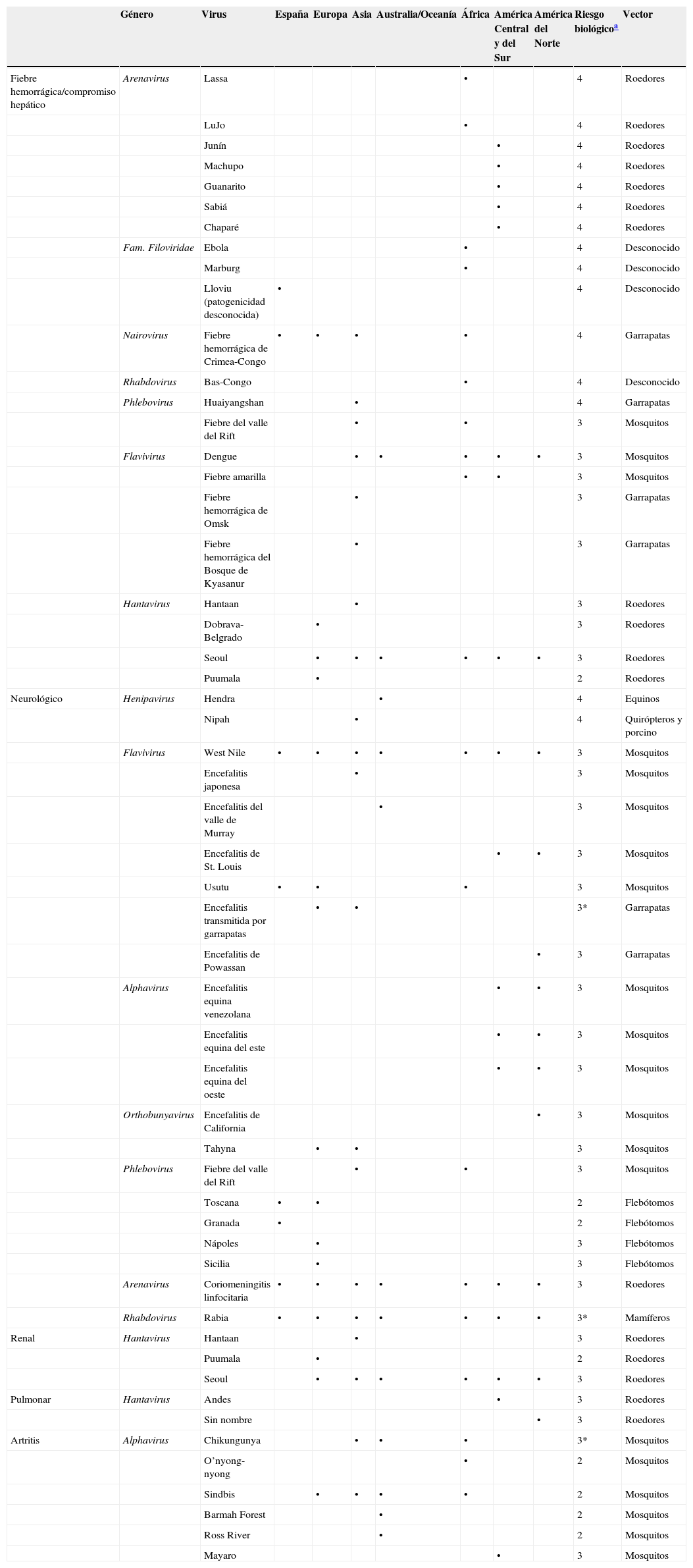
Los virus transmitidos por artrópodos (arbovirus) y los transmitidos por roedores (robovirus) o por otros animales se engloban bajo el epígrafe «virus transmitidos por vector» (VTV). La mayoría de los VTV no utilizan a los humanos en su ciclo de vida, aunque algunos de ellos pueden infectarlo y causar enfermedad de distinta gravedad, que abarca desde la infección asintomática al síndrome febril, acompañado o no de enfermedad neurológica, hepática, renal, pulmonar y/o hemorrágica. Son 3 los principales virus autóctonos transmitidos por vector que producen enfermedad humana en nuestro país: el virus Toscana (TOSV), el virus de la coriomeningitis linfocitaria (LCMV) y el virus West Nile (WNV). Los 3 se han asociado a enfermedad neurológica aguda y son transmitidos por vectores con una distribución geográfica específica en nuestro territorio (flebótomos, ratón común y mosquitos Culex, respectivamente). Existen otros VTV adaptados a nuestra especie capaces de producir ciclos urbanos de transmisión; se trata de los virus dengue (DENV), de la fiebre amarilla (YFV) y chikungunya (CHIKV), que todavía no circulan en nuestro país pero de los que existe un riesgo potencial de asentamiento debido a su presencia a través de viajeros infectados procedentes de zonas endémicas (tabla 1) y a la circulación de una de las especies de mosquito que le sirven como vector, Aedes albopictus, ampliamente distribuida por la zona del mediterráneo, incluida las Islas Baleares. Un tercer grupo de los VTV engloba a los de alto riesgo biológico, productores de fiebres hemorrágicas, cuya sospecha supone una situación de alerta debido a la gravedad de la patología que producen siendo capaces de transmitirse por contacto interpersonal.
Principales síndromes clínicos producidos por los virus transmitidos por vector, presencia en España y zonas endémicas
| Género | Virus | España | Europa | Asia | Australia/Oceanía | África | América Central y del Sur | América del Norte | Riesgo biológicoa | Vector | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| Fiebre hemorrágica/compromiso hepático | Arenavirus | Lassa | • | 4 | Roedores | ||||||
| LuJo | • | 4 | Roedores | ||||||||
| Junín | • | 4 | Roedores | ||||||||
| Machupo | • | 4 | Roedores | ||||||||
| Guanarito | • | 4 | Roedores | ||||||||
| Sabiá | • | 4 | Roedores | ||||||||
| Chaparé | • | 4 | Roedores | ||||||||
| Fam. Filoviridae | Ebola | • | 4 | Desconocido | |||||||
| Marburg | • | 4 | Desconocido | ||||||||
| Lloviu (patogenicidad desconocida) | • | 4 | Desconocido | ||||||||
| Nairovirus | Fiebre hemorrágica de Crimea-Congo | • | • | • | • | 4 | Garrapatas | ||||
| Rhabdovirus | Bas-Congo | • | 4 | Desconocido | |||||||
| Phlebovirus | Huaiyangshan | • | 4 | Garrapatas | |||||||
| Fiebre del valle del Rift | • | • | 3 | Mosquitos | |||||||
| Flavivirus | Dengue | • | • | • | • | • | 3 | Mosquitos | |||
| Fiebre amarilla | • | • | 3 | Mosquitos | |||||||
| Fiebre hemorrágica de Omsk | • | 3 | Garrapatas | ||||||||
| Fiebre hemorrágica del Bosque de Kyasanur | • | 3 | Garrapatas | ||||||||
| Hantavirus | Hantaan | • | 3 | Roedores | |||||||
| Dobrava-Belgrado | • | 3 | Roedores | ||||||||
| Seoul | • | • | • | • | • | • | 3 | Roedores | |||
| Puumala | • | 2 | Roedores | ||||||||
| Neurológico | Henipavirus | Hendra | • | 4 | Equinos | ||||||
| Nipah | • | 4 | Quirópteros y porcino | ||||||||
| Flavivirus | West Nile | • | • | • | • | • | • | • | 3 | Mosquitos | |
| Encefalitis japonesa | • | 3 | Mosquitos | ||||||||
| Encefalitis del valle de Murray | • | 3 | Mosquitos | ||||||||
| Encefalitis de St. Louis | • | • | 3 | Mosquitos | |||||||
| Usutu | • | • | • | 3 | Mosquitos | ||||||
| Encefalitis transmitida por garrapatas | • | • | 3* | Garrapatas | |||||||
| Encefalitis de Powassan | • | 3 | Garrapatas | ||||||||
| Alphavirus | Encefalitis equina venezolana | • | • | 3 | Mosquitos | ||||||
| Encefalitis equina del este | • | • | 3 | Mosquitos | |||||||
| Encefalitis equina del oeste | • | • | 3 | Mosquitos | |||||||
| Orthobunyavirus | Encefalitis de California | • | 3 | Mosquitos | |||||||
| Tahyna | • | • | 3 | Mosquitos | |||||||
| Phlebovirus | Fiebre del valle del Rift | • | • | 3 | Mosquitos | ||||||
| Toscana | • | • | 2 | Flebótomos | |||||||
| Granada | • | 2 | Flebótomos | ||||||||
| Nápoles | • | 3 | Flebótomos | ||||||||
| Sicilia | • | 3 | Flebótomos | ||||||||
| Arenavirus | Coriomeningitis linfocitaria | • | • | • | • | • | • | • | 3 | Roedores | |
| Rhabdovirus | Rabia | • | • | • | • | • | • | • | 3* | Mamíferos | |
| Renal | Hantavirus | Hantaan | • | 3 | Roedores | ||||||
| Puumala | • | 2 | Roedores | ||||||||
| Seoul | • | • | • | • | • | • | 3 | Roedores | |||
| Pulmonar | Hantavirus | Andes | • | 3 | Roedores | ||||||
| Sin nombre | • | 3 | Roedores | ||||||||
| Artritis | Alphavirus | Chikungunya | • | • | • | 3* | Mosquitos | ||||
| O’nyong-nyong | • | 2 | Mosquitos | ||||||||
| Sindbis | • | • | • | • | 2 | Mosquitos | |||||
| Barmah Forest | • | 2 | Mosquitos | ||||||||
| Ross River | • | 2 | Mosquitos | ||||||||
| Mayaro | • | 3 | Mosquitos |
a Definiciones de riesgo biológico:
Riesgo biológico 1: microorganismos cuyo riesgo de producir enfermedad en el trabajador es poco probable, no presentan riesgo de propagación a la colectividad y para los que no es necesario disponer de medidas profilácticas o terapéuticas adecuadas.
Riesgo biológico 2: microorganismos que pueden causar una enfermedad y constituir un riesgo para los trabajadores, el riesgo de propagación a la colectividad es poco probable y para los que hay disponibles medidas profilácticas o terapéuticas en la mayoría de los casos.
Riesgo biológico 3: microorganismos que pueden provocar una enfermedad grave y constituir un grave peligro para los trabajadores, el riesgo de propagación a la colectividad es probable y para los que hay disponibles medidas profilácticas o terapéuticas en la mayoría de los casos.
Riesgo biológico 3*: aun siendo de riesgo biológico 3, normalmente no se disemina por vía aérea.
Riesgo biológico 4: microorganismos que provocan una enfermedad grave y constituyen un serio problema para los trabajadores, el riesgo de propagación a la colectividad es elevado y no hay disponible profilaxis o tratamiento eficaz.
Los cambios introducidos por nuestra especie en los ecosistemas que ocupan estos virus, los cambios en el clima, y la velocidad y la frecuencia con la que viajan actualmente las personas y las mercancías están favoreciendo la rápida dispersión de los VTV, lo que provoca frecuentes amenazas a la salud pública.
Diagnóstico de las arbovirosis y robovirosisAntes de entrar en las particularidades del diagnóstico de las infecciones por los VTV hay que hacer hincapié en las consecuencias que puede tener para la salud pública la detección de un virus de elevado riesgo biológico (fiebre hemorrágica viral) o que pueda iniciar un ciclo de circulación autóctona en aquellas regiones en las que está presente su vector de transmisión.
En caso de sospecha de fiebre hemorrágica viral en España se debe contactar inmediatamente con las autoridades sanitarias y con el Laboratorio Nacional de Referencia (Centro Nacional de Microbiología [CNM], Instituto de Salud CarlosIII), que orientarán sobre la toma, procesamiento y envío de las muestras al laboratorio para su diagnóstico. Recientemente se ha aprobado un Protocolo de vigilancia de las fiebres hemorrágicas víricas (FHV); así, cuando se detecte un caso probable de FHV, el servicio de Vigilancia de la comunidad autónoma lo comunicará de forma urgente al Centro de Coordinación de Alertas y Emergencias Sanitarias (CCAES) del Ministerio de Sanidad y Servicios Sociales e Igualdad y al Centro Nacional de Epidemiología. El CCAES valorará junto con las comunidades autónomas afectadas las medidas a tomar y si fuera necesario su notificación al Sistema de Alerta y Respuesta Rápida de Unión Europea y a la Organización Mundial de la Salud, de acuerdo con el Reglamento Sanitario Internacional de 2005.
Un laboratorio de microbiología debe asumir también la responsabilidad de notificar cualquier hallazgo de un virus importado en un paciente que puede haber iniciado un ciclo de transmisión autóctona en un área en la que está presente su vector de transmisión (DENV, CHIKV) y de los casos de infección por los VTV autóctonos para establecer las medidas de control de la infección. La confirmación de los resultados por parte del Laboratorio Nacional de Referencia es, además de obligatoria, de crucial importancia para las actividades de vigilancia microbiológica a escalas nacional e internacional. Finalmente, si el laboratorio no dispone de la metodología de diagnóstico del patógeno sospechado, debe enviar muestras al Laboratorio Regional o Nacional de Referencia, que se hará cargo del diagnóstico etiológico y de la notificación de los resultados. El Laboratorio Nacional de Referencia apoyará a los laboratorios de microbiología que lo demanden no solo en estos casos, sino también transfiriendo metodología para el diagnóstico de los VTV y facilitando el control de calidad de los reactivos utilizados.
En el diagnóstico de los VTV es imprescindible conocer información sobre posibles viajes realizados por el paciente, así como conocer antecedentes en la exposición a animales. Esta información orienta al laboratorio en la selección de análisis para identificación del agente causal, así como en la interpretación de los resultados de diagnóstico obtenido. Otra información muy preciada consiste en conocer la fecha de inicio de los síntomas, dado que orientará en la selección del tipo de métodos, directos o indirectos, a emplear en el laboratorio.
El papel del microbiólogo en los hospitales es esencial para reforzar no solo el diagnóstico, sino la vigilancia y el control de estas enfermedades. Dada la importancia de la vigilancia microbiológica de las enfermedades víricas transmitidas por vector, el CNM ha puesto en marcha un programa que no tiene coste para el hospital, basado en el estudio de los casos sospechosos.
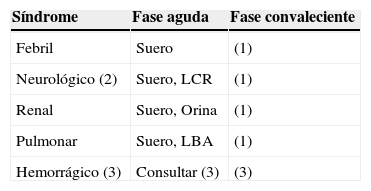
Recogida y transporte de muestrasLa muestra de elección es el suero, y las muestras adicionales que resultan apropiadas para la investigación de los VTV se seleccionarán en función del síndrome clínico observado (tabla 2). El estudio de muestras tomadas en la fase convaleciente puede ayudar en algunos casos a establecer un diagnóstico.
Muestras empleadas para el diagnóstico en función del tipo de síndrome clínico observado
| Síndrome | Fase aguda | Fase convaleciente |
|---|---|---|
| Febril | Suero | (1) |
| Neurológico (2) | Suero, LCR | (1) |
| Renal | Suero, Orina | (1) |
| Pulmonar | Suero, LBA | (1) |
| Hemorrágico (3) | Consultar (3) | (3) |
LBA: lavado bronco alveolar.
(1) El estudio de esta muestra puede ser necesario para establecer el diagnóstico etiológico final si los resultados obtenidos en la fase aguda de la enfermedad no fueran concluyentes.
(2) Suero y LCR deben obtenerse simultáneamente para poder estudiar la producción intratecal de anticuerpos. Este estudio es necesario en determinadas situaciones, como la presencia de una patología neurológica asociada a la vacunación frente la fiebre amarilla.
(3) Las muestras obtenidas se enviarán urgentemente al CNM siguiendo la normativa de materiales biológicos clase A. El laboratorio recomienda tomar las siguientes muestras, que servirán para la detección urgente de virus de elevado biológico, y de antígenos de plasmodio y de leptospira y para la realización de estudios complementarios posteriores: sangre anticoagulada con citrato: 2 viales de 10ml; sangre coagulada: 2 viales de 10ml (no centrifugar para evitar la formación de aerosoles en el laboratorio); orina: 2 viales con 10ml, y en caso de enfermedad que afecte preferentemente a un órgano, consultar con el CNM.
El suero debe tomarse en fase aguda de la enfermedad, en los primeros días tras el inicio de los síntomas (hasta el quinto a séptimo día), y en fase convaleciente 10 a 14días después. El LCR debe tomarse en las primeras 72h tras el inicio de los síntomas y enviarse inmediatamente al laboratorio para su procesamiento.
Es imprescindible una buena conservación de las muestras hasta su análisis (a 4°C si se tarda en procesar menos de 48-72h y a –70°C si el proceso se demora más), con objeto de asegurar la infectividad de las partículas víricas y la integridad del ARN viral. Las muestras destinadas a estudios serológicos pueden conservarse a –20°C.
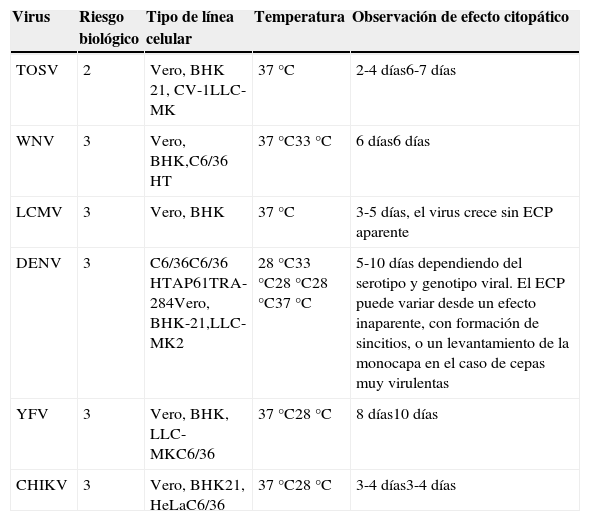
Aislamiento mediante cultivoEl aislamiento en el laboratorio de los VTV a partir de las muestras clínicas se puede realizar mediante técnicas de cultivo celular en los laboratorios con instalaciones de seguridad biológica adecuados para el virus sospechado. Así, el cultivo de los WNV, LCMV, DENV y CHIKV analizados en este documento requiere disponer de un laboratorio de nivel 3 de bioseguridad (tablas 1 y 3). Sin embargo, la manipulación de muestras clínicas que no implique la replicación de los virus se puede realizar en un laboratorio de nivel 2 de bioseguridad, con prácticas adecuadas. El aislamiento del virus en cultivo, aparte de su interés diagnóstico permite realizar estudios de caracterización biológica, antigénica, molecular y genética de los nuevos virus que hayan sido detectados.
Arbovirus y robovirus, nivel de riesgo biológico, líneas celulares y tiempo de observación del efecto citopático
| Virus | Riesgo biológico | Tipo de línea celular | Temperatura | Observación de efecto citopático |
|---|---|---|---|---|
| TOSV | 2 | Vero, BHK 21, CV-1LLC-MK | 37°C | 2-4 días6-7 días |
| WNV | 3 | Vero, BHK,C6/36 HT | 37°C33°C | 6 días6 días |
| LCMV | 3 | Vero, BHK | 37°C | 3-5 días, el virus crece sin ECP aparente |
| DENV | 3 | C6/36C6/36 HTAP61TRA-284Vero, BHK-21,LLC-MK2 | 28°C33°C28°C28°C37°C | 5-10 días dependiendo del serotipo y genotipo viral. El ECP puede variar desde un efecto inaparente, con formación de sincitios, o un levantamiento de la monocapa en el caso de cepas muy virulentas |
| YFV | 3 | Vero, BHK, LLC-MKC6/36 | 37°C28°C | 8 días10 días |
| CHIKV | 3 | Vero, BHK21, HeLaC6/36 | 37°C28°C | 3-4 días3-4 días |
En el caso de emplear muestra de suero o sangre es necesario que haya sido recogida, en general, durante los 5-7primeros días de inicio de los síntomas. El LCR no es una muestra en la que sea fácil recuperar virus en cultivo dadas las bajas cargas virales y la escasa vida media de los virus envueltos en esta muestra biológica. Para alguno de los virus mencionados, como el LCMV, el rendimiento del cultivo en líneas celulares es muy bajo.
Los VTV son capaces de infectar y producir efecto citopático (ECP) en varias líneas celulares de vertebrados, como: Vero (riñón de mono verde africano), HeLa (tumor uterino humano), BHK-21 (riñón de hámster), CV-1(riñón de mono Cercopithecus aethiops) y LLC-MK2 (riñón de mono Rhesus). También se pueden aislar los VTV en ratones inoculados intracerebralmente. Para el aislamiento de los virus transmitidos por mosquitos (WNV, DENV, YFV y CHIKV) se utilizan además líneas celulares de mosquito, como son las líneas C6/36 HT (derivadas de Ae.albopictus), AP61 (derivadas de Ae.pseudoscutellaris), y para DENV, además, se utiliza la línea TRA-284, derivada de Toxorhynchites amboinensis, entre otras (tabla 3). Tras la detección de ECP, la identificación específica del virus se realiza por técnicas de biología molecular, por pruebas de neutralización del crecimiento viral o por inmunofluorescencia (IF) usando anticuerpos específicos, aunque esta última técnica tiene limitaciones en cuanto a su posible especificidad. A continuación se apuntan algunas características que han de ser consideradas ante la identificación del agente etiológico empleando técnicas inmunológicas:
Virus West NileEl WNV antigénicamente está muy relacionado con los virus del grupo de la encefalitis japonesa, grupo al que pertenecen el propio virus de la encefalitis japonesa (JEV), el virus Usutu (USUV) o el virus de la encefalitis de San Luis (SLEV). Existe un cierto grado de reactividad cruzada en los ensayos de fijación del complemento e IF, y la forma de identificarlos inequívocamente mediante métodos serológicos consiste en la obtención de diferencias significativas (4 veces) en la prueba de neutralización del crecimiento viral.
Virus ToscanaEl TOSV antigénicamente está muy relacionado con el virus Nápoles (SFNV), con el que existe un alto grado de reactividad cruzada en determinadas pruebas como son la reacción de fijación del complemento e IF. No obstante, ambos virus, además de presentar diferencias biológicas, presentan diferencias inmunológicas mediante el test de neutralización de reducción de placas. El tratamiento con dimetilsulfóxido (DMSO) no afecta al tamaño de las placas que produce el TOSV, a diferencia de lo que ocurre con el SFNV, que con la adición de DMSO aumentan tanto el tamaño como el número de placas formadas.
Virus dengueLa prueba de IF o IF indirecta (IFI) es la técnica más empleada para la identificación del DENV en cultivo. Para la IF directa se dispone de un anticuerpo policlonal marcado con fluoresceína (FITC). Para la IFI, se dispone de anticuerpos monoclonales que están disponibles comercialmente en forma de complejo anti-dengue (Chemicon Int. Inc, Temecula, CA, EE.UU.), el cual detecta de forma genérica todos los serotipos o, en forma individual, para la tipificación de cada uno de los serotipos.
Virus chikungunyaPara la identificación inmunológica del CHIKV hay que tener en cuenta la reactividad cruzada con otros miembros del serogrupo al que pertenece, fundamentalmente con el virus O’Nyong nyong (ONNV), que produce también un cuadro clínico similar y con el que comparte la distribución geográfica en África y el vector de transmisión. Durante la fase aguda de la enfermedad este virus produce viremias de alto título, por lo que debe extremarse el cuidado en su manejo para evitar contaminaciones.
Detección de antígenoLas tecnologías basadas en el reconocimiento de un componente viral (proteína o ácido nucleico) o de la partícula del virus en su totalidad son útiles en etapas tempranas de la infección, antes de que la respuesta inmune haga desaparecer las partículas virales, por lo que la fecha de la toma de muestra, principalmente suero, ha de ser, en general, entre los 5-7 primeros días de inicio de los síntomas.
Los métodos basados en la detección de antígeno presentan la ventaja de permitir obtener un resultado rápido. Como contrapartida, estos resultados pueden ser difíciles de interpretar debido a la reactividad antigénica cruzada que existe entre las especies del mismo género viral como se ha comentado anteriormente, interpretación de resultados que se complica si distintas especies del mismo género viral circulan en la misma zona geográfica (tabla 1).
Las técnicas de detección de antígeno basadas en métodos inmunohistoquímicos han sido especialmente útiles en los casos en que no se ha podido establecer el diagnóstico por otras vías o para demostrar la infección viral en síndromes complejos, como el síndrome visceral tras vacunación frente al YFV o en infecciones asociadas a trasplante de órganos por el LCMV o por el WNV.
El desarrollo y la comercialización de métodos de detección de antígeno de los VTV se han centrado principalmente en el DENV y los VTV neurotropos americanos (WNV, SLEV, virus de la encefalitis equina del este [EEEV], virus de la encefalitis equina venezolana [VEEV] y virus de la encefalitis equina del oeste [WEEV]). En Estados Unidos se comercializa un método para la vigilancia en mosquitos de la circulación de los VTV neurotropos. El método usa una tira recubierta con anticuerpos frente a los WNV, SLEV y VEEV, y es rápido y de fácil manejo, aunque menos sensible que el uso de métodos moleculares.
Para la detección de antígeno del WNV también se ha desarrollado un sistema dirigido a la proteínaE basado en el reconocimiento a partir de un nanobiosensor electroquímico utilizando una membrana de alúmina, y se ha observado que el límite de detección lo hace comparable a las técnicas de reacción en cadena de la polimerasa (PCR).
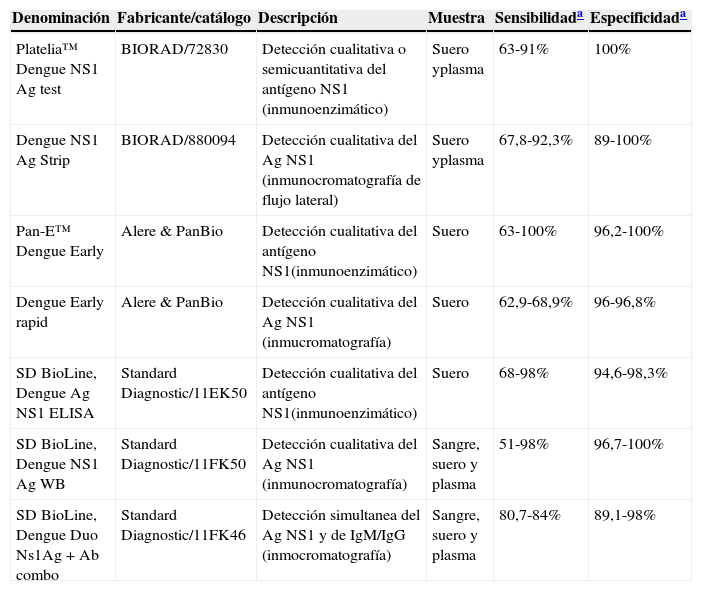
Sin embargo, actualmente, tanto para la detección del DENV como para la de otros flavivirus, el antígeno NS1 suele ser la diana de elección, por tratarse de un antígeno soluble que generalmente se encuentra de forma abundante durante la infección. En este caso, además, los ensayos suelen generar resultados específicos, con niveles muy bajos de reactividad cruzada entre los diferentes flavivirus. En el caso de la detección de la proteína NS1 del DENV puede realizarse desde el día1 hasta el día11 de inicio de los síntomas, y están disponibles numerosos equipos comerciales basados en técnicas inmunoenzimáticas así como inmunocromatográfícas (tabla 4).
Métodos comerciales disponibles para la detección del antígeno NS1 del virus dengue
| Denominación | Fabricante/catálogo | Descripción | Muestra | Sensibilidada | Especificidada |
|---|---|---|---|---|---|
| Platelia™ Dengue NS1 Ag test | BIORAD/72830 | Detección cualitativa o semicuantitativa del antígeno NS1 (inmunoenzimático) | Suero yplasma | 63-91% | 100% |
| Dengue NS1 Ag Strip | BIORAD/880094 | Detección cualitativa del Ag NS1 (inmunocromatografía de flujo lateral) | Suero yplasma | 67,8-92,3% | 89-100% |
| Pan-E™ Dengue Early | Alere & PanBio | Detección cualitativa del antígeno NS1(inmunoenzimático) | Suero | 63-100% | 96,2-100% |
| Dengue Early rapid | Alere & PanBio | Detección cualitativa del Ag NS1 (inmucromatografía) | Suero | 62,9-68,9% | 96-96,8% |
| SD BioLine, Dengue Ag NS1 ELISA | Standard Diagnostic/11EK50 | Detección cualitativa del antígeno NS1(inmunoenzimático) | Suero | 68-98% | 94,6-98,3% |
| SD BioLine, Dengue NS1 Ag WB | Standard Diagnostic/11FK50 | Detección cualitativa del Ag NS1 (inmunocromatografía) | Sangre, suero y plasma | 51-98% | 96,7-100% |
| SD BioLine, Dengue Duo Ns1Ag+Ab combo | Standard Diagnostic/11FK46 | Detección simultanea del Ag NS1 y de IgM/IgG (inmocromatografía) | Sangre, suero y plasma | 80,7-84% | 89,1-98% |
Las técnicas moleculares basadas en la detección del genoma viral han supuesto un enorme avance también en el diagnóstico de las infecciones producidas por los VTV, dada la rapidez en la obtención de resultados, su especificidad y su sensibilidad. Son consideradas de elección en el diagnóstico rápido en los primeros días de la enfermedad, aunque han de ser complementadas con los resultados procedentes de los métodos serológicos, dada la corta viremia que caracteriza a la mayoría de las infecciones producidas por este grupo de virus.
Se han desarrollado numerosos métodos basados en la PCR, tanto en el formato PCR convencional o tiempo final como en el formato PCR en tiempo real. Para ambos tipos de métodos se requiere un paso de transcripción reversa, dado que se trata de virus con genoma de ARN. Este paso se puede acoplar a la PCR (one-step) o bien puede realizarse independientemente, en una reacción previa a la reacción de amplificación genómica. La PCR en tiempo real comienza a desplazar a la RT-PCR convencional, que muchas veces requiere la aplicación de una PCR secuencial (PCR anidada o nested-PCR) para alcanzar una sensibilidad similar, lo que comporta más carga de trabajo, un incremento del tiempo necesario hasta la obtención de los resultados y un mayor riesgo de contaminaciones.
Hay disponibles métodos comerciales de PCR en tiempo real para los WNV, DENV, CHIKV, JEV, hantavirus y virus de la fiebre hemorrágica de Crimea-Congo, entre otros. La mayoría de ellos utilizan sondas de hibridación TaqMan como sistema de marcaje específico del producto amplificado; sin embargo, la mayoría de los métodos desarrollados siguen siendo técnicas «caseras» que ofrecen resultados óptimos de sensibilidad y especificidad. Aunque para el diagnóstico en el laboratorio la PCR en tiempo real se ha impuesto a las clásicas técnicas de PCR, para la realización de estudios de epidemiología molecular y variabilidad genética es preferible la utilización de la PCR convencional que permite la obtención de fragmentos genómicos de mayor tamaño portadores de información filogenética de contrastada calidad.
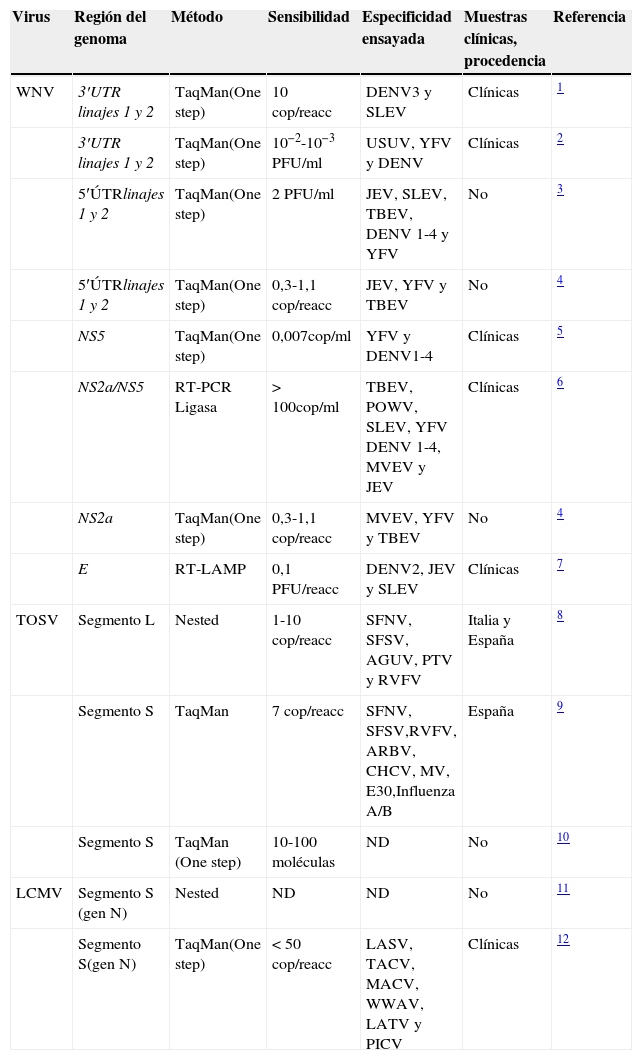
Otras estrategias metodológicas desarrolladas para la detección de genoma de los VTV utilizan técnicas de amplificación isotérmica tales como Nucleic Acid Sequence-Based Amplification (NASBA), Transcription-Mediated Amplification (TMA) y Retrotranscription Loop-mediated isothermal AMPlification (RT-LAMP). Las técnicas NASBA y TMA consisten en la amplificación del genoma durante un proceso isotérmico, a una temperatura entre 41 y 60°C, en presencia de 3 enzimas: transcriptasa, polimerasa y RNAasa. La técnica LAMP consiste también en un proceso isotérmico en el que la síntesis de nuevas cadenas se consigue utilizando una enzima con capacidad de desplazar la hebra de ADN y 4 cebadores cuyo diseño permite la obtención de una horquilla o loop de ADN. Alguna de esta metodología se ha desarrollado para detectar los WNV, DENV y CHIKV. En Estados Unidos se ha comercializado una TMA para el análisis de la presencia de genoma del WNV en muestras de donantes de sangre y de órganos, y recientemente esta metodología se está ensayando para la detección del DENV en donantes de sangre en Puerto Rico. En la tabla 5 se muestra una selección de métodos de detección de ácidos nucleicos para los virus mencionados en el documento.
Métodos moleculares basados en la amplificación de genoma viral
| Virus | Región del genoma | Método | Sensibilidad | Especificidad ensayada | Muestras clínicas, procedencia | Referencia |
|---|---|---|---|---|---|---|
| WNV | 3′UTR linajes 1 y 2 | TaqMan(One step) | 10 cop/reacc | DENV3 y SLEV | Clínicas | 1 |
| 3′UTR linajes 1 y 2 | TaqMan(One step) | 10−2-10−3 PFU/ml | USUV, YFV y DENV | Clínicas | 2 | |
| 5′ÚTRlinajes 1 y 2 | TaqMan(One step) | 2 PFU/ml | JEV, SLEV, TBEV, DENV 1-4 y YFV | No | 3 | |
| 5′ÚTRlinajes 1 y 2 | TaqMan(One step) | 0,3-1,1 cop/reacc | JEV, YFV y TBEV | No | 4 | |
| NS5 | TaqMan(One step) | 0,007cop/ml | YFV y DENV1-4 | Clínicas | 5 | |
| NS2a/NS5 | RT-PCR Ligasa | > 100cop/ml | TBEV, POWV, SLEV, YFV DENV 1-4, MVEV y JEV | Clínicas | 6 | |
| NS2a | TaqMan(One step) | 0,3-1,1 cop/reacc | MVEV, YFV y TBEV | No | 4 | |
| E | RT-LAMP | 0,1 PFU/reacc | DENV2, JEV y SLEV | Clínicas | 7 | |
| TOSV | Segmento L | Nested | 1-10 cop/reacc | SFNV, SFSV, AGUV, PTV y RVFV | Italia y España | 8 |
| Segmento S | TaqMan | 7 cop/reacc | SFNV, SFSV,RVFV, ARBV, CHCV, MV, E30,Influenza A/B | España | 9 | |
| Segmento S | TaqMan (One step) | 10-100 moléculas | ND | No | 10 | |
| LCMV | Segmento S (genN) | Nested | ND | ND | No | 11 |
| Segmento S(genN) | TaqMan(One step) | < 50 cop/reacc | LASV, TACV, MACV, WWAV, LATV y PICV | Clínicas | 12 |
| DENV | E/NS1 | Nested | 100 cop/reacc | No especificado | Clínicas | 13 |
| C/preM | Nested | < 103 TCDI50 | WNV, JEV,SLEV, YFV y EGV | Puerto Rico y Sudeste Asiático | 14 | |
| C | Nested | 1-50 PFU/ml | DENV 1-4 | Clínicas | 15 | |
| 3′NCR | TaqMan | 0,4-1,2×106 PFU/ml | JEV, SLEV WNV, YFV, VEEV, EEEV EEEV y WEEV | No | 16 | |
| 3′NCR | TaqMan | 100-5.000 cop/reacc | WNV, YFV, LIV y JEV | Clínicas | 17 | |
| 3′NCR | TaqMan | 0-8.000 cop/ml | ND | Clínicas | 18 | |
| CHIKV | E1 | RT-PCR convencional | 100 cop/ml | ND | India | 19 |
| nsP4 | TaqMan | 10,5 cop/ml | RRV, ONNV, MAYV, SFV, SINV,WEEV, EEEV y VEEV | India, África e islas del Indico | 19,20 | |
| E1 | RT-LAMP | 20 cop | DENV1-4, JEV, WNV y SLEV | India | 21 | |
| E1 | TaqMan(One step) | 3 cop/reacc | EV, HIV, HCV,HBV CMV, EBV, HSV, VZV, DENV y WNV | Clínicas | 20 | |
| nsP1 | SybrGreen. | 1 PFU/ml | ND | No | 22 | |
| E1, nsP1, 5′NTR, nsP2 | TaqMan(One step) | 28 PFU0,3 PFU0,3 PFU0,3 PFU | SINV, SFV, MAYV, UNAV, OCKV, AURAV, RRV, NDUV, VEEV y EEV | No | 23 | |
| nsP1 | TaqMan | 3,8-5,3 cop/reacc | RRV, SINV, SFV,MAYV, VEEV, WEEV, EEEV, ONNV y BFV | Clínicas | 24a | |
| YFV | Gen E | Seminested | 89 PFU/ml | DENV y SLEV | Brasil | 25 |
| NS5 | SybrGreen(One Step) | 8,9 PFU/ml | DENV y SLEV | Brasil | 25 | |
| NS5 | TaqMan (One step) | 12 cop/reacc | DENV, TBEV y JEV | No | 26 | |
| 5ÚTR | TaqMan | 25,9 PFU/ml | ND | Costa de Marfily Brasil | 27 | |
| 5ÚTR | TaqMan(One Step) | 10 cop/reacc | DENV, WNV, JEV, TBEV, VEEV, WEEV, CHIKV y ONNV | No | 28 |
AGUV: Aguacate virus; ARBV: Arbia virus; AURAV: Aura virus; BFV: Barmah Forest virus; CHCV: Chagres virus; CMV: citomegalovirus; E30: echovirus 30; EBV: Epstein-Barr virus; EHV: Edge Hill virus; EV: enterovirus; HBV: hepatitisB virus; HCV: hepatitisC virus; HSV: herpes simplex virus; ATV: Latino virus; LIV: Louping ill virus; MACV: Machupo virus; MV: Mumps virus; MYV: Mayaro virus; ND: no hay datos disponibles; NDUV: Ndumu virus; OCKV: Ockelbo virus; PICV: Pichinde virus; POWV: Powasan virus; PTV: Punta Toro virus; RRV: Ros River virus; SFV: Semliki Forest virus; SINV: Sindbis virus; TACV: Tacaribe virus; TBEV: Tick-borne encephalitis virus; UNA: Una virus; VZV: varicella-zoster virus; WWAV: Whitewater arroyo virus.
Desde pocos días después del inicio de los síntomas empieza a ser detectable una respuesta de anticuerpos específicos. En un primer momento son del isotipo IgM, anticuerpos que son de rápida aparición (normalmente entre 5 y 7días desde el comienzo de la sintomatología) y, en general, de corta duración (2-4meses), seguidos por anticuerpos del isotipo IgG, que permanecen detectables durante toda la vida. Para muchos de los VTV, dado su corto periodo de viremia, la serología basada en la detección de la respuesta de anticuerpos es el método diagnóstico de elección.
La muestra más adecuada para hacer el diagnóstico serológico es el suero, aunque en ocasiones se puede realizar sobre muestra de plasma. La detección de IgM es el marcador elegido para el diagnóstico de una infección reciente, en tanto que la respuesta IgG aislada es un indicador de una infección pasada, o de vacunación en su caso, siendo el marcador de aplicación para estudios de seroprevalencia. Cuando las muestras son tomadas muy cerca del comienzo de la enfermedad es frecuente la ausencia de respuesta detectable de IgM, por lo que es preciso analizar otra muestra tomada 7-10días después; analizando las muestras pareadas se confirma la infección demostrando seroconversión de IgM o IgG específica. La respuesta IgM puede ser detectable varios meses después de la infección por los TOSV, WNV, DENV y CHIKV, lo que origina dificultades para interpretar el resultado como una infección aguda. Los flavivirus muestran un alto grado de reacción cruzada, y la intensidad de esta reactividad depende del grado de purificación del antígeno empleado, lo que supone que con frecuencia se puedan detectar reactividades múltiples. En la interpretación de los resultados serológicos frente a los flavivirus además ha de considerarse el antecedente de vacunación frente a alguno de ellos (JEV, Tick-borne encephalitis virus [TBEV] y YFV). En estas situaciones son especialmente útiles los ensayos de avidez de IgG, de aplicación para el WNV y el DENV. De cualquier forma, para la caracterización última de la respuesta serológica específica es preciso realizar la técnica de neutralización tanto para los flavivirus como para el TOSV y los alphavirus, considerando en este último grupo de virus que tanto el CHIKV como el ONNV pueden ocasionar síndromes articulares. La técnica de neutralización se debe realizar en laboratorios expertos y adecuadamente equipados en términos de seguridad biológica, dado el nivel 3 de riesgo biológico en el que se incluyen la mayoría de estos virus (tablas 1 y 3).
Dado el tropismo neurológico de alguno de los VTV, la demostración de producción intratecal de anticuerpos es una herramienta aplicable para confirmar la infección del sistema nervioso central por los WNV, TOSV, LCMV, TBEV, DENV y para el CHIKV.
Como ya se ha comentado, la especificidad de los ensayos depende en gran medida del grado de purificación de los antígenos empleados. Básicamente, para el diagnóstico serológico de las infecciones por estos virus se emplean tanto técnicas de IFI, en general empleando células infectadas por virus, como de ELISA, con extractos de células infectadas, o más adecuadamente antígenos recombinantes. El uso de antígenos recombinantes es la aproximación más adecuada para conseguir minimizar las reactividades heterólogas, aunque no las evita totalmente.
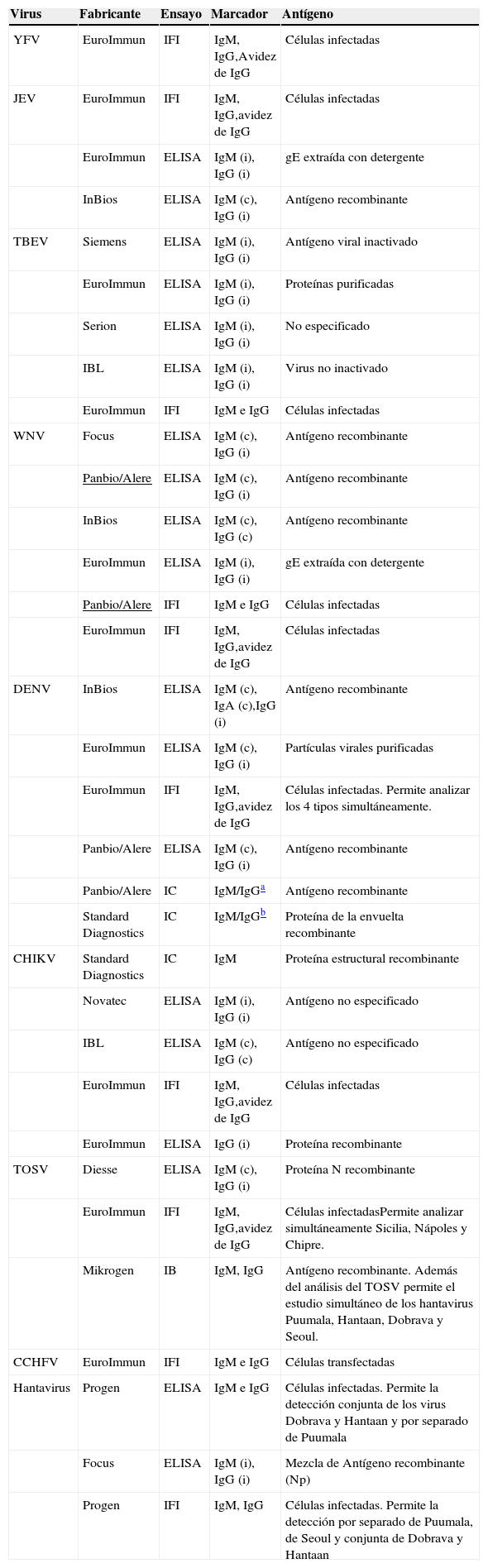
Existen numerosos ensayos disponibles comercialmente para identificar la respuesta serológica frente a algunos de estos virus, basados en diferentes metodologías (IFI, ELISA, inmunocromatografía IC). Se carece sin embargo de métodos comerciales para otros muchos, entre ellos el LCMV; en estos casos se pueden producir reactivos caseros utilizando antígenos obtenidos en laboratorios especializados y adecuadamente equipados. En la tabla 6, que no pretende ser exhaustiva, se presenta una lista de estos ensayos que pueden estar disponibles en los laboratorios españoles. Los fabricantes de algunos de estos ensayos contemplan su aplicación no solo a la determinación de anticuerpos en muestras de suero o plasma, sino a la caracterización de la avidez de IgG específica y a la confirmación de producción intratecal de anticuerpos, mediante el ensayo de muestras de LCR. Un aspecto importante para seleccionar el método más adecuado para cada laboratorio es conocer las características de los ensayos disponibles, mediante estudios de validación realizados en laboratorios experimentados.
Métodos comerciales serológicos basados en la detección de anticuerpos
| Virus | Fabricante | Ensayo | Marcador | Antígeno |
|---|---|---|---|---|
| YFV | EuroImmun | IFI | IgM, IgG,Avidez de IgG | Células infectadas |
| JEV | EuroImmun | IFI | IgM, IgG,avidez de IgG | Células infectadas |
| EuroImmun | ELISA | IgM (i), IgG (i) | gE extraída con detergente | |
| InBios | ELISA | IgM (c), IgG (i) | Antígeno recombinante | |
| TBEV | Siemens | ELISA | IgM (i), IgG (i) | Antígeno viral inactivado |
| EuroImmun | ELISA | IgM (i), IgG (i) | Proteínas purificadas | |
| Serion | ELISA | IgM (i), IgG (i) | No especificado | |
| IBL | ELISA | IgM (i), IgG (i) | Virus no inactivado | |
| EuroImmun | IFI | IgM e IgG | Células infectadas | |
| WNV | Focus | ELISA | IgM (c), IgG (i) | Antígeno recombinante |
| Panbio/Alere | ELISA | IgM (c), IgG (i) | Antígeno recombinante | |
| InBios | ELISA | IgM (c), IgG (c) | Antígeno recombinante | |
| EuroImmun | ELISA | IgM (i), IgG (i) | gE extraída con detergente | |
| Panbio/Alere | IFI | IgM e IgG | Células infectadas | |
| EuroImmun | IFI | IgM, IgG,avidez de IgG | Células infectadas | |
| DENV | InBios | ELISA | IgM (c), IgA (c),IgG (i) | Antígeno recombinante |
| EuroImmun | ELISA | IgM (c), IgG (i) | Partículas virales purificadas | |
| EuroImmun | IFI | IgM, IgG,avidez de IgG | Células infectadas. Permite analizar los 4 tipos simultáneamente. | |
| Panbio/Alere | ELISA | IgM (c), IgG (i) | Antígeno recombinante | |
| Panbio/Alere | IC | IgM/IgGa | Antígeno recombinante | |
| Standard Diagnostics | IC | IgM/IgGb | Proteína de la envuelta recombinante | |
| CHIKV | Standard Diagnostics | IC | IgM | Proteína estructural recombinante |
| Novatec | ELISA | IgM (i), IgG (i) | Antígeno no especificado | |
| IBL | ELISA | IgM (c), IgG (c) | Antígeno no especificado | |
| EuroImmun | IFI | IgM, IgG,avidez de IgG | Células infectadas | |
| EuroImmun | ELISA | IgG (i) | Proteína recombinante | |
| TOSV | Diesse | ELISA | IgM (c), IgG (i) | Proteína N recombinante |
| EuroImmun | IFI | IgM, IgG,avidez de IgG | Células infectadasPermite analizar simultáneamente Sicilia, Nápoles y Chipre. | |
| Mikrogen | IB | IgM, IgG | Antígeno recombinante. Además del análisis del TOSV permite el estudio simultáneo de los hantavirus Puumala, Hantaan, Dobrava y Seoul. | |
| CCHFV | EuroImmun | IFI | IgM e IgG | Células transfectadas |
| Hantavirus | Progen | ELISA | IgM e IgG | Células infectadas. Permite la detección conjunta de los virus Dobrava y Hantaan y por separado de Puumala |
| Focus | ELISA | IgM (i), IgG (i) | Mezcla de Antígeno recombinante (Np) | |
| Progen | IFI | IgM, IgG | Células infectadas. Permite la detección por separado de Puumala, de Seoul y conjunta de Dobrava y Hantaan |
(c) captura; (i) indirecto; IB inmunoblot.
Los autores declaran no tener ningún conflicto de intereses.