Las infecciones causadas por parásitos del tracto digestivo humano representan un problema de salud pública global. En países industrializados, sus particulares características epidemiológicas (baja prevalencia general de enteroparásitos), económicas (elevados costes laborales) y clínicas (incremento constante del número de muestras y determinaciones diagnósticas a realizar) han causado que las técnicas moleculares estén progresivamente reemplazando a la microscopía convencional como método de diagnóstico de primera línea de estos patógenos en el laboratorio clínico moderno. Los métodos basados en PCR, particularmente los desarrollados para la detección simultánea de varios agentes que causan la misma etiología (diagnóstico sindrómico), representan ya una opción coste-efectiva que permite automatizar procesos, optimizar flujos de trabajo, comparar resultados entre diferentes laboratorios y facilitar la acreditación de procedimientos diagnósticos. En esta revisión se detalla de forma clara y concisa el estado actual del diagnóstico molecular de las principales especies de parásitos intestinales humanos, particularmente de los protozoos entéricos causantes de diarrea (Cryptosporidium spp., Giardia duodenalis, Entamoeba histolytica), de los miembros más destacados de los filos Microsporidia (Enterocytozoon bieneusi) y Stramenopiles (Blastocystis sp.), así como de los helmintos transmitidos por suelo (Ancylostoma spp., Ascaris lumbricoides, Necator americanus, Strongyloides stercoralis y Trichuris trichiura) y alimentos (Anisakis spp., Clonorchis sinensis, Fasciola spp., Taenia solium, y Trichinella spiralis). Especial atención se ha prestado a la descripción de técnicas y formatos disponibles, a sus prestaciones diagnósticas y a los marcadores genéticos más usados, tanto para la detección en laboratorios clínicos como para el genotipado en centros de referencia e investigación.
Infections causes by parasites of the gastrointestinal tract are a global public health problem. In industrialised countries, their particular epidemiological (low general prevalence of enteroparasites), economic (high labour costs) and clinical characteristics (constant increase in the number of samples and diagnostic determinations to be performed) have led molecular techniques to progressively replace conventional microscopy as the first-line diagnostic method of these pathogens in modern clinical laboratories. PCR-based techniques, particularly those developed for the simultaneous detection of the various agents that can cause the same infectious disease (syndromic diagnosis), already represent a cost-effective option that allow process automisation, workflow optimisation, and comparison of results among different laboratories, and facilitate accreditation of diagnostic procedures. This review clearly and concisely discusses the current situation of the molecular diagnosis of the main species of intestinal parasites in humans, particularly the enteric protozoans causing diarrhoea (Cryptosporidium spp., Giardia duodenalis, Entamoeba histolytica), the most important members the Microsporidia phyla (Enterocytozoon bieneusi) and Stramenopiles phyla (Blastocystis sp.), as well as the helminths transmitted by soil (Ancylostoma spp., Ascaris lumbricoides, Necator americanus, Strongyloides stercoralis and Trichuris trichiura) and food (Anisakis spp., Clonorchis sinensis, Fasciola spp., Taenia solium, and Trichinella spiralis). Special attention is paid to the description of available techniques and formats, to their diagnostic benefits and the most widely used genetic markers for their detection, both in clinical laboratories and genotyping in referral and research centres.
Las infecciones por parásitos intestinales suponen un importante problema de salud pública global1,2. Cryptosporidium spp., Giardia duodenalis y Entamoeba histolytica se consideran los agentes causantes de diarrea de origen protozoario más relevantes en todo el mundo, con tasas de morbimortalidad considerables asociadas principalmente a poblaciones pediátricas en áreas desfavorecidas con pobres condiciones higienicosanitarias1. Estas 3 especies representan el 70% de las muestras positivas a cualquier parásito intestinal diagnosticadas en hospitales europeos anualmente3. Además, G. duodenalis y Cryptosporidium spp. son causa frecuente de brotes de enfermedad gastrointestinal de transmisión hídrica4. Por su parte, Enterocytozoon bieneusi y, en menor medida, Enterocytozoon intestinalis, son los microsporidios que más comúnmente infectan a los humanos, principalmente (pero no exclusivamente) individuos inmunocomprometidos y pacientes sometidos a trasplante de órgano sólido5. Otros protistas frecuentes en el tracto intestinal humano, pero de patogenicidad incierta, son Dientamoeba fragilis y Blastocystis sp.6,7.
Por otra parte, se estima que 1 de cada 6 personas en el mundo está infectada por alguna especie de geohelminto incluyendo Ancylostoma spp., Necator americanus, Ascaris lumbricoides, Trichuris trichiura o Strongyloides stercoralis8. En casos graves estos patógenos están asociados a retrasos en el desarrollo físico y cognitivo de niños infectados, son causa de mala absorción de nutrientes, anemia y obstrucción de las vías intestinales y biliares, lo que perjudica la calidad de vida de los afectados9.
Entre los helmintos transmitidos por alimentos destacan la anisakiasis y la triquinelosis producidas, respectivamente, por los nematodos Anisakis spp. y Trichinella spp., así como las taeniasis causadas por los cestodos Taenia solium y Taenia saginata. En todos ellos, la infección es adquirida por el consumo de carne o pescado crudo, ahumado o insuficientemente cocinado, que contiene los estadios larvarios de estos parásitos y causa trastornos intestinales (diarrea, dolor abdominal) y extraintestinales, principalmente reacciones alérgicas10–12. También de importancia creciente son los trematodos Clonorchis sinensis, Fasciola spp. y Opistorchis spp., que afectan primariamente a órganos internos (hígado y pulmón) de los individuos parasitados, causando problemas gastrointestinales, reacciones tóxicas y alergias. La opistorquiasis además se ha relacionado con el desarrollo de colangiocarcinoma en vías biliares. Todos ellos son hallazgos relativamente poco frecuentes en laboratorios clínicos europeos, normalmente asociados a pacientes o viajeros provenientes de áreas endémicas13.
2Diagnóstico convencional. LimitacionesEl examen microscópico de preparaciones en fresco obtenidas a partir de muestras fecales concentradas, sin modificar o teñidas con colorantes específicos, sigue siendo la técnica de referencia en muchos de los laboratorios clínicos de todo el mundo14. Debido a su bajo coste y mínimo equipamiento requerido, este método es particularmente adecuado para su uso en áreas endémicas con escasos recursos, donde la alta prevalencia parasitaria hace que la utilización de técnicas con mejores prestaciones diagnósticas no sea coste-efectiva. Sin embargo, la microscopía requiere personal técnico cualificado y tiempos elevados de procesamiento y análisis, presenta importantes problemas de sensibilidad y especificidad diagnóstica y, en la mayoría de los casos, no permite la diferenciación morfológica de especies/genotipos. La microscopía de inmunofluorescencia solventa alguno de estos inconvenientes, pero es costosa y dependiente de la experiencia del observador15. En los últimos años se han desarrollado y comercializado un gran número de técnicas de diagnóstico rápidas basadas en inmunocromatografía y ELISA que proporcionan resultados en 10-240min16,17. Aunque prácticos y relativamente asequibles, estos métodos generan con frecuencia tasas elevadas de falsos resultados positivos y negativos18,19, por lo que su uso está recomendado para el cribado de muestras, no para el diagnóstico definitivo.
3Diagnóstico molecularEn países industrializados, en los que las tasas de infección por parásitos entéricos en la población autóctona son típicamente bajas o muy bajas2, la sensibilidad de las técnicas de detección adquiere una relevancia crítica. Este hecho, unido a los elevados costes laborales, al incremento constante del volumen de muestras y peticiones diagnósticas, y la imperiosa necesidad de optimizar los flujos de trabajo de los laboratorios clínicos, hace necesario un cambio radical en la estrategia diagnóstica a adoptar20,21. Esta situación explica por qué la microscopía convencional está siendo rápidamente reemplazada por técnicas moleculares basadas en la reacción en cadena de la polimerasa (PCR) como primera línea para el diagnóstico de patógenos entéricos en los laboratorios clínicos más modernos, principalmente en Europa y Estados Unidos17,21. Esta tecnología, no solo mejora considerablemente el rendimiento diagnóstico de la microscopía22,23, sino que además permite un elevado grado de automatización21,24, posibilita la comparación directa de resultados interlaboratorios y facilita el proceso de acreditación de técnicas y procedimientos diagnósticos por agencias evaluadoras.
A pesar de su diversidad y sus particularidades epidemiológicas, muchos de los parásitos intestinales tienen como denominador común la excreción de alguna forma parasitaria (quistes/ooquistes, huevos, larvas, proglótides) junto con las heces del hospedador que parasitan. Debido a que las técnicas de diagnóstico molecular (PCR) son extremadamente sensibles a la calidad y cantidad de ADN, es esencial disponer de métodos eficaces que maximicen el proceso de rotura del material parasitario de partida, la extracción y purificación de ADN y la eliminación de substancias inhibidoras de la reacción de amplificación25.
Por otra parte, el rendimiento (sensibilidad, especificidad, cantidad de ADN amplificado, reproducibilidad, robustez) y utilidad práctica (diagnóstico clínico, cribado de muestras en estudios epidemiológicos, caracterización molecular de aislados) de cada protocolo de PCR va a depender en gran medida del formato utilizado, el gen y la región seleccionado que se debe amplificar, el diseño de los cebadores y la calidad de los reactivos (p. ej., polimerasa) utilizados26. Actualmente existe una amplia variedad de PCR para la detección de protistas y helmintos entéricos, incluyendo PCR directas, anidadas, semianidadas, en tiempo real (qPCR, únicas o múltiples), asociada al análisis del polimorfismo de longitud de fragmentos de restricción (PCR-RFLP) y basadas en mecanismos de amplificación isotérmica mediada por bucle (PCR-LAMP), entre otras (tablas 1 y 2). Las PCR anidadas y semianidadas ofrecen mejores rendimientos (en términos de sensibilidad y cantidad de ADN amplificado) que las PCR directas, y se utilizan tanto para la detección como para el genotipado de patógenos protozoarios. Las qPCR, debido a su elevada sensibilidad, relativa sencillez de manejo y rapidez, están específicamente diseñadas para fines diagnósticos y se utilizan cada vez más en laboratorios clínicos (tablas 1 y 2), sobre todo en su formato múltiple (tabla 3). La elevada flexibilidad de este diseño permite el desarrollo de ensayos adaptados a la epidemiología de un área geográfica o un grupo de población concreto, así como su uso en el diagnóstico sindrómico de patógenos causantes de diarrea, incluyendo agentes virales y bacterianos, de manera coste-efectiva27,28. La PCR-RFLP es una técnica en desuso debido a la dificultad de interpretar con claridad los perfiles de bandas diagnósticas obtenidos tras la digestión de los amplicones generados, particularmente en el caso de infecciones mixtas. Finalmente, LAMP está inicialmente diseñada para emplearse como técnica de cribado en estudios epidemiológicos de campo en condiciones adversas y no suele ser una opción práctica en el laboratorio clínico.
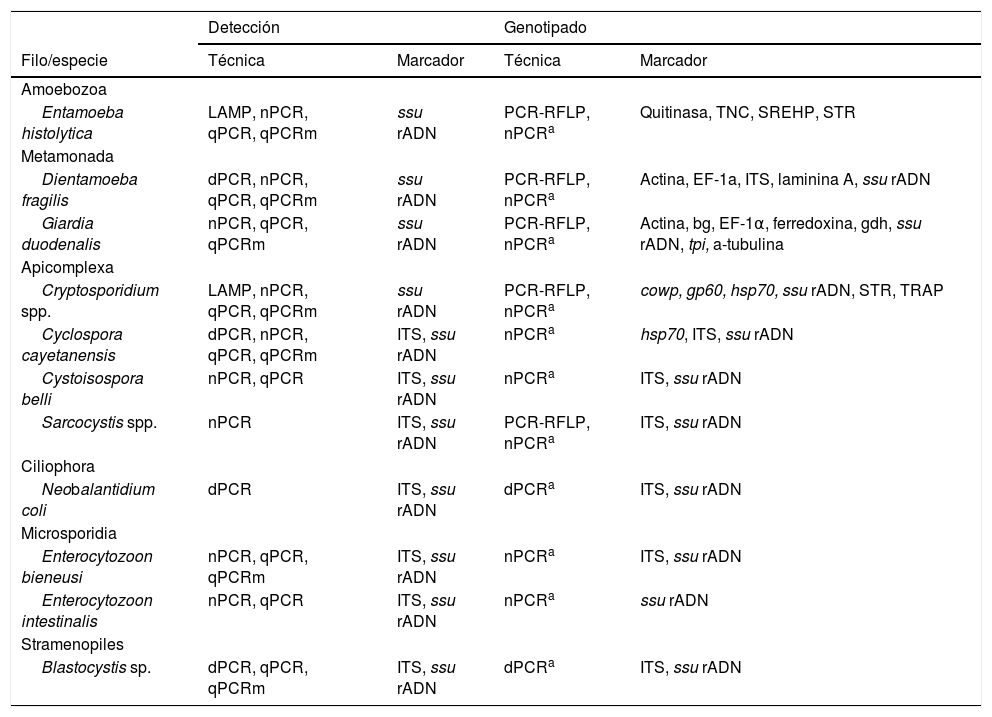
Principales técnicas y marcadores genéticos utilizados para la detección y caracterización molecular de los protistas parásitos entéricos más relevantes en salud humana.
| Detección | Genotipado | |||
|---|---|---|---|---|
| Filo/especie | Técnica | Marcador | Técnica | Marcador |
| Amoebozoa | ||||
| Entamoeba histolytica | LAMP, nPCR, qPCR, qPCRm | ssu rADN | PCR-RFLP, nPCRa | Quitinasa, TNC, SREHP, STR |
| Metamonada | ||||
| Dientamoeba fragilis | dPCR, nPCR, qPCR, qPCRm | ssu rADN | PCR-RFLP, nPCRa | Actina, EF-1a, ITS, laminina A, ssu rADN |
| Giardia duodenalis | nPCR, qPCR, qPCRm | ssu rADN | PCR-RFLP, nPCRa | Actina, bg, EF-1α, ferredoxina, gdh, ssu rADN, tpi, a-tubulina |
| Apicomplexa | ||||
| Cryptosporidium spp. | LAMP, nPCR, qPCR, qPCRm | ssu rADN | PCR-RFLP, nPCRa | cowp, gp60, hsp70, ssu rADN, STR, TRAP |
| Cyclospora cayetanensis | dPCR, nPCR, qPCR, qPCRm | ITS, ssu rADN | nPCRa | hsp70, ITS, ssu rADN |
| Cystoisospora belli | nPCR, qPCR | ITS, ssu rADN | nPCRa | ITS, ssu rADN |
| Sarcocystis spp. | nPCR | ITS, ssu rADN | PCR-RFLP, nPCRa | ITS, ssu rADN |
| Ciliophora | ||||
| Neobalantidium coli | dPCR | ITS, ssu rADN | dPCRa | ITS, ssu rADN |
| Microsporidia | ||||
| Enterocytozoon bieneusi | nPCR, qPCR, qPCRm | ITS, ssu rADN | nPCRa | ITS, ssu rADN |
| Enterocytozoon intestinalis | nPCR, qPCR | ITS, ssu rADN | nPCRa | ssu rADN |
| Stramenopiles | ||||
| Blastocystis sp. | dPCR, qPCR, qPCRm | ITS, ssu rADN | dPCRa | ITS, ssu rADN |
Tipos de PCR (reacción en cadena de la polimerasa): dPCR: PCR directa; nPCR: PCR anidada o semianidada; PCR-RFLP: PCR asociada al análisis del polimorfismo de longitud de fragmentos de restricción; qPCR: PCR en tiempo real; qPCRm: PCR en tiempo real en formato múltiple.
Marcadores génicos:bg: beta giardina; cowp: proteína de la pared del ooquiste; EF-1a: factor de elongación 1a; gdh: glutamato deshidrogenasa; gp60: glucoproteína de 60 kDa; hsp70: proteína de choque térmico de 70 kDa; ITS: espaciador transcrito interno; LAMP: amplificación isotérmica mediada por bucle; SREHP: proteína de Entamoeba histolytica rica en serina; ssu rADN: ADN codificante del ARN de la subunidad ribosomal pequeña; STR: microsatélites; TNC: transcrito no codificante; tpi: triosa fosfato isomerasa; TRAP: proteína relacionada con la trombospondina adhesiva.
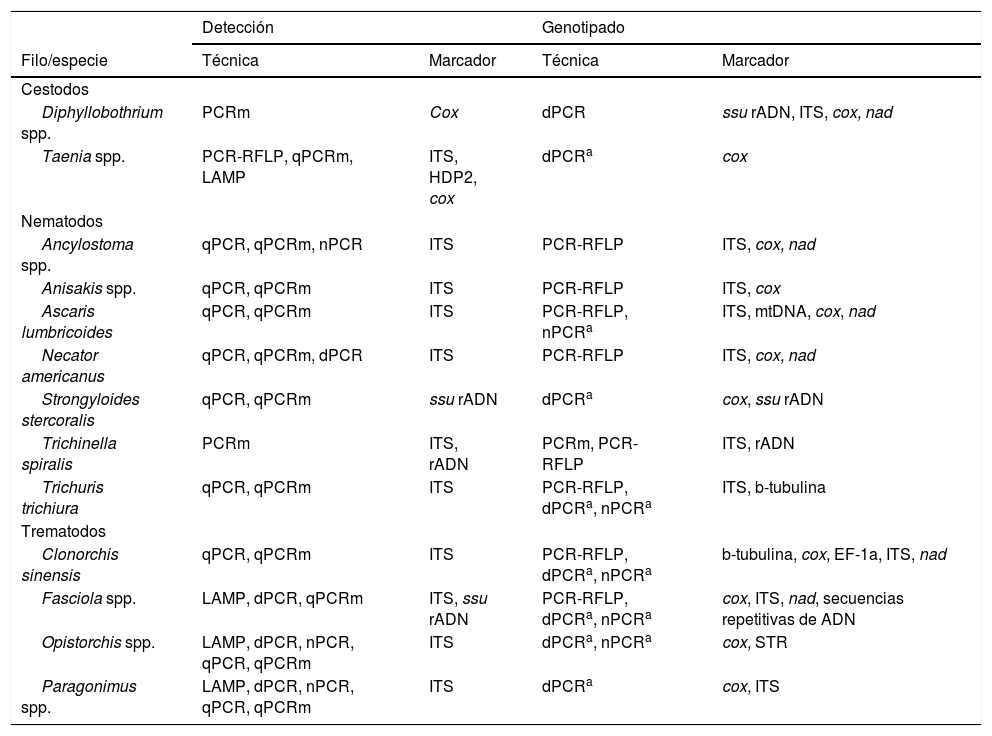
Principales técnicas y marcadores genéticos utilizados para la detección y caracterización molecular de los helmintos parásitos entéricos más relevantes en salud humana.
| Detección | Genotipado | |||
|---|---|---|---|---|
| Filo/especie | Técnica | Marcador | Técnica | Marcador |
| Cestodos | ||||
| Diphyllobothrium spp. | PCRm | Cox | dPCR | ssu rADN, ITS, cox, nad |
| Taenia spp. | PCR-RFLP, qPCRm, LAMP | ITS, HDP2, cox | dPCRa | cox |
| Nematodos | ||||
| Ancylostoma spp. | qPCR, qPCRm, nPCR | ITS | PCR-RFLP | ITS, cox, nad |
| Anisakis spp. | qPCR, qPCRm | ITS | PCR-RFLP | ITS, cox |
| Ascaris lumbricoides | qPCR, qPCRm | ITS | PCR-RFLP, nPCRa | ITS, mtDNA, cox, nad |
| Necator americanus | qPCR, qPCRm, dPCR | ITS | PCR-RFLP | ITS, cox, nad |
| Strongyloides stercoralis | qPCR, qPCRm | ssu rADN | dPCRa | cox, ssu rADN |
| Trichinella spiralis | PCRm | ITS, rADN | PCRm, PCR-RFLP | ITS, rADN |
| Trichuris trichiura | qPCR, qPCRm | ITS | PCR-RFLP, dPCRa, nPCRa | ITS, b-tubulina |
| Trematodos | ||||
| Clonorchis sinensis | qPCR, qPCRm | ITS | PCR-RFLP, dPCRa, nPCRa | b-tubulina, cox, EF-1a, ITS, nad |
| Fasciola spp. | LAMP, dPCR, qPCRm | ITS, ssu rADN | PCR-RFLP, dPCRa, nPCRa | cox, ITS, nad, secuencias repetitivas de ADN |
| Opistorchis spp. | LAMP, dPCR, nPCR, qPCR, qPCRm | ITS | dPCRa, nPCRa | cox, STR |
| Paragonimus spp. | LAMP, dPCR, nPCR, qPCR, qPCRm | ITS | dPCRa | cox, ITS |
Tipos de PCR (reacción en cadena de la polimerasa): dPCR: PCR directa; nPCR: PCR anidada o semianidada; PCR-RFLP: PCR asociada al análisis del polimorfismo de longitud de fragmentos de restricción; qPCR: PCR en tiempo real; qPCRm: PCR en tiempo real en formato múltiple.
Marcadores génicos:cox: citocromo oxidasa; EF-1a: factor de elongación 1a; ITS: espaciador transcrito interno; LAMP: amplificación isotérmica mediada por bucle; nad: NADH deshidrogenasa; ssu rADN: ADN codificante del ARN de la subunidad ribosomal pequeña; STR: microsatélites.
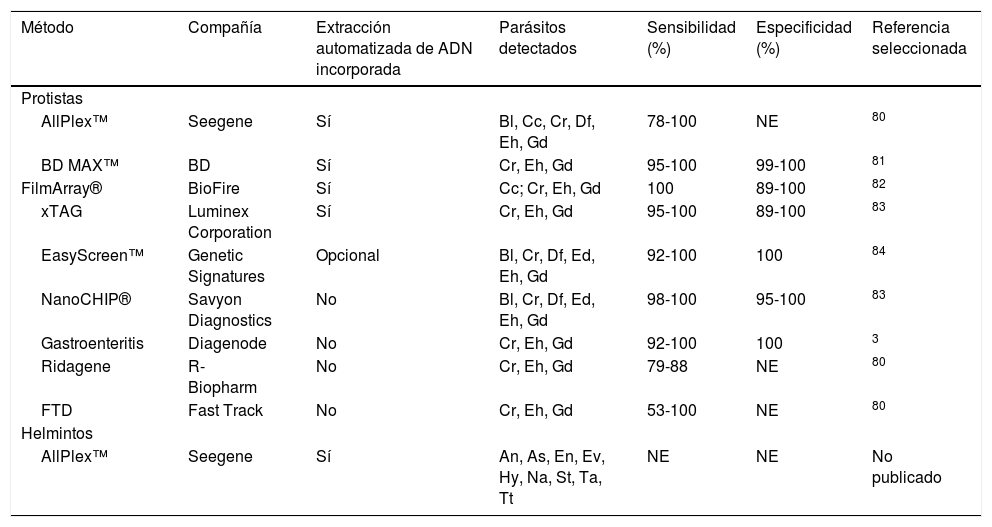
Ensayos de reacción en cadena de la polimerasa (PCR) en tiempo real disponibles comercialmente para la detección simultánea de parásitos entéricos.
| Método | Compañía | Extracción automatizada de ADN incorporada | Parásitos detectados | Sensibilidad (%) | Especificidad (%) | Referencia seleccionada |
|---|---|---|---|---|---|---|
| Protistas | ||||||
| AllPlex™ | Seegene | Sí | Bl, Cc, Cr, Df, Eh, Gd | 78-100 | NE | 80 |
| BD MAX™ | BD | Sí | Cr, Eh, Gd | 95-100 | 99-100 | 81 |
| FilmArray® | BioFire | Sí | Cc; Cr, Eh, Gd | 100 | 89-100 | 82 |
| xTAG | Luminex Corporation | Sí | Cr, Eh, Gd | 95-100 | 89-100 | 83 |
| EasyScreen™ | Genetic Signatures | Opcional | Bl, Cr, Df, Ed, Eh, Gd | 92-100 | 100 | 84 |
| NanoCHIP® | Savyon Diagnostics | No | Bl, Cr, Df, Ed, Eh, Gd | 98-100 | 95-100 | 83 |
| Gastroenteritis | Diagenode | No | Cr, Eh, Gd | 92-100 | 100 | 3 |
| Ridagene | R-Biopharm | No | Cr, Eh, Gd | 79-88 | NE | 80 |
| FTD | Fast Track | No | Cr, Eh, Gd | 53-100 | NE | 80 |
| Helmintos | ||||||
| AllPlex™ | Seegene | Sí | An, As, En, Ev, Hy, Na, St, Ta, Tt | NE | NE | No publicado |
An: Ancylostoma spp.; As: Ascaris spp.; Bl: Blastocystis; Cc: Cyclospora cayetanensis; Cr: Cryptosporidium; Df: Dientamoeba fragilis; Ed: Entamoeba dispar; Eh: Entamoeba histolytica; En: Enterocytozoon spp./Encephalitozoon spp.; Ev: Enterobius vermicularis; Gd: Giardia duodenalis; Hy: Hymenolepis spp.; Na: Necator americanus; St: Strongyloides spp.; Ta: Taenia spp.; Tt: Trichiuris trichiura; NE: no evaluado.
Tabla adaptada de referencia 17.
Existe una gama creciente de protocolos de qPCR disponibles comercialmente para la detección de patógenos entéricos, con un claro interés en el desarrollo de ensayos para el diagnóstico simultáneo (sindrómico) de agentes causantes de enfermedad gastrointestinal de origen viral, bacteriano o parasitario17,24. Aunque con cierto retraso comparativo, la misma tendencia empieza a observarse también en el caso de geohelmintos (tabla 3). Algunos de estos métodos han recibido la aprobación de agencias de salud pública en varios países y se están incorporando progresivamente en los algoritmos diagnósticos de muchos laboratorios clínicos, particularmente los que atienden a grandes poblaciones pediátricas, pacientes inmunocomprometidos o inmigrantes y viajeros procedentes de áreas endémicas29.
La elección del marcador génico a amplificar es también un factor clave en el diseño de PCR y se debe tener en cuenta en función del uso al que se quiera destinar el ensayo. Así, el gen codificante del ARN de la subunidad ribosomal pequeña (ssu rADN), incluyendo regiones del espaciador transcrito interno (ITS), es el marcador de elección en las PCR para el diagnóstico de protistas y helmintos entéricos (tablas 1 y 2). Al ser ssu rADN (e ITS) un gen multicopia, existen multitud de dianas disponibles en la reacción inicial de amplificación, lo que aumenta considerablemente la sensibilidad del ensayo. Por el mismo motivo, ambos marcadores se usan frecuentemente en PCR de cribado en estudios epidemiológicos con gran número de muestras. El principal inconveniente de ssu rADN e ITS es que sus secuencias nucleotídicas están relativamente bien conservadas entre especies evolutivamente cercanas, por lo que pueden generar amplificaciones inespecíficas (falsos positivos). Este problema se puede detectar secuenciando el producto de amplificación obtenido y confirmando la especie de origen.
Para el genotipado de muestras ya identificadas como positivas al parásito de interés, el enfoque debe ser diferente, se necesitan marcadores moleculares que muestren una variabilidad en su secuencia nucleotídica lo suficientemente amplia como para permitir la diferenciación entre distintos genotipos y subgenotipos. El principal inconveniente de este tipo de marcadores es que suelen ser genes de copia única, por lo que la sensibilidad de las PCR que los utilizan no es elevada. Esto explica que un porcentaje considerable de muestras positivas por ssu-PCR o ITS-PCR no se puedan amplificar en las PCR de genotipado correspondientes.
4Infecciones por protistasExisten más de 15 géneros de protistas capaces de infectar el tracto digestivo humano. Los patógenos de mayor relevancia en salud pública se distribuyen en seis filos incluyendo Amebozoa, Metamonada, Apicomplexa, Ciliophora, Microsporidia y Stramenopiles, todos ellos transmitidos principalmente por la ruta fecal-oral. Como se ha indicado anteriormente, la detección de este grupo de patógenos mediante PCR está casi exclusivamente limitada a los marcadores ssu rADN e ITS (tabla 1).
Entre los amebozoos, E. histolytica es la única especie que causa enfermedad en humanos. Estudios recientes han demostrado que solamente un número limitado de infecciones por E. histolytica progresa hasta causar síntomas clínicos, y existen diferencias moleculares en las cepas aisladas de individuos sintomáticos y asintomáticos. Estas diferencias se han estudiado en secuencias de los genes codificantes de las proteínas quitinasa y SREHP, en transcritos no codificantes y en microsatélites (tabla 1)30. Dentro del filo Metamonada, D. fragilis es un protozoo de patogenicidad incierta, cuya diversidad genética parece estar limitada a tan solo 2 genotipos (1 y 2) reconocidos del parásito. Estas variantes se han identificado usando marcadores bien conservados (ssu rADN, ITS) y genes constitutivos incluyendo actina y EF-1a, entre otros (tabla 1)31. En cuanto a G. duodenalis, se han descrito 8 genotipos (A-H), de los cuales A y B son los que mayoritariamente causan giardiasis en humanos. La diferenciación de variantes inter e intragenotípicas se ha basado en el análisis de genes codificantes de proteínas enzimáticas (EF-1a, ferredoxina, gdh, tpi), estructurales (actina, a-tubulina, bg), nucleosomales (histonas) o chaperoninas (hsp60), entre otras32.
Entre los apicomplejos, Cryptosporidium spp. es el género mejor estudiado molecularmente. De las 15 especies de Cryptosporidium capaces de infectar humanos, C. hominis y C. parvum son responsables del 90% de los casos reportados globalmente. La diversidad molecular intraespecie de Cryptosporidium se ha investigado a partir de varios marcadores (tabla 1), pero es sin duda la glucoproteína de 60 kDa (gp60) el que se utiliza más comúnmente debido a su alta variabilidad nucleotídica. Sobre la base de las secuencias de gp60 se han identificado al menos 10 (Ia-Ik) familias subgenotípicas en C. hominis y 16 (IIa-IIp) en C. parvum33. Aunque ampliamente utilizado, gp60 presenta problemas de discriminación en algunas familias, como la Ib de C. hominis34. La epidemiología molecular de otras especies pertenecientes al filo Apicomplexa, incluyendo Cyclospora cayetanensis,Cystoisospora belli y Sarcocystis spp., es mucho menos conocida y los pocos datos disponibles son los obtenidos a partir de marcadores relativamente conservados como ssu rADN e ITS (tabla 1). Por lo tanto, hay una necesidad de desarrollar y validar marcadores genotípicos para estos patógenos. Entre los candidatos evaluados se encuentran algunas regiones mitocondriales de C. cayetanensis35, o los genes codificantes de las subunidades de la enzima citocromo oxidasa y de la ARN polimerasa de Sarcocystis spp.36.
Neobalantidium coli es el único protozoo ciliado que infecta humanos, entre otros animales homeotermos37. Debido a que su principal reservorio son los suinos, la balantidiosis se considera una enfermedad ocupacional que afecta principalmente a personas en estrecho contacto con estos animales o sus excrementos. A nivel molecular N. coli es una especie genéticamente muy conservada, al menos en los marcadores ssu rADN e ITS. Los estudios orientados a la identificación de posibles genotipos o subgenotipos del parásito son muy limitados38.
E. bieneusi es el microsporidio que con mayor frecuencia produce microsporidiosis, afecta principalmente a individuos inmunodeprimidos, pero también a inmunocompetentes. Actualmente, la identificación y caracterización de E. bieneusi está basada en la amplificación y secuenciación de la región ITS, a partir de la cual se han podido identificar más de 250 genotipos, con una amplia diversidad genética entre ellos39. Estos genotipos se han dividido en 9 grupos, y los grupos 1 y 2 contienen los genotipos infectivos para el hombre y/o con potencial zoonósico40.
Blastocystis sp. se considera el protista más frecuente en heces humanas. Aunque su patogenicidad es cuestionada por algunos autores, hay estudios que relacionan este protista con diversos trastornos intestinales (incluyendo síndrome de colon irritable) y urticaria. Blastocystis sp. posee una gran diversidad genética que ha permitido la identificación de al menos 17 subtipos, de los cuales ST1-9 y ST12 se han descrito en humanos41. Al igual que en el caso de E. bieneusi, el método más extendido para detectar y genotipar (incluso a nivel de alelo) las variantes genéticas de Blastocystis es el análisis de secuencias ITS42.
5Infecciones por geohelmintosExisten más de 340 especies de helmintos asociadas a humanos, la mayoría son zoonosis poco frecuentes, destacan A. lumbricoides, A. duodenale, N. americanus y S. stercoralis (clase Chromadorea) y T. trichiura (clase Enoplea) como principales especies causantes de las geohelmintiasis en el ser humano43. El mecanismo de transmisión es a través de la ingesta de huevos infectantes o por penetración a través de la piel de larvas infectivas. Existen diferentes métodos de diagnóstico basado en PCR, y destacan la qPCR dirigida a la amplificación de ADN ribosomal, la secuencias ITS y el ADN mitocondrial de estos parásitos (tabla 2). A lo largo de los últimos años ha cobrado gran importancia la técnica qPCR en formato múltiple utilizando sondas de hidrólisis (TaqMan)44.
Ascaris lumbricoides es el geohelminto más prevalente y se ubica en el intestino delgado de su hospedador. Se dispone de numerosas técnicas moleculares para la detección de este parásito, la mayoría dirigidas a la amplificación de la región ITS en formato múltiple utilizando sondas TaqMan44,45, o combinadas con la plataforma Luminex®46. Se han realizado estudios de genotipado de ITS y genes mitocondriales para estudiar la epidemiología de Ascaris spp. y evaluar su posible potencial zoonósico. Estos estudios han revelado que existen haplotipos comunes para A. suum y A. lumbricoides que generan numerosos híbridos, por lo que algunos autores proponen agrupar a las 2 especies en una única47.
Trichuris trichiura es la principal especie causante de trichuriasis en el ser humano y generalmente se localiza en el ciego. Los protocolos de detección molecular son escasos debido a la dificultad de extracción del ADN parasitario por la robustez de los huevos48. Destacan 2 qPCR dirigidas a la amplificación de un fragmento del gen ITS, una en formato simple49 y otra múltiple, en las que además se incluye la detección de otros geohelmintos44. Gracias a la secuenciación de un fragmento del gen ITS se ha podido describir que el ser humano se puede infectar por la especie Trichuris suis, específica del ganado porcino50. Morfológicamente esta especie es indistinguible de T. trichiura, pero se han llevado a cabo análisis de los genes ssu rADN e ITS que muestran que son especies genéticamente diferentes, aunque estrechamente relacionadas51,52.
Ancylostoma duodenale y N. americanus son los principales parásitos causantes de la uncinariasis. Se caracterizan por la anemia grave que producen en el hospedador, y llegan a ingerir grandes cantidades de sangre de este53. Los huevos producidos por ambos parásitos son muy lábiles e indistinguibles por microscopía, por lo que la aplicación de técnicas moleculares está muy extendida, siendo el principal marcador el ITS tanto en formato simple49,54 como en formato múltiple44,55,56. La diversidad genética de las especies de uncinarias se ha estudiado principalmente mediante la secuenciación de los marcadores ITS y cox (tabla 2), que proporciona puntos determinantes a la hora de abordar las estrategias de control, así como la resistencia a fármacos y el desarrollo de posibles vacunas57.
Strongyloides stercoralis y S. fuelleborni son las 2 únicas especies descritas que causan la estrongiloidiasis humana. Este parásito, a diferencia del resto de los geohelmintos, tiene la peculiaridad de que puede producir ciclos de autoinfección, lo que favorece la persistencia de la enfermedad durante décadas. Además, principalmente en individuos inmunodeprimidos, puede desarrollarse el síndrome de hiperinfección e infección diseminada58. La dificultad del diagnóstico parasitológico ha llevado a un importante desarrollo de los protocolos moleculares, y la diana más frecuente es el ssu rADN amplificada mediante qPCR en formatos simple59–61 y múltiple44,45. Los estudios de genotipado son escasos y los que mayor información proporcionan son los realizados a partir de larvas del nematodo62.
6Infecciones por nematodos transmitidos por alimentosEntre las infecciones por nematodos transmitidas por alimentos destaca la anisakidosis causada por los géneros Anisakis spp., Pseudoterranova spp., Hysterothylacium spp. y Contracaecum spp. Dentro del género Anisakis spp. Se han reportado 3 especies como causa de la anisakiasis en humanos: Anisakis simplex sensu stricto, A. pegreffii y A. physeteris63, las dos primeras especies son las de mayor importancia zoonósica64. A pesar del extenso uso del diagnóstico serológico, en los últimos años se han desarrollado ensayos moleculares de detección múltiple que permiten identificar además los otros géneros de anisákidos65. Los estudios de secuenciación del gen cox han mostrado que es altamente polimórfico en Anisakis spp. y, por lo tanto, es particularmente útil para identificar las especies involucradas en la producción de híbridos (tabla 2)10.
La triquinelosis es una zoonosis de distribución cosmopolita causada por nematodos del género Trichinella. Actualmente se han descrito 9 especies y 3 genotipos, en el ser humano la especie más frecuente es T. spiralis. A nivel diagnóstico, las técnicas serológicas son las más empleadas, pero la identificación de especie, imprescindible en la investigación y gestión de brotes, únicamente puede realizarse con la utilización de técnicas moleculares. En este sentido destaca el desarrollo de una PCR multiplex basada en la amplificación de un fragmento del gen ITS y de una región del segmento V del rADN, que permite la detección y discriminación de 7 especies (T. spiralis, T. nativa, T. britovi, T. pseudospiralis, T. murrelli, T. nelsoni y T. papua) y 2 genotipos (T6 y T11) (tabla 2)11.
7Infecciones por cestodos transmitidos por alimentosLas principales especies de cestodos que afectan al ser humano son Taenia solium, T. saginata y Diphyllobothrium latum. Las infecciones por estos patógenos se adquieren por el consumo de carne o pescado crudo o poco cocinado que contiene los estadios larvarios (p. ej., cisticercos) de estos parásitos. El riesgo de desarrollar cisticercosis que conlleva la infección por T. solium en el hombre hace imprescindible disponer de herramientas diagnósticas que permitan la diferenciación entre especies, ya que morfológicamente los huevos de ténidos son indistinguibles mediante microscopía. Existe una tercera especie (T. asiatica) que también causa teniasis en el ser humano pero, a diferencia de las anteriores, está geográficamente restringida a países del sudeste asiático. Para la detección de las diferentes especies de ténidos se dispone de numerosas técnicas moleculares, entre las que se encuentran la PCR convencional, la PCR-RFLP, la qPCR y la LAMP, y las más desarrolladas son las de formato múltiple, que permiten diferenciar entre las diferentes especies (tabla 2).
Existen protocolos de PCR para el diagnóstico diferencial de T. solium y T. saginata basados en la amplificación del fragmento HDP2 a partir de ADN procedente tanto de proglótides como de huevos en muestras de heces24. Además, se ha desarrollado, a partir del ADN obtenido de muestras de heces, un ensayo qPCR que amplifica un fragmento del marcador ITS y gracias a la utilización de sondas de hidrólisis TaqMan permite identificar las diferentes especies reportando altos valores de sensibilidad y especificidad66. Sin embargo, actualmente es más frecuente la aplicación de los protocolos de PCR múltiple destinados a la detección de las 3 especies (T. solium, T. saginata y T. asiatica) con la amplificación de un fragmento del gen cox (tabla 2)67–69. También hay protocolos de PCR-RFLP que amplifican genes de proteínas mitocondriales a partir únicamente de proglótides, por lo que su uso está limitado para las muestras de heces70,71. Por último, se han propuesto protocolos basados en LAMP para la detección simultánea de las 3 especies, que se basan en la amplificación del gen cox72.
Dentro del género Diphyllobothrium existen 14 especies que son capaces de causar difilobotrioisis en humanos. La especie más común es D. latum, aunque cada vez es más frecuente la descripción de casos por las especies D. pacificum, D. denditricum y D. nihonkaiense73. A pesar de que la clínica y el tratamiento son similares, es importante conocer la especie para tener una mayor información epidemiológica. Por este motivo, para su detección molecular se ha desarrollado una PCR multiplex capaz de diferenciar estas especies mediante la amplificación de un fragmento del gen cox (tabla 2)73. No obstante, también existen protocolos que permiten la identificación de otras especies diferentes mediante la amplificación y secuenciación de regiones de los genes ssu rADN, ITS, cox y nad (tabla 2)74,75.
8Infecciones por trematodos transmitidos por alimentosAl menos 80 especies de trematodos, muchas de ellas zoonósicas, se pueden transmitir por alimentos crudos o poco cocinados que contienen las formas infectivas (metacercarias) de estos patógenos24. Su epidemiología y diversidad molecular son poco conocidas, por lo que no existen protocolos estandarizados para el genotipado de estos parásitos. Las infecciones por Opistorchis spp. (principalmente O. viverrini y O. felineus) son relativamente comunes en áreas endémicas del sudeste asiático. Estudios recientes mediante electroforesis de enzimas multilocus y análisis de microsatélites, han demostrado que O. viverrini es en realidad una especie críptica constituida por 2 especies morfológicamente muy similares pero molecularmente diferentes que presentan variaciones genéticas entre diferentes poblaciones76. Los estudios de secuenciación multilocus llevados a cabo con C. sinensis, una especie emparentada evolutivamente con el género Opistorchis, también indican un elevado grado de diversidad en las secuencias analizadas que probablemente reflejan los patrones de distribución geográfica del parásito77. Estos estudios coinciden en indicar que ITS es el marcador más fiable para el estudio de la epidemiología molecular de C. sinensis (tabla 2).
Se dispone de algo más de información en el caso de las infecciones causadas por Fasciola spp., donde la detección, diferenciación (F. hepatica frente a F. gigantica) y genotipado de aislados del parásito se han investigado en un número significativo de marcadores moleculares, entre los que destacan ITS y diversos genes mitocondriales (tabla 2). Los estudios que se han realizado hasta la fecha parecen indicar que Fasciola es un género relativamente bien conservado evolutivamente, con una limitada diversidad genética, al menos en los marcadores analizados78. Finalmente, la detección de infecciones por especies del género Paragonimus está restringida al uso del marcador ITS, y apenas hay estudios sobre la variabilidad molecular de este trematodo. Uno de los pocos que hay, basado en el análisis de secuencias de ITS y varios genes mitocondriales, apunta a la existencia de 3 genotipos diferentes dentro de P. westermani79.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.