Introducción
La determinación de la sensibilidad a los antimicrobianos es una de las tareas del laboratorio de microbiología clínica de mayor impacto en el manejo del paciente. Además, los estudios de sensibilidad son fundamentales para conocer las tendencias de resistencia en los microorganismos de importancia clínica y para definir la política de utilización de antimicrobianos. Estos estudios, por otra parte, suponen una de las principales cargas financieras en el laboratorio. Por todo ello, muchos de los cambios ocurridos durante las últimas décadas en la microbiología clínica están relacionados con los estudios in vitro de sensibilidad a los antimicrobianos.
La mayoría de los comentarios que siguen se refieren sólo a los antibacterianos, pero podrían aplicarse también al estudio de los antifúngicos, antivirales y antiparasitarios, que no se considerarán específicamente.
Parámetros para evaluar la actividad in vitro de los antimicrobianos
La categoría clínica de "resistencia" es más fiable para predecir el fracaso terapéutico que la categoría clínica de "sensibilidad" para predecir el éxito, por lo que la falsa sensibilidad tiene peores consecuencias para el enfermo que la falsa resistencia. Por ello, en la actualidad se está poniendo especial énfasis en el desarrollo de métodos que permiten detectar la existencia de mecanismos de resistencia1.
Aún se debate si para establecer las categorías clínicas de sensible, intermedio o resistente basta con la información proporcionada con el método de difusión con disco o si es necesario determinar el valor de la concentración inhibitoria mínima (CIM), y en este último caso, si dicho valor debe determinarse siempre o sólo en algunos casos. Hay varios elementos que aconsejarían determinar el parámetro cuantitativo de CIM:
1. Varios estudios han demostrado una correlación entre fracaso terapéutico y valor de CIM. En uno de ellos2 la tasa de fracaso terapéutico con cefoperazona fue del 4% cuando la CIM era inferior a 1 mg/l, entre 8 y 16% cuando era de 2-16 mg/l y entre 32 y 64% cuando la CIM era 32 mg/l. En otro estudio3 se comprobó una relación directa entre riesgo de fracaso del tratamiento con cefotaxima y un menor diámetro de halo en la prueba de difusión con disco. Las cepas implicadas en casos de fracaso clínico requieren CIM mayores que las cepas en las que el tratamiento tiene éxito: en un estudio sobre 50 pacientes con infecciones por bacterias gramnegativas el valor medio de la CIM de ciprofloxacino para las cepas de los pacientes en los que se logró el éxito terapéutico fue de 0,08 mg/l, mientras que la CIM media para las cepas en las que hubo fracaso terapéutico fue de 0,58 mg/l4.
2. Los estudios farmacodinámicos de la última década han demostrado que la determinación del valor de la CIM tiene importancia terapéutica porque las proporciones entre área bajo la curva/CIM, pico máximo/CIM y tiempo sobre la CIM ayudan a establecer pautas de dosificación más eficaces5.
3. El seguimiento de la evolución de los valores de CIM frente a una determinada especie bacteriana es de interés desde el punto de vista epidemiológico, porque ayuda a observar las tendencias de resistencia y a reconocer la aparición de cepas con mecanismos de bajo nivel de resistencia.
Estandarización
La fiabilidad de los estudios de sensibilidad in vitro exige la estandarización de los métodos empleados. En la actualidad se han desarrollado normas para la preparación de los medios de cultivo empleados en las pruebas de sensibilidad6, para la realización de técnicas de difusión con disco7 y de dilución en caldo y agar8, y para la interpretación de los resultados obtenidos con estas técnicas9-12. Para la mayoría de las bacterias de importancia clínica los resultados obtenidos en distintos laboratorios que sigan los mismos protocolos estandarizados debieran ser reproducibles y comparables. No existen, sin embargo, estándares para el método de difusión en gradiente (Etest) ni para la aplicación de métodos moleculares.
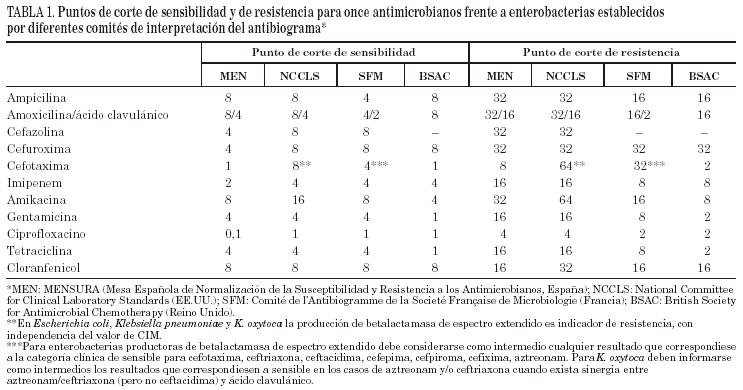
El organismo con mayor influencia en la aplicación de criterios de estandarización es el National Committee for Clinical Laboratory Standards, de Estados Unidos. Sin embargo, existen otros muchos comités y agencias, fundamentalmente en Europa y con frecuencia de carácter nacional, que han venido contribuyendo al desarrollo paralelo de programas de estandarización de las técnicas de estudio de sensibilidad y, sobre todo, de la interpretación de los resultados obtenidos10-12. A modo de ejemplo, en la tabla 1 se recogen las variaciones en los puntos de corte propuestos por varios comités para la interpretación de la sensibilidad de las enterobacterias. Las diferencias entre distintos comités no sólo tienen un componente académico, sino que suponen, en ocasiones, serios problemas para estimar la importancia de la resistencia en distintos países. La Sociedad Europea de Microbiología Clínica y Enfermedades Infecciosas, a través del EUCAST (que representa sociedades científicas de la mayoría de los países europeos), ha publicado recientemente varios documentos sobre terminología, metodología de los estudios de sensibilidad in vitro, y sobre el establecimiento de puntos de corte de sensibilidad y resistencia para algunos nuevos antimicrobianos13,14.
Quizá las dos diferencias más relevantes entre los comités europeos (sobre todo MENSURA y CA-SFM) y el National Committee for Clinical Laboratory Standards (NCCLS) han sido la definición de puntos de corte en función del tipo de microorganismo (que finalmente ha recogido el NCCLS en sus últimos suplementos) y la definición de puntos de corte de resistencia basados en mecanismos de resistencia, más que en la CIM. Este último aspecto ha tenido una claro exponente en el caso de la producción de betalactamasas de espectro extendido, cuya existencia no fue considerada de importancia clínica por el NCCLS, con independencia de valores de CIM, hasta 1999.
Se ha avanzado poco en el establecimiento de puntos de corte en función del cuadro clínico, probablemente por la escasa información de la que se dispone. Las recientes modificaciones de los criterios de interpretación de los resultados de antibiograma para las neumonías y las meningitis producidas por Streptococcus pneumoniae9 podrían significar un primer paso en esta dirección.
Sería necesario desarrollar criterios estandarizados para medir de forma objetiva el impacto (clínico, social, económico, etc.) del tratamiento antimicrobiano. Ello podría realizarse definiendo una escala basada en el estudio protocolizado del tratamiento antimicrobiano en entidades clínicas como endocarditis, bacteriemia en neutropénicos o meningitis, en las que se evalúe de manera cuantitativa la progresión temporal de signos, síntomas y exploraciones complementarias15.
Aspectos metodológicos
Técnica de difusión en gradiente (Etest)
La técnica de difusión en gradiente (Etest) ha permitido solventar la limitación de la técnicas de difusión con disco para obtener directamente valores de CIM. Infinidad de estudios han demostrado la fiabilidad y la reproducibilidad del Etest, que ofrece, en general, resultados similares a los obtenidos con un método estandarizado de dilución16. Por desgracia, el precio de las tiras limita su uso habitual en el laboratorio clínico, por lo que, en la práctica, la mayoría de los laboratorios disponen del Etest como alternativa para la comprobación de los resultados obtenidos con otras pruebas, o para estudio de especies (sobre todo bacterias de crecimiento difícil) para las que suele evaluarse un número limitado de antimicrobianos.
Pruebas de sensibilidad para bacterias de crecimiento difícil
Una de las principales limitaciones de los métodos estandarizados para bacterias de crecimiento rápido es la dificultad para su aplicación a microorganismos de crecimiento difícil, o que no se pueden cultivar. Aunque en los últimos años se han logrado notables avances en la automatización del estudio de la sensibilidad de S. pneumoniae y Haemophilus influenzae17, aún deben desarrollarse técnicas automatizadas para Neisseria, Mycobacterium, o bacterias anaerobias, por citar algunos ejemplos. Otras veces no existen metodologías estandarizadas que permitan establecer las condiciones ideales para el antibiograma, como ocurre, por ejemplo, en bacterias corineformes, Campylobacter, Mycoplasma, Chlamydia, etc.
Es de esperar que la aplicación de las técnicas moleculares a este tipo de microorganismos (v. posteriormente) aportará importantes beneficios desde el punto de vista clínico.
Métodos automáticos
Los principales objetivos actuales en esta área incluyen desarrollar sistemas con alto nivel de automatización y conseguir métodos que permitan generar resultados de antibiograma en el mismo día de incubación de los paneles.
Las innovaciones introducidas en microprocesadores y robótica, junto con ligeras modificaciones en la metodología empleada (incremento del inóculo, modificación de la concentración de antimicrobiano, elección de medios de cultivo alternativos, uso de compuestos fluorescentes en el caldo de cultivo, etc.) han permitido diseñar sistemas que ofrecen datos de sensibilidad a los antimicrobianos en menos de 4 h. Existen ya varias opciones disponibles, incluyendo los sistemas VITEK 2 System (bioMérieux-Vitek, Hazelwood, EE.UU.), WalkAway System (Dade Behring Inc., EE.UU.), Phoenix Automated Microbiology System (BD Biosciences EE.UU.), o Sensititre ARIS18.
El empleo de paneles comerciales con antimicrobianos deshidratados o liofilizados (en lugar de congelados) ha facilitado su almacenamiento, pero siguen sin resolverse los problemas planteados por antimicrobianos poco estables (carbapenemas, ácido clavulánico), cuya inactivación durante el proceso de producción del panel o a lo largo de su almacenamiento pueden determinar falsos resultados de resistencia19,20.
Antes de su aplicación en el laboratorio clínico sería conveniente desarrollar programas de control encaminados a verificar la fiabilidad de los resultados obtenidos con estos sistemas. Hasta el momento sólo se exigen estos estudios de premarketing en Estados Unidos, bajo control de la Food and Drug Administration (FDA).
Los programas de evaluación deberían incluir cepas de referencia, microorganismos con mecanismos de resistencia conocidos y cepas clínicas aisladas recientemente. Debe esperarse que con estos sistemas automáticos de antibiograma de incubación corta los porcentajes de falsa sensibilidad sean inferior o igual a 1,5% y los de falsa resistencia inferior o igual a 3%, y que el acuerdo esencial (en ± 1 dilución con respecto al método de referencia) sea superior o igual al 90,0%.
Sistemas digitales de lectura automatizada
Un área en expansión creciente en el estudio automatizado de sensibilidad a los antimicrobianos es el uso de sistemas de vídeo o escáner que realizan análisis digitales de placas con discos de antimicrobianos o de paneles de microdilución. Mediante programas informáticos adicionales las imágenes se traducen en valores de diámetros de halos de inhibición o de CIM y, finalmente, en categorías clínicas.
El sistema WIDER (Soria Melguizo SA, Madrid) permite la lectura tanto de halos de inhibición en placas como paneles de microdilución, incluyendo paneles específicamente diseñados para S. pneumoniae y H. influenzae17. Otros sistemas son: BIOMIC VIDEO (Giles Scientifis, Inc, Nueva York, EE.UU.), (Berke I, JCM), AccuZone system (AccuMed International, Inc., West Lake, EE.UU.), Aura Image (Oxoid, Reino Unido), Mastascan Elite (Mast, Reino Unido), SIRSCAN (SIRSCAN, Montpellier, Francia) y OSIRIS (Bio-Rad, Marnes la Coquette, Fancia)18.
Ocasionalmente, los halos leídos con estos sistemas difieren de manera significativa de los obtenidos por un observador a simple vista, y aunque ello no siempre implica cambios de categoría clínica, sí puede tener relevancia en determinados casos (p. ej., detección de resistencia a meticilina en Staphylococcus). Otra importante limitación es la frecuente dificultad de analizar resultados para Enterococcus u otros microorganismos con crecimiento tenue, o para diferenciar zonas de crecimiento marginal en la periferia de los halos de inhibición.
Métodos moleculares
El uso de métodos moleculares para el estudio de la actividad de los antimicrobianos está orientado, esencialmente, hacia el estudio de genes que determinan mecanismos de resistencia.
Las principales ventajas potenciales del empleo de estos métodos podrían resumirse en21:
1. Detección del gen de resistencia en la propia muestra clínica, lo que permite estudiar microorganismos no cultivables o de crecimiento lento.
2. En el caso de mecanismos de bajo nivel de resistencia con trascendencia clínica, la detección de genes de resistencia es, en ocasiones, más fiable que la detección del fenotipo de resistencia.
3. Suponen un menor riesgo biológico que los métodos que requieren cultivo.
Estos métodos también plantean importantes inconvenientes, como:
1. Baja sensibilidad, si el inóculo inicial es bajo, y baja especificidad, si la detección de un gen no implica resistencia de interés clínico.
2. Para cada antimicrobiano se requiere, al menos, una prueba, y debido al carácter multifactorial de la resistencia, pueden necesitarse múltiples pruebas para la detección de los distintos mecanismos de resistencia frente a un antimicrobiano concreto.
3. Algunos fenotipos de resistencia pueden depender de mecanismos aún no conocidos.
4. Podrían ocurrir casos de falsos positivos como consecuencia de la contaminación cruzada entre muestras, sobre todo cuando se emplean técnicas de amplificación de ácidos nucleicos (reacción en cadena de la polimerasa, PCR).
5. Estos métodos aún no están estandarizados.
Los métodos moleculares para el estudio de la resistencia a los antimicrobianos incluyen la detección de genes de resistencia mediante sondas o técnicas de amplificación (PCR, "branched DNA", etc.), la detección de mutaciones en genes previamente amplificados (polimorfismo de fragmentos de restricción, polimorfismo conformacional de cadena sencilla, PCR en tiempo real, secuenciación, etc.) o la aplicación de microchips de ADN. Pueden encontrarse detalles específicos en la revisión recientemente publicada por Cockerill22.
Esta metodología se emplea sistemáticamente en laboratorios de investigación, y algunas técnicas también se están usando ya de forma habitual en algunos laboratorios de microbiología clínica. Es posible que el futuro desarrollo de microchips de ADN favorezca el empleo de los métodos moleculares en el laboratorio de microbiología clínica, pero no parece que ello vaya a ocurrir a corto plazo.
Muchos de los problemas inherentes a esta metodología aún no están resueltos, en especial los derivados de las condiciones técnicas del ensayo y la interpretación de los resultados. Sirvan a título orientativo las discrepancias observadas por 2 grupos de investigación en la aplicación de chips de E. coli del mismo origen comercial: al estudiar la expresión de genes regulados por MarA (relacionado con la resistencia múltiple a los antimicrobianos), un grupo observó la modulación de 88 genes23 y el otro de sólo 6224, y sólo 21 de estos genes coincidían en ambos estudios.
Los chips de ADN suelen contener múltiples (incluso miles) fragmentos de cADN depositados de forma precisa mediante técnicas de microdispensación, o múltiples oligonucleótidos (50-1.000 bases de longitud) sintetizados in situ mediante un proceso fotolitográfico. Obviamente, con el mismo dispositivo pueden obtenerse, a la vez, la identificación y el perfil de resistencia de un microorganismo, lo que hace más atractiva la opción de su empleo en el laboratorio clínico.
La técnica de amplificación por desplazamiento de hebra permite la detección de cantidades mínimas de ADN (bastan unas 100 copias por muestra), y se ha usado con éxito en la detección de mecA25 y en la caracterización de Campylobacter jejuni resistentes a quinolonas.
Otros métodos
Se ha descrito un método rápido de microdilución en agar empleando portaobjetos con una delgada lámina de medio de cultivo que permite determinar la CIM mediante lectura microscópica en tan sólo 4 h26. Este método, sin embargo, no se ha popularizado hasta ahora. El método de gradiente en espiral tampoco ha alcanzado una gran implantación en laboratorios de microbiología clínica, aunque sí constituye una herramienta eficaz en algunos laboratorios de investigación27.
Otras técnicas fenotípicas de determinación de la sensibilidad incluyen:
1. Incorporación de azul de tetrazolio o de hemolisina en el medio de cultivo para diferenciar bacterias viables y no viables, y favorecer la interpretación del punto final de crecimiento en tan sólo 4 h.
2. Aplicación de las técnicas de citometría de flujo28.
3. Empleo de bacteriófagos con un gen marcador de luciferasa que al atacar sólo las bacterias viables traducen el crecimiento bacteriano en una señal luminosa.
4. Medición de los niveles de ATP como marcador del estado metabólico bacteriano.
5. Medición de la cantidad de ácidos micólicos en un cultivo bacteriano empleando cromatografía líquida de alta presión.
6. Uso de sustratos fluorescentes para medir el crecimiento bacteriano por espectrofluorometría.
La existencia de genes de virulencia que sirven de dianas de nuevos antimicrobianos29 exige la puesta a punto de nuevas metodologías para el estudio de la sensibilidad in vitro. Puesto que estos genes se expresan preferentemente in vivo, las técnicas fenotípicas actuales basadas en el crecimiento in vitro del microorganismo resultan, por definición, poco útiles. El uso modelos con cultivos celulares que permitan examinar la interferencia del antimicrobiano en los procesos de adherencia, colonización e invasión o de métodos moleculares son alternativas adecuadas en este campo.
Determinación del poder bactericida y de sinergia
La utilidad clínica del poder bactericida del suero ha quedado claramente establecida en la endocarditis infecciosa y se ha recomendado su determinación para la monitorización terapéutica en osteomielitis y artritis infecciosas30. Es un método más útil que el estudio de sinergia para evaluar el tratamiento de pacientes inmunodeprimidos en los que se empleen combinaciones de antimicrobianos30. Sería conveniente estudiar la utilidad de esta técnica en otros cuadros graves como la meningitis, o como indicador para iniciar tratamientos orales tras el uso de pautas parenterales. La estandarización de la prueba31 ayudará a definir su utilidad.
Las indicaciones de los estudios de sinergia para orientar el tratamiento en un paciente concreto son difíciles de precisar.
El empleo de combinaciones para evitar la aparición de resistencias es una práctica bien fundamentada en la tuberculosis, pero en otros muchos casos (combinaciones de betalactámicos, fluoroquinolonas y aminoglucósidos en infecciones por gramnegativos) pueden encontrarse en la literatura tanto datos a favor de su uso como en contra del mismo.
La aparición de antimicrobianos con muy amplio espectro ha limitado las indicaciones para aumentar el espectro de tratamiento. Hay también contadas circunstancias en las que es conveniente combinar antimicrobianos para disminuir su efecto tóxico, y en este caso no hay necesidad clara de evaluar previamente la combinación in vitro.
Finalmente, aunque existen indicaciones del uso de combinaciones para favorecer la acción bactericida, no se requiere estudiar in vitro la propia combinación, sino los mecanismos de resistencia de la cepa evaluada (la resistencia de alto nivel a aminoglucósidos en Enterococcus elimina su acción sinérgica con antimicrobianos activos a nivel de la pared).
Persisten importantes limitaciones en la obtención de una correlación clínica de los resultados de los estudios de sinergia por la técnicas del tablero de ajedrez y de las curvas de muerte. Aunque la primera técnica es más fácil de realizar (especialmente en su forma miniaturizada), los resultados de ambas técnicas no tienen suficiente concordancia y, en general, la técnica de las curvas de muerte indica sinergia con más frecuencia que la prueba del tablero de ajedrez. Algunos autores, incluso, consideran que los resultados de sinergia mediante el tablero de ajedrez son muy poco útiles o, cuando menos, deben tomarse con precaución. Sería conveniente desarrollar un método estandarizado y basándose en éste realizar estudios clínicos que sustenten la utilidad real de los estudios de interacción de antimicrobianos para un paciente concreto.
Antibiograma directo
Los resultados de un estudio sobre datos de sensibilidad obtenidos mediante inoculación directa de los paneles comerciales con el crecimiento bacteriano obtenido en frascos de hemocultivos indicaron concordancias superiores al 90%, en comparación con los datos obtenidos tras el subcultivo del frasco de hemocultivo32. Sin embargo, en otro estudio reciente en el que se inocularon directamente los paneles convencionales con microorganismos crecidos directamente en frascos de hemocultivos se obtuvieron cifras inaceptables de error, en comparación con los determinados mediante un método estandarizado33. También se han publicado diversos estudios sobre antibiograma directo de orina, aunque en este caso se hace necesario estudiar cultivos monomicrobianos, y debe tenerse en cuenta la posible interferencia de los antimicrobianos que estén presentes en la propia muestra de orina.
Lectura interpretada del antibiograma y sistemas expertos
El antecedente más significativo de la lectura interpretada del antibiograma es considerar que las cepas de S. aureus resistentes a meticilina por producción de PBP2a son resistentes a todos los betalactámicos. Más recientemente el NCCLS ha aceptado que las cepas de E. coli y K. pneumoniae productoras de betalactamasa de espectro extendido deben considerarse resistentes a penicilinas, cefalosporinas y monobactamas, con independencia del valor de su CIM.
En 1992 se introdujo formalmente el concepto de lectura interpretada del antibiograma34, y desde entonces en muchos laboratorios clínicos el análisis del antibiograma se realiza observando el fenotipo de resistencia del microorganismo, infiriendo a partir del mismo los mecanismos de resistencia subyacentes y definiendo en función de estos las categorías de sensible, intermedio y resistente35. La interpretación debe realizarse en función de la identificación, porque la mayoría de las especies presentan fenotipos de resistencia basal o adquirida que difieren de otros microorganismos filogenéticamente próximos.
La lectura interpretada del antibiograma exige la labor activa del microbiólogo, quien no sólo debe elaborar informes clínicos, sino que debe mantener una actitud alerta para (una vez descartados los errores metodológicos) detectar la aparición de patrones de resistencia poco frecuentes o "imposibles".
Uno de los últimos logros en el área de interpretación del antibiograma ha sido el uso de programas informáticos (sistemas expertos) que facilitan la interpretación clínica de los resultados obtenidos. Estos sistemas expertos pueden constituir una herramienta pedagógica eficaz que permite actualizar los conocimientos del microbiólogo clínico en el análisis de los fenotipos de resistencia, pero también pueden generar una inadecuada sensación de fiabilidad que comprometa la significación clínica de los resultados obtenidos.
El software de los sistemas expertos se basa, obviamente, en los fenotipos de resistencia conocidos en el momento de su programación. Continuamente se están descubriendo nuevos mecanismos de resistencia y se analiza su impacto en el riesgo de fracaso terapéutico. Los últimos estudios, tanto en cepas de laboratorio como de origen clínico, están demostrando que la resistencia es un fenómeno multifactorial, lo que dificulta conocer tanto la importancia individual de cada mecanismo como su trascendencia clínica, en especial cuando se trata de mecanismos de bajo nivel de resistencia. Por otra parte, persisten importantes lagunas en el conocimiento de los mecanismos de resistencia, ya sea por dificultades metodológicas (p. ej., estudio de las alteraciones de las proteínas fijadoras de penicilina en la resistencia de los bacilos gramnegativos) o por la ausencia de estudios clínicos que demuestren la trascendencia de los hallazgos microbiológicos (p. ej., relevancia clínica de los bajos niveles de resistencia a fluoroquinolonas).
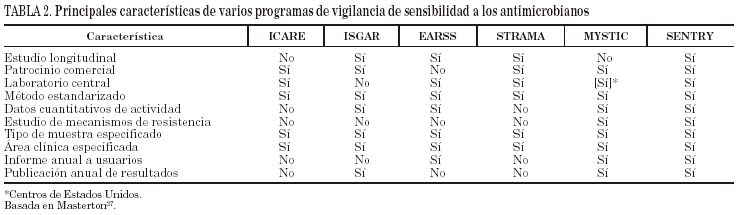
Aunque los resultados con algunos de estos sistemas han sido muy satisfactorios36, aún se han realizado pocas evaluaciones sobre su fiabilidad en condiciones de trabajo reales. En la práctica totalidad de los estudios se han identificado áreas en las que el microbiólogo clínico sigue realizando una interpretación clínica más fiable que el sistema experto (tabla 2).
Programas de vigilancia de evolución de las resistencias a los antimicrobianos
Existen varios programas de vigilancia de resistencia a los antimicrobianos patrocinados por agencias de tipo supranacional (WHONET y GSAP de la OMS, ENARE [ya concluido] y ERAS, de la Unión Europea) nacional (ICARE, GIPS y NARMS de EE.UU., ISGAR de Italia, STRAMA de Suecia, etc.), o por entidades privadas (Alexander Project, MYSTIC, SENTRY, SMART, TSN, ZAP). En la tabla 3 se recogen las características de algunos de estos programas.
Se están desarrollando, básicamente, dos tipos de programas:
1. Los que evalúan los resultados de sensibilidad de microorganismos seleccionados, empleando para ello métodos estandarizados (la práctica totalidad de los estudios en curso).
2. Programas de vigilancia basados en los resultados diarios proporcionados por laboratorios de microbiología clínica, la mayor parte de ellos obtenidos con sistemas comerciales de determinación de la sensibilidad. De entre estos programas el de mayor implantación es la red TSN, en la que automáticamente se vuelcan cada día más de 75.000 resultados procedentes de laboratorios clínicos localizados en Estados Unidos, Canadá, Europa y Australia, una cifra entre 40 y 1.000 veces superior a la obtenida con otros sistemas de vigilancia basados en el estudio de cepas clínicas en laboratorios centralizados38.
La capacidad de almacenamiento informático aumenta unas 10 veces cada 3 o 4 años. Se estima que los sistemas bioinformáticos actuales permiten el almacenamiento de hasta 3-4 billones de datos, una cifra que supera con creces los datos obtenidos en cientos de laboratorios que determinaran cada día las CIM de 15-20 antimicrobianos frente a 30-50 microorganismos.
En vez de realizar estudios para cada uno de los antimicrobianos evaluados es más adecuado (de acuerdo con la tendencia actual de interpretación del antibiograma), realizar análisis porcentuales y de variación temporal de fenotipos de resistencia (S. aureus resistentes a meticilina, enterobacterias productoras de betalactamasas de espectro extendido, etc.), facilitando así la aplicación epidemiológica de estos resultados. Se han desarrollado varios sistemas computarizados (CIDM, Germwatcher, DMZ) que permiten evaluar los resultados del antibiograma desde el punto de vista epidemiológico.
Otra de las aplicaciones de la informática para un mejor uso de los datos microbiológicos de sensibilidad es el desarrollo de programas que en función del informe de sensibilidad permiten identificar pacientes con tratamiento antimicrobiano inadecuado. También están en desarrollo diversos sistemas de alerta para reconocer en el momento de su ingreso hospitalario, a los pacientes en los que previamente se identificó un microorganismo implicado en brotes nosocomiales epidémicos.
Impacto de los resultados rápidos de antibiograma
Varios trabajos realizados en torno a 1980-1981 demostraron que los resultados de las pruebas de sensibilidad antimicrobiana no tenían una influencia significativa en las decisiones del médico sobre la elección de tratamiento antimicrobiano. En particular, si un tratamiento iniciado antes de recibir la información microbiológica se consideraba eficaz, en pocos casos se cambiaba a una opción más adecuada. Otros autores han demostrado que la información rápida al clínico responsable del paciente de los informes de sensibilidad tiene un impacto significativo en la reducción de la morbimortalidad y en la duración y el coste de la estancia hospitalaria.
Hay poca información aún sobre la importancia del reconocimiento rápido de microorganismos multirresistentes implicados en la aparición de brotes epidémicos nosocomiales.
En un estudio en pacientes bacteriémicos se demostró un ahorro significativo por paciente (158 dólares de 1989) cuando los resultados de sensibilidad se informaron al responsable del enfermo el mismo día en que se constató el crecimiento en los frascos de hemocultivo en vez de cuando el informe se realizó a las 24-48 h del aislamiento39. Se ha demostrado, igualmente, una disminución en el uso inadecuado de antimicrobianos, en la realización de técnicas diagnósticas complementarias, y en la mortalidad en el grupo de pacientes para los que se dispuso de antibiograma el mismo día que en el grupo de pacientes para los que los resultados de sensibilidad se obtuvieron según la metodología tradicional. Estos datos apoyan la utilidad del empleo de sistemas automatizados que obtengan resultados de antibiograma el mismo día en que se realiza la inoculación de los paneles.
No sólo debe perseguirse una disminución en el tiempo de obtención de resultados en el laboratorio, sino que para incrementar el rendimiento de esta información es necesario mejorar los métodos de comunicación con el clínico, lo que, probablemente, obligue a reestructurar a corto plazo las rutinas de trabajo e, incluso, los horarios de actividad, del laboratorio de microbiología clínica. En un estudio40 en el que se combinaron un método rápido de antibiograma y el envío inmediato de la información mediante un sistema informático adecuado, se comprobó una disminución de la estancia hospitalaria y en el coste variable por paciente (tabla 4). El impacto económico positivo de este programa (que en conjunto se cifró en casi 5 millones de euros/años para el conjunto de pacientes hospitalizados) se relacionó con un cambio hacia un tratamiento antimicrobiano más adecuado como consecuencia de la información recibida por el clínico.