La resistencia a glucopéptidos en las especies de Enterococcus spp. es un problema clínico importante debido a su rápida diseminación, a la posible transferencia de la resistencia a vancomicina a patógenos más virulentos, como Staphylococcus aureus, y a las limitadas posibilidades terapéuticas de las infecciones causadas por estos microorganismos. En este estudio se caracterizaron 10 cepas de Enterococcus faecium resistentes a vancomicina (ERV) de 10 pacientes distintos, aisladas en este hospital entre 2004 y 2005.
MétodosSe determinó el gen implicado en la resistencia a glucopéptidos mediante la técnica de la reacción en cadena de la polimerasa. El análisis molecular de los aislados clínicos se determinó mediante electroforesis en campo pulsante (ECP). Se estudió la presencia de genes previamente asociados a epidemicidad y virulencia (esp, hyl, asa1, gel, cyl). Se realizó un estudio de casos y controles para analizar los factores de riesgo.
ResultadosSe detectó de manera mayoritaria el gen vanA. El análisis mediante ECP reveló 5 genotipos distintos (A-E). Resultaron mayoritarios los genotipos A (n=3) y B (n=3). Se detectó en todas las cepas la presencia de los genes esp (proteína de superficie) e hyl (hialuronidasa) excepto en los genotipos B y D. En todas las cepas se demostró la presencia del alelo purK1 asociado al complejo clonal 17.
ConclusiónLa administración previa de cefalosporinas, aminoglucósidos y vancomicina (solos o en combinación) se asocia significativamente a la colonización e infección por ERV.
Glycopeptide resistance in Enterococcus spp. is a clinical problem because of the rapid dissemination of these microorganisms, possible transfer of vancomycin resistance to more virulent pathogens, such as Staphylococcus aureus, and the limited therapeutic possibilities for the infections they cause. In this study, 10 strains of vancomycin-resistant Enterococcus faecium isolated from 10 different patients in our hospital during 2004 to 2005 were characterized.
MethodsPCR was used to analyze the gene implicated in glycopeptide resistance. Molecular analysis of clinical isolates was performed by pulsed-field gel electrophoresis (PFGE). The presence of genes previously associated with epidemicity/virulence (esp, hyl, asa1, gel, cyl) was also studied. Risk factors were determined in a case-control study.
ResultsThe most commonly detected gene was vanA. PFGE analysis revealed 5 different genotypes (A-E), with a predominance of genotype A (n=3) and B (n=3). The esp (surface protein) and hyl (hyaluronidase) genes were detected in all strains, with the exception of genotypes B and D. The purK1 allele, associated with clonal complex 17 was demonstrated in all strains.
ConclusionPrior administration of cephalosporins, aminoglycosides and vancomycin alone or in combination was significantly associated with colonization/infection by vancomycin-resistant Enterococcus faecium.
En los últimos años, los Enterococcus spp. han llegado a ser una causa importante de infección nosocomial debido a la selección de clones resistentes a la mayoría de los agentes antimicrobianos disponibles en el arsenal terapéutico1. Los enterococos resistentes a vancomicina (ERV), o más genéricamente resistentes a glucopéptidos, se aislaron inicialmente en 1986 en Francia y el Reino Unido y, poco después, en Estados Unidos2. Desde entonces, se han descrito ERV como causantes de infecciones nosocomiales en todo el mundo, aunque con una incidencia variable según el área geográfica y las instituciones hospitalarias2,3,4,5,6. Debido a su rápida diseminación, la posible transferencia de la resistencia a vancomicina a otros patógenos de mayor impacto clínico, como Staphylococcus aureus7, y las posibilidades terapéuticas limitadas para el tratamiento de las infecciones causadas por ERV éstos se han convertido en un problema clínico importante.
Actualmente se conocen 5 tipos de resistencia adquirida a vancomicina, cada uno asociado a un gen ligasa diferente (vanA, vanB, vanD, vanE y vanG)2,6. Los enterococos con genotipo vanA son generalmente resistentes a vancomicina y teicoplanina mientras que los genotipos vanB, vanD, vanE y vanG suelen asociarse a grados moderados de resistencia a vancomicina y sensibilidad a teicoplanina6. Los genotipos vanA y vanB son los más comunes y están ampliamente diseminados en Estados Unidos, Europa y Australia2,6. Ambos fenotipos se han identificado en enterococos aislados de muestras clínicas, veterinarias y productos alimenticios para consumo humano.
La resistencia a glucopéptidos en Enterococcus es más frecuente en aislados de Enterococcus faecium que en otras especies del género y ha causado numerosos brotes de infecciones hospitalarias en distintas áreas geográficas5,6. Aunque en menor proporción, se han descrito brotes causados por Enterococcus faecalis y Enterococcus raffinosus resistentes a glucopéptidos5,8.
El estudio de la estructura poblacional de E. faecium y E. faecalis ha revelado la asociación de determinados complejos clonales, como el CC17 de E. faecium y los CC2, CC9, CC87 y CC21 de E. faecalis, a distintos huéspedes y nichos ecológicos. La mayoría de los aislados clínicos resistentes a antibióticos descritos están asociados a estos complejos clonales9. Aunque la patogénesis de las infecciones enterocócicas no se ha demostrado, se han descrito algunos genes que pueden contribuir a la virulencia de estas especies y que están asociados a la epidemicidad de estos clones9,10,11,12,13. En E. faecalis estos factores incluyen la sustancia de agregación (asa), implicada en los fenómenos de conjugación y que facilita la adhesión e internalización y supervivencia en las células humanas14, una gelatinasa (gel), una metaloproteasa extracelular dependiente de zinc, una citolisina (cyl) con actividad tóxica en células eucariotas15 y una proteína de superficie (esp) que favorece la adhesión a células epiteliales, la formación de biofilms y la evasión de la respuesta inmune y que está codificada por un gen localizado en una isla de patogenicidad12,15,16,17.
Los factores asociados a la epidemicidad o virulencia de E. faecium son una proteína con homología a la hialuronidasa (hyl) involucrada en procesos de virulencia en otras especies, y una proteína de superficie análoga a la de E. faecalis pero codificada por un gen localizado en una isla de patogenicidad diferente17,18. Ambos se han identificado exclusivamente en CC17, con lo que se ha establecido una asociación entre la presencia de esp y clones causantes de brotes nosocomiales13,18,19,20.
El objetivo del presente trabajo fue caracterizar molecular y epidemiológicamente los aislados clínicos de E. faecium resistentes a glucopéptidos identificados en este hospital durante el período de 2004 a 2005 y determinar las variables significativas asociadas a infección y colonización por ERV.
Material y métodosCepas bacterianasSe analizaron 10 aislados clínicos de Enterococcus spp. resistentes a glucopéptidos correspondientes a 10 pacientes que ingresaron en este hospital durante el período de junio de 2004 a diciembre de 2005. Se seleccionó un aislado por paciente (el primer aislamiento en todos los casos, y de adquisición nosocomial). Se identificaron todas las cepas mediante la utilización del sistema comercial semiautomatizado MicroScan Walk-Away y los paneles “Positive Combo Panel 22S” (Dade Internacional Inc., West Sacrament, CA, EE. UU.) y API 20 STREP (BioMeriux, Marcy l`Etoile, Francia) y se emplearon como cepas control E. faecalis CECT 407 y E. faecium CECT 4931 y 410.
Susceptibilidad antimicrobianaSe estudiaron todas las cepas por microdilución en MicroScan Walk-Away con los paneles “Positive Combo Panel 22S” (Dade International Inc., West Sacrament, CA, EE. UU.). La sensibilidad a los glucopéptidos vancomicina y teicoplanina y a linezolid y ciprofloxacino se confirmó por la técnica de difusión en disco y mediante la utilización de tiras de E-test. La sensibilidad a eritromicina, clindamicina, rifampicina, synercid y cotrimoxazol se confirmó por la técnica de difusión en disco. Se siguieron las normas del Clinical Laboratory Standards Institute (documentos M2 y M7)21 y se emplearon como cepas control E. faecalis CECT 407 y E. faecium CECT 4931 y 410.
Caracterización de los genes Van y confirmación de la identificación en cuanto a la especieEl ácido desoxirribonucleico (ADN) de las cepas estudiadas utilizado como molde en la reacción en cadena de la polimerasa (PCR) se obtuvo mediante la técnica de boiling, resuspendiendo colonias en 500 μml de agua destilada y manteniendo esta suspensión en ebullición durante 10min para posteriormente separar por centrifugación (5min a 13.000rpm) el sobrenadante que contiene el ADN. Se usaron los pares de oligodesoxinucleótidos que amplifican los genes vanA, vanB, vanC1, vanC2, vanC3, ddlE.faecalis y ddlE.faecium de acuerdo con las condiciones experimentales descritas para cada caso22,23. Como controles positivos para la amplificación de los genes vanA, vanB, vanC1 y vanC2/C3 se utilizaron las cepas control E. faecalis resistente a glucopéptidos portador del gen vanA24, E. faecium CKU12, Enterococcus gallinarum CECT 970, Enterococcus casseliflavus CECT 969 y Enterococcus flavescens CECT 4481, respectivamente.
Análisis de la clonalidadLa relación clonal entre los aislados se estableció mediante el análisis de los perfiles de macrorrestricción del ADN cromosómico obtenidos por electroforesis de campo pulsante (ECP) tras digestión enzimática con la enzima smaI (New England Biolabs, Boston, MA, EE. UU.), según los procedimientos y criterios previamente publicados25,26.
La identificación de la secuencia de purK (uno de los 7 genes utilizados en el esquema de multilocus sequence typing) se realizó mediante la utilización de los pares de oligodesoxinucleótidos purk1 (5′-GCA GAT TGG CAC ATT GAA AGT-3′) y purk2 (5′-tac ata aat ccc gcc tgt tty-3) y las condiciones de PCR descritas previamente18. El gen purK codifica la subunidad fosforibosilaminoimidazol carboxilasa adenosintrifosfatasa y su alelo 1 se considera marcador de E. faecium CC1713,19.
Detección de genes de virulenciaLos genes de virulencia esp, asa1, gelE, cyl e hyl se amplificaron mediante la utilización de los oligonucleótidos descritos previamente y las siguientes condiciones de PCR: desnaturalización inicial a 95°C durante 10min; 40 ciclos a 94°C durante 1min, 56°C durante 1min y 72°C durante 1min; y extensión a 72°C durante 10min27. Se utilizaron como cepas control E. faecalis OG1RF (esp-, asa1-, gelE+, cyl-, hyl-) y E. faecalis TX400 (esp-, asa1+, gelE-, cyl-, hyl-), cedidas por B. E. Murray; E. faecalis MMH594 (esp+, asa1+, gelE+, cyl+, hyl-), cedida por N. Shankar, University of Oklahoma, y E. faecalis C68 (esp+, asa1-, gelE-, cyl-, hyl+), cedida por L. B. Rice, University of Cleveland.
Estudio de casos y controlesPor cada caso se eligieron aleatorizadamente al menos 2 controles entre los pacientes que ingresaron durante el período de estudio en el mismo servicio que el caso correspondiente, a los que se les hubiera tomado una muestra igual o similar en origen (anatómica y microbiológica) a la que fue positiva para ERV en el caso y con una estancia hospitalaria previa a la toma de la muestra similar a la del caso.
Estudio de variables para el análisis estadísticoSe estudiaron las siguientes variables tanto para los casos como para los controles: edad, sexo, enfermedad de base (enfermedad autoinmune, enfermedad hematológica, diabetes mellitus, tumor sólido, trasplante, enfermedad digestiva, enfermedad cardiovascular y enfermedad pulmonar obstructiva crónica), índice APACHE, índice de comorbilidad de Charlson, presencia de sonda nasogástrica, presencia de sonda vesical o catéter urinario, presencia de catéter intravenoso, nutrición parenteral, ventilación mecánica, cirugía previa, fallecimiento, tratamiento inmunosupresor, tratamiento antibiótico previo (betalactámicos, cefalosporinas, carbapenémicos, vancomicina, quinolonas, aminoglucósidos, antifúngicos) y días de estancia previos al aislamiento.
Análisis estadísticoEste estudio incluye el análisis univariante y de regresión logística condicional múltiple para determinar variables significativas asociadas a colonización e infección por ERV resistente a glucopéptidos. Los resultados se consideraron estadísticamente significativos para p<0,05. Las variables cuantitativas se expresan como media ± desviación típica. Las variables cualitativas se expresan como valor absoluto y porcentaje. La comparación de variables continuas se realizó por medio del test de la U de Mann-Whitney. La asociación de variables cualitativas entre sí se estimó por medio del test estadístico de la χ2 y el cálculo, a su vez, de la odds ratio (OR) con su intervalo de confianza (IC) del 95%. Para determinar las variables asociadas de manera independiente a la adquisición de ERV se realizó un análisis de regresión logística condicional. En este análisis se utilizaron como covariables aquellas variables que en el análisis univariado definían un caso, o aquellas variables consideradas clínicamente relevantes. El tamaño muestral de este estudio (10 casos y 22 controles) permite con una seguridad del 95%, un poder estadístico del 80%, una OR mayor o igual a 2 ante una frecuencia de exposición de los casos del 70%, y una exposición de los controles del 18%.
ResultadosCepas bacterianas y sensibilidad a antibióticosLa tasa de ERV en este hospital durante el período de 2001 a 2007 se mantuvo desde el brote del año 2001(entre mayo y septiembre) por debajo del 1% para E. faecalis, y ha aumentado del 0 al 6% para E. faecium; se observó un pico de incidencia del 21% en 2005, año del aislamiento de las cepas del estudio. Los 10 aislados clínicos de E. faecium se identificaron a partir de muestras de orina (n=4), catéteres intravenosos (n=3) y líquidos orgánicos (peritoneal, ascítico y drenaje peritoneal, n=3). En la mayoría de los aislados (8 de 10) se amplificó el gen vanA; en 2 aislados se amplificó el gen vanB, y en ninguno de ellos se amplificaron los genes vanC1 y vanC2/C3.
Uno de los aislados estudiados (aislado número 2, en tabla 1), correspondiente a una muestra de heces de un paciente infectado con ERV, fue sensible a vancomicina y teicoplanina y no contenía ninguno de los genes van. Las cepas identificadas como portadoras del gen vanA exhibieron resistencia a vancomicina y teicoplanina (concentración mínima inhibitoria [CMI] superior o igual a 32μg/ml) mientras que las 2 cepas portadoras del gen vanB fueron resistentes a vancomicina (CMI superior o igual a 32μg/ml) y sensibles a teicoplanina (CMI<8μg/ml). Todas las cepas fueron resistentes a penicilina, ampicilina, eritromicina, clindamicina, rifampicina y ciprofloxacino y sensibles a linezolid. Todas fueron sensibles a synercid, excepto la cepa 8, y sensibles a cotrimoxazol, excepto las cepas 4 y 8 (genotipo vanB). La mayoría de los aislados presentaban alto grado de resistencia a gentamicina y estreptomicina, excepto las cepas 5, 7 y 8 que presentaron bajo grado de resistencia a gentamicina y las cepas 5, 7, 9 y 11 que presentaron bajo grado de resistencia a estreptomicina. Las características de los aislados se exponen en la tabla 1.
Tabla 1. Aislamientos de Enterococcus faecium implicados en este estudio y resultados de los datos de sensibilidad antibiótica, genes de resistencia a glucopéptidos, tipificación mediante electroforesis de campo pulsante y detección de los genes de virulencia
| Cepa a | Servicio | Muestra | Fecha aislamiento | CMI b AMP | CMI CIP | CMI c GEN | CMI VAN | CMI TEI | CMI LZD | CMI d SYN | Genotipo | PGFE | esp | hyl | cyl | gelE | asa |
| 1 | UCI | Orina | 27/04/2005 | >8 | >32 | >256 | >256 | 48 | 2 | >21(S) | vanA | A1 | (+) | (+) | (-) | (-) | (-) |
| 2 | UCI | Heces | 18/05/2005 | >8 | >32 | >256 | 1,5 | 1,5 | 2 | >21(S) | A2 | (+) | (+) | (-) | (-) | (-) | |
| 3 | UCI | Orina | 14/06/2005 | >8 | >32 | >256 | >256 | 48 | 2 | >21(S) | vanA | B1 | (+) | (+) | (-) | (-) | (-) |
| 4 | Reanimación | Drenaje | 17/06/2005 | >8 | >32 | >256 | 32 | 1,5 | 1.5 | >21(S) | vanB | C | (+) | (-) | (-) | (-) | (-) |
| 5 | UCI | Catéter | 13/01/2005 | >8 | >32 | 8 | >256 | 24 | 1.5 | >21(S) | vanA | D | (-) | (-) | (-) | (-) | (-) |
| 6 | UCI | Catéter | 24/01/2005 | >8 | >32 | >256 | >256 | 32 | 2 | >21(S) | vanA | A2 | (+) | (+) | (-) | (-) | (-) |
| 7 | UCI | Catéter | 28/01/2005 | >8 | >32 | 8 | >256 | 32 | 2 | >21(S) | vanA | D | (-) | (-) | (-) | (-) | (-) |
| 8 | Nefrología | Orina | 14/03/2005 | >8 | >32 | 8 | >256 | 2 | 2 | <10(R) | vanB | B2 | (+) | (+) | (-) | (-) | (-) |
| 9 | Reanimación | Orina | 20/06/2004 | >8 | >32 | >256 | >256 | 32 | 1.5 | >21(S) | vanA | E | (+) | (+) | (-) | (-) | (-) |
| 10 | Cirugía General A | Líquido peritoneal | 05/12/2005 | >8 | >32 | >256 | 96 | 24 | 1.5 | >21(S) | vanA | B1 | (+) | (+) | (-) | (-) | (-) |
| 11 | Reanimación | Líquido ascítico | 30/11/2005 | >8 | >32 | 4 | >256 | 32 | 2 | >21(S) | vanA | A3 | (+) | (+) | (-) | (-) | (-) |
AMP: ampicilina; CIP: ciprofloxavino; CMI: concentración mínima inhibitoria; GEN: gentamicina, LZD: linezolid; SYN: synercid; TEI: teicoplanina; UCI: unidad de cuidados intensivos; VAN:vancomicina.
a Cepas 1 y 2 se aislaron del mismo paciente.
b Concentración mínima inhibitoria en mg/l.
c Bajo grado de resistencia a gentamicina: cepas 5, 7 y 8. Bajo grado de resistencia a estreptomicina: cepas 5, 7, 9 y 11.
d Concentración mínima inhibitoria en mm (interpretación).
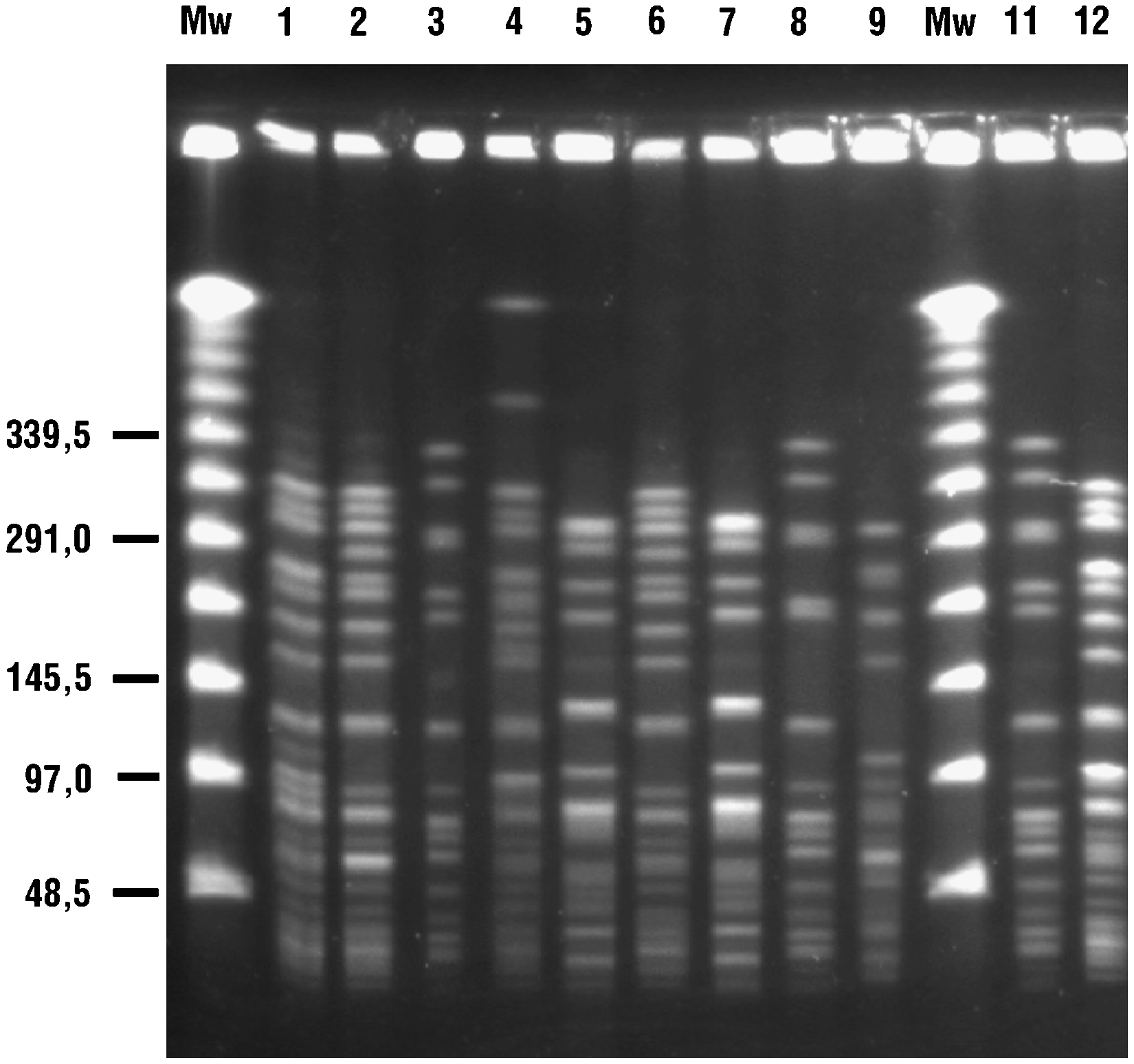
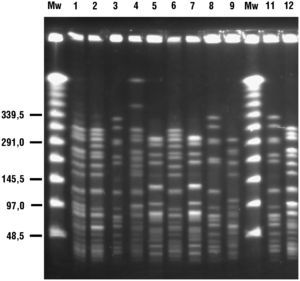
Se identificaron 5 genotipos distintos correspondientes a los 10 ERV estudiados: A (n=3, subtipos A1 y A3), B (n=3, subtipos B1 y B2), C (n=1), D (n=2) y E (n=1) mediante la técnica de ECP (tabla 1 y figura 1). En un paciente se obtuvieron 2 aislados con diferente sensibilidad a glucopéptidos. La cepa 1 fue resistente a glucopéptidos y portadora del gen vanA (subtipo A1) mientras que la cepa 2 fue sensible a glucopéptidos y no amplificó para ninguno de los genes de resistencia estudiados (subtipo A2).
Figura 1. Estudio mediante electroforesis en campo pulsante de las cepas de enterococo resistente a la vancomicina detalladas en la tabla 1 . Se indica cada cepa con el número sobre el gel Mw, marcadores de peso molecular.
Los subtipos B1 y B2 correspondían a aislados de distintos pacientes con los genotipos vanA y vanB, respectivamente. El alelo purK-1, específico de E. faecium-CC17, fue identificado en todos los aislados estudiados (datos no mostrados).
Factores de epidemicidad o virulenciaSe demostró la presencia de esp y de hyl para todos los aislados de los genotipos A, B y E (cepas 1–3, 6, 8–11). Los aislados del genotipo C (cepa 4) amplificaron solamente esp y los del genotipo D (cepas 5 y 7) no amplificaron ninguno de los genes analizados (tabla 1). Todos los aislados fueron negativos para los genes cyl, gelE y asa.
Análisis estadístico y de regresión logística multipleLa media de edad de los 10 pacientes estudiados (11 cepas) fue de 45,2 años, con una media de estancia previa al aislamiento de ERV de 35 días. La media de edad de los 22 pacientes control que ingresaron durante el mismo período en el que se aislaron las cepas del estudio fue de 62 años, con una media de ingreso previo al aislamiento de 52 días. Las características de los casos y los controles se muestran en la tabla 2. No se encontraron diferencias entre casos y controles en referencia a la enfermedad de base. Ninguno de los casos presentó como enfermedad de base diabetes mellitus, enfermedad pulmonar obstructiva crónica, enfermedad cardiovascular, enfermedad digestiva o enfermedad hematológica, y aproximadamente el 40% de los casos y controles presentaron enfermedad hepatorrenal o tumor sólido. La inmunosupresión no fue una variable estadísticamente significativa (tabla 2). Cabe resaltar que 2 de los casos presentaron politraumatismo frente a ninguno de los controles.
Tabla 2. Características de los pacientes del estudio (estudio de casos y controles)
| Variable | Casos (n=10) | Controles (n=22) | ||
| Media (DT) | Media (DT) | p | ||
| Edad | 45,5 (22,9) | 62,1 (16,4) | 0,058 | |
| Días de ingreso | 34,8 (16,4) | 51,95 (72,3) | 0,889 | |
| Índice APACHE | 11,20 | 12,77 | 0,37 | |
| Índice de comorbilidad de Charlson | 4,5 (2,4) | 4,6 (2,4) | 0,92 | |
| n (%) | n (%) | p | OR (IC del 95%) | |
| Sexo | 0,47 | 1,75 (0,4–7,9) | ||
| Varón | 5 (26,3) | 14 (73,7) | ||
| Fallecimiento (Sí) | 4 (40,0) | 9 (40,9) | 0,96 | 0,96 (0,2–4,4) |
| Cirugía | 3 (30) | 10 (45,5) | 0,41 | 0,5 (0,1–2,53) |
| Tratamiento previo | 10 (100) | 22 (100) | – | – |
| Vancomicina | 8 (80) | 8 (36,4) | 0,022 | 7 (1,19–41,36) |
| Aminoglucósidos | 8 (80) | 8 (36,4) | 0,022 | 7 (1,19–41,36) |
| Betalactámicos | 3 (30) | 12 (54,5) | 0,197 | 0,36 (0,07–1,75) |
| Cefalosporinas | 7 (70) | 4 (18,2) | 0,004 | 10,5 (1,86–59,4) |
| Carbapenémicos | 6 (60) | 12 (54,5) | 0,773 | 1,25 (0,27–5.7) |
| Quinolonas | 1 (10) | 4 (18,2) | 0,555 | 0,5 (0,049–5,15) |
| Antifúngicos | 4 (40) | 4 (18,2) | 0,186 | 3 (0,57–15,87) |
| Cefalosporinas+aminoglucósidos | 5 (50) | 3 (13,6) | 0,028 | 6,33 (1,11–35,9) |
| Vancomicina+aminoglucósidos | 6 (60) | 9 (9,1) | 0,002 | 15 (2,18–103) |
| Vancomicina+cefalosporinas | 5 (50) | 2 (9,1) | 0,009 | 10 (1,48–67,55) |
| Inmunosupresión | 4 (40) | 4 (18,2) | 0,186 | 3 (0,57–15,87) |
| Catéter urinario/sonda vesical | 7 (70) | 14 (63,6) | 0,725 | 1,33 (0,27–6,65) |
| Sonda nasogástrica | 8 (80) | 15 (68,2) | 0,49 | 1,87 (0,31–11,19) |
| Catéter intravenoso | 10 (100) | 20 (90,9) | 0,325 | 1,5 (0,12–44,30) |
| Ventilación mecánica | 5 (50) | 10 (45,5) | 0,81 | 1,2 (0,27–5,36) |
| Nutrición parenteral | 7 (70) | 11 (50) | 0,29 | 2,33 (0,48–11,44) |
DT: desviación típica; IC: intervalo de confianza; OD: odds ratio.
Sí hubo diferencias significativas entre los casos y los controles respecto al tratamiento previo con los antibióticos vancomicina, cefalosporinas y aminoglucósidos y la asociación entre ellos. Se encontró una mayor asociación con la exposición previa a cefalosporinas (OR de 10,5; IC del 95%, rango de 1,86 a 59,4), seguida de la exposición previa a vancomicina o aminoglucósidos. En ambos casos la OR fue de 7 (IC del 95%, rango de 1,19 a 41,36). La combinación entre vancomicina y aminoglucósidos presentó la mayor asociación (OR de 15; IC del 95%, rango de 2,18 a 103).
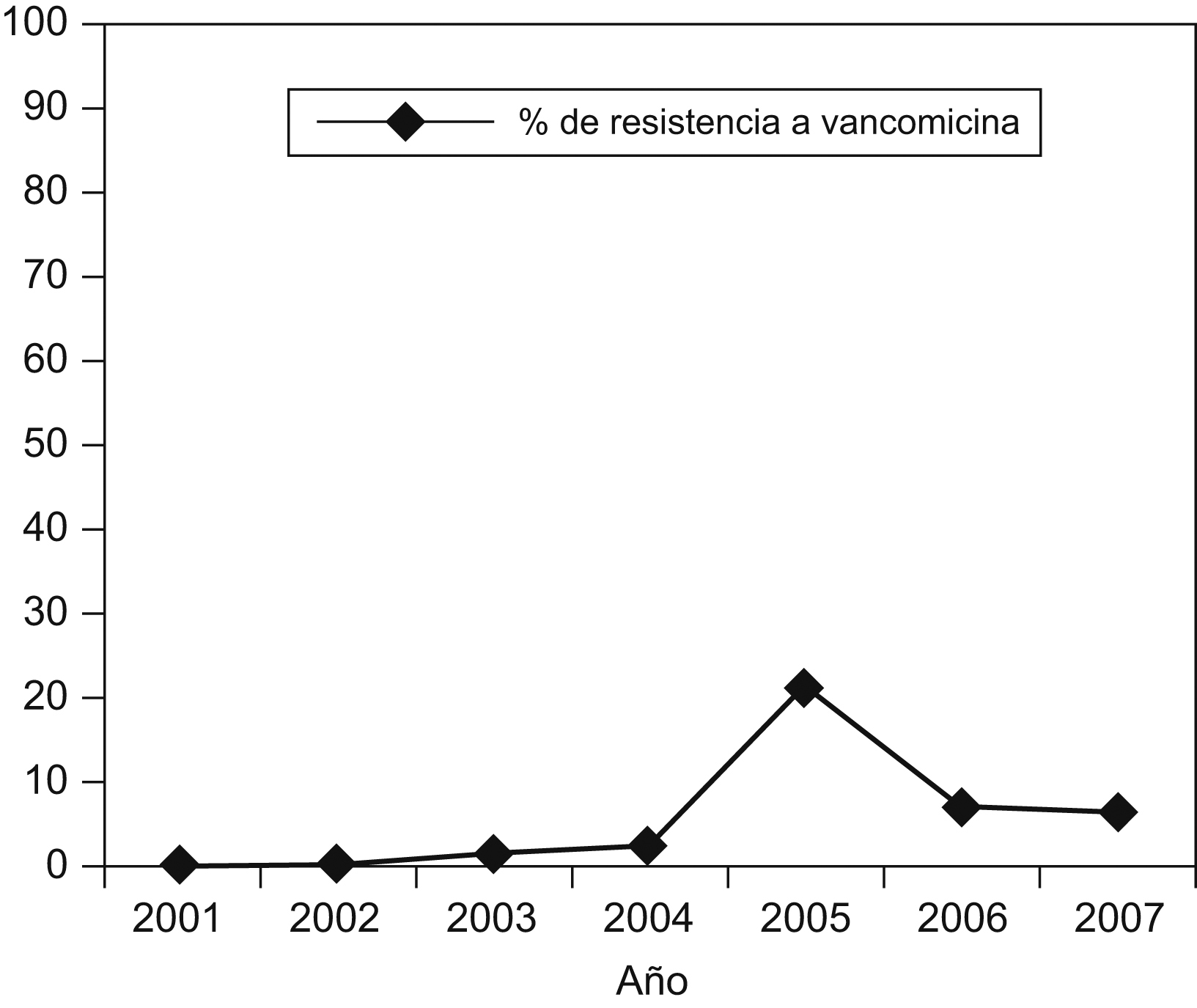
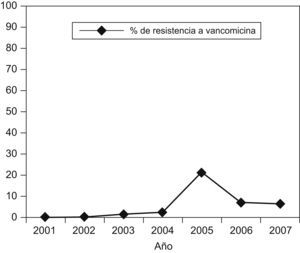
DiscusiónLa incidencia de ERV en los hospitales de Europa ha aumentado en los últimos años con tasas de resistencia superiores al 10% en 6 países ( http://www.rivm.nl/earss ). La prevalencia de ERV en España, al igual que en algunos estados europeos, permanece baja, con valores inferiores al 5% (2,2% de E. faecium resistente a vancomicina en España en 2006), aunque se han descrito de forma esporádica brotes monoclonales y policlonales causados por E. faecalis y E. faecium de los genotipos vanA o vanB en Valencia, Santander, Palma de Mallorca, La Coruña, La Rioja y Soria, y todos estos han sido autolimitados28,29,30,31,32,33. En este hospital, la tasa de ERV en el período de 2001 a 2007 aumentó del 0 a 6% a expensas de E. faecium (figura 2) y se observó un pico de incidencia del 21% en 2005, correspondiente al año del aislamiento de las cepas del estudio y que, de forma análoga a lo descrito en otras instituciones, no se ha mantenido en el tiempo.
Figura 2. Evolución de la resistencia a vancomicina (en %) de los aislamientos de Enterococcus faecium en este hospital durante los últimos 6 años.
Distintos clones de E. faecium-purk1 de los genotipos vanA y vanB, aislados en distintas áreas del hospital causaron el aumento de casos de E. faecium resistente a vancomicina que se describe en el presente estudio; la mayoría de estos clones presentaron factores de epidemicidad o virulencia (7 de 10, genotipos A, B y E). La mayor parte de las cepas de ERV causantes de brotes hospitalarios descritas pertenecen a CC17 y las características asociadas son resistencia a ampicilina y ciprofloxacino, el gen de virulencia esp y a veces también hyl9,13. La heterogeneidad observada en el presente estudio puede reflejar la selección endógena a partir de la propia flora del paciente debido al tratamiento antibiótico recibido. Sin embargo, otros estudios han descrito que la mayoría de las cepas adquiridas a partir de la flora de los pacientes hospitalizados son sensibles a los antibióticos ampicilina, aminoglucósidos y quinolonas y no presentan factores de virulencia13. Otra posibilidad es la adquisición de elementos genéticos de resistencia a glucopéptidos por parte de cepas epidémicas o endémicas de CC17-E. faecium resistentes a ampicilina y sensibles a glucopéptidos, productoras de esp como se ha descrito en otras instituciones de países con altas y bajas tasas de ERV, como Portugal y Polonia o España, Holanda, Finlandia y Noruega, respectivamente13,34,35,36. Aunque la población de E. faecium resistentes a antibióticos en esta institución no se ha analizado, la selección de clones predominantes de CC17 resistentes a ampicilina no puede descartarse. La identificación de aislados productores de factores de epidemicidad con un variable contenido de genes van, como los subtipos A1 (vanA) y A2 (sensible a glucopéptidos y no portador de genes van), o los subtipos B1 (vanA) y B2 (vanB), reflejan la adquisición de distintos elementos genéticos por parte de cepas determinadas y la importancia de la transferencia horizontal en la diseminación y persistencia de ERV en el ámbito hospitalario.
En relación con la presencia de genes de epidemicidad o virulencia, todas las cepas del presente estudio amplificaron para el gen esp, excepto el genotipo D. La presencia de los genes esp se ha visto asociada a brotes hospitalarios y a la capacidad de formar biofilms10,11,13,17,19, aunque este gen no es exclusivo de cepas epidémicas11,18,37,38,39,40. Además, la presencia variable del gen esp dentro de clones ampliamente distribuidos puede explicarse por la transferencia horizontal de la isla de patogenicidad en la que se encuentra localizado13,18,38. El gen hyl asociado a cepas implicadas en brotes hospitalarios de América y Europa5,13 fue identificado en todas las cepas, excepto en las de genotipo C y D. Los demás factores de virulencia, como los genes asa y cyl, sólo se han descrito para E. faecali, y el gelE puede estar presente en cepas de E. faecium, pero ninguno de ellos se relaciona directamente con resistencia a glucopéptidos o brotes hospitalarios11,13.
El estudio de los factores de riesgo de la infección o colonización, a pesar del bajo número de casos analizados (que limita de manera considerable la detección de otros factores de riesgo), reveló una asociación estadísticamente significativa de la adquisición de cepas ERV con el uso previo de antimicrobianos pertenecientes a 3 familias distintas, solos o administrados de forma conjunta, en confirmación con otros estudios11,13. Los casos y los controles son similares en edad, días de ingreso, índice APACHE e índice de comorbilidad. Aunque entre los casos es más frecuente la presencia de inmunosupresión, catéter urinario o sonda vesical, sonda nasogástrica, catéter intravenoso, traqueotomía o intubación y nutrición parenteral, las diferencias respecto a los controles no alcanzan la significación estadística. Tras ajustar por diferentes covariables (edad, índice APACHE, vancomicina, aminoglucósidos, cefalosporinas, inmunosupresión), la variable que tuvo un efecto independiente de todas las demás para predecir por sí misma el hecho de infección o colonización por E. faecium resistente a vancomicina en el presente estudio fue la utilización previa de cefalosporinas. Se trata de un factor de riesgo bien conocido para la adquisición e infección por cepas de ERV, como han mostrado otros estudios, y que subraya la importancia de estos antimicrobianos como potenciales coselectores de estos patógenos28,40.
En resumen, los autores de este artículo describen un aumento en el número de aislamientos de E. faecium-purk1 de los genotipos vanA y vanB, mayoritariamente productores de esp durante el período de 2004 a 2005. La identificación de ERV y de cepas sensibles de genotipo similar a alguno de los ERV enfatiza no sólo el posible papel de determinados marcadores como esp en la virulencia o epidemicidad de la especie estudiada sino también la posible presencia de cepas endémicas sensibles a glucopéptidos que podrían servir como sustratos para la adquisición de ERV. Los datos que se recogieron en el presente estudio ponen también de relieve el papel del tratamiento antibiótico de amplio espectro en la selección de cepas ERV del área hospitalaria y la importancia de una correcta política de antibióticos para evitar la selección de determinados clones epidémicos y probablemente la adquisición de determinantes de resistencia por parte de los éstos.
Agradecimientos
Este estudio ha sido financiado por el Ministerio de Sanidad y Consumo, Instituto de Salud Carlos III, Red Española de Investigación en Patología Infecciosa (REIPI RD06/0008).
Recibido 27 Junio 2008
Aceptado 18 Septiembre 2008
Autor para correspondencia. germanbou@canalejo.org