En julio de 2005 se aisló el primer Enterococcus faecium resistente a vancomicina (EFRV) genotipo vanA en el Hospital Universitario de Canarias en un paciente trasplantado renal ingresado en la Unidad de Nefrología. En los meses siguientes se aisló EFVR en otros 15 pacientes trasplantados renales ingresados en la misma unidad. Nuestro objetivo fue estudiar este primer brote de EFVR vanA en el Hospital Universitario de Canarias y el vínculo epidemiológico con los aislamientos de EFVR vanA encontrados también en otros 2 hospitales universitarios ubicados en las Islas Canarias.
Material y métodosSe estudiaron mediante métodos microbiológicos y moleculares un total de 22 aislamientos de EFRV y las historias clínicas de los 22 pacientes de los que se aislaron.
Resultados y conclusionesSe confirmó mediante electroforesis de campo pulsante que los aislamientos del brote pertenecían a una misma clona y, a su vez, se confirmó el vínculo epidemiológico con los primeros aislamientos en los otros 2 hospitales universitarios relacionando los resultados moleculares con los datos epidemiológicos. Todos los aislamientos pertenecían a la secuencia tipo ST18 mediante multilocus sequence typing, asociado al complejo clonal 17. El complejo clonal 17 es un linaje que comprende una población policlonal que se ha adaptado bien al ambiente hospitalario y que se aísla en los 5 continentes.
In July, 2005 the first vancomycin-resistant Enterococcus faecium (VREF) with a genotype vanA was isolated in Hospital Universitario de Canarias (HUC). From September to December 2005, VREF vanA was isolated from another 15 patients (3 nosocomial infections and 12 rectal carriers). All of them were kidney transplant patients hospitalized in the Nephrology ward. This study describes the first VREF vanA outbreak in the HUC and the epidemiological link of the first VREF vanA isolates found in another two university hospitals in the Canary Islands.
Materials and methodsWe studied a total of 22 VREF isolates by microbiological and molecular methods. Epidemiological and clinical data of the patients involved were collected.
Results and conclusionsWe confirmed that these VREF isolates belonged to the same clone using pulse-field gel electrophoresis (PFGE). In November 2005 and February 2006, the first VREF were isolated in two other University Hospitals in the Canary Islands and we also confirmed the link with the HUC cluster by comparison of patient-related information with the molecular typing data. These VREF isolates belonged to the ST18 associated to the Clonal Complex-17 (CC17). CC17 is the major hospital adapted lineage, representing a polyclonal population and associated to VREF outbreaks and infections in the five continents.
Los primeros enterococos resistentes a la vancomicina (ERV) fueron aislados en el Reino Unido y Francia hace no más de 2 décadas y un poco después en Estados Unidos1–3. Desde entonces, se ha informado ERV como causa importante de infección hospitalaria en muchos países del mundo4. Mientras que en Estados Unidos las infecciones y colonizaciones hospitalarias se han incrementado drásticamente produciendo brotes en unidades de cuidados intensivos, en Europa la prevalencia permanece baja en general, y produce brotes ocasionales en plantas de hospitalización de Nefrología y Hematología, principalmente5–7. Los pacientes trasplantados renales tienen un alto riesgo de colonización e infección por ERV debido a diferentes factores de riesgo, incluida la administración frecuente de antibióticos, particularmente vancomicina, antes o después del trasplante8,9.
Varios estudios de epidemiología molecular demuestran que la mayoría de los brotes hospitalarios y aislamientos clínicos de Enterococcus faecium resistente a la vancomicina (EFRV) pertenecen al complejo clonal 17 (CC17) que representa la primera línea clonal adaptada a los hospitales y diseminada de forma global. Este complejo clonal se caracteriza por ser resistente a la ampicilina y a las quinolonas y por la presencia en su genoma de islotes de virulencia que codifican la proteína de superficie del enterococo (gen esp) y una hialuronidasa (gen hyl) en la mayoría de los aislamientos10–12.
El Hospital Universitario de Canarias (HUC) ha sido el hospital de referencia para la realización de trasplantes renales en toda Canarias durante muchos años. El objetivo de este estudio es describir un brote de EFRV que se produjo en la Unidad de Nefrología y que afectó a pacientes trasplantados renales, y su diseminación posterior a otros 2 hospitales ubicados también en Canarias. Estos aislamientos de EFRV con fenotipo VanA fueron los primeros aislados en nuestro hospital y en toda Canarias.
Material y métodosDatos demográficos los pacientes y medidas de controlEl HUC es un hospital universitario de 655 camas ubicado en Tenerife que ha sido el hospital de referencia en Canarias para la realización de trasplantes renales. Este hospital realiza entre 100–130 trasplantes renales al año. Entre julio y octubre de 2005, 4 pacientes se infectaron por EFRV en la Unidad de Nefrología. Se aplicaron medidas de control de la infección, incluido aislamiento de contacto, recogida de muestras perianales una vez por semana a todos los pacientes ingresados en la unidad, implantación de un programa de educación del personal sanitario y traslado definitivo (previamente planeado) de los pacientes a otra planta de hospitalización el 24 de octubre de 200513,14. Se estudiaron mediante métodos microbiológicos y moleculares un total de 22 aislamientos de EFRV, incluidos los primeros EFVR aislados en otros 2 hospitales de Canarias: el Hospital Universitario Nuestra Señora de la Candelaria (HUNSC) de 960 camas ubicado también en Tenerife y el Hospital Universitario Insular de Gran Canaria (HUIGC) de 718 camas situado en Las Palmas de Gran Canaria. La recogida de datos epidemiológicos de las historias clínicas de los 22 pacientes se realizó como parte del programa de control de la infección. Se siguieron las definiciones de infección o colonización, hospitalaria o comunitaria del Centers for Disease Control15. Un brote se definió como un incremento temporal en la incidencia de infección producida por un determinado microorganismo en la población estudiada, o, alternativamente, como un incremento temporal de la colonización con o sin incremento de la incidencia de la infección16.
CribadoDesde el 17 de octubre de 2005 hasta el 13 de marzo de 2006 todos los pacientes ingresados en la Unidad de Nefrología se cribaron una vez por semana con cultivos de muestras perianales en busca de colonización por ERV. Se recogieron un total de 391 muestras perianales que se cultivaron en 2 tipos diferentes de medios de cultivo sólidos en placa para detectar el mayor número posible de portadores. Estos medios de cultivo fueron los siguientes: Slanetz and Bartley (OXOID, Hampshire, Inglaterra) con un disco de 30 μg de vancomicina (bioMérieux, Marcy l’Etoile, Francia) y campylosel (bioMérieux, Marcy l’Etoile, Francia). Se trabajaron entre 3–5 colonias sospechosas por paciente y medio, que se subcultivaron posteriormente para la realización de identificación y antibiograma. En septiembre de 2007 se realizó otro cribado en la Unidad de Nefrología y se recogieron 32 muestras perianales de 19 pacientes, que se cultivaron en agar chromID VRE (bioMérieux, Marcy l’Etoile, Francia).
Identificación y antibiogramaSe realizó a través de Vitek 2 (bioMérieux, Marcy l’Etoile, Francia). Los antibióticos testados fueron los siguientes: ampicilina, ciprofloxacino, clindamicina, eritromicina, levofloxacino, norfloxacino, teicoplanina, tetraciclina, vancomicina, quinupristina-dalfopristina, linezolid, gentamicina y estreptomicina. La concentración mínima inhibitoria (CMI) de vancomicina y teicoplanina se determinó por Etest (AB Biodisk, Solna, Suecia) de acuerdo con las instrucciones del fabricante. La tipificación molecular de la resistencia a glucopéptidos se realizó mediante reacción en cadena de la polimerasa (PCR) múltiple, ya descrita previamente17.
Tipificación por electroforesis de campo pulsanteLa extracción del ADN cromosómico de los aislados de EFRV se realizó en bloques de agarosa de acuerdo con el protocolo descrito por Smith et al18. Los insertos se digirieron con 30U de enzima de restricción SmaI (Promega, Madison, EEUU) y los fragmentos de ADN se separaron en un gel de agarosa al 1% mediante electroforesis de campo pulsante (PFGE); se utilizó un aparato CHEF-DRII (Bio-Rad Laboratories, California, EEUU) bajo las siguientes condiciones: 20h de pulsos de 0,5 a 15s, temperatura de 14°C y 6V/cm. Terminada la electroforesis, los geles se tiñeron con bromuro de etidio y se visualizaron y fotografiaron bajo luz UV. Los patrones de bandas se compararon de acuerdo con los criterios de Tenover et al19 y se analizaron mediante el programa informático InfoQuest FP (Bio-Rad). Los patrones obtenidos se compararon mediante el método unweighted pair group method with arithmetic averages utilizando el coeficiente de Dice.
Multilocus sequence typing y la amplificación de los genes hyl y espUn subconjunto de aislamientos de EFRV representativos de cada PFGE tipo se estudió mediante multilocus sequence typing (MLST), y se utilizó el esquema previamente descrito para su realización20. A cada secuencia diferente se le asignó un número de alelo y a cada perfil alélico se le asignó una secuencia tipo (STs); se consultó la base de datos de MLST para E. faecium (http://www.efaecium.mlst.net). La presencia de los genes hyl y esp se investigó mediante PCR y se utilizaron los cebadores específicos previamente descritos21.
ResultadosDescripción del brote, medidas de control y características de los pacientesEn julio de 2005, un EFRV con fenotipo VanA se aisló por primera vez en nuestro hospital, procedente de un paciente con una infección del sitio quirúrgico ingresado en la Unidad de Nefrología. Este paciente murió al día siguiente. En septiembre, se detectó una segunda infección del sitio quirúrgico por EFRV VanA, y en octubre se detectaron 2 casos más de infección (una del tracto urinario y una del sitio quirúrgico) en la misma unidad. Estas infecciones evolucionaron favorablemente con tratamiento antibótico (linezolid). Se adoptaron medidas de control de la infección, por lo que se aplicó aislamiento de contacto, y el 17 de octubre se realizó el primer cribado con muestras perianales a todos los pacientes (21 pacientes) ingresados en la Unidad de Nefrología. Se encontraron en ese momento 9 pacientes colonizados por EFRV VanA, incluidos 3 de los 4 pacientes con infección hospitalaria (al primer paciente no se lo pudo recoger, pues había fallecido). Después de este primer barrido y hasta el 15 de diciembre de 2005 se realizó el cribado a todos los pacientes ingresados en la unidad una vez por semana, y en este período se encontraron 6 pacientes más colonizados. A partir de este momento y hasta marzo de 2006, cuando se finalizó con los cribados, no se encontró ningún paciente más. Las colonias de ERV se aislaron indistintamente en los 2 medios de cultivo utilizados en este momento aunque, en nuestra experiencia, Slanetz and Bartley las diferenciaba mejor. A los pacientes colonizados por EFRV se les aplicó aislamiento de contacto sin tratamiento de su estado de portador. El 24 de octubre de 2005, la Unidad de Nefrología se trasladó definitivamente a otra planta de hospitalización.
Los 16 pacientes en los que se aisló EFRV VanA, ya sea por colonización o por infección hospitalaria, eran trasplantados renales y a la mayoría de ellos se los había intervenido quirúrgicamente. La media de edad de los pacientes fue de 53,5 años (rango: 35–61 años): 10 pacientes (62,5%) eran mujeres y 6 pacientes (37,5%) eran hombres. A todos se les había realizado hemodiálisis antes del trasplante renal, con una duración media de 43 meses (rango: 7–128 meses). El 100% de los pacientes había recibido tratamiento antibiótico múltiple los meses previos al aislamiento de EFRV, y concretamente el 44% había recibido glucopéptidos en el último mes: 5 pacientes recibieron vancomicina y 2 pacientes recibieron teicoplanina. La media de días de hospitalización previos al aislamiento de EFRV fue de 35 días (rango: 4–83 días).
En el mismo período otros 3 pacientes con aislamiento de EFRV VanA se detectaron por primera vez en otros 2 hospitales universitarios de Canarias. En el HUIGC se aisló EFRV VanA en una paciente de 16 años a la que se había trasplantado riñón en nuestro hospital (HUC) en septiembre de 2005. En el HUNSC se detectaron 2 pacientes más con aislamientos de EFRV VanA en febrero y mayo de 2006. El primer paciente era un varón de 37 años ingresado en la Unidad de Cirugía General del HUNSC, y coincidió en tiempo y lugar con uno de los pacientes involucrados en el brote por EFRV del HUC. El segundo paciente era otro varón de 81 años de edad hospitalizado en la Unidad de Digestivo del HUNSC sin aparente relación con los casos anteriores. Ante los casos de EFRV, tanto en el HUIGC como en el HUNSC se adoptaron medidas de control de la infección, entre ellas el aislamiento de contacto y la toma de muestras de cribado de los pacientes infectados para descartar colonización rectal.
A partir de diciembre de 2005 no se aislaron EFRV en la Unidad de Nefrología de nuestro hospital (HUC). Aún así, se decidió realizar un nuevo cribado con muestras perianales en los pacientes ingresados en esta planta en septiembre de 2007, donde se encontró sólo un paciente colonizado por Enterococcus gallinarum. Sin embargo, en marzo de 2007 se detectaron 2 nuevos casos de EFRV en otras unidades de nuestro hospital: un paciente de 72 años con una infección por VanB, y otro de 84 años con una infección por EFRV VanA hospitalizado en Cirugía General. En el HUNSC, en mayo de 2007, también se aisló otro EFRV VanA en un paciente de 41 años de edad.
Identificación y resistencia antibióticaTodos los aislados de ERV de este estudio se identificaron como E. faecium mediante Vitek 2. Los aislamientos de los 16 pacientes involucrados en el brote del HUC y los pacientes del HUIGC y el HUNSC fueron resistentes a ampicilina, ciprofloxacino, clindamicina, eritromicina, levofloxacino, norfloxacino, teicoplanina, tetraciclina, vancomicina y sensibles a quinupristina-dalfopristina y linezolid. Ninguno presentaba alto nivel de resistencia a gentamicina o estreptomicina. Las CMI calculadas por Etest de todos los aislamientos fueron las siguientes: vancomicina superior a 256μg/ml y teicoplanina de 32μg/ml. El fenotipo VanA se confirmó por la presencia de vanA mediante PCR múltiple.
El EFRV VanA que se aisló en el HUC en 2007 (CMI: vancomicina superior a 256μg/ml y teicoplanina de 32μg/ml) fue resistente a los mismos antibióticos que los precedentes pero sensible a tetraciclina, y presentaba alto nivel de resistencia a gentamicina y estreptomicina. El EFRV VanB aislado en el HUC (CMI: vancomicina superior a 256μg/ml y teicoplanina inferior a 0,5μg/ml) también tenía un perfil de resistencia igual a los anteriores pero sensible a teicoplanina, y presentaba alto nivel de resistencia a la estreptomicicna. La presencia de los genes vanA y vanB confirmó los fenotipos de estos aislados, mediante PCR múltiple.
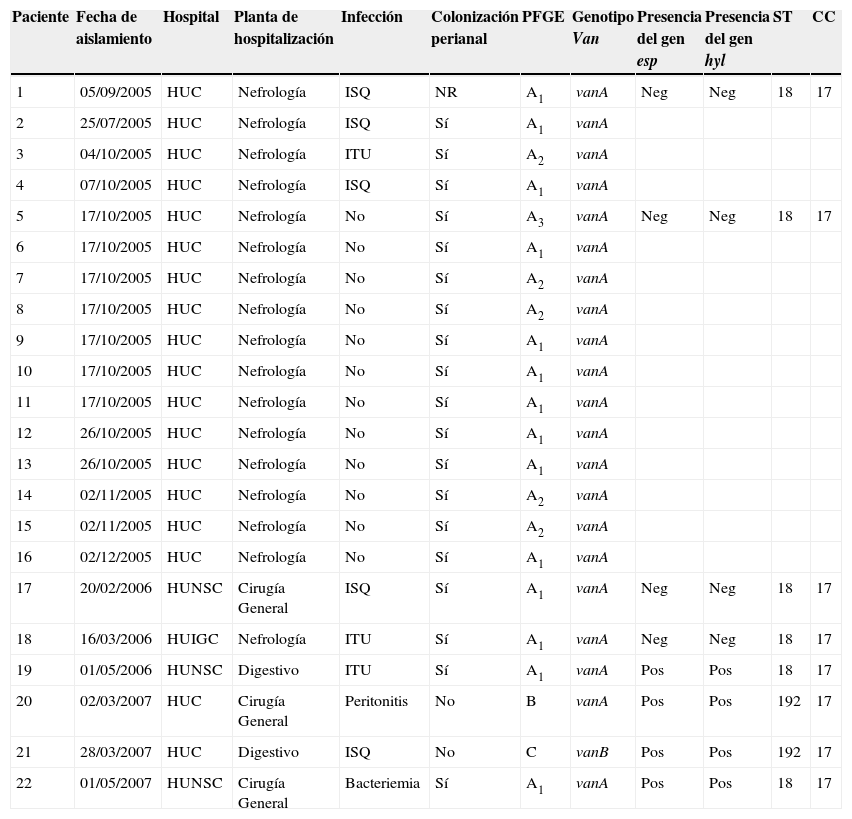
Tipificación molecularLos resultados de PFGE revelan que los 16 EFRV aislados en el brote producido en 2005 en el HUC y los aislamientos de los otros 2 hospitales (HUIGC y HUNSC) se agruparon en un mismo PFGE tipo con 3 subtipos (A1, A2 y A3) estrechamente relacionados (2 o 3 bandas de diferencia entre ellos y más del 90% de similitud), por lo que consideramos que todos ellos, probablemente, formarían parte de un brote. Los 3 pacientes colonizados rectales con una infección hospitalaria por EFRV en el HUC estaban colonizados por el mismo subtipo de PFGE. Los aislamientos de EFRV encontrados en el HUC en el año 2007 y estudiados por PFGE eran diferentes entre ellos y diferentes de los involucrados en el brote de 2005 (B y C). El resto de los resultados obtenidos por las técnicas de epidemiología molecular junto con otros datos epidemiológicos de los pacientes se observan en la tabla 1.
Características epidemiológicas de los pacientes con infección o colonización por Enterococcus faecium resistente a vancomicina y características moleculares de los aislados
| Paciente | Fecha de aislamiento | Hospital | Planta de hospitalización | Infección | Colonización perianal | PFGE | Genotipo Van | Presencia del gen esp | Presencia del gen hyl | ST | CC |
| 1 | 05/09/2005 | HUC | Nefrología | ISQ | NR | A1 | vanA | Neg | Neg | 18 | 17 |
| 2 | 25/07/2005 | HUC | Nefrología | ISQ | Sí | A1 | vanA | ||||
| 3 | 04/10/2005 | HUC | Nefrología | ITU | Sí | A2 | vanA | ||||
| 4 | 07/10/2005 | HUC | Nefrología | ISQ | Sí | A1 | vanA | ||||
| 5 | 17/10/2005 | HUC | Nefrología | No | Sí | A3 | vanA | Neg | Neg | 18 | 17 |
| 6 | 17/10/2005 | HUC | Nefrología | No | Sí | A1 | vanA | ||||
| 7 | 17/10/2005 | HUC | Nefrología | No | Sí | A2 | vanA | ||||
| 8 | 17/10/2005 | HUC | Nefrología | No | Sí | A2 | vanA | ||||
| 9 | 17/10/2005 | HUC | Nefrología | No | Sí | A1 | vanA | ||||
| 10 | 17/10/2005 | HUC | Nefrología | No | Sí | A1 | vanA | ||||
| 11 | 17/10/2005 | HUC | Nefrología | No | Sí | A1 | vanA | ||||
| 12 | 26/10/2005 | HUC | Nefrología | No | Sí | A1 | vanA | ||||
| 13 | 26/10/2005 | HUC | Nefrología | No | Sí | A1 | vanA | ||||
| 14 | 02/11/2005 | HUC | Nefrología | No | Sí | A2 | vanA | ||||
| 15 | 02/11/2005 | HUC | Nefrología | No | Sí | A2 | vanA | ||||
| 16 | 02/12/2005 | HUC | Nefrología | No | Sí | A1 | vanA | ||||
| 17 | 20/02/2006 | HUNSC | Cirugía General | ISQ | Sí | A1 | vanA | Neg | Neg | 18 | 17 |
| 18 | 16/03/2006 | HUIGC | Nefrología | ITU | Sí | A1 | vanA | Neg | Neg | 18 | 17 |
| 19 | 01/05/2006 | HUNSC | Digestivo | ITU | Sí | A1 | vanA | Pos | Pos | 18 | 17 |
| 20 | 02/03/2007 | HUC | Cirugía General | Peritonitis | No | B | vanA | Pos | Pos | 192 | 17 |
| 21 | 28/03/2007 | HUC | Digestivo | ISQ | No | C | vanB | Pos | Pos | 192 | 17 |
| 22 | 01/05/2007 | HUNSC | Cirugía General | Bacteriemia | Sí | A1 | vanA | Pos | Pos | 18 | 17 |
CC: complejo clonal; HUC: Hospital Universitario de Canarias; HUIGC: Hospital Universitario Insular de Gran Canaria; HUNSC: Hospital Universitario Nuestra Señora de la Candelaria; ISQ: infección del sitio quirúrgico perianal; ITU: infección del tracto urinario; NR: no realizado; PFGE: electroforesis en campo pulsante (y macrorrestricción); ST: secuencia tipo.
Los brotes epidémicos por ERV en España no son frecuentes y los que están descritos en la literatura médica son brotes por ERV vanB22–24. Los aislamientos de ERV en el HUC antes de julio de 2005 se producían de forma esporádica. En este estudio describimos el primer aislamiento y el primer brote de EFRV vanA ocurrido en el HUC, en el que estuvieron involucrados pacientes trasplantados renales, y describimos su relación con los primeros aislamientos de EFRV encontrados en otros 2 hospitales universitarios de las Islas Canarias.
La infección y colonización por ERV es un problema creciente, sobre todo en grupos específicos de pacientes, tales como los trasplantados renales. Estos pacientes tienen un alto riesgo de tener una colonización gastrointestinal y de que esta colonización persista en el tiempo8,9,25. Los factores de riesgos descritos para una colonización rectal en trasplantados renales o pacientes con fallo renal crónico son la hemodiálisis, el tratamiento previo con vancomicina o cefalosporinas, y otros factores de riesgo similares a los de otros grupos de pacientes con riesgo de adquisición de ERV, tales como multitratamiento antibiótico, hospitalizaciones frecuentes y duraderas y enfermedades crónicas subyacentes8,26–30. Varios estudios demuestran que la mayoría de los pacientes en los que se aísla ERV están colonizados más que infectados, y demuestran que por cada caso clínico de infección por ERV, en pacientes con riesgo, existen de 10 a 20 casos de colonización4,9,27,30. Estas observaciones son consistentes con nuestros resultados: todos los pacientes involucrados en el brote del HUC eran trasplantados renales con factores de riesgo para la adquisición de ERV, como hemodiálisis, tratamiento con antibióticos y prolongada hospitalización, previos al aislamiento de ERV, y en el brote se produjeron más colonizaciones por EFRV que infecciones (4 pacientes con infección frente a 15 colonizaciones). A partir del 17 de octubre de 2005, cuando se adoptaron las medidas de control de la infección, no se detectaron más infecciones hospitalarias por EFRV en la planta de hospitalización de Nefrología de nuestro hospital, y a partir de diciembre de ese mismo año y hasta el momento actual tampoco se han detectado más colonizaciones. En octubre de 2005 la planta de Nefrología se trasladó definitivamente a otra planta de hospitalización, planeada previamente como parte de una reorganización del hospital. Creemos que este traslado de ubicación de los pacientes fue beneficioso para el control del brote, ya que no debemos olvidar que los enterococos pueden contaminar el ambiente en torno al paciente y sobrevivir durante largos períodos de tiempo sobre las superficies secas14,31.
La tipificación molecular por PFGE, combinada con la información epidemiológica obtenida de los pacientes confirmó, por un lado, que estábamos ante un brote por EFRV vanA, y, por otro, su vínculo con los primeros EFRV vanA aislados en los otros 2 hospitales universitarios ubicados también en Canarias. Por MLST, se agruparon en una secuencia tipo ST18 que pertenece al CC17 y que es un linaje importante de EFRV adaptado a los hospitales. El CC17 representa una población policlonal que está relacionada con brotes e infecciones por EFRV en los 5 continentes y que se caracterizan por ser, en su mayoría, resistentes a la ampicilina y a las quinolonas, y por contener los genes de virulencia esp y hyl, hecho este último que se ha propuesto como marcador del CC1721,32. En nuestro caso, este marcador no nos hubiese servido, ya que los aislamientos de EFRV que produjeron el brote en el HUC no contenían estos genes de virulencia. Dos de los aislados ST18 del HUNSC y los ST192 aislados en el HUC en el año 2007, también pertenecientes al CC17, sí los contenían. Algunos estudios indican que se puede producir una transferencia horizontal de estos islotes de virulencia que podría explicar la aparición de estos genes en los últimos aislamientos de EFRV de nuestro estudio. También la adquisición de los genes vanA o vanB situados en transposones parece deberse a una transferencia horizontal hacia los linajes de E. faecium adaptados a los hospitales con potencial pandémico, y ambas, la diseminación clonal y la transferencia horizontal de la resistencia a la vancomicina, podrían desempeñar un papel importante. La diseminación del CC17 resistente a glucopéptidos se ha descrito en muchos países europeos y en otros continentes, y ha contribuido al incremento de las infecciones y colonizaciones de EFRV en todo el mundo10–12,33–41.
En este estudio hemos querido describir el primer brote de EFRV vanA en las Islas Canarias. El HUC se ha caracterizado en la última década por tener una alta endemicidad por Staphylococcus aureus resistente a la meticilina42,43. El control para evitar la emergencia y diseminación de clones de ERV adaptados a los hospitales pertenecientes a linajes como CC17 es esencial, especialmente si pensamos en la posibilidad de la transferencia horizontal de los transposones que contengan vanA o vanB y que transformen a los S. aureus resistentes a la meticilina en resistentes a la vancomicina11,14.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
FinanciaciónEste estudio ha sido subvencionado, en parte, por el proyecto de investigación de la Fundación Canaria de Investigación y Salud (FUNCIS) (PI43/07).
Los autores de este trabajo querríamos agradecer al Dr. Eduardo Saludo Ruiz, director del Laboratorio de la Unidad de Investigación del Hospital Universitario de Canarias, la aportación de sus conocimientos técnicos en este manuscrito. A su vez, estamos muy agradecidos por la ayuda prestada por parte del personal de la planta de Nefrología del Hospital Universitario de Canarias, y, en especial, al Dr. Aurelio Rodríguez Hernández y al Dr. José Manuel González Posadas.