El aumento de la prevalencia de infecciones causadas por enterobacterias productoras de betalactamasas de espectro extendido (BLEE), tanto en la comunidad como en el ámbito hospitalario, plantea la necesidad, en determinadas investigaciones epidemiológicas de brotes nosocomiales, de disponer de métodos rápidos para la detección de portadores de estos microorganismos1.
En los análisis microbiológicos de flora entérica, el empleo de medios de cultivo selectivos y diferenciales permite acortar el periodo de análisis. Hay descritos en la literatura diferentes medios de este tipo para la detección de enterobacterias productoras de BLEE, siendo los más habituales los que están suplementados con cefotaxima o ceftazidima. Los medios base a los que se añaden estos antibióticos suelen ser agar MacConkey, agar de Driglasky o agar nutritivo con vancomicina y anfotericina B2.
El medio chromID ESBL (bioMérieux) contiene una mezcla de antibióticos que incluye cefpodoxima, además de sustratos cromogénicos que permiten la identificación directa de las enterobacterias productoras de ESBL más frecuentes: Escherichia coli, especie productora de betaglucuronidasa, el grupo de géneros que producen betaglucosidasa como Klebsiella, Enterobacter, Serratia y Citrobacter y, finalmente, los géneros Proteus, Providencia y Morganella, que producen una desaminasa. Mientras que la identificación en cuanto a especie o grupo es directa, la producción de ESBL debe confirmarse con una o más pruebas adicionales2,3.
Nuestro objetivo fue estudiar la rentabilidad de este medio para detectar portadores en la unidad de neonatología y compararlo con el medio habitual utilizado en nuestro laboratorio (agar MacConkey con 4mg/l de cefotaxima).
Se procesaron 80 muestras de frotis rectales procedentes de pacientes ingresados en la unidad de neonatología, que se inocularon simultáneamente en agar MacConkey con 4mg/l de cefotaxima (MCC) y en el medio chromID ESBL (bioMérieux). Las placas a 35°C se incubaron y se efectuaron lecturas a las 24 y a las 48 h. Como cepas control se utilizaron E. coli ATCC 25922 (no productor de BLEE) y K. pneumoniae ATCC 700603 (productor de BLEE). La identificación de los aislados se realizó mediante pruebas bioquímicas (oxidasa, TSI, ureasa e indol) y galerías API (bioMérieux)3. El cribado de la producción de BLEE se llevó a cabo mediante la técnica de doble disco con ceftazidima, ceftazidima clavulánico y cefotaxima, cefotaxima clavulánico en agar Mueller Hinton4 y, cuando se sospechaba la hiperproducción de AmpC, en agar Mueller Hinton con 200mg/l de cloxacilina. La evaluación de la hiperproducción de AmpC se realizó comparando los halos de cefotaxima, cefotaxima clavulánico y ceftazidima ceftazidima clavulánico en medio Mueller Hinton con y sin 200mg/l de cloxacilina. Si se observaba un aumento del halo en los cuatro discos en presencia de cloxacilina, se consideraba hiperproducción de AmpC5.
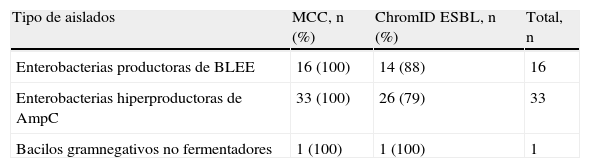
Se detectó crecimiento bacteriano a las 24 h en 50 muestras (62%), correspondiendo a enterobacterias productoras de BLEE en 16 muestras (20%). En total, se detectaron 18 aislados productores de BLEE, siendo las especies identificadas: 11 (61%) E. coli, 4 (22%) K. pneumoniae, 1 (5,5%) K. oxytoca, 1 (5,5%) C. freundii y 1 (5,5%) Enterobacter spp. En dos muestras (12,5%) se recuperaron dos especies productoras de BLEE (C. freundii y K. pneumoniae en una y K. pneumoniae y K. oxytoca en otra). No se detectó ningún aislado adicional en la lectura de las 48 h. La sensibilidad para detectar enterobacterias BLEE fue mayor con el MCC que con chromID (tabla 1). Los aislados hiperproductores de AmpC se identificaron como E. cloacae, detectándose con menos frecuencia en el medio chromID que en el MCC.
Comparación de los resultados en los dos medios selectivos según el tipo de aislados recuperados
| Tipo de aislados | MCC, n (%) | ChromID ESBL, n (%) | Total, n |
| Enterobacterias productoras de BLEE | 16 (100) | 14 (88) | 16 |
| Enterobacterias hiperproductoras de AmpC | 33 (100) | 26 (79) | 33 |
| Bacilos gramnegativos no fermentadores | 1 (100) | 1 (100) | 1 |
El medio chromID permitió la identificación directa de E. coli en 24 h sin pruebas adicionales, con un 100% de concordancia con las pruebas bioquímicas. Este medio mostró una sensibilidad del 88% y una especificidad del 58% para el cribado en 24 h de portadores de enterobacterias BLEE y fue más selectivo para el crecimiento de hiperproductores de cefalosporinasas que el medio que se usa actualmente en nuestro laboratorio. Los resultados obtenidos fueron similares a los de otros estudios6. La utilización del medio chromID ESBL identifica directamente E. coli BLEE en 24 h y permite descartar de forma rápida a portadores de este microorganismo en programas de vigilancia activa de patógenos nosocomiales, donde habitualmente interesa detectar a portadores de Klebsiella spp. y Enterobacter spp. productores de BLEE. Este aspecto es útil dada la prevalencia observada en nuestro país de portadores comunitarios de E. coli BLEE7. No obstante, es necesario una identificación ampliada para todas las enterobacterias excepto en el caso de colonias betaglucuronidasa positiva (rojo-púrpura), que son directamente identificadas como E. coli8.
Adelina López y Carmen Lupión del Hospital Virgen Macarena de Sevilla por el trabajo técnico realizado.