El incremento de la prevalencia de aislados resistentes a los medicamentos antituberculosos clásicos ha hecho necesario encontrar alternativas terapéuticas, y a este respecto las fluoroquinolonas son uno de los grupos de medicamentos más prometedores; sin embargo, el uso de fluoroquinolonas en el tratamiento de otras infecciones ha derivado en un incremento en los porcentajes de resistencia en muchos patógenos1. Pretendemos determinar la frecuencia de la resistencia a fluoroquinolonas en nuestro medio durante 18años. Para ello hemos estudiado 317 aislados de Mycobacterium tuberculosis reunidos en el área de salud de Elche (Alicante) entre 1992 y 2010.
Se determinó la presencia de mutaciones en las regiones de los genes gyrA y gyrB asociadas a la resistencia a fluoroquinolonas (QRDR), ya que la concordancia entre métodos fenotípicos (sensibilidad in vitro a levofloxacino y a moxifloxacino) y genotípicos es muy alta2,3.
El ADN se extrajo usando el sistema descrito por Van Embden et al.4, y se amplificaron las regiones QRDR de los 2 genes en un termociclador GenAmp® PCR System (Applied Biosystems, Foster City, California, EE.UU.) con primers diseñados en nuestro laboratorio:
gyrA_F_TBC: 5’-cagctacatcgactatgcga-3’, gyrA_R_TBC: 5’-gggcttcggtgtacctcat-3’, gyrB_F_TBC: 5’-accgacatcggtggattgcc-3’ y gyrB_R_TBC: 5’-actgcgctgccacttgagtt-3’
Las secuencias de los aislados fueron comparadas con la secuencia wild type (nativa) (GenBank:L27512.1), usando la base de datos TB Drug Resistance Mutation Database (http://www.tbdreamdb.com/). Se utilizó como control una cepa de M.tuberculosis ATCC 413235.
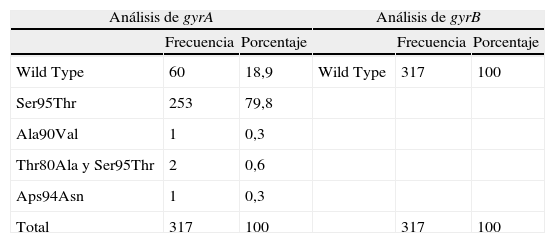
Detectamos la presencia de 4 aislados con mutaciones en el gen gyrA asociadas con la resistencia a fluoroquinolonas, genéticamente no relacionadas entre sí. No se encontraron mutaciones en el gen gyrB, y por tanto el porcentaje de resistencia en nuestro medio fue del 1,26% (tabla 1).
Análisis de los fragmentos QRDR de los genes gyrA y gyrB en Mycobacterium tuberculosis
| Análisis de gyrA | Análisis de gyrB | ||||
| Frecuencia | Porcentaje | Frecuencia | Porcentaje | ||
| Wild Type | 60 | 18,9 | Wild Type | 317 | 100 |
| Ser95Thr | 253 | 79,8 | |||
| Ala90Val | 1 | 0,3 | |||
| Thr80Ala y Ser95Thr | 2 | 0,6 | |||
| Aps94Asn | 1 | 0,3 | |||
| Total | 317 | 100 | 317 | 100 | |
El polimorfismo más común (79,81% de los aislados) fue Ser95Thr en el codón 95, pero este no se asocia con la resistencia a fluoroquinolonas; el 98,7% de estos aislados tuvieron ACC (treonina) y el 2% AGC (serina). No se encontró una asociación estadísticamente significativa entre este polimorfismo y las variables estudiadas, a excepción del lugar pulmonar de infección (riesgo de 1,939; intervalo de confianza al 95%: 1,031-3,646).
Nuestros datos muestran que a pesar del amplio uso hecho de estos compuestos, el porcentaje de resistencia de M.tuberculosis en nuestro medio es bajo y, por tanto, las fluoroquinolonas continúan siendo una opción terapéutica válida cuando los antibióticos antituberculosos clásicos no pueden ser usados; esto puede explicarse por la gran estabilidad genética de M.tuberculosis en el paciente hospedador, tal y como se ha demostrado en estudios de hipermutabilidad6.
Nuestro trabajo muestra que hay una baja frecuencia de resistencia a estos compuestos, como ya se ha descrito previamente en otras regiones, como Estados Unidos o África7-10; sin embargo, para evitar el incremento de la misma sería recomendable implementar medidas que ayuden a utilizar correctamente estos compuestos.
FinanciaciónEste trabajo fue financiado por la Fundación para la Investigación en Salud (FIS) del Gobierno Español (subvención PI071158), por la Fundación de la Comunidad Valenciana para la investigación biomédica, la docencia y la cooperación internacional y para el desarrollo del Hospital General Universitario de Elche (FIBELX09/09) y por subvención de la Conselleria d’Educació de la Comunitat Valenciana (BFPI/2008/115).