Hemocultivos
Situación actual de los sistemas de hemocultivos
En Estados Unidos se diagnostican anualmente alrededor de 250.000 episodios de bacteriemia nosocomial, número que prácticamente se duplica si se consideran aquellas de origen comunitario1. El incremento de pacientes inmunocomprometidos justifica parcialmente el aumento de este tipo de infecciones. La mortalidad cruda de este proceso oscila del 25 al 50% de las que un tercio aproximadamente son atribuibles a la infección. La instauración de un tratamiento correcto y precoz disminuye la mortalidad atribuible a estas infecciones y es un factor de gran relevancia en el pronóstico del enfermo. El enorme incremento de la resistencia a antimicrobianos de numerosos microorganismos han limitado y dificultado el uso de tratamientos empíricos eficaces.
A pesar del desarrollo en los últimos años de diferentes tecnologías basadas en la detección, amplificación y/o secuenciación de los ácidos nucleicos, los hemocultivos continúan teniendo una gran relevancia como técnica diagnóstica en las bacteriemias y fungemias2. Uno de los principales motivos radica en los diferentes patrones de sensibilidad de los microorganismos que se aíslan en sangre, lo que requiere de manera indefectible la realización de estudios de sensibilidad a los agentes antimicrobianos de los microorganismos aislados para poder instaurar un tratamiento efectivo.
El procesamiento de los hemocultivos ha sufrido modificaciones muy importantes en las últimas décadas que han permitido incrementar el número de bacteriemias verdaderas diagnosticadas, acortar los tiempos de detección y mejorar todos los aspectos relacionados con la bioseguridad del personal de laboratorio.
La desinfección de la piel sigue siendo de extraordinaria importancia para evitar la contaminación de los hemocultivos con flora de la piel. El uso de alcohol isopropílico al 70% seguido de algún yodóforo sigue siendo tan eficaz como la mayoría de los sistemas comerciales de desinfección disponibles3.
El número de hemocultivos que se debe tomar sigue siendo un tema controvertido. Desde la publicación de los trabajos realizados por Washington et al en la Clínica Mayo de Estados Unidos donde se demostraba que la realización de 3 tandas de hemocultivos aumentaba de manera significativa la sensibilidad de esta técnica, se han desarrollado numerosos sistemas automatizados de hemocultivos que al mejorar la recuperación de microorganismos de sangre podrían permitir la disminución del número de tomas de hemocultivos a dos4. Otros autores (E. Bouza, comunicación personal) consideran que el uso de sólo 2 tandas de hemocultivos supone la pérdida de hasta el 20% de las bacteriemias. Sólo en procesos especiales (endocarditis, etc.) estaría justificada la utilización de un número más elevado de hemocultivos. Aunque la mayoría de los diagnósticos se establecen con la primera tanda de hemocultivos, la importancia clínica actual de bacterias como estafilococos coagulasa negativo u otros contaminantes de piel se verá confirmada cuando se aíslen en varios hemocultivos consecutivos. De hecho, actualmente existen numerosos laboratorios donde no se admiten hemocultivos realizados con una sola extracción, porque ofrecen una baja sensibilidad y gran dificultad de interpretación. Es importante, por lo tanto, dejar de hablar de hemocultivos seriados y no seriados y admitir que los hemocultivos son seriados por definición.
Existen pocos estudios que evalúen de manera sistemática cuándo y con qué intervalos deben tomarse los hemocultivos. En casos de sepsis se ha establecido de manera arbitraria intervalos de 30-60 min, aunque existen estudios que no han encontrado diferencias entre realizar todas las tomas seguidas o con intervalos establecidos durante un período5 de 24 h.
Se ha descrito una relación significativa entre el volumen de sangre extraído para un hemocultivo y la rentabilidad de los mismos. Se recomienda un volumen por toma en adultos de 20-30 ml. Extraer volúmenes superiores es innecesario y puede provocar anemia nosocomial3. En caso de niños se recomiendan 2-4 ml para mayores de 2 años, 2-3 ml para menores de 2 años y 1-2 ml para recién nacidos, que suelen sembrarse en una sola botella.
Existe una gran cantidad de medios de cultivo comerciales y ninguno de ellos permite el crecimiento de todos los microorganismos. En general, todos los medios de cultivo comerciales tienen una base de caldo tripticasa de soja o infusión cerebro-corazón y ofrecen excelentes resultados, aunque la comparación entre ellos es difícil al adicionar los fabricantes diferentes sustancias para mejorar la recuperación de microorganismos3. Entre estas sustancias se encuentran una serie de aditivos dirigidos principalmente a la inactivación de los antimicrobianos. En nuestro país los más utilizados son los medios con resinas del sistema Bactec (Frascos Plus; Becton Dickinson) y los medios con carbón activado del sistema BacT/Alert (Frascos FAN; Organon Teknika). En estudios comparativos ambos medios ofrecen resultados similares cuando son utilizados en sus respectivos aparatos de monitorización continua6,7. Uno de los inconvenientes del carbón activado radica en que dificulta la interpretación de las tinciones de Gram de los frascos positivos.
Otro tema controvertido es la atmósfera de incubación de los frascos. El uso sistemático de un frasco para anaerobios en cada toma es cuestionado por numerosos microbiólogos que prefieren restringir su uso a pacientes con riesgo de sufrir infecciones por este tipo de microorganismos.
Los protocolos tradicionales de incubación de frascos de hemocultivos recomendaban un mínimo de 7 días, que se incrementaba hasta 4 semanas en el caso de endocarditis, fungemias, brucelosis y casos especiales. Los sistemas actuales de monitorización continua y agitación permanente de los frascos han disminuido de manera significativa el tiempo de incubación. En nuestro hospital, en los primeros 6 meses de 2001, el 70% de los frascos positivos se detectaron en las primeras 24 h de incubación (tabla 1) utilizando el sistema BacT/Alert. De hecho, la mayoría de los sistemas de monitorización continua sugieren un tiempo de incubación de 5 días o menos. En un estudio de 18.000 hemocultivos procesados en frascos FAN del sistema BacT/Alert en Estados Unidos los porcentajes de aislamientos a los días 1, 2, 3, 4 y 5 fueron 71, 22, 5, 1 y 1%, respectivamente8. De hecho, el 97,5% de los hemocultivos se detectaron a los 3 días de incubación. En 13 de los 14 pacientes en los que se detectó el hemocultivo positivo en los días 4 y 5 de incubación, el tratamiento antimicrobiano no se modificó a consecuencia del resultado del hemocultivo. Los autores defienden que no sería necesario incubar los frascos FAN durante mas de 3 días. Estudios similares se han publicado utilizando otros sistemas de monitorización continua9.
Existen numerosos sistemas de detección del crecimiento de microorganismos en frascos de hemocultivos. Los sistemas manuales, según un estudio mundial de hemocultivos realizado en 1992, siguen siendo utilizados por el 30% de los laboratorios de microbiología7. Con este sistema, la detección del crecimiento bacteriano se hace macroscópicamente mediante visualización de turbidez, de hemólisis, de producción de gas o formación de colonias. Su principal desventaja es la laboriosidad, que dificulta su utilización en grandes hospitales. Existen variaciones de los sistemas manuales dirigidos al ahorro de tiempo como Oxoid Signal System (Oxoid), Septi-Chek (Becton Dickinson) o lisis-centrifugación, que han ofrecido buenos resultados en diferentes estudios comparativos10.
Los sistemas instrumentales de detección de crecimiento fueron introducidos en la década de 1970. Los primeros sistemas eran radiométricos, lo cual dificultaba su utilización en el laboratorio. Estos sistemas se basan en el marcaje mediante isótopos de algún hidrato de carbono del medio de cultivo, que al ser utilizado metabólicamente por los microorganismos producía la liberación de dióxido de carbono radiactivo que era detectado por el aparato. En la siguiente década se sustituyeron los sistemas radiométricos por sistemas que detectaban la producción de dióxido de carbono mediante infrarrojos, lo que evitaba el uso de isótopos radiactivos. Ambos sistemas permitían la lectura de todos los frascos cada 12 h.
Actualmente existen en el mercado una gran cantidad de sistemas de hemocultivos de monitorización continua. Uno de los principales problemas en la actualidad radica en la elección del sistema más adecuado a nuestras necesidades. La realización de estudios en paralelo bien diseñados es muy difícil, entre otras cosas porque alrededor del 90% de los hemocultivos son negativos y el 3% corresponden a contaminaciones. Un estudio bien diseñado debe incluir más de 20 especies microbianas, incluyendo levaduras, anaerobios y microorganismos fastidiosos. Los instrumentos se considerarán fiables cuando detecten más del 98% de todos los verdaderos positivos demostrados mediante subcultivo. Los microbiólogos franceses recomiendan evaluar al menos 1.000 hemocultivos con realización de subcultivos ciegos antes de validar un sistema11. La Food and Drug Administration (FDA) americana requiere estudiar 600 aislamientos clínicamente valorables de 370 episodios sépticos diferentes, lo que obliga a evaluar alrededor de 8.000 sets de hemocultivos asumiendo un rango de positividad del 6-16%. La FDA valida un sistema de hemocultivos cuando detecta más del 95% de los microorganismos detectados con el sistema de referencia12.
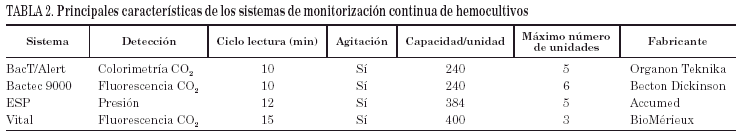
La selección de un sistema se basa en gran medida en los estudios publicados en la literatura científica de calidad, aunque se recomienda realizar algunos estudios con inoculación de frascos en cada laboratorio como una importante manera de verificación de un sistema antes de adoptarlo de manera definitiva. Uno de los principales criterios, que como veremos posteriormente justifica en gran medida el procesamiento de los hemocultivos dentro de los hospitales, es el tiempo necesario para la detección de crecimiento. La mayoría de los estudios consideran el tiempo de positividad contando desde el momento en que un frasco se introduce en el aparato de lectura, sin tener en cuenta el tiempo que transcurre desde la obtención del hemocultivo hasta su introducción en el aparato de lectura que puede variar extraordinariamente en función de la forma de trabajar de cada institución. Como veremos posteriormente, el intervalo entre la toma de un hemocultivo y su introducción en el sistema de monitorización continua puede ser importante en la eficiencia de estos sistemas. Como criterios de aceptabilidad el sistema a elegir debe ofrecer menos del 1% de falsos negativos, menos del 2% de falsos positivos, menos del 3% de contaminaciones y un tiempo medio de detección inferior a 18 h. Es importante que tenga un sistema de lectura continua, que se pueda conectar bidireccionalmente con el sistema de gestión del laboratorio, que evite el uso de agujas y que ofrezcan una buena relación capacidad de frascos/tamaño. La mayoría de los sistemas disponibles han mostrado una buena eficacia en estudios independientes10. Algunas de sus principales características se recogen en la tabla 2. En un estudio europeo realizado en 1996 en 466 laboratorios se observó que los sistemas de hemocultivos más utilizados eran: Bactec (34%), BacT/Alert (21%), Signal (17%), Convencional (11%), Sentinel (5%), Septi-Chek (5%), Vital (5%), Hémoline (2%) y Vacutainer (0,5%)13.
Por qué se deben procesar los hemocultivos en los laboratorios intrahospitalarios
Existen numerosos estudios en la literatura científica que evalúan el impacto de los métodos realizados en el laboratorio de microbiología en el tratamiento del paciente infeccioso y en el ahorro de costos económicos14. Es necesario destacar sin embargo que la mayoría de estos estudios se llevan a cabo en países donde existe un control exhaustivo de los costes generados en los hospitales y se determina la metodología con criterios de eficiencia, lo que no siempre ocurre en algunos centros españoles. Doern et al15 demostraron que el informe rápido al clínico de la identificación y sensibilidad de un agente etiológico disminuía de manera significativa la mortalidad, el número de pruebas complementarias de los pacientes, los días de ventilación mecánica y de ingreso en cuidados intensivos. El ahorro estimado era de unos 4.200 dólares por paciente.
La sepsis constituye una de las infecciones más graves en los hospitales debido principalmente a su alta mortalidad asociada. En un estudio realizado por Bouza et al16 en 22 países europeos se observó que la mortalidad de los 266 pacientes incluidos en el estudio fue del 19%, siendo el porcentaje de muerte atribuible del 7,1%. A pesar del desarrollo de nuevas técnicas diagnósticas basadas en detección de ácidos nucleicos, los hemocultivos constituyen hoy por hoy la técnica de elección para el diagnóstico de la sepsis. Es por lo tanto esencial mejorar los protocolos y sistemas de tal manera que permitan acortar significativamente los tiempos requeridos para la emisión de informes sobre el agente causal.
En un estudio mundial sobre procesamiento de hemocultivos publicado en 1992 se demostró que el número de bacteriemias por 1.000 admisiones era considerablemente superior en los hospitales de Estados Unidos (36,7) que en los europeos (15,2) o españoles (21,0). En aquel año, la tasa en el Hospital Universitario Virgen Macarena fue de 17,0. Una de las principales razones era que en Estados Unidos existía mayor "cultura" de toma de hemocultivos en los pacientes hospitalizados. De hecho, las cifras de hemocultivos /1.000 admisiones fueron de 0,8, 0,25 y 0,17 para Estados Unidos, Europa y España, respectivamente17. En nuestro hospital el número de hemocultivos/1.000 admisiones fue de 0,20. En el estudio paneuropeo publicado por Bouza et al16 en 1999 se observa un aumento de la tasa de bacteriemias por 1.000 admisiones en los hospitales europeos (27,2), sin que se observaran diferencias entre los países de la Unión Europea y los demás. Este estudio concluye que la situación de las septicemias en Europa demuestra cambios importantes en la etiología, morbilidad y mortalidad, lo que requiere futuras intervenciones.
Una de las principales tendencias que ha sido observada por otros autores es el aumento de los microorganismos grampositivos, que en este estudio supusieron el 52,9% de los casos frente a los gramnegativos, hongos y anaerobios que supusieron el 41,2, 4,6 y 1,3%, respectivamente. Este fenómeno se debe a varios factores entre los que destacan la aparición de nuevos mecanismos de resistencia, mejor control y tratamiento de las infecciones por gramnegativos y la existencia de poblaciones más susceptibles de sufrir infecciones por grampositivos como pacientes con material protésico de cualquier tipo o drogadictos por vía parenteral.
Tiempo de detección de cultivos positivos
La realización y el procesamiento de los hemocultivos en los hospitales puede disminuir de una manera significativa el tiempo de detección de crecimiento de microorganismos. La mayoría de los sistemas de detección continua de microorganismos permite la introducción de frascos anónimos, es decir, sin introducir los datos de filiación del paciente, aunque no haya personal de presencia física en los laboratorios las 24 h del día. En el caso de laboratorios externos, los frascos pasarían una serie de horas, bien a temperatura ambiente o a 35 °C, antes de ser introducidos en el sistema de monitorización de frascos. En nuestra experiencia con Bactec y BacT/Alert, la preincubación a temperatura ambiente o a 35 °C de frascos inoculados con microorganismos de crecimiento rápido (Escherichia coli y Streptococcus pneumoniae) durante tiempos inferiores a 8 h no modificó la capacidad de estos sistemas para detectar crecimiento bacteriano18,19. Sin embargo, para tiempos superiores y determinados microorganismos, como bacilos gramnegativos no fermentadores, sí se ha observado una disminución en la capacidad de estos sistemas para detectar crecimiento ofreciendo resultados falsamente negativos20.
Emisión de informes
Otra ventaja del procesamiento de hemocultivos en los laboratorios hospitalarios radica en la rapidez de emisión de los informes provisionales. En nuestro centro, cuando un hemocultivo es positivo, un facultativo informa pesonalmente (nunca telefónicamente) de los resultados obtenidos en la tinción de Gram y se rellena un protocolo de bacteriemias. En caso de no encontrar al médico responsable del enfermo se escribe el resultado preliminar en la hoja de evoluciones de la historia clínica del paciente. Ello representa que el 20-25% de los hemocultivos positivos son informados personalmente por un microbiólogo en las primeras 8 h y que este porcentaje aumenta al 75% a las 24 h. El impacto de esta conducta en el pronóstico del paciente se está evaluando en la actualidad, aunque existen experiencias previas que demuestran la relevancia que tiene una información provisional en la mortalidad, tiempo de hospitalización y costes adicionales21.
Existen diversas publicaciones que demuestran los beneficios derivados de una información precoz del resultado del hemocultivo al clínico. Ello permite la elección de fármacos de menos espectro, dosis adecuadas, cambio a tratamientos orales y/o ambulatorios21. En el Hospital Gregorio Marañón de Madrid (E. Bouza, comunicación personal) se ha realizado un estudio prospectivo y aleatorio en el que durante 6 meses se ha comparado el impacto de diferentes sistemas de información de hemocultivos en 297 episodios de bacteriemias:
1. Informe telefónico precoz más informe microbiológico final.
2. Escrito de alerta con recomendación terapéutica seguido de informe final.
3. Igual que el anterior, pero con entrevista con el médico responsable del enfermo.
El porcentaje de días con tratamiento adecuado fue significativamente superior en los episodios informados activamente (por escrito, 92,2%) que a los informados de manera convencional (66,3%). En este estudio se efectuaron cambios de tratamiento en el 41,8% de los episodios informados de forma escrita y en el 50,6% de los informados de manera oral-comentada. La duración del tratamiento y el número de dosis diaria definida (DDD) de antimicrobianos utilizados de manera correcta fueron también parámetros en los que la información activa ofreció ventajas significativas. Finalmente, el gasto medio por episodio, generado por terapéutica antimicrobiana incorrecta, fue sensiblemente mayor en los episodios no informados por escrito. Todas estas ventajas se ven favorecidas cuando el centro hospitalario tiene una red informática interna que permite el vuelco de resultados, preliminares o definitivos, de manera directa en la historia del enfermo con un sistema de alarma que advierta al clínico de la relevancia de la información enviada.
Evidentemente, en el caso de un laboratorio externo, que además atendería a diferentes centros hospitalarios esta conducta sería absolutamente inviable. La experiencia con estos centros es que no se emiten informes provisionales de hemocultivos, sino que se espera a tener la identificación y antibiograma final, lo que atrasa los resultados más de 48 h en el mejor de los casos. No se dispone de datos sobre el efecto del transporte de hemocultivos a centros externos en la tasa de falsos negativos.
Situaciones clínicas especiales
Un problema que se ha puesto de manifiesto en los últimos años es el importante número de bacteriemias detectadas en pacientes dados de alta desde el servicio de urgencias de los hospitales, que llega a ser el 14% del total de las bacteriemias comunitarias. Javaloyas et al22 concluyen que para resolver este problema es importante centrarse en la selección de los pacientes con sospecha de bacteriemia, en la revisión de los criterios de ingresos y en la definición del perfil de los enfermos con probable bacteriemia candidatos a un seguimiento ambulatorio y a la mejora de la prescripción de antibióticos. A todo lo anterior nosotros sugeriríamos el uso de métodos que permitan disminuir el tiempo de detección de crecimiento de microorganismos en sangre y establecer un sistema de información oral al clínico de resultados preliminares. Todo ello permitiría disminuir estos porcentajes de manera significativa, mejorando la eficiencia de los hemocultivos.
Otro problema importante son las bacteriemias por estafilococos coagulasa negativos. Estos microorganismos constituyen los principales contaminantes de los hemocultivos y determinar la significación clínica de un aislamiento es con frecuencia difícil. Tradicionalmente muchos laboratorios de microbiología han basado el potencial valor de un aislamiento en función del número de frascos con crecimiento bacteriano23. Sin embargo, en un estudio reciente se demuestra que el número de frascos positivos no es un buen predictor de la significación clínica de un aislamiento en sangre de estafilococos coagulasa negativos y que es necesario valorar clínicamente cada paciente. Realizar esta función desde fuera de un hospital es una tarea imposible teniendo en cuenta el elevado número de aislamientos de estafilococos coagulasa negativos en sangre.
Modificaciones de protocolos
Una vez establecido que la rapidez en la emisión de los informes es esencial para el pronóstico del paciente con sepsis, se está intentando en la actualidad buscar métodos que permitan disminuir los tiempos de emisión de los resultados. Una de las posibilidades consiste en la inoculación directa de los paneles del sistema de identificación y antibiograma utilizado en el laboratorio con inóculos bacterianos obtenidos directamente del frasco de hemocultivos. Nuestra experiencia y la de otros autores en los últimos años con la inoculación directa de paneles del sistema MicroScan (DADE) con inóculos obtenidos de frascos plus del sistema Bactec ha sido que la identificación y antibiograma de los bacilos gramnegativos es bastante fiable y que el porcentaje de errores mayores en los resultados de sensibilidad a los antimicrobianos es mínimo. Sin embargo, los cocos grampositivos presentan mayores dificultades. Actualmente estamos evaluando la inoculación directa de paneles del sistema Vitek II a partir de inóculos directos de frascos plus (Bactec) y FAN (BacT/Alert). La combinación de un sistema de monitorización continua de hemocultivos con un sistema de identificación y antibiograma rápido podría permitir la emisión de informes provisionales con datos de sensibilidad en tiempos inferiores a las 24 h desde la toma de los hemocultivos para microorganismos de crecimiento rápido24. El efecto de esta combinación en el pronóstico del enfermo está por demostrar, pero en cualquier caso es necesario validar adecuadamente estos métodos antes de utilizarlos de manera rutinaria en los laboratorios.
Otro factor importante es la posibilidad de utilizar los sistemas de monitorización continua para funciones diferentes del procesamiento de hemocultivos. Por ejemplo, se ha demostrado que las diferencias en tiempos de detección de hemocultivos positivos tomados a través de un catéter central frente a una vía periférica del mismo paciente permiten hacer un diagnóstico presuntivo de bacteriemias asociadas al uso de catéteres intravasculares.
Futuro en el procesamiento de hemocultivos
En los últimos años se están desarrollando numerosas técnicas diagnósticas basadas en la detección y/o amplificación de los ácidos nucleicos y existen numerosas revisiones al respecto2,25. Las principales aplicaciones clínicas son las infecciones sistémicas muy graves, aquellas causadas por agentes etiológicos de difícil crecimiento o detección por métodos convencionales, infecciones en pacientes inmunodeprimidos en los que los métodos convencionales son poco efectivos y detección rápida de determinantes de resistencia. La ventaja de estos métodos es que tienen mayor sensibilidad y podrían aplicarse directamente a la sangre del paciente sin pasar por un período de cultivo intermedio, lo que acortaría de manera significativa el tiempo de obtención de resultados. Las principales desventajas radican en que al no obtener el microorganismo causal completo no permite realizar estudios de sensibilidad a los agentes antimicrobianos, lo que resulta actualmente esencial con el incremento general de las resistencias. El diagnóstico molecular desempeña un papel cada vez más relevante en la detección e identificación rápida de microorganismos en muestras clínicas. La variación genética de los genes ribosómicos en las bacterias ofrece una interesante alternativa a los cultivos clásicos. Estos genes, como el gen del ARNr 16S, presenta regiones conservadas y variables que los convierten en dianas útiles para la identificación de especies. Mediante tecnología sencilla basada en la reacción en cadena de la polimerasa (PCR) como la SSCP (single strand conformation polymorphism) o técnicas similares se han detectado numerosas bacterias, levaduras y hongos filamentosos, si bien muchos de los estudios se efectúan sobre frascos de hemocultivos crecidos y se desconoce su utilidad aplicadas directamente en sangre del paciente25. Hay que tener en cuenta que diversas sustancias de la sangre, o incluso el polianetol sulfonato sódico que llevan numerosos medios de cultivo, pueden interferir con las reacciones de amplificación, lo que obliga al uso de protocolos de extracción de ácidos nucleicos especiales. Otra limitación importante de estos métodos es su escasa utilidad para diagnosticar infecciones mixtas. Mediante técnicas de PCR es fácil determinar adicionalmente genes de resistencia como mecA, gen responsable de la resistencia a meticilina en Staphylococcus spp., los genes responsables de resistencia a glucopépticos en enterococos o los genes responsables a betalactámicos en bacterias gramnegativas.
Como se ha señalado anteriormente, una de las principales dificultades para el uso de técnicas moleculares en el diagnóstico de la sepsis es el amplio rango de microorganismos y genotipos de resistencia que podemos encontrar, lo que dificulta la aplicabilidad de estas técnicas. Para paliar este problema se están desarrollando actualmente sistemas basados en microchips o microarrays que básicamente consisten en soportes diminutos que albergan miles de sondas de ácidos nucleicos que además de la identificación permiten detectar potencialmente los principales genes de resistencia bacteriana para una gran cantidad de microorganismos de manera simultánea y automatizada26. A pesar de que el uso de esta tecnología no ha sido aprobado todavía por la FDA, su relevancia en los laboratorios de microbiología clínica en la actual década será incuestionable.
En resumen, el uso de las técnicas microbiológicas convencionales basadas en el cultivo y tinción de Gram siguen teniendo una gran relevancia en el diagnóstico etiológico de la sepsis. Tenemos herramientas, sin embargo, que permiten acortar de manera significativa el tiempo necesario para la obtención de resultados preliminares. El papel del microbiólogo clínico es esencial para una correcta interpretación de los resultados preliminares y definitivos. La aplicación de la tecnología molecular acortará indudablemente los tiempos necesarios para el diagnóstico etiológico de las infecciones graves en un futuro próximo.
Líquido cefalorraquídeo
Las meningitis infecciosas constituyen una urgencia médica debido a la alta morbilidad y mortalidad de alguna de ellas. Las meningitis bacterianas aguda pueden llegar a tener una mortalidad del 18%27. En los últimos años se han producido cambios importantes en la epidemiología de las meningitis bacterianas. En primer lugar, la introducción de la vacuna conjugada frente a Haemophilus influenzae tipo b (Hib) en muchos países ha producido una disminución de las infecciones invasivas y concretamente en las meningitis producidas por este microorganismo. En Estados Unidos, la incidencia de meningitis por Hib ha disminuido el 96% desde 1986 a la actualidad. Posiblemente la introducción de la nueva vacuna conjugada frente a Neisseria meningitidis grupo C producirá una disminución significativa de las meningitis producidas por este serogrupo, si bien actualmente se desconoce si su uso puede producir un aumento de la prevalencia de cuadros producidos por otros serogrupos.
La principal causa de meningitis aséptica o viral son los enterovirus, que producen del 80 al 92% de estas infecciones28. En Estados Unidos se declaran más de 10.000 casos anuales de meningitis por enterovirus, pero se estima que su incidencia es superior a 75.000 casos anuales. Otros virus productores de estos cuadros son el virus de la parotiditis, virus herpes, arbovirus, virus de la coriomeningitis linfocitaria, virus de la inmunodeficiencia humana (VIH), adenovirus, virus de la gripe, etc. El desarrollo de técnicas moleculares de gran sensibilidad ha permitido relacionar a otros patógenos con infecciones del sistema nervioso central, no necesariamente meningitis, como ha sido el caso de rotavirus29.
La disminución de la incidencia de las meningitis por Hib ha cuestionado el valor de las técnicas rápidas para el diagnóstico de las meningitis agudas. En un estudio con 901 muestras de líquido cefalorraquídeo (LCR) se describió que sólo en 4 de 26 pacientes se modificó el tratamiento en función de los resultados de una técnica rápida de detección de antígenos en LCR30. Sin embargo, existen diferentes factores que justifican la realización de un diagnóstico rápido. En primer lugar, el incremento de la resistencia a antimicrobianos de uso empírico en meningitis bacterianas por determinados patógenos. Por ejemplo, la resistencia a cefalosporinas de tercera generación por S. pneumoniae. En segundo lugar, la posibilidad de tratar cuadros que antes no disponían de tratamiento específico, como la meningitis por enterovirus y el pleconaril. Finalmente, la necesidad a veces de instaurar medidas profilácticas y epidemiológicas en cuadros de gran impacto social, como la meningitis meningocócica, aconsejan disponer de un diagnóstico precoz. De hecho, en nuestro centro hospitalario, aproximadamente el 90% de las llamadas al personal facultativo de guardia se deben a sospechas de meningitis de diferente naturaleza.
Presente y futuro del diagnóstico de las meningitis
A pesar de la gran cantidad de agentes etiológicos involucrados se intentarán enfatizar las novedades más relevantes. El procesamiento de los LCR en los laboratorios de microbiología constituyen una urgencia médica. Las muestras deben enviarse al laboratorio y procesarse rápidamente. El tiempo medio de emisión de un informe preliminar en nuestro laboratorio es de 45 min en caso de tinción de Gram y de 75 min cuando se realiza detección de antígeno. A pesar de la gravedad de la meningitis bacteriana se ha descrito un considerable retraso en la instauración de tratamiento empírico y para evitarlo la información preliminar activa del laboratorio puede desempeñar un papel relevante27.
En el caso de las meningitis bacterianas agudas, la tinción de Gram de una muestra de LCR sigue siendo la técnica de elección. El uso de una citocentrífuga mejora el rendimiento de la tinción de Gram. La sensibilidad de esta tinción de Gram oscila del 60 al 90% con una especificidad del casi el 100%. La sensibilidad de la técnica depende del microorganismo llegando a ser del 90% en el caso de S. pneumoniae (3 p592). La sensibilidad es inferior al 50% en casos de meningitis por Listeria monocytogenes y cuadros nosocomiales tras intervenciones neuroquirúrgicas.
La utilización sistemática de un medio de enriquecimiento líquido para el cultivo de LCR es controvertida. De hecho, la mayoría de microoganismos que crecen en medio líquido y no en los sólidos suelen ser falsos positivos.
Existen numerosas técnicas rápidas (enzimoinmunoanálisis [ELISA], látex, coaglutinación, etc.) para detectar antígenos bacterianos en LCR. La batería suele incluir Hib, N. meningitidis, S. pneumoniae, E. coli K1 y S. agalactiae. La mayoría de los sistemas comerciales no incluyen al meningococo del grupo B por la baja inmunogenicidad de su cápsula. La sensibilidad de estas técnicas es variable en función de los estudios y oscila del 50 al 100% dependiendo de la técnica y el microorganismo30. En un estudio de 103 meningitis bacterianas agudas la sensibilidad total de estos métodos fue del 33%, pero disminuyó al 9% en casos donde no visualizaron microorganismos en la tinción de Gram del LCR31. Por todo ello, el uso de estas técnicas se debe reservar para aquellos casos de sospecha de meningitis bacteriana en los que la tinción de Gram es negativa y los cultivos a las 48 h no revelan ningún crecimiento. Estos casos suelen corresponder a pacientes con meningitis tratados con antimicrobianos antes de obtener la muestra de LCR. Actualmente existen técnicas inmunocromatográficas con sensibilidad y especificidad del 100% para la detección de antígeno neumocócico en orina y LCR de pacientes con meningitis32. El uso de métodos físicos, como el ultrasonido, incrementa significativamente la sensibilidad de técnicas de látex hasta niveles alcanzables exclusivamente por técnicas de amplificación de ácidos nucleicos (PCR)33.
Existen numerosos estudios en la literatura que evalúan diferentes técnicas de PCR para la detección de ácidos nucleicos de bacterias en LCR. Algunas permiten la detección simultánea de varios microorganismos. Se ha descrito una técnica de "nested" PCR que amplifica el gen que codifica la proteína fijadora de penicilina 2B de neumococo, lo que permite además de detectar el microorganismo, conocer al mismo tiempo su sensibilidad o resistencia a penicilina. No se describen en este estudio falsos positivos. Se han descrito técnicas de PCR para el diagnóstico de meningitis producidas por Treponema pallidum y Borrelia burgdorferi que se encuentran en fase experimental, pero que parecen superiores al estudio de secreción intratecal de anticuerpos. La detección de ADN de Mycobacterium tuberculosis en LCR es más sensible que el cultivo. En el caso de sospecha de meningitis tuberculosa o micótica debe realizarse siempre el cultivo de la muestra de LCR a pesar de disponer de un diagnóstico presuntivo por alguna de las técnicas rápidas disponibles.
Para la meningitis producida por enterovirus, la técnica de referencia actualmente es la PCR con transcriptasa inversa que se ha mostrado más sensible que el cultivo viral (tabla 3). En primer lugar, acorta extraordinariamente el tiempo de detección que para el cultivo puede ser de 7 días, y además permite la detección de virus defectivos o inviables. Otras meningitis virales se siguen diagnosticando mediante técnicas serológicas o cultivos, aunque se han descrito técnicas de PCR para detectar virus de la parotiditis, herpes zóster, herpes simple tipo 2, y algunos arbovirus30.
En nuestra opinión, el futuro del diagnóstico de cuadros de meningitis a partir de LCR puede pasar por el uso de microarrays. Las técnicas tradicionales de PCR múltiples son laboriosas y requieren realizar discriminación de amplicones mediante el uso de electroforesis en agarosa, hibridación y secuenciación. Los microarrays simplifican extraordinariamente este proceso. En casos de meningitis bacterianas donde el número de agentes causales y de genes de resistencia es limitado, el uso de esta tecnología es especialmente atractivo, si bien su uso de manera sistemática requerirá una disminución de los costes que actualmente son muy elevados.