Introducción
La gastroenteritis es la manifestación más común de la salmonelosis. Suele tratarse de un cuadro leve y autolimitado que no requiere tratamiento antibiótico. Sin embargo, en aquellos casos cada vez más habituales por el aumento del número de pacientes inmunocomprometidos y su mayor supervivencia, en que la infección es extraintestinal o cursa con bacteriemia es esencial la instauración precoz de un tratamiento antibiótico eficaz1. Desafortunadamente, el tratamiento empírico de este tipo de infecciones se ha visto complicado por el progresivo aumento de la resistencia a antibióticos considerados tradicionalmente activos como cotrimoxazol (T/S), amoxicilina-ácido clavulánico, cefalosporinas de tercera generación o fluoroquinolonas que han experimentado las cepas de Salmonella enterica y, en particular, las pertenecientes al serotipo Typhimurium2-4.
La resistencia a betalactámicos en S. enterica suele estar mediada por betalactamasas plasmídicas clásicas2,3,5,6. Se ha comprobado que los genes que codifican algunas betalactamasas y muchos otros genes involucrados en la resistencia a distintos grupos de antimicrobianos, pueden localizarse en una nueva familia de elementos genéticos denominados integrones, que a menudo están contenidos en transposones o en plásmidos de multirresistencia2,6-8.
Los integrones, descritos por primera vez por Stokes y Hall en 1989, son estructuras genéticas aparentemente móviles cuyo tamaño no supera los 4.000 pb, capaces de integrar o movilizar genes individuales de resistencia a antibióticos. Los integrones están constituidos por dos regiones de ADN muy conservadas, la 59CS y la 39CS, y otra variable situada entre las dos anteriores, en la que se pueden insertar hasta cinco determinantes genéticos de resistencia.
Hasta el momento se han descrito varias clases de integrones, siendo los de clase 1 los que se encuentran en la mayoría de aislados clínicos. La acumulación y extraordinaria capacidad de recombinación e intercambio que poseen los genes de resistencia dentro de los integrones y la gran aceptación de estos últimos por parte de las bacterias, debido a que no es necesario que almacenen grandes piezas de material genético para hacerse resistentes a los antibióticos, podrían haber contribuido a la continua aparición y diseminación de enterobacterias y otros gramnegativos multirresistentes9, como ha sucedido en S. ser Typhimurium10-13.
De particular relevancia es el caso de S. ser Typhimurium DT104 multirresistente que, desde que se identificó en el Reino Unido en 1984 en ganado y seres humanos, se ha diseminado ampliamente dando lugar a una verdadera epidemia mundial relacionada con el consumo de alimentos de origen animal contaminados10. Las cepas de S. ser Typhimurium DT104 causantes de esta epidemia comparten un mismo patrón de resistencia (ampicilina, tetraciclina, cloranfenicol, estreptomicina y sulfamidas), asociado a un locus cromosómico de resistencia (SGI1) en el que están presentes dos integrones de clase 1. La presencia de integrones en el serotipo Enteritidis y en otros serotipos menos comunes, aunque menos habitual que en el serotipo Typhimurium, cada vez se comunica con mayor frecuencia7,8,14,15.
En nuestro laboratorio hemos podido constatar un creciente incremento en el porcentaje de aislados de S. ser Typhimurium que producen betalactamasas del tipo OXA y PSE-1, en detrimento de aquellos que producen TEM-1, prevalentes hasta mediados de la década de los noventa (datos no publicados) y consideramos que sería interesante investigar si este extremo podría estar relacionado con el hecho de que los genes que codifican las dos primeras betalactamasas se han localizado en integrones, lo que habría facilitado su diseminación, mientras que esto nunca ha sucedido con el gen que codifica TEM-1.
El objetivo de nuestro trabajo es estudiar la frecuencia de integrones de clase 1 entre aislados de S. enterica de origen humano productores de diferentes tipos de betalactamasas recuperados en la región sanitaria de Tortosa, así como intentar determinar los genes de resistencia insertados en dichos integrones.
Métodos
Aislados
Entre junio de 2000 y octubre de 2001 se obtuvieron en nuestro laboratorio (Laboratorio de Microbiología del Hospital de Tortosa Verge de la Cinta) 233 aislados clínicos de S. enterica (121 S. enterica ser Enteritidis, 92 ser Typhimurium y 20 de otros serotipos) que procedían de pacientes atendidos tanto en nuestro hospital como en cualquiera de los 34 centros de atención primaria de la región sanitaria de Tortosa. Se incluyeron en el estudio todos los aislados resistentes a ampicilina (30 aislados de S. enterica ser Enteritidis, 56 ser Typhimurium, 4 ser Hadar, 3 ser [4,5,12:i:-], 2 ser Panama, 1 ser inmóvil [4,12:-:-], 1 ser Virchow, 1 ser Grumpensis, 1 ser Give y 1 ser Mbadanka); en aquellos casos en que se tenía constancia de que varios aislados estaban epidemiológicamente relacionados se incluyó sólo uno de ellos en el estudio. Se recuperaron 98 aislados a partir de coprocultivos, uno a partir de un hemocultivo y otro a partir de un urocultivo.
Identificación y pruebas de sensibilidadin vitro
La identificación de especie y la sensibilidad in vitro por microdilución en caldo a ampicilina, cefotaxima, ceftazidima, C, T/S, ácido nalidíxico, ciprofloxacino, gentamicina, tobramicina, tetraciclina y colistina se estudiaron mediante los paneles comerciales 95W y 93W (Soria Melguizo, Madrid, España). La sensibilidad a estreptomicina, kanamicina y sulfamidas se llevó a cabo por la técnica de difusión en agar de Kirby-Bauer. Las cepas se clasificaron como sensibles, intermedias o resistentes siguiendo los criterios del National Committee for Clinical Laboratory Standards (NCCLS)16 y se incluyeron las cepas de referencia Escherichia coli ATCC 35218 y E. coli ATCC 25922 como controles. La serotipificación y fagotipificación se realizaron en el Centro Nacional de Microbiología (Majadahonda, Madrid).
Identificación de betalactamasa e investigación de genes codificadores de betalactamasas
Los extractos para los ensayos de betalactamasas se obtuvieron a partir de un cultivo reciente de cada aislado en fase de crecimiento exponencial, por sonicación en baño de hielo a 160 W, durante 3 min, a intervalos de 1 min, y posterior centrifugación a 14.000 rpm y 4 °C durante 30 min. Las betalactamasas se identificaron por su pI, que se determinó por isoelectroenfoque en un equipo LKB Multiphor II (Pharmacia LKB, Uppsala, Suecia) con geles de poliacrilamida con un rango de pH de 3.5 a 9.5 (Pharmacia LKB Uppsala, Suecia) siguiendo un protocolo descrito previamente17. Las betalactamasas se visualizaron tras tinción con nitrocefina (500 μg/ml) y su pI se determinó por comparación con las enzimas patrón TEM-1 (pI 5,4), TEM-2 (pI 5,6), PSE-1 (pI 5,7), SHV-1 (pI 7,6), OXA-1 (pI 7,4) y OXA-2 (pI 7,7 / 7,5). Por otro lado, se investigó en todas las cepas la presencia de los genes tem-1, oxa-1 y pse-1 que codifican las betalactamasas TEM-1, OXA-1 y PSE-1 respectivamente, mediante PCR. La reacción de PCR se llevó a cabo en un volumen final de 50 μl que contenía Cl2 Mg, 1,5 mM; tampón PCR 1x; 0,2 mM de cada uno de los nucleósidos trifosfato, 0,5 mM de cada uno de los correspondientes cebadores (tem-1: 59-TTG GGT GCA CGA GTG GGT-39 y 59-TAA TTG TTG CCG GGA AGC-3918,19; oxa-1: 59-ACC AGA TTC AAC TTT CAA-39 y 59-TCT TGG CTT TTA TGC TTG-3919; pse-1: 59-AAT GGC AAT CAG CGC TTC-39 y 59-GGG GCT TGA TGC TCA CTC-3918,19), 1,5 U de taqpolimerasa (Ecogen, Barcelona, España) y 25 μl de lisado bacteriano, obtenido tras someter a ebullición a 100 °C durante 10 min una suspensión de tres colonias, procedentes de un cultivo fresco en agar sangre, en 50 μl de agua bidestilada estéril y posterior centrifugación a 14.000 rpm durante 20 s. La mezcla de reacción se amplificó en un termociclador GeneAmp PCR System 2400 (Perkin Elmer) empleando las siguientes condiciones: 1 ciclo inicial de 5 min a 94 °C; 30 ciclos de 94 °C durante 30 s, 55 °C durante 30 s y 72 °C durante 30 s; 1 ciclo final de 72 °C durante 5 min. Los productos finales de cada PCR se analizaron por electroforesis en geles de agarosa al 1,5% con 0,5 mg/ml de bromuro de etidio.
Investigación de integrones de clase 1
Se investigó la presencia de integrones de clase 1 en todos los aislados mediante amplificación por PCR y posterior análisis de los amplicones generados por electroforesis en geles de agarosa, empleando una mezcla de reacción de composición idéntica a la indicada en el apartado anterior y usando los cebadores 59CS (59-GGC ATC CAA GCA GCA AGC-39) y 39CS (59-AAG CAG ACT TGA CCT GAT-39)2. El programa de amplificación fue un ciclo inicial de 5 min a 94 °C; 30 ciclos de 94 °C durante 30 s, 55 °C durante 30 s y 72 °C durante 2 min y 30 s; 1 ciclo final de 72 °C durante 5 min. El tamaño de los amplicones generados se calculó por comparación con el patrón de tamaño molecular Supperladder-Mid 1 100 pb (Gensura, San Diego, EE.UU.).
Investigación de los genes de resistencia incluidos
en la región variable de los integrones
Se investigó la presencia de los genes de resistencia aadA1, aadA2 (resistencia a estreptomicina-espectinomicina), dfrA1 (resistencia a trimetoprima) y tem-1, oxa-1 y pse-1 (resistencia a betalactámicos) mediante una segunda PCR, utilizando como ADN molde una dilución al 1:10 de los productos generados en la PCR para detección de integrones, con la finalidad de determinar los genes de resistencia incluidos en la región variable de éstos. Las condiciones de la PCR fueron las indicadas anteriormente en el apartado de investigación de genes codificadores de betalactamasa. Los cebadores utilizados para detectar la presencia de los tres primeros genes fueron 59-GTG GAT GGC GGC CTG AAG CC-39 y 59-ATT GCC CAG TCG GCA GCG-39 (aadA1)19; 59-TGT TGG TTA CTG TGG CCG TA-39 y 59-GCT GCG AGT TCC ATA GCT TC-39 (aadA2)7 y 59-GTG AAA CTA TCA CTA ATG G-39 y 59-CCC TTT TGC CAG ATT TGG-39 (dfrA1)19. Los cebadores empleados en el presente estudio para amplificar aadA2 son específicos para este gen, pero no así los que usamos para amplificar aadA1, que amplifican también aadA2, por lo que consideramos que en un integrón estaba presente el gen aadA1 sólo cuando se obtenía amplificación con los cebadores para aadA1 pero no con los específicos para aadA2.
Definición diferentes perfiles de integrones
Los diferentes perfiles de integrones obtenidos al analizar los aislados estudiados se definieron de acuerdo con el número, tamaño de los amplicones generados y genes de resistencia detectados en la región variable de los integrones.
Resultados
Caracterización de betalactamasas
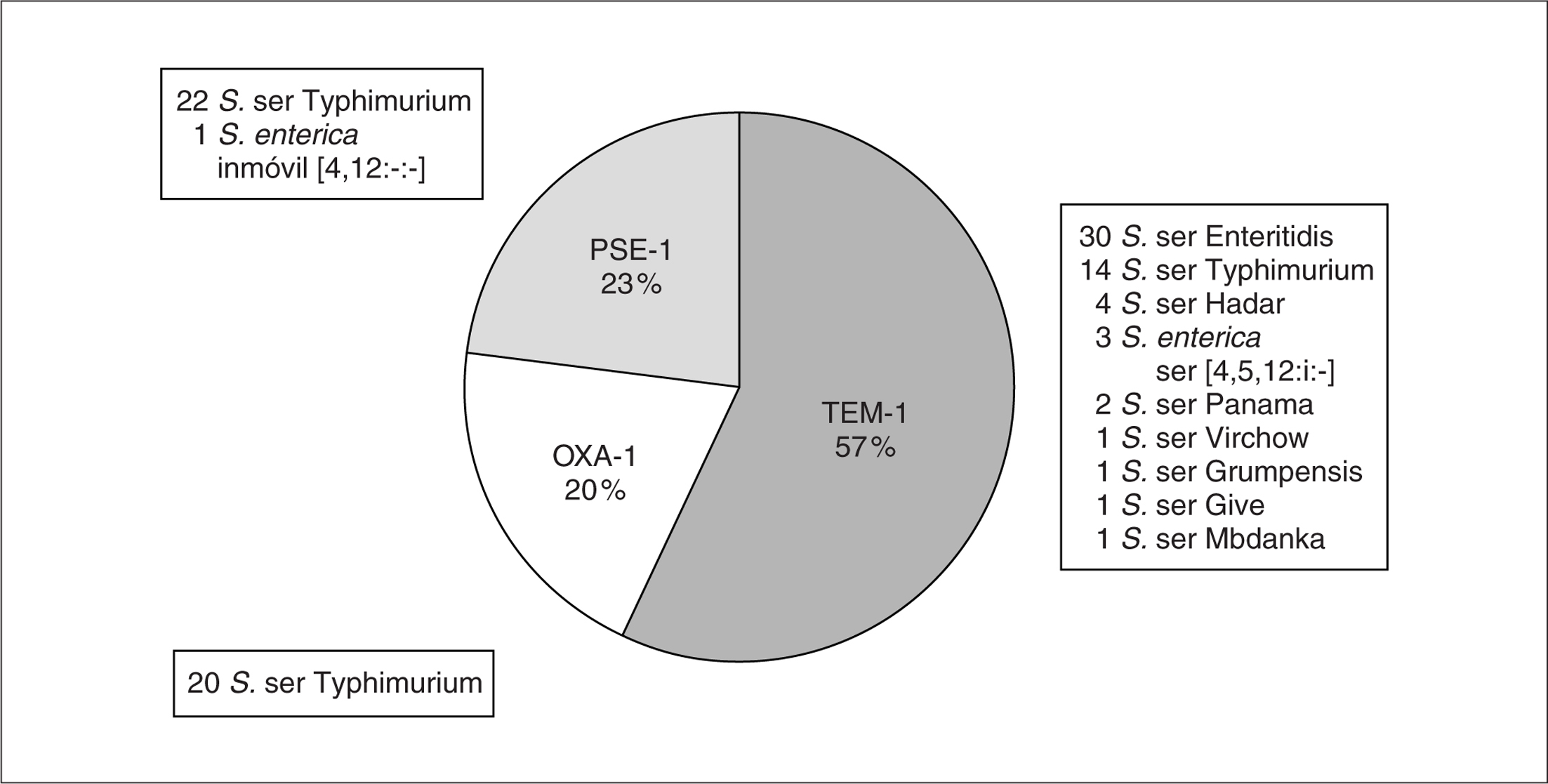
Los diferentes tipos de betalactamasas producidos por los 100 aislados estudiados aparecen reflejados en la figura 1. Los aislados que producían una betalactamasa de tipo TEM-1 (pI 5,4 y PCR positiva para tem-1) fueron los más frecuentes, seguidos por aquellos que albergaban una enzima de tipo PSE-1 (pI 5,7 y PCR positiva para pse-1) o de tipo OXA-1 (pI 7,4 y PCR positiva para oxa-1).
Figura 1. Distribución de los diferentes tipos de betalactamasas producidas por los 100 aislados de Salmonella enterica estudiados.
Patrones de resistencia
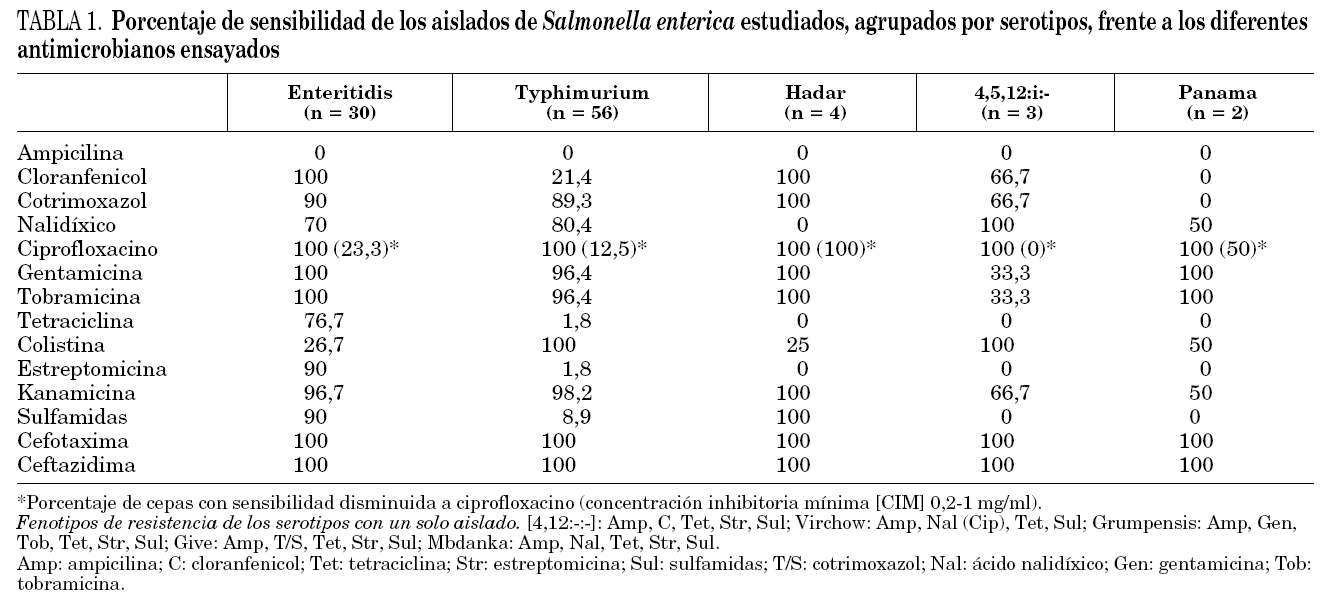
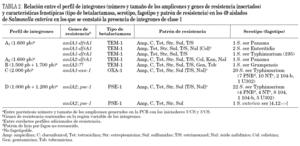
a)La sensibilidad de los 100 aislados de S. enterica investigados, agrupados por serotipos, frente a los diferentes antimicrobianos ensayados aparece reflejada en la tabla 1.
b)Aislados productores de TEM-1: Entre los aislados pertenecientes al serotipo Enteritidis productores de esta betalactamasa, el 83,3% resultaron resistentes a más de dos grupos de antibióticos y el 20% fueron multirresistentes (resistencia a cuatro o más grupos de antimicrobianos); se observaron hasta 11 patrones distintos de resistencia siendo las asociaciones más frecuentes ampicilina-colistina-ácido nalidíxico (40%) y ampicilina-colistina (16,7%). En cuanto a los aislados del serotipo Typhimurium el 100% fueron resistentes a más de dos grupos de antimicrobianos y el 64% a más de tres; se encontraron 8 tipos de patrones de resistencia y los más habituales fueron ampicilina-tetraciclina-estreptomicina-sulfamidas (42,8%) y ampicilina-tetraciclina-estreptomicina (21,4%). La totalidad de los aislados pertenecientes al resto de serotipos identificados fueron multirresistentes y el patrón de resistencia más frecuente fue ampicilina-tetraciclina-estreptomicina-ácido nalidíxico (38,5%).
c)Aislados productores de OXA-1: Todos los aislados productores de OXA-1 pertenecían al serotipo Typhimurium y mostraban el patrón de resistencia ampicilina-C-estreptomicina-tetraciclina-sulfamida. Dos aislados presentaban resistencia adicional a ácido nalidíxico y uno a ácido nalidíxico y T/S.
d)Aislados productores de PSE-1: La totalidad de los aislados productores de PSE-1 (22 serotipo Typhimurium y 1 serotipo inmóvil [4,12:-:-]) presentaban el patrón de resistencia ampicilina-C-estreptomicina-tetraciclina-sulfamida. Siete aislados del serotipo Typhimurium eran también resistentes a ácido nalidíxico y uno a T/S.
Frecuencia de integrones de clase 1 y detección de los genes de resistencia insertados
Se detectó la presencia de integrones de clase 1 en casi el 50% (49/100) de los aislados estudiados (10,5% en los aislados productores de TEM-1 y 100% en los productores de OXA-1 y PSE-1). De éstos, el 51% albergaban un único integrón y el resto dos integrones. Si se analiza la frecuencia de integrones de tipo 1 según el serotipo de S. enterica se observa que éstos estaban presentes en el 76% (43/56) de los aislados del serotipo Typhimurium, en el 6,7% (2/30) de los de serotipo Enteritidis, en los dos aislados pertenecientes al serotipo Panama, en el de serotipo [4,12:-:-] y en el único del serotipo Grumpensis de nuestra serie; no se detectaron integrones en ninguno de los aislados pertenecientes al resto de serotipos a pesar de ser todos ellos multirresistentes.
Las características fenotípicas (tipo de betalactamasa, serotipo, fagotipo y patrón de resistencia) y el perfil de integrones de los 49 aislados en los que se constató la presencia de integrones se resumen en tabla 2. Se identificaron cinco perfiles diferentes de integrones: perfil A1 (1.600 pb/aadA1-dfrA1) que se detectó en dos aislados de S. enterica ser Enteritidis, en uno de S. ser Panama y en otro de S. ser Typhimurium fagotipo 195; el perfil A2 (1.600 pb/aadA2-dfrA1) se observó en el otro aislado del serotipo Panama; perfil B (1.500 pb + 1.700 pb, en los que además de aadA2 presumiblemente deben estar ubicados otros determinantes de resistencia distintos a los que investigamos) que apareció en la única cepa de serotipo Grumpensis; perfil C (2.000 pb/aadA1-oxa-1) que se constató en 20 aislados del serotipo Typhimurium, la mayoría no fagotipables o de patrón de lisis no reconocido; perfil D (1.000 pb/aadA2 + 1.200 pb/pse-1) que se observó en 23 aislados, todos ellos, excepto uno del serotipo inmóvil [4,12:-:-], pertenecientes al serotipo Typhimurium, de los fagotipos 104, 104-b y U302 o bien no fagotipificables o de patrón de lisis no reconocido (los aislados de S. Typhimurium que no poseían integrones eran no fagotipificables o pertenecían a los fagotipos 98, 193, 308 o 312).
Discusión
A pesar de existir pocos datos publicados acerca de la prevalencia de diferentes betalactamasas en S. enterica nuestros resultados coinciden con los de otros autores3,5,6 en cuanto a que la betalactamasa producida con mayor frecuencia por los aislados de S. enterica recogidos en nuestra región geográfica fue TEM-1. En efecto, TEM-1 fue la única betalactamasa detectada en los aislados del serotipo Enteritidis, coincidiendo con lo reflejado en otros trabajos14, y también en los aislados del resto de serotipos incluidos en nuestra serie a excepción de los del serotipo Typhimurium y del único aislado del serotipo inmóvil [4,12:-:-]. En el caso de S. ser Typhimurium, PSE-1 fue la betalactamasa mayoritaria (39,3%) seguida a poca distancia por OXA-1 (35,7%), mientras que TEM-1 sólo representaba el 25% de las betalactamasas identificadas. Es interesante destacar que el porcentaje de aislados de S. ser Typhimurium productores de OXA-1 en la región sanitaria de Tortosa es sensiblemente más alto que el descrito por otros investigadores: en un estudio realizado en Francia en 1996 con aislados de S. ser Typhimurium de origen tanto humano como animal, Casini et al6 encuentran una frecuencia similar de aislados productores de enzimas tipo TEM y el 75% de tipo CARB, pero sólo descubren un aislado productor de una enzima tipo OXA; también en Francia, Llanes et al20 en un estudio de vigilancia llevado a cabo entre 1987 y 1994 refieren aproximadamente el 52% de aislados de S. enterica ser Typhimurium productores de PSE-1, el 45% productores de TEM-1 y sólo el 4% de productores de OXA-1, en España Gallardo et al estudian 23 aislados de S. Typhimurium resistentes a ampicilina procedentes de pacientes atendidos en 1999 en consultas externas de un hospital de Barcelona y el 39,1% de ellas albergan PSE-1, el 8,7% OXA-1 y el resto TEM-13.
A diferencia de otros trabajos, nosotros no hallamos en el período comprendido en el estudio ningún aislado que produjese betalactamasa TEM-2 o de tipo SHV5,6 y tampoco betalactamasas de espectro extendido21, aunque anterior y posteriormente a este período sí se han identificado algunos aislados de S. enterica ser Enteritidis y S. ser Virchow productores de este último tipo de betalactamasa.
Respecto a la sensibilidad antimicrobiana, tal y como cabía esperar2,19,22, las cepas de los serotipos Hadar y Typhimurium, en particular las que producían OXA-1 o PSE-1, resultaron ser las que presentaban porcentajes más elevados de resistencia, al igual que sucedió con las del serotipo recientemente descrito [4,5,12:i:-], siendo tetraciclina, estreptomicina y sulfamida los antimicrobianos frente a los que se detectó mayor número de aislados resistentes. El porcentaje de resistencia a ácido nalidíxico, por el contrario, fue más elevado en el serotipo Enteritidis que en el Typhimurium, y coincidiendo con lo descrito en otros estudios2,14,22 no hubo ningún aislado resistente a ciprofloxacino, si bien el 77,8% de los aislados resistentes a ácido nalidíxico presentaban sensibilidad disminuida a esta fluoroquinolona.
La frecuencia de integrones entre los aislados de S. enterica estudiadas fue muy diferente según el tipo de betalactamasa que producían: esta era muy baja entre aislados productores de TEM-1 (10,1%) (aproximadamente la misma frecuencia que encontramos en nuestro laboratorio para aislados de S. enterica no productores de betalactamasa [datos no publicados]), mientras que la presencia de integrones fue constante en los aislados productores de OXA-1 y PSE-1. Por otro lado, los genes de resistencia que codifican estas enzimas siempre se localizaron en la región variable de integrones de clase 1; sin embargo, el gen que codifica TEM-1 no se encontró en ninguna ocasión formando parte de uno de estos elementos genéticos.
En cuanto a los genes insertados en la región variable de los integrones, los más prevalentes fueron, como ya han señalado otros autores2,7,23, los que codifican enzimas modificantes de aminoglucósidos. En nuestro estudio todos los integrones contenían los genes aadA1 o aadA2.
Los perfiles de integrones A1 (1.600 pb/aadA1-dfrA1), A2 (1.600 pb/aadA2-dfrA1), C (2.000 pb/aadA1-oxa-1) y D (1.000 pb/aadA2 + 1.200 pb/pse-1) ya habían sido previamente descritos en S. enterica2,7,8,14,15, si bien no se puede descartar que, debido a los cebadores empleados, en algún caso los genes que nosotros hemos denominado oxa-1 o dfrA1 no sean tales, sino genes estrechamente relacionados. Parece interesante resaltar la presencia de un integrón 1.600 pb/aadA2-dfrA1 en uno de los dos aislados de S. ser Panama; no hemos encontrado ninguna referencia que mencione la presencia de un integrón de dichas características en este serotipo, en el que es frecuente el integrón 1.600 pb/aadA1-dfrA1, pero sí en una cepa de S. enterica ser Brandenburg que ocasionó un brote epidémico en Italia15. También llama la atención la ausencia de aislados con integrones del tipo 1.000 pb/aadA1, en contraste con lo observado en otros estudios realizados en España en que era el integrón más prevalente entre cepas de S. ser Enteritidis2,12, y la baja frecuencia de aislados pertenecientes al serotipo emergente [4,5,12:i:-], así como la ausencia del integrón 1.900 pb/dfAr12-aadA2; en este último serotipo, en el que según un estudio realizado por Guerra et al19 con aislados procedentes del Principado de Asturias su presencia era constante. En cuanto al perfil B (1.500 pb + 1.700 pb), que sólo apareció en la única cepa del serotipo Grumpensis de nuestra serie, será necesario recurrir a la secuenciación para caracterizar los casetes de resistencia contenidos en los integrones pues sólo obtuvimos amplificación para el gen aadA2; hasta donde sabemos nunca antes se había descrito la presencia de integrones en este serotipo.
La uniformidad en el patrón de resistencia y perfil de integrones que mostraban los aislados productores de OXA-1 y los de PSE-1 que, por otro lado, pertenecían en todos los casos menos uno al serotipo Typhimurium y se agrupaban en unos fagotipos concretos, sugiere que su propagación se debe fundamentalmente a la diseminación de un número limitado de clones. En el caso de S. ser Typhimurium DT 104 multirresistente, la particularidad de que los integrones que llevan insertados los genes pse-1 y aadA2 se hayan integrado en el cromosoma bacteriano formando parte de un locus cromosómico de resistencia denominado Salmonella Genomic Island 1 (SGI1), en el que también están presentes los determinantes de resistencia a tetraciclina (tetA) y a florfenicol y cloranfenicol (flo)11,12, les permite perpetuarse incluso en ausencia de presión antibiótica sin constituir un coste adicional para el microorganismo y ha conducido a su diseminación estable y de amplia distribución geográfica3,6,10,20,24-26. Por el contrario, la gran diversidad de perfiles de resistencia y la escasa incidencia de integrones entre los aislados productores de TEM-1 parece indicar que su aparición se debe principalmente a la transferencia genética entre aislados de distinto origen.
La constatación del patrón de resistencia y el perfil de integrones D (1.000 pb/aadA2 + 1.200 pb/pse-1) típicos de la cepa epidémica de S. ser Typhimurium DT104 de distribución universal en aislados del fagotipo U302, no fagotipificables o de patrón de lisis no reconocido e incluso del serotipo inmóvil [4,12:-:-] podría deberse a transmisión horizontal de estructuras genéticas de multirresistencia. Un estudio realizado en Grecia que demuestra que en este país los aislados de S. typhimurium DT104 multirresistentes están agrupados en varios clones diferentes27 y otro llevado a cabo en Bélgica en que se informa de varios aislados de S. enterica ser Agona con una agrupación genética de multirresistencia idéntica a la de S. ser Typhimurium DT10428, sugieren la posibilidad de transmisión horizontal de esta estructura de resistencia, que se cree que podrían formar parte de un transposón. De confirmarse este extremo nos encontraríamos ante una seria amenaza para la salud pública, ya que facilitaría la diseminación de genes de resistencia entre aislados de S. ser Typhimurium no relacionados genéticamente e incluso entre Salmonella de otros serotipos y otras especies de gramnegativos. Esta amenaza es tanto más preocupante en tanto en cuanto existen datos que sugieren que las infecciones producidas por S. ser Typhimurium DT104 multirresistentes tienden a producir cuadros infecciosos más graves que otros serotipos o fagotipos10. Otra posible explicación sería que algunos de nuestros aislados de S. ser Typhimurium DT104 hayan desarrollado una alteración en la susceptibilidad a los fagos empleados en la tipificación, tal y como han demostrado otros investigadores29 y, por lo tanto, a pesar de ser genéticamente indistinguibles de las cepas clásicas, hayan sido consideradas como pertenecientes a otros fagotipos al mostrar un patrón de lisis atípico. Por lo que respecta a los aislados portadores del integrón 2.000 pb/aadA1-oxa-1, cuya emergencia junto a la de los aislados con el integrón 2.000 pb/aadA1-oxa-30 ya se ha constatado en algunas zonas de España y Portugal2,13, resultaron sorprendentemente frecuentes en nuestra serie y estaban agrupados en diversos fagotipos (no fagotipificables, de fagotipo no reconocido, DT104-b y U302). Por lo tanto, tampoco puede excluirse la hipótesis de que, a pesar de haberse comprobado mediante técnicas de epidemiología molecular que los aislados de los dos anteriores países comparten un mismo perfil genómico13,30, en algunos casos haya tenido lugar una diseminación horizontal de las estructuras genéticas de resistencia; además, Tosini et al12 y Guerra et al30 demostraron en aislados del serotipo Typhimurium de pacientes albaneses y españoles que un integrón aparentemente idéntico al de nuestros aislados era transportado por un plásmido conjugativo12, 30.
En resumen, en nuestro ámbito geográfico la frecuencia de integrones en aislados de S. enterica productores de TEM-1 es muy baja, mientras que todos los aislados productores de betalactamasas de tipo OXA y PSE, que en su práctica totalidad pertenecen al serotipo Typhimurium, poseen integrones en cuya región variable están ubicados los determinantes de resistencia que codifican estas enzimas, circunstancia que podría haber facilitado su diseminación y explicar el incremento de aislados multirresistentes de S. ser Typhimurium portadoras de PSE-1 y especialmente de OXA-1 que hemos constatado en nuestro laboratorio en los últimos años. También cabe destacar la presencia de integrones, hasta ahora no descrita, en la única cepa de S. ser Grumpensis de nuestra serie.
Agradecimientos
Este trabajo ha sido parcialmente financiado por la Beca de Investigación de la Fundació Dr. Ferrán del año 2000. Agradecemos la inestimable colaboración técnica de la Sra. M. Pilar Cid Ventura y de la Sra. Mercé Escobedo Fontanet.