Introducción
En la actualidad hay descritas y reconocidas más de 100 especies del género Mycobacterium, aunque sólo un pequeño número de ellas se consideran patógenas estrictas para el hombre o los animales1. Las especies del complejo tuberculoso son las responsables de la mayoría de las enfermedades producidas por el género Mycobacterium. El uso de medios de cultivo más sensibles y más ricos que permiten el aislado de cepas de difícil crecimiento, el empleo cada vez más frecuente de técnicas agresivas en medicina, la utilización de fármacos inmunosupresores, la mayor supervivencia de los pacientes con inmunodeficiencias clásicas y, sobre todo, la aparición del virus de la inmunodeficiencia humana (VIH) han sido factores que han hecho aumentar sensiblemente la incidencia de enfermedad en los últimos años por especies micobacterianas no incluidas en el grupo M. tuberculosis complex llamadas micobacterias no tuberculosas (MNT)2,3.
M. kansasii se define como un patógeno oportunista que causa mayoritariamente enfermedad pulmonar, pudiendo ocasionar una infección diseminada que raramente se produce en pacientes no inmunodeprimidos, pero con frecuencia asociada con el sida4-9.
El reservorio natural de M. kansasii es aún desconocido. Algunos investigadores postulan que el agua es su hábitat natural por haber sido aislado ocasionalmente en muestras procedentes de grifos, duchas y sistemas de distribución de agua corriente, medios en los que se ha demostrado que es capaz de sobrevivir durante largos períodos de tiempo10-12. En muy pocas ocasiones se ha recuperado de agua de ríos o de lagos13 y excepcionalmente se ha aislado a partir de muestras procedentes de suelo o de animales14.
El análisis de la secuencia de la región 16S-23S rRNA15, y del gen 16S rRNA16, la amplificación mediante reacción en cadena de la polimerasa (PCR) y posterior digestión del gen hsp65 (PRA)17-19 y la detección de un fragmento de inserción IS165220 subdividen a esta especie en siete genotipos.
Durante los años 2000-2003, el Laboratorio de Referencia de Micobacterias ha identificado 3.086 cepas como micobacterias no tuberculosas, lo que representa el 27,8% de las micobacterias recibidas para su estudio. Las especies identificadas más frecuentemente fueron M. avium (562 cepas), M. gordonae (523), M. fortuitum (298) y M. kansasii (298).
El objeto de este trabajo es la caracterización tanto fenotípica (características morfológicas, de cultivo, bioquímicas y de sensibilidad a los diferentes fármacos antituberculosos) como genotípica (determinar la frecuencia de los siete genotipos mediante PRA) de estas cepas aisladas en España y enviadas al Centro Nacional de Microbiología para su estudio durante los últimos 4 años.
Métodos
Cepas
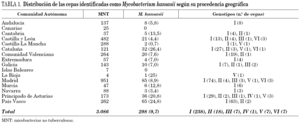
Se estudiaron 298 cepas (aisladas de 298 pacientes) identificadas como M. kansasii. La mayoría de los aislados (92,6%) procedían de muestras de origen respiratorio, y sólo en 2 pacientes con más de un aislado, las cepas se obtuvieron de diferentes localizaciones (pulmonar y ganglionar). Estas cepas fueron enviadas por 56 hospitales de 14 comunidades autónomas. La distribución geográfica de los aislados se muestra en la tabla 1. En 19 pacientes se obtuvieron aislados múltiples, y aunque sólo se incluyó en el estudio el primero, todos fueron analizados para conocer la reproducibilidad y estabilidad de las técnicas empleadas.
Como cepas tipo se incluyeron dos cepas de M. kansasii ATCC 25101 y ATCC 25100.
Caracterización morfológica y bioquímica
El Laboratorio de Micobacterias identificó todas las cepas como M. kansasii mediante la aplicación de métodos clásicos. El panel usado incluye las características del cultivo sobre medio de Löwenstein-Jensen (LJ): velocidad de crecimiento, producción de pigmento en oscuridad o después de fotoinducción, capacidad de crecimiento a 22, 30, 37 y 45 °C y sobre medios de cultivo hostiles (LJ con 5% de NaCl; LJ con 5 μg/ml de hidralazida del ácido tiofencarboxílico [TCH]; LJ con 10 μg/ml de tiosemicarbazona [TSC] y sobre agar MacConkey sin cristal violeta) y pruebas bioquímicas: producción de niacina, actividad catalásica a 68 °C, reducción de nitratos y telurito potásico, arilsulfatasa (3 días), hidrólisis del Tween 80 (lectura a los 1, 3 y 5 días) y ureasa1.
Pruebas de sensibilidad
Los estudios de sensibilidad se realizaron mediante el método de las proporciones descrito por Canetti et al21 sobre medio de Löwenstein-Jensen, elaborado en el Centro Nacional de Microbiología, frente a isoniazida (H) (0,2, 0,5 y 1 μg/ml), estreptomicina (S) (4 y 8 μg/ml), etambutol (E) (1,5 y 2 μg/ml); rifampicina (R) (20 y 40 μg/ml); PAS (P) (0,5 y 1 μg/ml); cicloserina (C) (30 y 40 μg/ml); kanamicina (K) (20 y 30 μg/ml), y etionamida (T) (20 y 30 μg/ml). La sensibilidad frente a pirazinamida (Z) (50) μg/ml se realizó por el método radiométrico Bactec (Becton Dickinson, EE.UU.) según las indicaciones del fabricante.
Análisis de los patrones de restricción del gen hsp65 (PRA)
Se resuspende un asa de bacteria obtenida a partir de un cultivo sobre LJ en 500 μl de agua destilada y se calienta a 100 °C en baño durante 5 min. Las muestras se centrifugan a 12.000 g durante 5 min y 5 μl del sobrenadante es utilizado para realizar la PCR.
Para la amplificación de un fragmento de 439 pb del gen hsp65 se utilizaron los primers Tb11 (ACCAACGATGGTGTGTCCAT) y Tb 12 (CTTGTCGAACCGCATA CCCT), previamente descritos por Telenti et al18. La PCR se realizó usando el kit comercial PureTaq Ready-To-Go PCR Beads (Amersham Biosciences, Piscataway NJ, EE.UU.) y las condiciones de la PCR se modificaron de las descritas previamente para mejorar la amplificación: un ciclo inicial de desnaturalización de 5 min a 94 °C, seguido por 35 ciclos de 1 min a 95 °C, 1 min a 60 °C, y 90 s a 72 °C. Diez μl del producto de amplificación fueron digeridos con BstEII y otros 10 μl con HaeIII (MBI Fermentas, España). Los fragmentos se analizaron por electroforesis en 3% de agarosa (8066; Pronadisa, Madrid, España), visualizándose con luz UV tras tinción con bromuro de etidio. Como marcador de peso molecular se utilizó el de 50 pb (Amersham Bioscences). Los patrones que corresponden a cada genotipo se muestran en la tabla 2.
El análisis estadístico se realizó para variables cualitativas mediante el test de la ji cuadrado (x2), con el programa informático Epi-Info.
Resultados
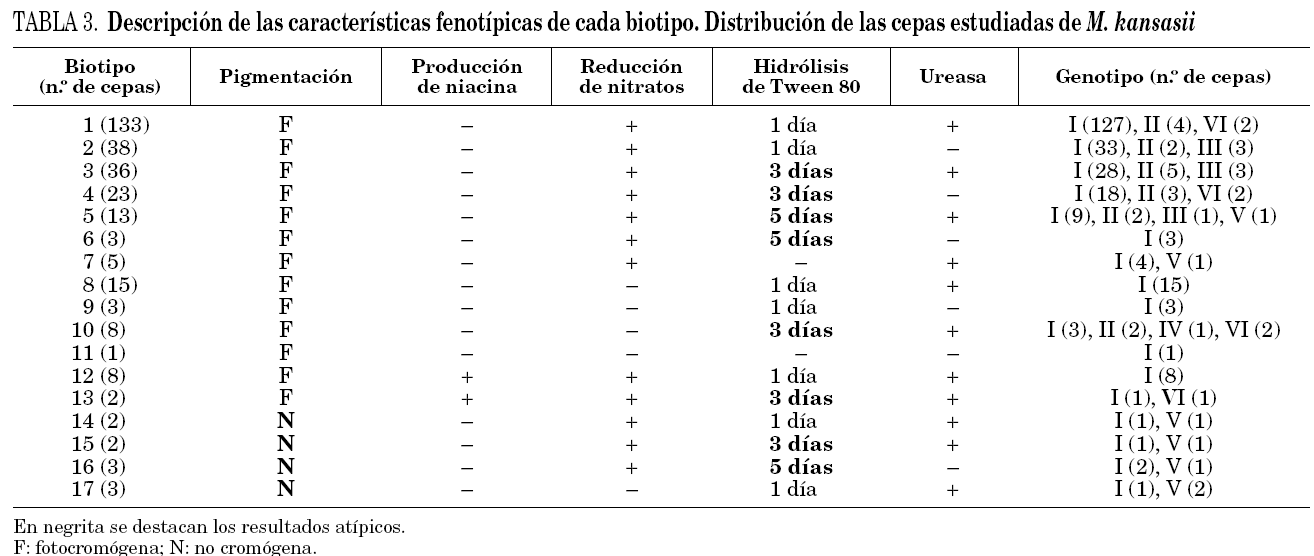
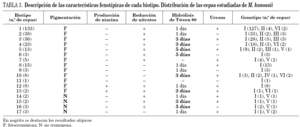
El panel utilizado para la identificación de las cepas demostró que el 100% de ellas crecían en más de 7 días, siendo 37 °C la temperatura óptima de crecimiento, inhibiéndose cuando las cepas se incubaron a 22 y 45 °C, crecieron en LJ con 5 μg/ml de TCH, y no se detectó crecimiento en presencia de 10 μg/ml de TSC, en 5% de NaCl ni en agar MacConKey sin cristal violeta. Todas ellas presentaron una fuerte actividad catalásica a 68 °C, ninguna redujo el telurito potásico, ni se detectó arilsulfatasa a los 3 días. Los resultados del resto de las pruebas estudiadas fueron variables lo que permitió definir 17 biotipos (tabla 3) con un índice de discriminación de Simpson del 0,77122. En 127 cepas (42,6%) se obtuvieron resultados que son atípicos con respecto a los que definen la especie, 10 cepas no produjeron pigmento tras fotoinducción, 10 produjeron niacina, 30 no redujeron los nitratos, 6 no hidrolizaron el Tween 80 y 90 lo hicieron más lentamente.
Se identificaron 6 de los 7 genotipos previamente descritos según la distribución que se muestra en la tabla 2. El índice de discriminación de Simpson fue de 0,24622.
Las cepas estudiadas que se aislaron en varias muestras clínicas de un mismo paciente mantuvieron el mismo biotipo y genotipo a lo largo del tiempo.
Los patrones de resistencia fueron bastante homogéneos según los datos que aparecen en la tabla 4. El 80,2% de las cepas tuvieron un mismo patrón, fueron resistentes a H, S, Z, P y K. Las cepas presentaron una resistencia moderada a H, todas ellas fueron sensibles a una concentración de H de 1 μg/ml, tuvieron una resistencia intermedia a 0,5 μg/ml, y fueron resistentes a 0,2 μg/ml.
En caso de aislados múltiples, los patrones de sensibilidad se modificaron tras el tratamiento en 4 de los 19 pacientes. En los 4 casos el patrón de resistencia para el primer aislado fue HSRZK, la cepa obtenida de una segunda muestra con una diferencia superior a 2 meses del primer aislado, además fue resistente a etambutol.
Discusión
Como se observa en la tabla 1, la distribución del porcentaje de aislados de M. kansasii con respecto al número total de MNT identificados en cada Comunidad Autónoma no es homogénea, con porcentajes que varían desde el 26,4% en Cataluña al 0,7% en Castilla-La Mancha. Esta diferencia podría ser debida a las diferentes características de cada comunidad. Las que tienen un mayor porcentaje de aislados poseen una actividad industrial mayor y los núcleos de población se agrupan en grandes zonas urbanas, en el otro extremo, en general, se encuentran comunidades con mayor actividad agrícola donde la población se agrupa en núcleos rurales más pequeños.
La variabilidad fenotípica de M. kansasii es bastante grande, lo que nos ha permitido definir 17 biotipos con un índice de discriminación de 0,771. El 42,6% de las cepas estudiadas tienen características fenotípicas diferentes de las definidas para esta especie, algunas de ellas claves para su identificación como son la producción de pigmento tras la exposición a la luz, o la de niacina23, ya descrita previamente, característica bioquímica casi exclusiva de M. tuberculosis. No existen en la literatura especializada estudios donde se combinen datos sobre la morfología y bioquímica, que permitan conocer la variabilidad fenotípica de esta especie, por lo que no se pueden comparar estos resultados.
Este gran número de cepas atípicas puede dificultar la identificación correcta de esta especie si sólo se utilizan las técnicas convencionales por lo que proponemos además la utilización de métodos moleculares.
La existencia de varios genotipos en M. kansasii está muy documentada en la literatura médica9,16,19,24,25. Como se observa en la tabla 2, nuestros resultados confirman esa heterogeneidad, pero con una distribución nada homogénea. La mayoría de las cepas (86,6%) pertenecían al genotipo I, no encontrando ninguna cepa perteneciente al genotipo VII recientemente descrito por Taillard et al9. El grado de variabilidad encontrado es superior a otros datos publicados previamente con cepas de M. kansasii de zonas geográficas concretas de España. En un estudio realizado en Bilbao por Gaafar et al26, el 99,2% de los aislados identificados como M. kansasii pertenecieron al genotipo I, y en un estudio anterior de Alcaide et al24 con cepas procedentes de muestras clínicas de Barcelona sólo identificaron cepas de M. kansasii del genotipo I. Por el contrario, la prevalencia de cepas de M. kansasii de origen humano en otros países de Europa, pertenecientes a otros genotipos distintos del I, es muy superior. En un estudio realizado en Suiza por Taillard et al9, sólo el 67% de las cepas pertenecían al genotipo I, el 21% pertenecían al genotipo II, el 8% al genotipo III y ya únicamente el 4% pertenecieron a los genotipos IV, VI y VII. En otro estudio similar llevado a cabo por Picardeau et al19, en Francia, en el genotipo I se agruparon el 44% de las cepas, el 33% en el genotipo II, el 5% en el genotipo III, el 10% en el genotipo IV, y el 8% en el genotipo V.
El bajo índice de discriminación que se obtuvo (0,246) hace pensar que el genotipado en España no es un buena técnica para estudios con fines epidemiológicos que permitan conocer el reservorio y las fuentes de infección, pero tiene gran utilidad para identificar correctamente cepas atípicas de M. kansasii.
Al analizar los resultados que se muestran en la tabla 3, sí parece existir una relación entre biotipo y genotipo. El 32,9% de las cepas del genotipo I presentaron características atípicas, mientras que en el resto de los genotipos este porcentaje fue del 72,5% (p = 0,0002). La relación entre las características de las cepas y el genotipo se hace más evidente al analizar los estudios de sensibilidad (tabla 4). Las cepas más sensibles pertenecieron a los genotipos V y III (p = 0,0000001).
Como conclusión cabe destacar que se ha obtenido una mayor discriminación para la diferenciación intraespecífica de M. kansasii cuando se analizan las características fenotípicas que las genotípicas. Creemos necesario la utilización conjunta de métodos convencionales y de métodos moleculares para la correcta identificación de cepas de M. kansasii por el alto número de cepas con características fenotípicas atípicas, algunas de ellas muy específicas para la especie como la fotocromogenicidad.