Rilpivirina (RPV) es un nuevo inhibidor no nucleósido de la transcriptasa inversa (INNTI) de segunda generación, aprobado para el tratamiento de pacientes con infección por virus de la inmunodeficiencia humana (VIH)-1 naïve con carga viral plasmática basal ≤ 100.000 copias/ml, junto con 2 inhibidores de la transcriptasa inversa análogos de nucleósidos (ITIAN). RPV es un derivado diarilpirimidínico con potente actividad antiviral in vitro frente a cepas salvajes de VIH-1 y conserva actividad frente a diversas variantes virales con mutaciones de resistencia a los INNTI de primera generación, como la mutación K103N. Los estudios in vitro y los ensayos clínicos fase III han permitido identificar 16 mutaciones de resistencia a RPV: K101E/P, E138A/G/K/Q/R, V179L, Y181C/I/V, Y188L, H221Y, F227C y M230I/L. El riesgo de fracaso virológico en pacientes tratados con RPV y 2 ITIAN con carga viral basal ≤ 100.000 copias/ml es bajo, pero en un elevado porcentaje de estos pacientes se selecciona resistencia, tanto a RPV como a ITIAN. La mutación de resistencia a RPV más frecuente en estos casos es la E138K, que habitualmente emerge en combinación con M184I, debido a un doble efecto compensatorio de esta asociación, ocasionando resistencia a RPV y, al mismo tiempo, resistencia a lamivudina y emtricitabina. La selección de resistencia a RPV confiere resistencia cruzada al resto de INNTI, destacando el alto porcentaje de casos con resistencia a etravirina.
Rilpivirine (RPV) is a new second-generation nonnucleoside reverse transcriptase inhibitor (NNRTI) approved for use in combination with two nucleoside/nucleotide reverse transcriptase inhibitors (NRTI) as initial therapy in treatment-naïve HIV-1-infected patients with a baseline viral load ≤100,000 copies/mL. RPV is a diarylpyrimidine derivative with potent in vitro activity against multiple HIV-1 variants with resistance mutations to first-generation NNRTI such as K103N. In vitro studies and phase III clinical trials have allowed the identification of 16 mutations associated with resistance to RPV K101E/P, E138A/G/K/Q/R, V179L, Y181C/I/V, Y188L, H221Y, F227C and M230I/L. The risk of virologic failure in patients receiving RPV plus 2 NRTI with plasma viral load ≤100,000 copies/mL is low, but a high percentage of patients failing RPV develop resistance mutations to both RPV and NRTI. The most common resistance mutation that emerges in this setting is E138K. This mutation is usually associated with M184I due to a double compensatory effect of this combination, which confers resistance to RPV, as well as to lamivudine and emtricitabine. The emergence of RPV resistance confers cross-resistance to all NNRTI and, importantly, high percentages of cross-resistance to etravirine.
Rilpivirina (RPV) es un nuevo inhibidor no nucleósido de la transcriptasa inversa (INNTI) de segunda generación aprobado para el tratamiento de pacientes con infección por virus de la inmunodeficiencia humana (VIH)-1 naïve con carga viral (CV) plasmática basal ≤ 100.000 copias/ml, junto con 2 inhibidores de la transcriptasa inversa análogos de nucleósidos (ITIAN). Igual que etravirina (ETR), RPV es un derivado diarilpirimidínico con actividad antiviral potente frente a cepas salvajes de VIH-1. Gracias a sus características moleculares, tiene actividad antiviral también frente a algunas variantes de VIH-1 resistentes a INNTI de primera generación, efavirenz (EFV) y nevirapina (NVP), dependiendo del patrón mutacional seleccionado1. De forma similar al resto de INNTI, RPV se une de forma no competitiva a un pequeño bolsillo hidrofóbico cercano al sitio activo de la transcriptasa inversa del VIH-1 e inhibe de forma alostérica su actividad enzimática y, por tanto, la replicación viral2. Sin embargo, dentro del sitio de unión a la transcriptasa inversa del VIH-1, RPV tiene una mayor afinidad por aminoácidos en posiciones muy conservadas. Estos son, por ejemplo, el residuo Trp229, una posición donde se producen menos mutaciones que en otras posiciones fundamentales para la unión de EFV y NVP como son Tyr181 y Tyr 1883. Además, gracias a la flexibilidad que le confieren sus 3 anillos aromáticos, RPV es una molécula capaz de adoptar diferentes cambios de conformación y unirse al sitio diana, incluso en presencia de mutaciones de resistencia en posiciones clave que impiden la unión de los INNTI de primera generación. Así, por ejemplo, la presencia de la mutación K103N, la más frecuente en los pacientes que fracasan a un tratamiento con ITINAN de primera generación, sobre todo EFV, y que confiere resistencia completa a EFV y NVP, no compromete la actividad de RPV1–3.
La ausencia de afectación de la sensibilidad a RPV en presencia de variantes virales con una única mutación de resistencia a NVP o EFV en estudios in vitro, sugirió inicialmente que la barrera genética para la selección de resistencia del VIH-1 de RPV era mayor que en el caso de los INNTI de primera generación1. Sin embargo, los ensayos clínicos en los que se ha evaluado la eficacia de RPV en pacientes con infección por VIH-1 han puesto de manifiesto que en los pacientes tratados con 2 ITIAN y RPV, el riesgo de fracaso virológico y de selección de mutaciones de resistencia es mayor que en los tratados con 2 ITIAN y EFV, especialmente en el subgrupo de pacientes con una CV plasmática (ARN VIH-1) > 100.000 copias/ml4–8.
En este artículo se revisan los estudios in vitro y ensayos clínicos que han permitido definir el perfil de resistencia de RPV.
Estudios in vitroEl punto de corte establecido inicialmente para definir la resistencia fenotípica a RPV in vitro (biological cut-off, BCO) es un aumento de la CI50 respecto a la cepa salvaje (fold change, FC) de 3,7 veces1.
Actividad de rilpivirina frente a variantes de VIH-1 con resistencia a inhibidores no nucleósidos de la transcriptasa inversaEn estudios iniciales se evaluó la actividad de RPV in vitro frente a 55 variantes de VIH-1 con mutaciones de resistencia a INNTI obtenidas mediante mutagénesis dirigida1. RPV mantiene su actividad antiviral, de forma aislada, en presencia de la mayoría de mutaciones de resistencia a ITINAN: V90I, L100I, K101E/Q, K103N/S, V106A/M,V108I, E138A/G/K/Q/R/S, V179D/E/F/T, Y181C, Y188L, G190A/S, H221Y, M230I/L/V y M236L. La mutación Y181C no confiere por sí sola resistencia a RPV (FC 2,7) mientras que las mutaciones K101P (FC 51,7) y Y181I/V (FC 15,3 y 12, respectivamente) sí reducen la actividad de RPV de forma importante in vitro. La presencia de la mutación K103N no afecta a la actividad de RPV, pero en combinación con otras mutaciones sí puede aumentar la resistencia a RPV (p. ej., K103N+L100I que confiere un FC de 7 y, sobre todo, K103N+Y181I que confiere un FC de 95)1.
De las 16 variantes de VIH-1 estudiadas con 2 mutaciones de resistencia a ITINN, RPV mostró actividad frente a 7 de ellas (L100I+K101E, K101E+K103N, K103N+F227L, K103N+V108I, K103N+Y181C, V179I+Y181C, Y181C+G190S), mientras que se observó resistencia en el resto (L100I+K103N, K103N+Y181I, E138K+M230L, V179D+Y181C, V179E+Y181C, V179F+Y181C, V179F+Y181I, Y181C+F227L, Y181C+Y188L). Se estudiaron 8 variantes de VIH-1 con 3 mutaciones de resistencia a ITINN y todas ellas se mostraron resistentes a RPV1.
Por otro lado se estudió la actividad de RPV in vitro frente a 4.786 muestras clínicas con mutaciones de resistencia a INNTI de primera generación, EFV o NVP. Se observó que el 62% de las cepas virales mantenía sensibilidad a RPV, un porcentaje similar al de actividad de ETR, mientras que solo el 11% era sensible a EFV y el 5% a NVP1.
Más recientemente se ha estudiado el efecto de otras mutaciones sobre la actividad de RPV, tanto en muestras clínicas como en estudios de mutagénesis dirigida. La mutación E138A confiere resistencia a RPV de forma similar a E138G/K/Q9. Otro estudio reciente, en el que se analizaron genotipos y fenotipos de 20.004 muestras clínicas, ha demostrado que la mutación Y188L confiere resistencia a RPV con un FC de 9,2, situándose en cuarto lugar en cuanto al impacto sobre la actividad de RPV, por detrás de K101P, Y181I e Y181V. El estudio de la actividad de RPV frente a mutantes Y188L obtenidos mediante mutagénesis dirigida confirmó estos resultados10.
Emergencia de mutaciones de resistencia a rilpivirina en cultivos celularesEn estudios realizados en cultivos con células T linfoblásticas humanas infectadas con la cepa de VIH-1 IIIB, así como con virus recombinantes de muestras clínicas de diferentes subtipos de HIV-1 grupo M (A1, AE, AG, B, BG, C, D, F1, G, y H) con alta multiplicidad de infección (ratio de la cantidad de virus inoculado por célula del cultivo celular), no aparecieron mutaciones de resistencia después de 32 días de exposición a RPV en concentraciones ≥ 40nM (14,66ng/ml) (CI50 de RPV para cepa salvaje de VIH-1 y corregida por unión a proteínas plasmáticas: 13,5ng/ml; Ctrough de RPV en ensayos clínicos fase III: 80ng/ml)11. Tampoco se observaron nuevas mutaciones cuando RPV se expuso a variantes de VIH-1 con las mutaciones K103N o Y181C1. En cambio, concentraciones de RPV de 10nM (3,66ng/ml) permitieron la aparición de diferentes combinaciones de las mutaciones L100I, K101E, V106I, Y181C, Y181I y M230I.
Por otro lado, en experimentos con cepas salvajes de subtipos de VIH-1 AE, AG, B, C y D, y cepas resistentes de subtipo B, con baja multiplicidad de infección y exposición a dosis crecientes de RPV, se observó emergencia de nuevas mutaciones de resistencia con concentraciones iniciales de RPV entre 5nM y 16μM. Un mayor número de mutaciones de resistencia se asoció con un nivel más alto de resistencia a RPV1.
En conjunto, los estudios genotípicos de las cepas virales emergentes bajo presión selectiva de RPV, realizados en experimentos con variantes de VIH-1 de diferentes orígenes y perfiles genéticos, ha mostrado un patrón de resistencia a RPV in vitro que incluye las siguientes mutaciones: V90I, L100I, K101E, V106A/I, V108I, E138G/K/Q/R, V179F/I, Y181C/I, Y188L, V189I, G190E, H221Y, F227C y M230I/L1.
Los estudios in vitro han mostrado que RPV tiene una barrera genética para la selección de resistencia in vitro superior a los INNTI de primera generación y que mantiene actividad frente a un gran número de variantes de VIH-1 resistentes a EFV y NVP. El patrón genotípico de resistencia a RPV muestra bastante similitud con el patrón de resistencia a ETR y, por tanto, es esperable que exista una amplia resistencia cruzada entre ambos fármacos1,12.
Resistencia a rilpivirina en ensayos clínicos en pacientes que reciben tratamiento triple con rilpivirina y 2 inhibidores de la transcriptasa inversa análogos de nucleósidosLa información acerca de la selección de mutaciones de resistencia a RPV y el impacto de estas mutaciones en la reducción de la actividad de RPV en pacientes tratados con este fármaco, procede principalmente de los ensayos clínicos fase III ECHO (Efficacy Comparison in Treatment-naive, HIV-infected Subjects of TMC278 and Efavirenz), THRIVE (TMC278 against HIV, in a once-daily regimen versus efavirenz) y STaR (Single-Tablet Regimen)4–5,8. En estos ensayos clínicos se comparó la eficacia y seguridad de RPV a dosis de 25mg 1 vez al día con EFV a dosis de 600mg al día, ambos administrados junto con 2 ITIAN (tenofovir/emtricitabina [TDF/FTC] en los estudios ECHO y STaR, y TDF/FTC o abacavir/lamivudina [3TC] o zidovudina/3TC en el estudio THRIVE). Se excluyó a los pacientes en los que se hubiera documentado, en el estudio genotípico previo al inicio del tratamiento, la presencia de alguna mutación de resistencia a INNTI entre un listado de 39 mutaciones (A98G, L100I, K101E/P/Q, K103H/N/S/T, V106A/M, V108I, E138A/G/K/Q/R, V179D/E, Y181C/I/V, Y188C/H/L, G190A/ C/E/Q/S/T, P225H, F227C, M230I/L, P236L, K238N/T e Y318F)4,5,8.
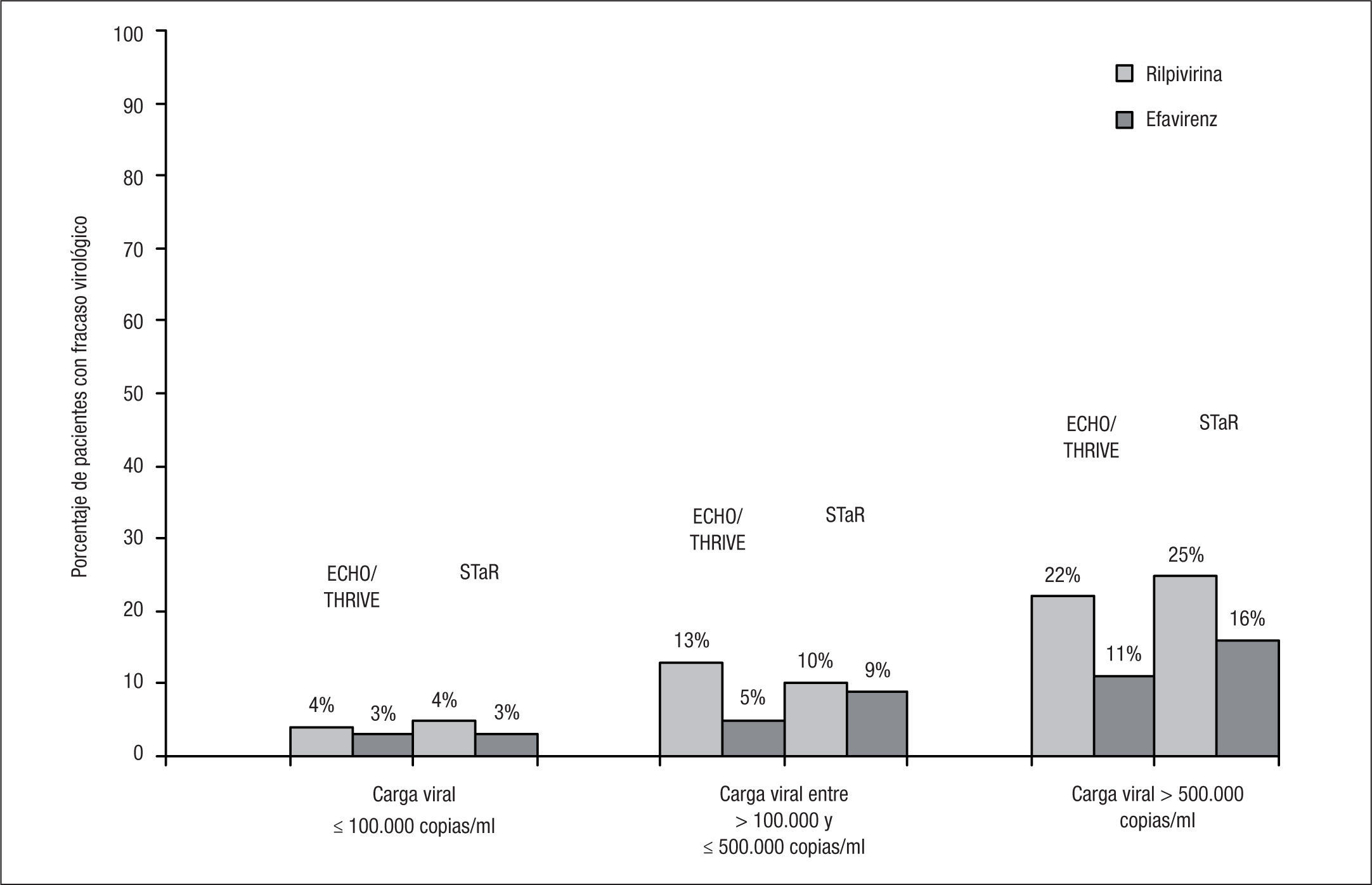
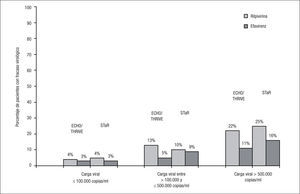
Los porcentajes de respuesta terapéutica a las 48 semanas en el análisis conjunto de los estudios ECHO y THRIVE, según el criterio de valoración principal (CV confirmada < 50 copias/ml en el análisis por intención de tratamiento-TLOVR) fueron el 84% en la rama de RPV y el 82% en la rama de EFV, cumpliéndose el criterio de no inferioridad de RPV respecto a EFV6. En el estudio STaR, la respuesta terapéutica a las 48 semanas (supresión virológica < 50 copias/ml según análisis snapshot) fue del 86% en la rama RPV/FTC/TDF y del 82% en la rama EFV/FTC/TDF, cumpliendo también el criterio de no inferioridad de RPV respecto a EFV8. Sin embargo, en los estudios ECHO/THRIVE, un mayor porcentaje de pacientes tratados con RPV presentó fracaso virológico (el 9 frente al 5%), que se agruparon especialmente en los sujetos con CV basal > 100.000 copias/ml. En estos pacientes, el porcentaje de fracasos virológicos ascendió al 17% en el grupo de RPV y al 7% en el grupo de EFV6. En el estudio STaR se observó una tasa significativamente mayor de fracaso virológico con RPV solo en sujetos con CV > 500.000 copias, aunque no en aquellos con 100.000–500.000 copias/ml8 (fig. 1).
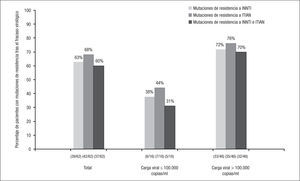
En los pacientes con fracaso virológico y estudio genotípico de resistencia disponible en los estudios ECHO y THRIVE, no se hallaron diferencias significativas en el porcentaje de nuevas mutaciones de resistencia a INNTI entre los pacientes tratados con RPV o con EFV (el 63 frente al 54%; p=0,49), mientras que el porcentaje de nuevas mutaciones de resistencia a ITIAN (principalmente M184I/V) fue significativamente superior en el grupo de RPV (el 68 frente al 32%; p=0,003). No hubo diferencias en los porcentajes de selección de mutaciones de resistencia en ambos grupos según se tratara de pacientes infectados por VIH-1 subtipo B o subtipo distinto al B7.
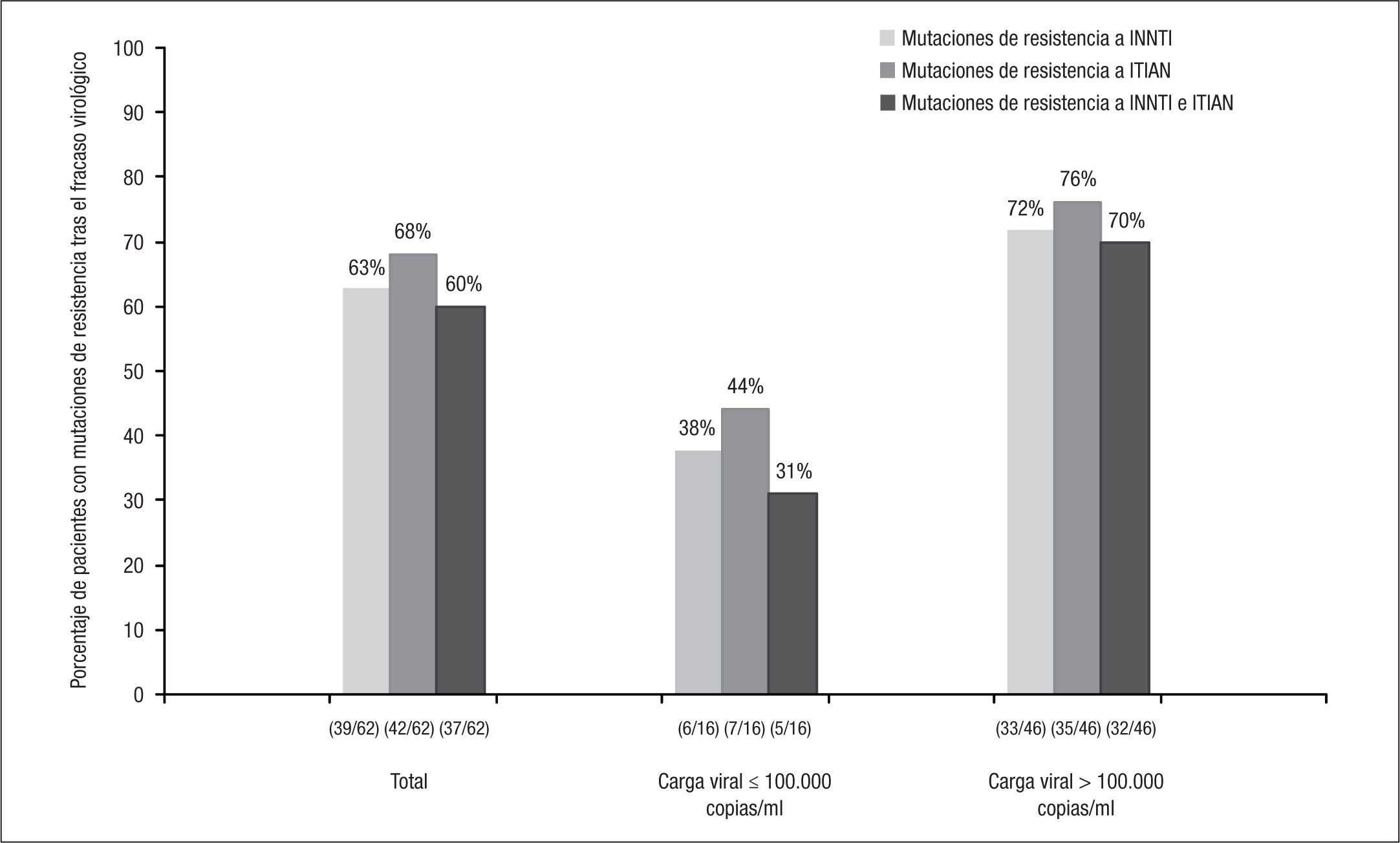
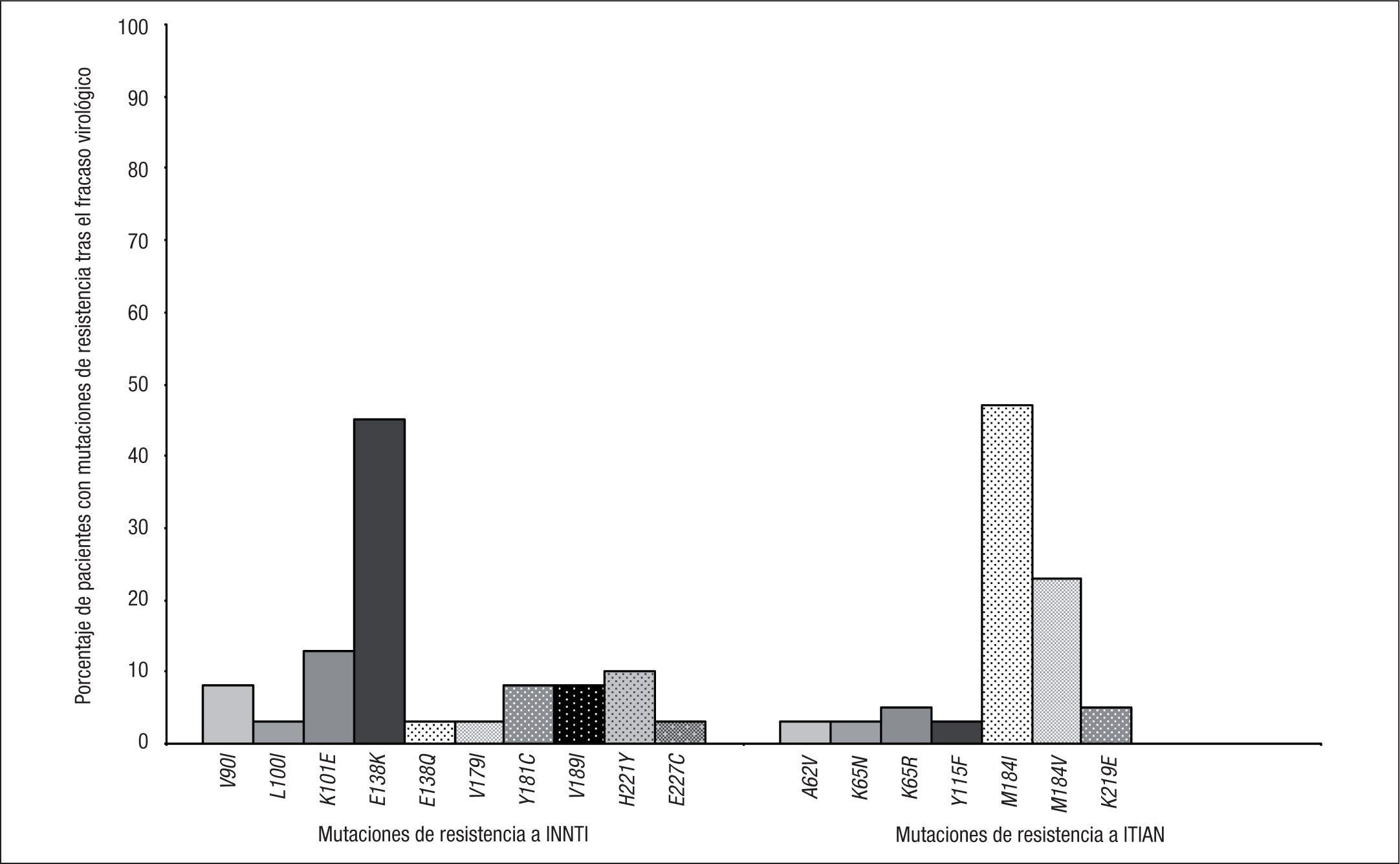
Se dispuso de estudio genotípico de resistencia en 62 de los 72 pacientes que presentaron fracaso virológico en los grupos de tratamiento con RPV + 2 ITIAN. De estos, en 44/62 (71%) se documentaron nuevas mutaciones de resistencia, en 39/62 (63%) mutaciones de resistencia a INNTI, en 42/62 (68%) a ITIAN y en 18/62 (29%) a ambas familias. Los porcentajes de mutaciones de resistencia a INNTI, ITIAN o a las 2 familias fueron superiores en los pacientes con CV basal > 100.000 copias/ml en comparación con los que tenían una CV basal ≤ 100.000 copias/ml (fig. 2). Las mutaciones de resistencia a INNTI más frecuentes (detectadas en al menos 2 pacientes) fueron E138K (45%), K101E (13%), H221Y (10%), V90I, Y181C, V189I (8% cada una), L100I, V179I, E138Q y F227L (3% cada una) (fig. 3). En cuanto a las mutaciones de resistencia a ITIAN, las más frecuentes tras el fracaso virológico a RPV fueron M184I (47%), M184V (23%), K219E, K65R (5% cada una), Y115F, K65N y A62V (3% cada una) (fig. 3). Estos datos fueron confirmados en el estudio STaR. En este estudio, los porcentajes de pacientes que desarrollaron resistencia a RPV tras el fracaso virológico fueron el 2, 5 y 19%, según la CV basal < 100.000, 100.000–500.000 y > 500.000 copias/ml, respectivamente, y las mutaciones de resistencia más frecuentes fueron E138K/Q, Y181C/I y K101E para los INNTI, y M184V/I para los ITIAN8.
La mutación de resistencia a RPV más frecuente en estos ensayos clínicos fue E138K, una mutación muy poco habitual en los genotipos de VIH-1 de pacientes tratados con otros INNTI (EFV, NVP o ETR) y cuyo impacto clínico no era bien conocido hasta el momento. Únicamente, un estudio realizado con muestras clínicas había mostrado resistencia de bajo grado a ETR en clones virales con la mutaciónE138K, sobre todo en subtipos de VIH-1 no B13. En los ensayos clínicos ECHO, THRIVE y STaR, la mutación E138K no se observó de forma aislada en ningún caso, apareciendo siempre en asociación con otras mutaciones de resistencia a INNTI y/o ITIAN. Por otro lado, la mutación de resistencia a ITIAN más frecuente fue M184I, una mutación que confiere resistencia de alto grado a 3TC y FTC, pero que no se detecta habitualmente, ya que aparece como mutación de transición en fases muy precoces del fracaso virológico y es sustituida posteriormente por M184V.
En los estudios ECHO/THRIVE, 37 pacientes (60%) de los que tuvieron fracaso virológico seleccionaron la combinación de al menos 1 mutación de resistencia a INNTI y otra a ITIAN. La combinación más frecuente fue E138K+M184I, presente en 17 pacientes (46%) y en 6 de ellos (16%) estas fueron las únicas mutaciones que aparecieron. Otras combinaciones frecuentes fueron E138K+M184V (22%) y E138K+K101E+M184I (11%)7.
No se observaron diferencias en los porcentajes de emergencia de mutaciones de resistencia a INNTI e ITIAN ni tampoco en la frecuencia de estas mutaciones o combinaciones de mutaciones en función del subtipo viral (VIH-1 subtipo B o subtipo no B)7.
La presencia basal de alguna de las 4 mutaciones de resistencia a INNTI permitidas para la inclusión en los ensayos clínicos ECHO y THRIVE (V90I, V106I, V179I y V189I) no se asoció con la respuesta clínica a las 48 semanas de tratamiento con RPV14.
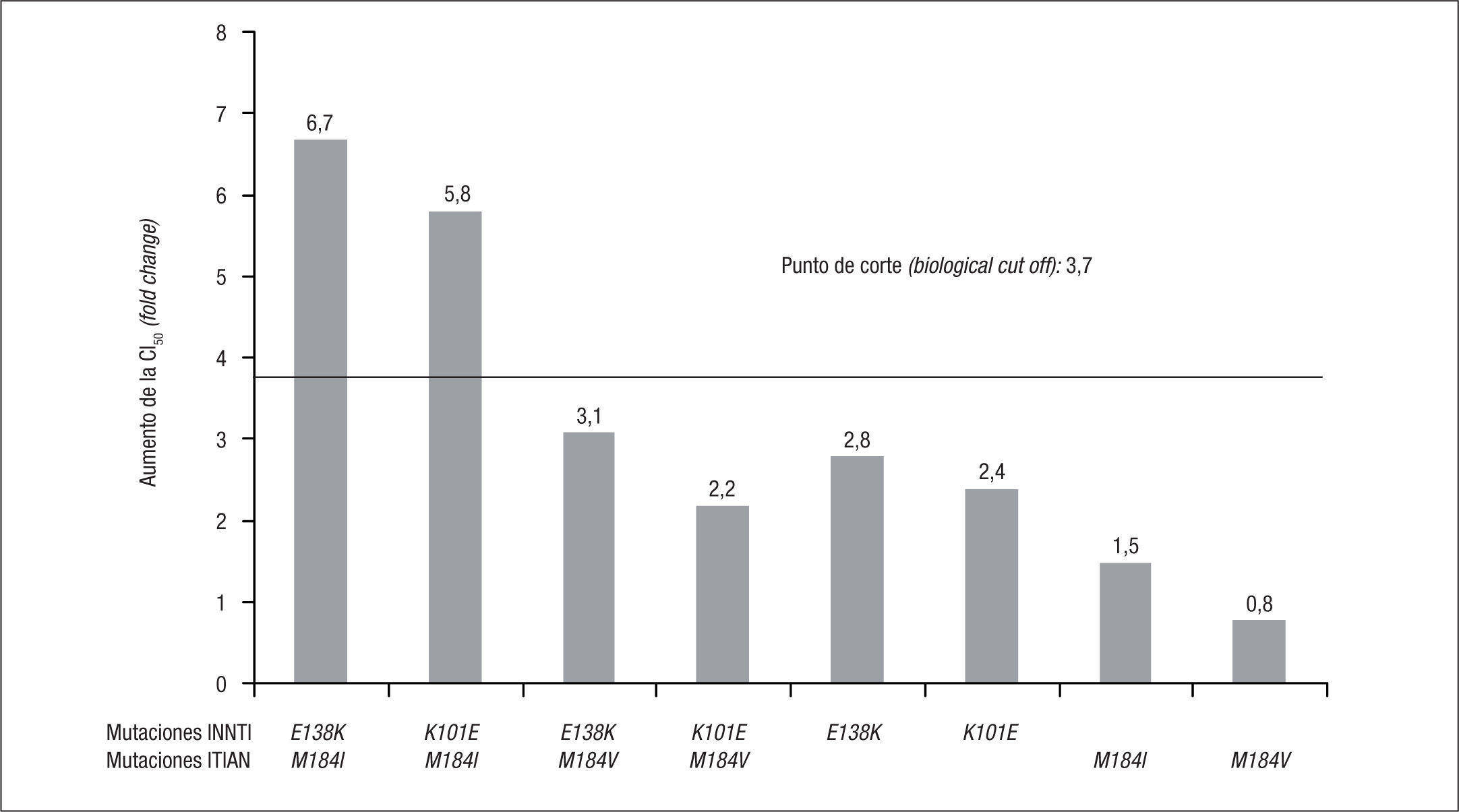
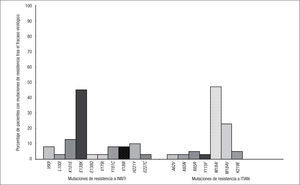
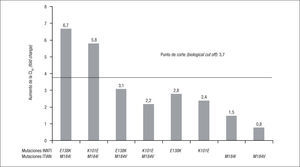
Datos fenotípicosLos estudios fenotípicos permiten una mejor interpretación de la resistencia, ya que los estudios genotípicos no pueden medir las interacciones que tienen lugar entre algunas mutaciones que pueden ser antagonistas, compensatorias o sinérgicas. Los estudios de resistencia en los que se correlaciona el genotipo y el fenotipo viral permiten conocer el nivel de resistencia al fármaco que confiere la presencia de cada mutación o asociación de mutaciones. En el estudio de resistencia realizado dentro de los ensayos clínicos ECHO y THRIVE se observó que en el 50% de los pacientes con fracaso virológico a RPV (31/62), el nivel de resistencia a RPV era superior al BCO (FC o aumento de la CI50 > 3,7 veces). En los pacientes con CV basal < 100.000 copias/ml, el porcentaje de resistencia fenotípica a RPV tras el fracaso virológico fue del 13% (2/16 pacientes) mientras que en aquellos con CV basal > 100.000, el porcentaje de resistencia fenotípica a RPV fue del 63% (29/46 pacientes)7. Para confirmar el impacto sobre la actividad de RPV de las mutaciones y combinaciones más frecuentes tras el fracaso virológico con RPV y 2 ITIAN, se realizaron estudios de sensibilidad fenotípica de cepas de VIH-1 mutantes obtenidas mediante mutagénesis dirigida. Las mutaciones E138K y K101E disminuyen la sensibilidad a RPV sin conferir resistencia completa (FC 2,8 y 2,4, respectivamente). Las combinaciones de E138K o K101E con M184I sí confieren resistencia a RPV (FC > 3,7), pero, en cambio, no ocurre lo mismo si estas mutaciones se asocian con M184V7 (fig. 4).
Nivel de resistencia a rilpivirina (RPV) que confieren las mutaciones más frecuentes en el fracaso virológico con RPV e inhibidores de la transcriptasa inversa análogos de nucleósidos (ITIAN). INNTI: inhibidores no nucleósidos de la transcriptasa inversa; VIH: virus de la inmunodeficiencia humana. Adaptado de Rimsky et al7.
Por otro lado, en los casos con resistencia fenotípica a RPV tras el fracaso virológico se observó resistencia cruzada con el resto de INNTI, destacando el alto porcentaje de casos con resistencia cruzada a ETR (EFV 87%, NVP 48%, ETR 90%)7.
Mecanismos de selección de resistencia a rilpivirinaAsociación de las mutaciones E138K y M184I tras el fracaso virológicoLas mutaciones de resistencia más frecuentes observadas tras el fracaso virológico con RPV y 2 ITIAN en los ensayos clínicos ECHO/ THRIVE y STaR fueron E138K y M184I, que muy frecuentemente emergen asociadas.
La combinación E138K+M184I y su impacto sobre la resistencia a RPV podría explicarse por un doble efecto sinérgico y compensatorio de ambas mutaciones. Por un lado, la mutación E138K confiere resistencia de bajo nivel a RPV (FC de 2,2 a 2,8)1,7,12,13. Por otro lado, la mutación M184I/V confiere resistencia de alto nivel a 3TC y FTC, pero no a RPV. Sin embargo, estudios in vitro han demostrado que la asociación E138K+M184I incrementa la resistencia a RPV (FC de 4,1 a 6,7), algo que no sucede con la combinación E138K+M184V7,15,16. La mutación E138K altera el equilibrio de unión/disociación de RPV resultando en un aumento de la tasa de disociación y una pérdida de afinidad por el sitio de unión en la transcriptas inversa17. El modo mediante el cual la combinación M184I aumenta la resistencia a RPV en presencia de E138K no es, de momento, bien conocido, pero podría estar relacionado con una mayor desestabilización del bolsillo hidrofóbico de la transcriptasa inversa y, por tanto, mayor dificultad de unión de RPV en presencia de las 2 mutaciones15.
Además, la mutación E138K podría compensar la pérdida de capacidad replicativa viral producida por M184I, pero no tanto en el caso de M184V, de forma que las variantes virales con las mutaciones E138K+M184I podrían tener cierta ventaja replicativa en comparación con E138K+M184V. Hu et al observaron que la capacidad replicativa de los dobles mutantes E138K+M184I y E138K+M184V era superior que la de virus con las mutaciones E138K, M184I o M184V de forma aislada y de virus wild type en presencia de diferentes concentraciones de RPV y de RPV+3TC. Además, este aumento de la capacidad replicativa era superior con la combinación E138K+M184I que con 138K+M184V16. Singh et al demostraron que la mutación M184I disminuye la actividad catalítica de la transcriptasa inversa y la incorporación de nucleótidos, y que la combinación E138K+M184I puede restaurar esta actividad enzimática17. Aunque este hecho no se ha confirmado en otros estudios15, ya se había observado también con mutantes E138K+M184I expuestos a ETR18,19.
Por otra parte se ha observado que, como resultado de la hipermutagénesis viral condicionada por la actividad de enzimas APOBEC3, podrían preexistir variantes de VIH-1 con la combinación de mutaciones E138K+M184I en el reservorio proviral de algunos pacientes sin exposición previa a RPV ni ETR20, lo que, en algunos casos, podría favorecer el fracaso virológico durante el tratamiento con rilpivirina y FTC o 3TC.
Antagonismo entre las mutaciones E138K y Y181CLas mutaciones E138K y Y181C pueden seleccionarse en presencia de RPV, y condicionan pérdida de actividad del fármaco aunque, por sí solas, no presentan resistencia completa1. En los genotipos virales estudiados de los pacientes con fracaso virológico en los ensayos clínicos ECHO y THRIVE se observó la mutación Y181C en 5 pacientes, siempre en asociación con otras mutaciones y solo en un paciente asociada con E138K7. Estudios in vitro han demostrado que la presencia de la mutación Y181C dificulta la emergencia de E138K y que, por otro lado, la combinación de Y181C+E138K no mejora la capacidad replicativa viral en comparación con la presencia de E138K sola21,22.
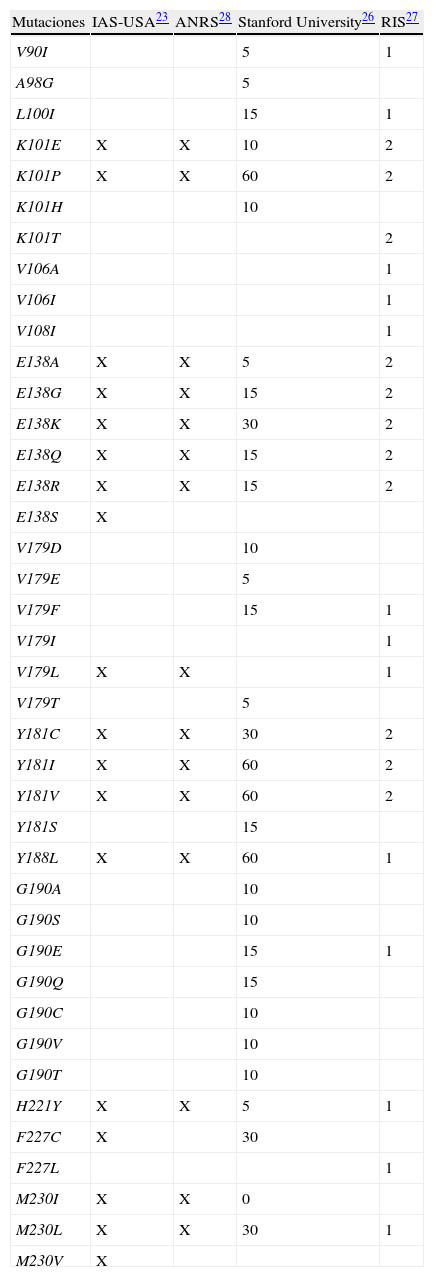
Valoración genotípica de resistencia a rilpivirina: algoritmos de interpretaciónTeniendo en cuenta de forma conjunta las mutaciones observadas tras el fracaso virológico con RPV en ensayos clínicos, las mutaciones seleccionadas tras la exposición a RPV in vitro y las mutaciones asociadas con resistencia fenotípica a RPV en los estudios in vitro, tanto en estudios de mutagénesis dirigida como en muestras clínicas, se han identificado 16 mutaciones de resistencia asociadas con disminución de la sensibilidad del VIH-1 a RPV: K101E/P, E138A/G/K/Q/R, V179L, Y181C/I/V, Y188L, H221Y, F227C, M230I/L. Este listado de mutaciones es el que considera la asociación IAS-USA como patrón de resistencia genotípica a RPV23 y es el que se contempla en la ficha técnica del fármaco (excluyendo Y188L)24,25. Otros algoritmos de interpretación de resistencia basados en el genotipo incluyen otras mutaciones con efecto demostrado sobre la actividad de RPV in vitro y también otras mutaciones que podrían reducir la actividad de RPV26–28. En la tabla 1 se resumen los listados de mutaciones de resistencia a RPV contemplados en las principales guías de interpretación genotípica. Los algoritmos de interpretación de la Universidad de Stanford26 y de la Red Española de Investigación en SIDA (RIS)27 asignan una puntuación a cada mutación en función del impacto sobre la actividad del fármaco, y se corrigen continuamente a medida que se generan nuevos datos.
Mutaciones de resistencia a rilpivirina en las guías y algoritmos de interpretación genotípica*
| Mutaciones | IAS-USA23 | ANRS28 | Stanford University26 | RIS27 |
| V90I | 5 | 1 | ||
| A98G | 5 | |||
| L100I | 15 | 1 | ||
| K101E | X | X | 10 | 2 |
| K101P | X | X | 60 | 2 |
| K101H | 10 | |||
| K101T | 2 | |||
| V106A | 1 | |||
| V106I | 1 | |||
| V108I | 1 | |||
| E138A | X | X | 5 | 2 |
| E138G | X | X | 15 | 2 |
| E138K | X | X | 30 | 2 |
| E138Q | X | X | 15 | 2 |
| E138R | X | X | 15 | 2 |
| E138S | X | |||
| V179D | 10 | |||
| V179E | 5 | |||
| V179F | 15 | 1 | ||
| V179I | 1 | |||
| V179L | X | X | 1 | |
| V179T | 5 | |||
| Y181C | X | X | 30 | 2 |
| Y181I | X | X | 60 | 2 |
| Y181V | X | X | 60 | 2 |
| Y181S | 15 | |||
| Y188L | X | X | 60 | 1 |
| G190A | 10 | |||
| G190S | 10 | |||
| G190E | 15 | 1 | ||
| G190Q | 15 | |||
| G190C | 10 | |||
| G190V | 10 | |||
| G190T | 10 | |||
| H221Y | X | X | 5 | 1 |
| F227C | X | 30 | ||
| F227L | 1 | |||
| M230I | X | X | 0 | |
| M230L | X | X | 30 | 1 |
| M230V | X |
X indica que la mutación esta incluida en cada uno de estos listados.
Los algoritmos de la Universidad de Stanford y de la RIS asignan una puntuación a cada mutación en función del grado de resistencia.
Algoritmo de la Universidad de Stanford: 0–9 puntos, sensible; 10–14 puntos, resistencia potencial de bajo nivel (el virus será sensible al fármaco pero las mutaciones indican exposición previa a fármacos de la misma clase); 15–29, resistencia de bajo nivel; 30–59, resistencia intermedia; ≥ 60, resistencia de alto nivel.
Algoritmo de la RIS: ≤ 2 puntos, sensible; ≥ 3 puntos, resistente.
La prevalencia de mutaciones de resistencia a RPV en pacientes naïve es baja. En los ensayos clínicos ECHO y THRIVE se realizó un cribado a 1.895 sujetos, 1.796 de ellos con estudio genotípico14. De estos se observaron mutaciones de resistencia a INNTI en el 21% y mutaciones de resistencia a RPV en el 5,2%, siendo la más frecuente E138A (2,7%), seguida de K101E y E138K (0,4%), aunque ninguna de ellas por separado confiere resistencia al fármaco. En un estudio realizado en Francia entre 2008 y 2011, la prevalencia de mutaciones de resistencia a RPV en pacientes naïve con infección por VIH-1 fue del 4,6% (el 3,7% en genotipos de subtipo B y el 6% en subtipo no B), y las mutaciones más frecuentes fueron E138A (3%), E138K (0,3%), H221Y (0,3%), E138G (0,3%) e Y181C (0,2%)29.
También se ha estudiado la prevalencia de mutaciones de resistencia a RPV en 15.991 muestras clínicas de pacientes en tratamiento antirretroviral remitidas para realizar estudio genotípico de resistencia en Estados Unidos, entre enero de 2010 y junio de 2012. La mayoría de mutaciones de resistencia a RPV aisladas o en combinación fueron poco frecuentes (≤ 3%) a excepción de Y181C (7%). La presencia de mutaciones de resistencia a RPV se asoció con disminución de actividad de RPV y también de ETR en estudios fenotípicos30.
Un estudio realizado en España ha evaluado la presencia de mutaciones de resistencia y la potencial actividad de RPV en pacientes con infección por VIH-1 con fracaso previo con INNTI24. Se analizaron genotipos procedentes de 1.064 pacientes con fracaso previo a EFV, NVP y ETR. Las mutaciones más frecuentes (> 1%) fueron: K101E (9,1%), K101P (1,4%), E138A (3,9%), Y181C (21,8%), H221Y (8,3%) y M230L (1,5%). La mutación E138K se observó en un 0,3%, la asociación K101E+M184I en el 1% y no se encontró la combinación E138K+M184I. Se estimó resistencia a RPV según el algoritmo de la RIS en el 19,3% de los pacientes, siendo menor en los pacientes con fracaso a EFV (14,5%) y mayor en aquellos con fracaso a NVP (25%) y ETR (27,6%)27.
Eficacia de rilpivirina en pacientes con infección por VIH-1 con mutaciones de resistencia a inhibidores no nucleósidos de la transcriptasa inversaA pesar de que RPV ha demostrado actividad in vitro frente a cepas de VIH-1 con una mutación de resistencia a INNTI y actividad frente a una gran variedad de cepas de VIH-1 con resistencia fenotípica a INNTI de primera generación, no se dispone de información suficiente de ensayos clínicos que hayan evaluado la eficacia de RPV en pacientes con fracaso previo y resistencia a INNTI. Se ha publicado únicamente un pequeño ensayo clínico fase II en el que se comparó la eficacia de RPV a dosis de 25, 50 y 150mg como sustituto de un inhibidor de la proteasa potenciado con ritonavir (IP/r) o un INNTI, ambos junto con 2 ITIAN, en pacientes con fracaso virológico y, al menos, una mutación de resistencia a INNTI. Al octavo día de tratamiento (objetivo primario) se observó una reducción de la CV plasmática de 0,71 log10 copias/ml cuando se sustituyó un INNTI, y de 1,19 log10 copias/ml cuando se sustituyó un IP/r31. Por tanto, no se dispone de información suficiente para poder evaluar la utilidad de RPV en este escenario clínico.
ConclusionesLos estudios in vitro y los datos de los ensayos clínicos fase III han permitido identificar 16 mutaciones asociadas con resistencia a RPV: K101E/P, E138A/G/K/Q/R, V179L, Y181C/I/V, Y188L, H221Y, F227C y M230I/L. La mayoría de estas mutaciones son poco prevalentes en pacientes que no han recibido tratamiento antirretroviral, y no suelen conferir resistencia de moderado o alto nivel por sí solas. Aunque el porcentaje de fracaso virológico en pacientes con CV basal ≤ 100.000 copias/ml tratados con RPV y 2 ITIAN es bajo y comparable al porcentaje de fracaso virológico con EFV y 2 ITIAN, el riesgo de selección de mutaciones de resistencia tras el fracaso, tanto a RPV como a ITIAN, es elevado. La mutación de resistencia a RPV más frecuente en estos casos es la E138K, que habitualmente emerge en combinación con M184I, debido al doble efecto compensatorio de esta asociación, ocasionando resistencia a RPV y, al mismo tiempo, resistencia a 3TC y FTC. La selección de resistencia a RPV confiere resistencia cruzada con el resto de INNTI, destacando el alto porcentaje de resistencia a ETR.
Por otro lado, aunque la actividad de RPV in vitro no se ve afectada por la presencia de algunas mutaciones frecuentes de resistencia a INNTI, como la mutación K103N, no hay información de la eficacia de RPV como tratamiento de rescate en fracasos con INNTI de primera generación.
Conflicto de interesesArkaitz Imaz ha realizado consultorías, actividades de formación o ha recibido ayudas para la investigación en relación con la infección por VIH de Abbott, Boehringer Ingelheim, Bristol-Myers Squibb, Gilead Sciences, Janssen-Cilag, Merck Sharp&Dohme y ViiV Healthcare.
Federico García ha realizado consultorías, actividades de formación o ha recibido ayudas para la investigación de Abbott, Gilead Sciences, Merck Sharp&Dohme y ViiV Healthcare.
Silvana di Yacovo ha realizado consultorías, actividades de formación o ha recibido ayudas para la investigación en relación con la infección por VIH de Abbott, Boehringer Ingelheim, Gilead Sciences, Merck Sharp&Dohme y ViiV Healthcare.
Josep M. Llibre ha realizado consultorías, actividades de formación o ha recibido ayudas para la investigación de Abbott, Boehringer Ingelheim, Bristol-Myers Squibb, Gilead Sciences, Janssen-Cilag, Merck Sharp&Dohme y ViiV Healthcare.