La tuberculosis es un problema de salud pública que se distribuye de forma diferenciada en regiones de México. El presente trabajo propone un panel, con el mínimo número de MIRU-VNTR (mycobacterial interspersed repetitive unit-variable number tandem repeats), para la diferenciación preliminar de aislamientos clínicos de Mycobacterium tuberculosis (M. tuberculosis).
MétodoSe realizó el panel completo de 24 MIRU-VNTR a 65 aislamientos clínicos de M. tuberculosis provenientes de diferentes regiones geográficas de México.
ResultadosSe presenta un panel de cinco loci MIRU-VNTR para discriminar aislamientos clínicos de M. tuberculosis de tres diferentes regiones geográficas de México.
ConclusionesLa utilización del panel MIRU-VNTR 5 podría utilizarse como tamizaje durante la caracterización genotípica de aislamientos clínicos de M. tuberculosis en México.
Tuberculosis is a public health problem across Mexico. This paper aims to select a panel, with a minimum number of repetitive elements (MIRU-VNTR) for genotypic characterization of Mycobacterium tuberculosis (M. tuberculosis) clinical isolates.
MethodIn this study, a full panel of 24 MIRU-VNTR loci was used to discriminate 65 clinical isolates of M. tuberculosis from three different geographical regions of Mexico. Those loci with the highest discriminatory power were subsequently selected.
ResultsThe panel, including five loci, was obtained by selecting the highest values of allelic diversity among the genotypes obtained. The dendrogram, generated by the panel MIRU-VNTR 5, showed a high discriminatory power with 65 unique genotype profiles and formed clusters according to the geographical region of origin.
ConclusionsThe panel MIRU-VNTR 5 can be useful for characterizing clinical isolates of M. tuberculosis in Mexico.
La tuberculosis (TB) es un problema de salud pública en México. La intensa migración poblacional entre distintas regiones de México podría favorecer la diseminación de cepas de Mycobacterium tuberculosis (M. tuberculosis). No obstante lo anterior, las diferentes regiones presentan distintos patrones de incidencia de TB. Por ejemplo, en el año 2010 Sonora, ubicada en la región noroeste del país y en colindancia con los Estados Unidos de Norteamérica, ocupó el 4.° lugar en incidencia de TB (31,2/100.000); por otro lado Veracruz, en el sudeste del país (aproximadamente a 2.500km de Sonora), ocupó la séptima posición con una incidencia de 26,7/100.000. Finalmente, el Estado de México, ubicado en el centro del país (a 2.000km de Sonora y 450km de Veracruz) se ubicó en el penúltimo lugar de incidencia de TB (5,1/100.000)1. Debe realizarse un esfuerzo importante en México para conocer mejor la distribución y los patrones de diseminación geográfica de las cepas de M. tuberculosis. Para lograr esto, es necesario simplificar los métodos moleculares para incrementar su uso sistemático.
El análisis de 24 loci MIRU-VNTR (mycobacterial interspersed repetitive unit-variable number tandem repeats) es un método de alta resolución para la genotipificación rápida de aislamientos de M. tuberculosis, no obstante, algunos estudios han mostrado que las combinaciones con un número menor de loci presentan un menor poder discriminatorio que el panel estándar de 24 loci2, sin embargo los formatos con pocos elementos MIRU-VNTR podrían ser de gran utilidad para el tamizaje de los aislamientos clínicos3. En este estudio se presenta un panel de cinco loci MIRU-VNTR seleccionados por su poder para discriminar adecuadamente una muestra poblacional de aislamientos de M. tuberculosis procedentes de tres diferentes regiones de México. El uso de este panel MIRU-VNTR para el tamizaje de aislamientos clínicos de M. tuberculosis, brindaría la posibilidad de optimizar recursos y estudiar con más precisión aspectos epidemiológicos de la TB en México.
MétodoSe utilizaron 65 aislamientos de M. tuberculosis, sin caracterización genotípica previa, recuperados entre 2006 y 2010 de residentes de tres diferentes regiones geográficas: Estado de México (n=20), Sonora (n=27) y Veracruz (n=18). Todos los aislamientos fueron identificados por métodos fenotípicos (pruebas bioquímicas convencionales) y genotípicos (gyrB-RFLP y RD12)4-6.
En Sonora, el ADN genómico de los aislamientos se obtuvo como se describió previamente7. En los aislamientos provenientes del Estado de México y Veracruz, el ADN se obtuvo mediante un procedimiento que involucra la utilización del detergente CTAB8.
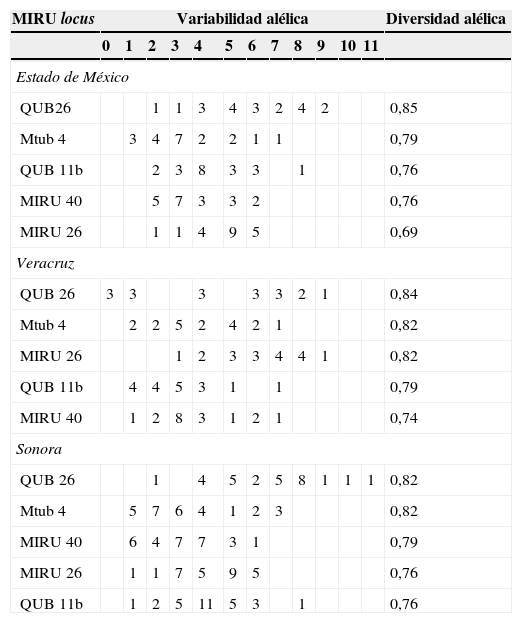
Para la genotipificación, se amplificaron los 24 loci MIRU-VNTR en reacciones de PCR individuales, utilizando los iniciadores y las condiciones descritos en estudios previos2,9. Posteriormente el tamaño de las bandas se determinó mediante el programa TotalLab TL100 (Nonlinear Dynamics, Ltd., United Kingdom). Los dendrogramas fueron generados por el método UPGMA y el coeficiente de correlación de Pearson en el programa Bionumerics versión 6.5 (Applied Maths, Belgium). La diversidad alélica (h) de cada locus fue calculada como se ha descrito previamente10; a partir de los resultados se seleccionaron los loci con mayor diversidad alélica para formar un panel de 5 loci MIRU-VNTR y así generar un nuevo dendrograma.
De los 24 elementos repetitivos analizados, se utilizaron los resultados de 12 loci MIRU para generar los códigos MIT y cinco VNTR, para obtener los respectivos códigos VIT, los mismos que fueron capturados en la base de datos SITVIT11.
ResultadosAl realizar el análisis genotípico de los 65 aislamientos clínicos de M. tuberculosis incluidos en este estudio, se observaron 65 genotipos, uno específico por cada aislamiento. Los loci que presentaron mayor diversidad alélica entre los 65 aislamientos, fueron QUB 26 (0,85), Mtub 04 (0,79), QUB 11b, MIRU 26 y MIRU 40 (tabla 1) y por esta razón fueron seleccionados para formar el panel MIRU-VNTR 5. Con este panel acortado, los 65 genotipos identificados con el panel MIRU-VNTR 24, formaron 9 grandes grupos relacionados casi exclusivamente, de acuerdo a cada una de las tres regiones geográficas de México estudiadas (fig. 1).
Loci MIRU-VNTR que presentaron mayor diversidad alélica en 65 aislamientos clínicos de Mycobacterium tuberculosis de México
| MIRU locus | Variabilidad alélica | Diversidad alélica | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | ||
| Estado de México | |||||||||||||
| QUB26 | 1 | 1 | 3 | 4 | 3 | 2 | 4 | 2 | 0,85 | ||||
| Mtub 4 | 3 | 4 | 7 | 2 | 2 | 1 | 1 | 0,79 | |||||
| QUB 11b | 2 | 3 | 8 | 3 | 3 | 1 | 0,76 | ||||||
| MIRU 40 | 5 | 7 | 3 | 3 | 2 | 0,76 | |||||||
| MIRU 26 | 1 | 1 | 4 | 9 | 5 | 0,69 | |||||||
| Veracruz | |||||||||||||
| QUB 26 | 3 | 3 | 3 | 3 | 3 | 2 | 1 | 0,84 | |||||
| Mtub 4 | 2 | 2 | 5 | 2 | 4 | 2 | 1 | 0,82 | |||||
| MIRU 26 | 1 | 2 | 3 | 3 | 4 | 4 | 1 | 0,82 | |||||
| QUB 11b | 4 | 4 | 5 | 3 | 1 | 1 | 0,79 | ||||||
| MIRU 40 | 1 | 2 | 8 | 3 | 1 | 2 | 1 | 0,74 | |||||
| Sonora | |||||||||||||
| QUB 26 | 1 | 4 | 5 | 2 | 5 | 8 | 1 | 1 | 1 | 0,82 | |||
| Mtub 4 | 5 | 7 | 6 | 4 | 1 | 2 | 3 | 0,82 | |||||
| MIRU 40 | 6 | 4 | 7 | 7 | 3 | 1 | 0,79 | ||||||
| MIRU 26 | 1 | 1 | 7 | 5 | 9 | 5 | 0,76 | ||||||
| QUB 11b | 1 | 2 | 5 | 11 | 5 | 3 | 1 | 0,76 | |||||
La búsqueda de los patrones 24-loci MIRU-VNTR en la base de datos SITVIT no arrojó resultados favorables, ya que no fue posible identificar similitudes ni asignar códigos MIT para 64 de los aislamientos clínicos (fig. 1). Solamente el aislamiento C-102 pudo asignarse al clado X2. Al introducir el código VIT, se obtuvieron códigos para 27 de los aislamientos (fig. 1), tres aislamientos fueron clasificados como huérfanos y el resto no pudo ser clasificado, al no encontrarse cepas registradas que compartieran los códigos obtenidos para estos aislamientos clínicos.
DiscusiónLa genotipificación mediante MIRU-VNTR es útil para identificar perfiles genotípicos microbianos pertenecientes a una misma región geográfica12. Por esta razón, en diversas regiones del mundo se realizan investigaciones para identificar los loci MIRU-VNTR con mayor poder de discriminación3. En este estudio, se encontró que el desempeño de un panel MIRU-VNTR con cinco loci, fue comparable al panel estándar MIRU-VNTR 24 en la discriminación de 65 aislamientos clínicos de M. tuberculosis provenientes de los estados mexicanos de Sonora, Estado de México y Veracruz.
Un estudio reciente realizado en México con el panel 12 loci MIRU-VNTR también demostró gran variabilidad alélica en 109 aislamientos clínicos obtenidos de 23 estados de México13.
Respecto a los loci con mayor poder discriminatorio (tabla 1) elegidos para formar el panel MIRU-VNTR 5, reportes provenientes de países latinoamericanos demostraron que dos de estos loci, MIRU 26 y MIRU 40, son de los más polimórficos y tienen un elevado poder de discriminación14,15. En regiones geográficamente muy distantes de Latinoamérica, como Eurasia, el locus MIRU26 también tuvo un poder de discriminación alto en cepas de MTB aisladas en Rusia16.
No se detectó la presencia de aislados idénticos (100% de similitud); esto podría deberse a la probablemente alta variabilidad alélica de las cepas de M. tuberculosis presentes en México. No obstante, el panel con cinco loci MIRU-VNTR propuesto en el presente trabajo (QUB 26, Mtub 04, QUB 11b, MIRU 26 y MIRU 40) tiene un buen poder de discriminación para los aislamientos clínicos de MTB de las tres regiones geográficas de México estudiadas (estado de México, Sonora y Veracruz), por lo que su empleo ayudaría a diferenciar presuntivamente el origen de las cepas de MTB de estas tres regiones geográficas y posiblemente de otras áreas del país.
Si bien, análisis posteriores con un número más grande de aislamientos permitirán fortalecer el valor de los hallazgos presentados en este trabajo, estos resultados permiten mostrar la utilidad del panel propuesto como un panel tamiz, no para sustituir MIRU 24, sino para utilizarlo en análisis preliminares de los aislamientos micobacterianos
FinanciaciónEste trabajo fue apoyado por «Apoyo a la Incorporación de Nuevos PTC», convocatoria 2010 y «Fortalecimiento de los Cuerpos Académicos», convocatoria 2011, del Programa para el Mejoramiento del Profesorado (PROMEP) de México.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Los autores desean agradecer la invaluable participación de la M. en C. Ansix Pérez-Mendoza, Q. B. C. María Georgina Ortiz-Arballo y la Q. B. C. Esmeralda Guadalupe Urías-Cázares en la caracterización genotípica de los aislamientos.