Introducción
El tratamiento de las infecciones sistémicas por Salmonella, así como la erradicación del estado de portador, no es fácil cuando la cepa es multirresistente. Las fluoroquinolonas y los nuevos betalactámicos constituyen una importante alternativa terapéutica frente a este microorganismo, tanto por su actividad bactericida como por sus excelentes propiedades farmacocinéticas1-3.
El principal mecanismo de resistencia a las fluoroquinolonas en las bacterias gramnegativas se debe a mutaciones en los genes de la ADN-girasa (gyrA y gyrB) y de la topoisomerasa IV (parC y parE)4. Otros mecanismos involucrados en la resistencia a estos antimicrobianos incluyen las alteraciones en la expresión de las porinas o del lipopolisacárido, que disminuyen la penetración del antimicrobiano al interior de la bacteria, la expresión de bombas de achique, que expulsan el antimicrobiano hacia el exterior5-7, y la presencia de la proteína Qnr, de origen plasmídico, que protege a la topoisomerasa de la acción de las quinolonas8,9. En el caso concreto de Salmonella se han descrito mutaciones en los genes que codifican la ADN-girasa1-3,10-13, y muy recientemente en parC14-16 y se ha comprobado la importancia de la hiperexpresión de bombas de expulsión activa3,17-19. Aunque también se han detectado alteraciones en las proteínas de la membrana externa, aún no se ha podido demostrar en Salmonella la correlación directa entre la ausencia de ciertas porinas y la resistencia a fluoroquinolonas3.
La resistencia a los betalactámicos en bacterias gramnegativas se debe, principalmente, a la expresión de betalactamasas4,20, contribuyendo también las alteraciones de la permeabilidad21, la presencia de bombas de achique6 y las alteraciones en las proteínas fijadoras de penicilina (penicillin binding proteins, PBP)22. En Salmonella enterica la resistencia a ampicilina se debe principalmente a la producción de las betalactamasas TEM-1, TEM-2, PSE-1, OXA-1 o SHV-1, habiéndose descrito una baja incidencia de betalactamasas de espectro extendido o de cefamicinasas plasmídicas23-25.
Si bien se han descrito cepas de Salmonella resistentes a betalactámicos19,23,24 o a quinolonas1,3,13,26-28, hasta el momento, las cepas de Salmonella con resistencia simultánea a ambas clases de antimicrobianos son infrecuentes, siendo también poco habitual el desarrollo de estas resistencias durante el tratamiento17,28-30.
Por otro lado, se han documentado fracasos terapéuticos tras tratamiento con fluoroquinolonas en caso de infección por cepas de Salmonella resistentes al ácido nalidíxico y con bajo nivel de resistencia a fluoroquinolonas31.
El objetivo de este estudio ha sido determinar los mecanismos implicados en la resistencia a las fluoroquinolonas y a los betalactámicos en cepas de S. enterica aisladas de 2 pacientes tratados con fluoroquinolonas.
Métodos
Paciente A
Varón de 44 años, seropositivo para el virus de la inmunodeficiencia humana (VIH), que tras una enteritis por S. enterica en Agosto de 1993 (cepa no disponible), tratada con amoxicilina, quedó como portador crónico, iniciándose por ello un tratamiento continuo con norfloxacino (500 mg/12 h) durante 3 años. Desde enero de 1994 hasta abril de 1996, el paciente presentó varios episodios de gastroenteritis, en cada uno de los cuales se aisló de las heces una cepa de S. enterica (cepas A1 a A10). De la última cepa aislada (A10) se seleccionó una mutante con alto nivel de resistencia a ciprofloxacino (A10M), mediante el método de gradiente de Szybalski32.
Paciente B
Varón de 68 años diagnosticado de colitis ulcerosa desde hace 30 años, con antecedentes patológicos de colescistectomía por colecistitis en 1985. En 1988, fue diagnosticado de colangitis esclerosante en fase inicial, realizándosele profilaxis con norfloxacino (500 mg/48 h). En agosto de 1998 fue ingresado en otro hospital por gastroenteritis por S. enterica (cepa no disponible), que fue tratada con ciprofloxacino (500 mg/12 h) durante una semana. En septiembre del mismo año ingresó por un cuadro séptico de probable foco biliar, aislándose S. enterica del hemocultivo (cepa no disponible) y de las heces (cepa B1). Se inició tratamiento con ciprofloxacino intravenoso (400 mg/8 h), por lo que presentó una buena evolución clínica. Al alta se continuó con norfloxacino como descontaminación intestinal selectiva (400 mg/48 h) para la prevención de recurrencias de los episodios de colangitis. Entre octubre y diciembre de 1998 reingresó en diversas ocasiones por síndrome febril de probable foco biliar. Durante todo este período se le administró profilácticamente, de forma continua, ciprofloxacino (500 mg/24 h). En diciembre de 1998 acudió al hospital por diarrea y fiebre y se aisló en distintos coprocultivos seriados S. enterica (cepas B2 y B3), detectándose, asimismo, toxina de Clostridium difficile. Este episodio se trató con amoxicilina-ácido clavulánico y metronidazol, continuándose la profilaxis con ciprofloxacino.
Serotipificación
La determinación de la serotipificación de las cepas se realizó en el Centro Nacional de Microbiología, Virología e Inmunología Sanitarias (Majadahonda, Madrid, España).
Electroforesis en campo pulsante
La extracción de ADN se realizó según Rasheed et al33, utilizando como enzima de restricción XbaI (Roche, Mannheim, Alemania). Las condiciones empleadas fueron de 21 h a 250V de 5 a 50 s, a 14 °C en un gel de agarosa al 1% en TBE al 0,5x (Trizma 0,1M, ácido bórico 0,1M y EDTA 0,2 mM) (Sigma, Madrid, España).
Determinación de la sensibilidad a los antimicrobianos
El estudio inicial de la sensibilidad se realizó por la técnica de disco-difusión siguiendo las recomendaciones del National Committee for Clinical Laboratory Standards (NCCLS)34,35. La determinación de la concentración inhibitoria mínima (CIM) se realizó mediante la técnica del Etest (Biodisk, Solna, Suecia), siguiendo las recomendaciones del fabricante. Se evaluaron los siguientes antimicrobianos: ácido nalidíxico, ciprofloxacino, clinafloxacino, levofloxacino, norfloxacino, ampicilina, cefotaxima, cefoxitina, ceftazidima, cefuroxima y tetraciclina. La CIM a cloranfenicol se determinó por la técnica de microdilución (Sensititre, Trek Diagnostic System, West Sussex, Inglaterra)35.
En otra serie de experimentos se evaluó la actividad del inhibidor de bombas de expulsión activa fenil-arginil-naftilamida (Sigma, Madrid, España). Para ello se determinó mediante microdilución (siguiendo las normas del NCCLS35) la CIM de ácido nalidíxico (Sigma), pefloxacino (Rhône-Poulenc, St. Antoine, Francia), ciprofloxacino (Sigma) y norfloxacino (Sigma) frente a 6 cepas del paciente A (A1, A3, A4, A6, A10 y A10M) y frente a las 3 cepas del paciente B, en ausencia y en presencia (20 mg/l) de dicho compuesto.
Detección de betalactamasas
Para la obtención del extracto de betalactamasas se siguió un protocolo previamente descrito36. La presencia de betalactamasas se detectó por colorimetría, mezclando 100 μl del extracto crudo de betalactamasas con 50 μl de una solución de 1 mg/ml de nitrocefin (Oxoid, Hampshire, Inglaterra).
Secuenciación de los genes gyrA, gyrB, parC y parE
Para la amplificación de las regiones que determinan resistencia a quinolonas (quinolone-resistance determining region, QRDR) de los genes gyrA, gyrB, parC y parE se utilizaron los iniciadores y las condiciones descritas por Giraud et al2. La secuenciación de los productos amplificados se realizó utilizando iniciadores marcados con una sonda fluorescente (Cy5 dideoxi) y el secuenciador ALF Express (Automatic Laser Fluorescent DNA Sequencer, Amersham Biosciences, Uppsala, Suecia). Las secuencias nucleotídicas obtenidas y las aminoacídicas deducidas se analizaron mediante el software disponible en la página web del National Center for Biotechnology Information (www.ncbi.nlm.nih.gov).
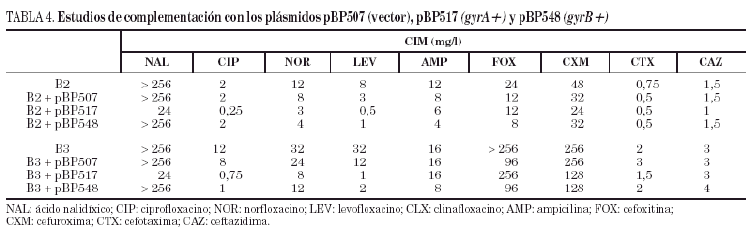
Estudios de complementación
Los plásmidos pBP507 (vector), pBP517 (gyrA+) y pBP548 (gyrB+) se introdujeron mediante transformación en las cepas de S. enterica B2 y B3. Estos plásmidos son portadores de los genes gyrA y gyrB salvajes, por lo que permiten restablecer la sensibilidad a las quinolonas en los correspondientes transformantes si la resistencia es consecuencia en la cepa original de mutaciones en gyrA o gyrB, respectivamente1,37.
Perfil de proteínas de la membrana externa (OMP)
Las OMP se obtuvieron a partir de un cultivo en fase exponencial de 40 ml de Mueller Hinton, empleando un método descrito previamente38. Las bacterias se separaron mediante centrifugación (5.000 x g, 4 °C, 15 min) y posteriormente se sonicaron. Las envolturas celulares se recuperaron mediante centrifugación (13.000 x g, 4 °C, 45 min) y se trataron con laurilsarcosinato (Sigma, Madrid, España). Finalmente y mediante una centrifugación adicional (13.000 x g, 4 °C, 45 min), se obtuvieron las OMP. Los perfiles de OMP se determinaron mediante electroforesis en geles de poliacrilamida con dodecilsulfato sódico y tinción con azul Coomassie. Como control se utilizó la cepa de Salmonella choleraesuis subsp. choleraesuis NCTC 5188.
Determinación de la acumulación de norfloxacino
La acumulación basal de norfloxacino se realizó siguiendo la metodología descrita previamente39. Las bacterias se separaron por centrifugación y se resuspendieron en tampón fosfato-salino a una densidad óptica (a 520 nm) de 1,5. La suspensión bacteriana se incubó con norfloxacino (10 mg/l) a 37 °C durante 30 min. Las bacterias se separaron del líquido extracelular mediante centrifugación diferencial a través de una capa de aceite de silicona (densidad, 1,029 g/cm3) y se lisaron en tampón 0,1 M de glicina-HCl (pH 3,0). Las muestras se centrifugaron a 12.800 x g durante 5 min y la cantidad de norfloxacino se determinó por espectrofluorometría (Hitachi 2000, Tokio, Japón). En otra serie de experimentos en paralelo, se evaluó el efecto del carbonil-cianuro-m-clorofenilhidrazona (CCCP, 0,1 mM, Sigma, Madrid, España) sobre la acumulación de norfloxacino. Para ello, tras la preincubación de las bacterias con norfloxacino durante 10 min (10 mg/l) se añadió el inhibidor metabólico y se realizó una incubación adicional de 20 min. Los experimentos se realizaron tres veces, en días diferentes. Cada día, las determinaciones se realizaron por duplicado.
Resultados
Todas las cepas aisladas en el paciente A pertenecían a la subespecie I de S. enterica, pero eran rugosas e inmóviles, por lo que no pudo determinarse su serotipo, mientras que las cepas aisladas en el paciente B pertenecían al serotipo Enteritidis. Mediante la técnica de electroforesis en campo pulsante se obtuvieron patrones de restricción idénticos entre las cepas de un mismo paciente, e idéntico patrón entre las cepas de ambos pacientes. Clínicamente no parece que haya relación alguna entre ambos pacientes.
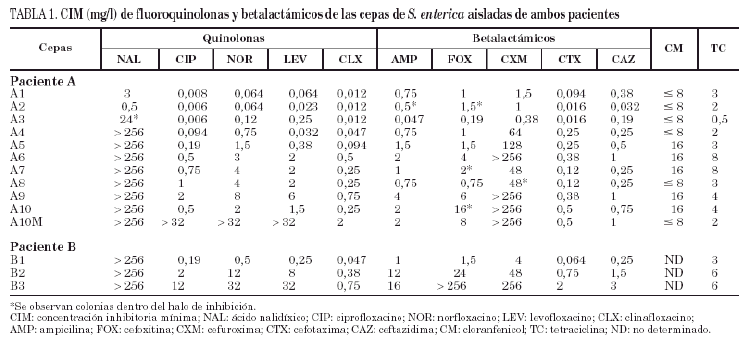
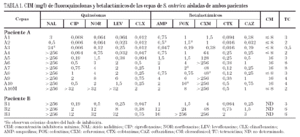
Los resultados del estudio de sensibilidad a los diferentes antimicrobianos evaluados se recogen en la tabla 1. Con las cepas del paciente A (A1-A10) pueden establecerse cuatro grupos: el primer grupo lo constituirían las cepas A1 y A2, aisladas en 1994, sensibles a todos los antimicrobianos. El segundo lo constituye la cepa A3, aislada en febrero de 1995, que con respecto a las cepas precedentes presenta resistencia moderada al ácido nalidíxico, discreta disminución de la sensibilidad al norfloxacino y levofloxacino (pero sin variación en la sensibilidad a ciprofloxacino y clinafloxacino) y leve aumento de la sensibilidad a betalactámicos. En el tercer grupo estarían las cepas A4 y A5, aisladas a comienzos de 1995, resistentes al ácido nalidíxico y con valores de CIM de fluoroquinolonas más elevados, mostrándose además resistentes a cefuroxima y presentando un moderado incremento de la CIM de cefotaxima y ceftazidima. Por último, el cuarto grupo incluye las cepas A6 a A10, aisladas a finales del mismo año y principio de 1996, donde es más evidente el incremento de la resistencia a fluoroquinolonas. En este último grupo también se observaron variaciones en la actividad de cefuroxima y de otros betalactámicos.
Todas las cepas del paciente B (B1-B3) presentaron resistencia al ácido nalidíxico, observándose en las cepas B2 y B3 un incremento de la CIM, tanto a fluoroquinolonas como a betalactámicos.
Para evaluar los posibles mecanismos implicados en la resistencia a betalactámicos y a fluoroquinolonas se realizó la siguiente aproximación:
1. Descartar la presencia de una betalactamasa, que explicaría la resistencia a los betalactámicos.
2. Buscar mutaciones en las regiones QRDR de los genes de la ADN-girasa y de la topoisomerasa IV, que explicarían la resistencia a fluoroquinolonas.
3.Estudiar la existencia de alteraciones en las proteínas de la membrana externa y/o en la expresión de un mecanismo de expulsión activa, que se relacionaría con resistencia a ambas familias de antimicrobianos. Los resultados de estos estudios se detallan en las tablas 2 y 3.
Ninguna de las cepas de los 2 pacientes evaluados presentó actividad betalactamasa.
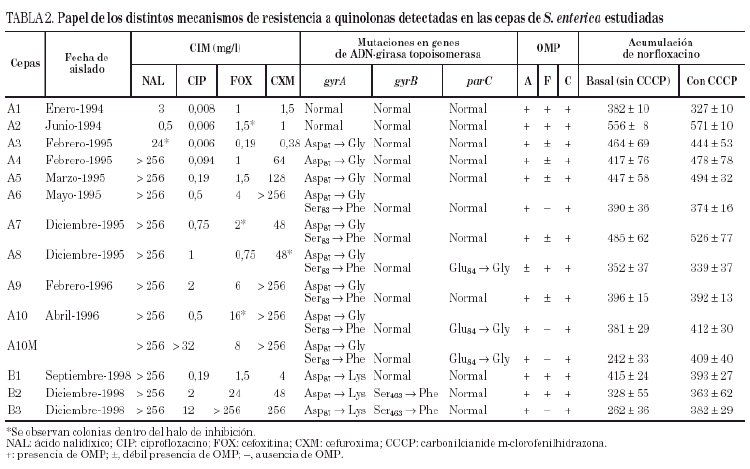
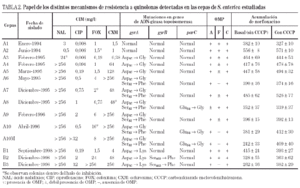
Las cepas A3, A4 y A5 mostraron una mutación en el gyrA (Asp87 → Gly). En las cepas A6, A7 y A9, pudo observarse además una segunda mutación en gyrA (Ser83 → Phe). Por último, las cepas A8 y A10 (y A10M) presentaron una tercera mutación en el gen parC (Glu84 →Gly).
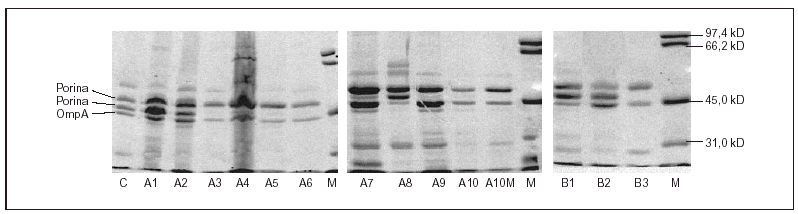
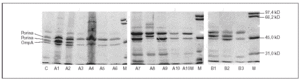
Todas las cepas presentaron dos o tres proteínas de membrana externa (fig. 1). En función de las movilidades electroforéticas, y por analogía con lo descrito en Salmonella y en otras enterobacterias, todas las cepas presentaron una banda que se corresponde con OmpA (la proteína de menor peso molecular) y una o dos porinas. La expresión de OmpA estaba reducida en la cepa A8. Las cepas A1, A2 y A8 presentaron dos porinas; en las cepas A3, A4, A5, A7 y A9 se observó una disminución de la expresión de la porina de menor peso molecular (correspondiente a OmpF) y en las cepas A6, A10 y A10M se produjo la pérdida completa de esta misma porina (fig. 1).
Figura 1. SDS-PAGE de las proteínas de la membrana externa de las distintas cepas de S. enterica aisladas de los pacientes A y B.
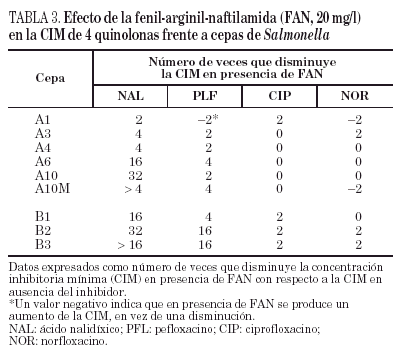
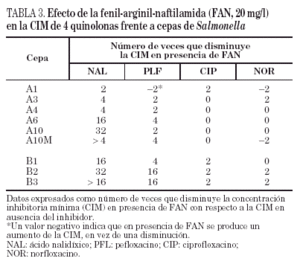
Por lo que se refiere a la acumulación de norfloxacino en las distintas cepas de este paciente, puede observarse cierta variabilidad en los valores basales, que oscilaron entre 328 ± 10 y 556 ± 8 ng/mg de peso bacteriano seco, correspondiendo el mayor valor de acumulación a la cepa A2 (tabla 2). Sólo en la cepa A10M pudo observarse un aumento significativo de la acumulación de norfloxacino en presencia de CCCP con respecto al valor basal. Los resultados de este experimento sugieren que en esta cepa existe un mecanismo de expulsión activa. En presencia de FAN, las CIM de norfloxacino no variaron (tres de las seis cepas evaluadas) o incluso aumentaron al doble (dos de las seis cepas). De forma análoga, las CIM de ciprofloxacino no variaron (cinco de las seis cepas) o sólo disminuyó dos veces (la cepa restante). Por el contrario, la FAN consiguió un descenso significativo (al menos cuatro veces) las CIM de ácido nalidíxico (cinco de las seis cepas) y las CIM de pefloxacino (dos de las seis cepas) (tabla 3).
Todas las cepas del paciente B presentaron una mutación en gyrA (Asp87 → Lys), observándose, además, en B2 y B3 una segunda mutación en gyrB (Ser463 →Phe). Sólo se observó la pérdida de OmpF y un aumento significativo en la acumulación de norfloxacino en presencia de CCCP respecto al valor basal en la cepa B3 (fig. 1) (tabla 2). La FAN disminuyó las CIM de ácido nalidíxico (16 a 32 veces) y de pefloxacino (4 a 16 veces), pero en presencia de este inhibidor las CIM de norfloxacino o ciprofloxacino no variaron o disminuyeron sólo a la mitad (tabla 3).
Los estudios de complementación demostraron que la introducción del plásmido pBP517 (con gyrA salvaje) en las cepas B2 y B3 disminuía la CIM de ácido nalidíxico, ciprofloxacino, norfloxacino y levofloxacino (tabla 4). De igual forma, la transformación de las cepas B2 y B3 con el plásmido pBP548 (que codifica gyrB salvaje) redujo ligeramente la CIM de norfloxacino y de levofloxacino en la cepa B2, y la CIM de ciprofloxacino y levofloxacino en B3. La CIM de las tres fluoroquinolonas ensayadas fueron de dos a tres veces mayores frente a los transformantes de la cepa B3 que frente a los transformantes de la cepa B2. La expresión de GyrA o GyrB salvajes a partir de los correspondientes plásmidos no afectó significativamente (como sería de esperar) la CIM de los betalactámicos estudiados.
En ninguna de las cepas estudiadas de ambos pacientes se encontraron mutaciones en el gen parE.
Discusión
Las infecciones por Salmonella continúan siendo un importante problema sanitario en nuestro país25,40,41. Es importante, por tanto, profundizar en el estudio de los mecanismos de resistencia a los antimicrobianos en las especies de este género y en el impacto terapéutico de los mismos.
En los dos pacientes incluidos en este estudio, que fueron tratados durante un largo período de tiempo con fluoroquinolonas, se obtuvieron cepas de salmonela con resistencia creciente a quinolonas y a betalactámicos. Las características fenotípicas, junto con las genotípicas (mutaciones detectadas), apoyan la relación clonal de las cepas aisladas en ambos pacientes.
Estudios previos han puesto de manifiesto que las alteraciones en las topoisomerasas no son suficientes para explicar la resistencia a quinolonas en S. enterica13. En consonancia con esta observación, nuestros resultados ponen de manifiesto que los mecanismos de resistencia involucrados en las cepas evaluadas son múltiples e incluyen mutaciones en las topoisomerasas (genes gyrA y quizá parC, en el paciente A; y gyrA y gyrB en el paciente B), alteraciones en la permeabilidad celular (principalmente debido a la disminución de la expresión de la porina OmpF, en ambos pacientes) y expresión de un mecanismo de eliminación activa.
Las mutaciones Asp87 →Gly y Ser83 →Phe en gyrA son las descritas con mayor frecuencia en S. enterica11. La mutación Asp87 →Gly parece conferir resistencia sólo al ácido nalidíxico. En Escherichia coli, la resistencia de alto nivel a fluoroquinolonas requiere una segunda mutación en el mismo gen y/o una mutación en parC37. En nuestro caso, en las cepas con dos mutaciones en gyrA el incremento de la CIM a ciprofloxacino oscila entre 2 y 4 veces respecto a la cepa A5, o de 8 a 12 veces respecto a la cepa A3, ambas con una sola mutación en gyrA. No parece que la mutación Glu84 xGly en parC observada en las cepas clínicas A8 y A10 tenga por sí misma un papel destacado en el nivel de resistencia, pues en ambas cepas no se observa ningún incremento de la resistencia con respecto a las cepas A7 o A9 (que carecen de ella).
En el paciente B, además de la mutación Asp87 → Lys en gyrA, se observa una segunda mutación en el gen gyrB de Ser463 → Phe. En este mismo codón, Gensberg et al10 describieron en S. typhimurium un cambio aminoacídico de Ser x Tyr. Mediante la prueba de complementación se ha demostrado la importancia de la mutación en gyrA en la resistencia de las cepas del paciente B tanto a ácido nalidíxico como a fluoroquinolonas. Con esta misma aproximación se ha observado que la mutación en gyrB también está relacionada con la resistencia a quinolonas. Estos resultados concuerdan con los encontrados por Heisig1, quien comprobó que la resistencia de alto nivel a fluoroquinolonas en Salmonella puede deberse a la presencia de las mutaciones tanto en gyrA como en gyrB.
Los resultados sobre el efecto de la expresión de porinas en el nivel de resistencia no son concluyentes. Si se compara la actividad de los antimicrobianos frente a dos pares de cepas que difieren en el nivel de expresión de OmpF (A6 y A7, por un lado y A9 y A10 por otro) puede observarse que las cepas deficientes en dicha porina son algo más resistentes a betalactámicos, pero no a quinolonas. En cualquier caso, la disminución de la expresión o la pérdida de OmpF no explica completamente el aumento de resistencia a cefuroxima, cefoxitina y cefotaxima que se observa en algunas cepas aisladas. Teniendo en cuenta que las cepas carecen de betalactamasa, es de suponer que éstas deben expresar mecanismos adicionales de resistencia a los betalactámicos (¿alteraciones de PBP?). En Klebsiella pneumoniae38,42 y en E. coli (datos no publicados) la pérdida de sólo una de las porinas principales tiene escasa relevancia clínica en la resistencia a betalactámicos y quinolonas, presumiblemente porque la expresión de la otra porina permite la entrada de suficiente antimicrobiano como para inhibir al microorganismo. Serían necesarios nuevos estudios, en los que por técnicas genéticas se reexprese la porina perdida, para conocer el impacto real de este mecanismo en la resistencia, y para evaluar la relevancia de estos canales en la penetración de diferentes familias de antimicrobianos.
El hecho de que en las cepas A10M (mutante obtenida in vitro) y B3 (seleccionada in vivo) haya un aumento en la acumulación de norfloxacino por inactivación de la bomba mediante CCCP sugiere que ambas presentan un incremento en la actividad del sistema de bombeo activo. Es interesante resaltar que aunque la expresión de un sistema de expulsión se relaciona con un incremento de la resistencia a quinolonas, no afecta a otras familias de antimicrobianos, como betalactámicos, tetraciclina o cloranfenicol. La observación de que los transformantes de la cepa B2 son más sensibles que los de la cepa B3 (tanto cuando se expresa gyrA salvaje como cuando se expresa gyrB salvaje) sugiere que la pérdida de una porina y la expresión de un mecanismo de expulsión activa en esta última causan una disminución de la acumulación de quinolonas y, por ello, una menor actividad de estos agentes. En E. coli6,17 y en S. typhimurium43 el sistema de expulsión activa más importante es AcrAB, sometido a múltiples mecanismos de regulación. Los operones marRAB y soxRS, que regulan acrAB, intervienen también en el control de la expresión de la porina OmpF. Se han descrito varias mutaciones en estos sistemas de regulación que causan incremento en la expresión de AcrAB y disminución en la de OmpF43. Esta situación es similar a la que se ha observado en este estudio en la cepa B3 del paciente B (fig. 1). El bajo impacto que tiene en la CIM la expresión de este doble mecanismo, ya ha sido descrito por Piddock et al27. La existencia de otras porinas, por donde los antimicrobianos pueden seguir penetrando en ausencia de OmpF explicaría esta situación, como recientemente se ha comprobado en cepas de K. pneumoniae44.
La disminución, en presencia de FAN, de las CIM de ácido nalidíxico frente a 5 de las 6 cepas del paciente A y frente a las 3 cepas del paciente B también indica que dichas cepas expresan un mecanismo de expulsión activa. Se ha apreciado un efecto similar, pero menos marcado, en relación con pefloxacino, pero las CIM de ciprofloxacino y de norfloxacino no variaron, o lo hicieron mínimamente. El hecho de que las disminuciones inducidas por FAN sean más marcadas para ácido nalidíxico y pefloxacino (dos de las quinolonas más hidrófobas) que para ciprofloxacino y norfloxacino es compatible con la teoría actualmente aceptada de que las bombas de expulsión reconocen de forma poco específica sustratos preferentemente hidrófobos. Existen pocos datos sobre el modo de interacción de CCCP y FAN con las bombas de expulsión activa, y tampoco se sabe a ciencia cierta si ambos inhibidores actúan sobre el mismo (o los mimos) sistema(s) de expulsión. Es posible que las diferencias en los efectos observados con FAN y con CCCP se deban a la distinta capacidad de ambos compuestos para inhibir distintos sistemas de expulsión activa que, a su vez, pueden estar implicados de diferente forma en la eliminación de algunas quinolonas y no de otras. También podría ocurrir que la disminución de la acumulación de norfloxacino en presencia de CCCP sólo se traduzca en un pequeño incremento de la CIM en cepas que tienen alteraciones en las dianas de las quinolonas (como se ha comprobado previamente en cepas de K. pneumoniae42), lo que a su vez explicaría el mínimo descenso de la CIM de norfloxacino en presencia de FAN. Los resultados de este estudio son similares a otros obtenidos en nuestro laboratorio con cepas de K. pneumoniae en las que se han caracterizado los mecanismos de resistencia a quinolonas (datos no publicados). Todo ello indica que son necesarios muchos más estudios para comprender los mecanismos de expulsión activa en enterobacterias y la verdadera significación de las distintas aproximaciones metodológicas empleadas para poner de manifiesto este tipo de mecanismo de resistencia.
Los valores basales de acumulación de norfloxacino en las cepas clínicas del paciente A (en las que no se observa expresión de un mecanismo de expulsión activa) presentan variaciones importantes. Es interesante resaltar que en la cepa A2, donde se produce la mayor acumulación basal de norfloxacino, los valores de CIM de ácido nalidíxico y de algunas fluoroquinolonas son los más bajos de toda la serie. Aunque es posible que ello sea debido a un fenómeno dependiente del método empleado (sometido a variación, como indican los valores de desviación estándar obtenidos), no debe descartarse que ello sea la consecuencia de una regulación fina de los sistemas de expulsión activa de Salmonella. Para aclarar este aspecto, y para valorar el impacto real de las bombas de expulsión en la resistencia a quinolonas en esta especie son necesarios estudios adicionales.