En el año 2018, todavía hay laboratorios de microbiología que no subtipan o no detectan virus de la gripe, uno de los principales agentes de la neumonía adquirida en la comunidad. Uno de los retos principales es acercar tecnologías de tipo multiplex a la mayor parte de laboratorios de diagnóstico virológico clínico, lo que facilita que el diagnóstico etiológico de la gripe y otros virus respiratorios sea factible en tiempo y forma en todos aquellos casos clínicos que lo precisen, y limita así tratamientos antibióticos. Otras herramientas diagnósticas, como los marcadores asociados con gravedad y la detección de resistencias, son retos pendientes de completar y expandir. El cultivo del virus, herramienta imprescindible en la vigilancia epidemiológica del virus, ha quedado relegado por las técnicas moleculares más sensibles y asequibles. La secuenciación del virus gripal junto con la caracterización antigénica y las técnicas de detección de anticuerpos frente a la hemaglutinina y la neuraminidasa acompañarán en un futuro a otras técnicas para detectar anticuerpos frente a otras proteínas estructurales, lo que ayudará a entender la compleja epidemiología de estos virus y la producción de nuevas vacunas y su evaluación. Información sobre el suplemento: este artí-culo forma parte del suplemento titulado «Programa de Control de Calidad Externo SEIMC. Año 2016», que ha sido patrocinado por Roche, Vircell Microbiologists, Abbott Molecular y Francisco Soria Melguizo, S.A.
© 2019 Elsevier España, S.L.U. y Sociedad Española de Enfermedades Infecciosasy Microbiología Clínica. Todos los derechos reservados.
In 2018 there are still microbiology laboratories that do not subtype or detect influenza viruses, one of the main agents of community-acquired pneumonia. A major challenge is to introduce multiplex-type technologies into most clinical virological diagnostic laboratories, increasing the feasibility of timely etiological diagnosis of influenza and other respiratory viruses whenever required and thus limiting antibiotic treatments. Other diagnostic tools such as markers of severity and the detection of resistance are pending challenges to complete and expand. Viral culture, an essential tool in the epidemiological surveillance of viruses, has been relegated by more sensitive and affordable molecular techniques. Sequencing of the influenza virus together with the antigenic characterisation and detection techniques of antibodies against hemagglutinin and neuraminidase will, in future, be used in tandem with other techniques to detect antibodies against other structural proteins, helping to elucidate the complicated epidemiology of these viruses and the production of new vaccines and their evaluation. Supplement information: This article is part of a supplement entitled «SEIMC External Quality Control Programme. Year 2016», which is sponsored by Roche, Vircell Microbiologists, Abbott Molecular and Francisco Soria Melguizo, S.A.
© 2019 Elsevier España, S.L.U. and Sociedad Española de Enfermedades Infecciosasy Microbiología Clínica. All rights reserved.
La gripe está en contra de dogmas y suposiciones. A lo largo de su historia ha sido uno de los virus más elusivos en cuanto al hecho de mostrar aspectos de su patología, epidemiología, respuesta inmunológica y correlatos de protección. Las características de su biología continúan siendo un reto de tal manera que en cada una de las áreas del conocimiento de la gripe existen lagunas que impiden ver completamente el rompecabezas de la infección gripal en su conjunto, incluidos los aspectos relativos a su diagnóstico.
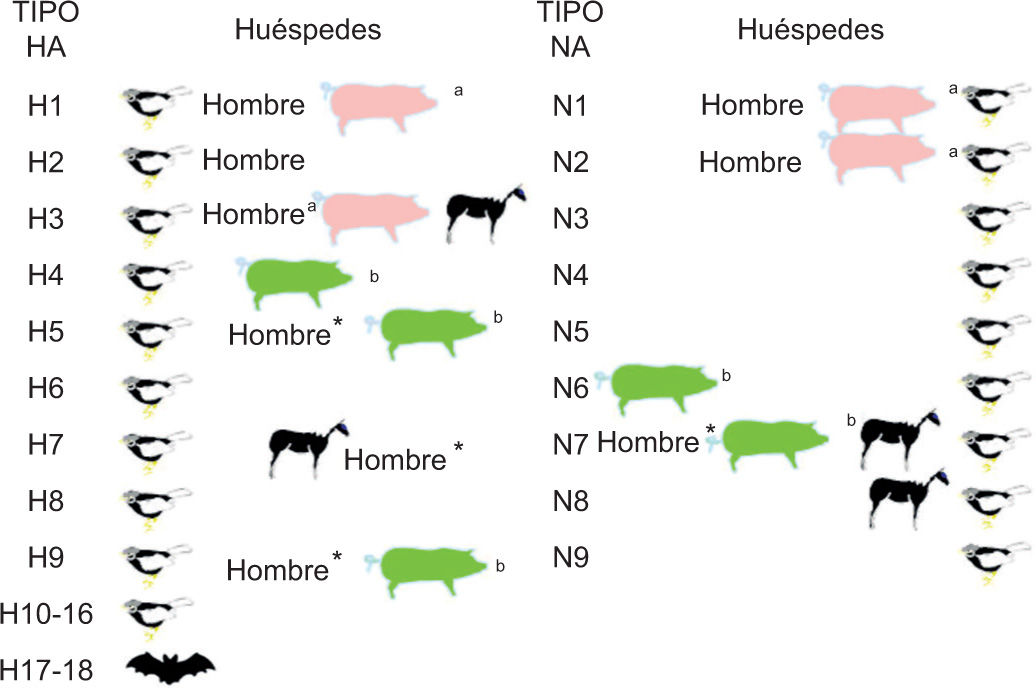
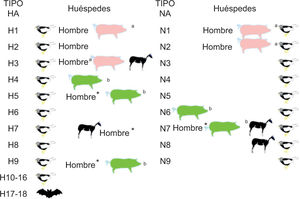
La gripe está producida fundamentalmente por los tipos de virus A y B ya que el virus C produce solo casos esporádicos, en general leves o asociados con otros virus respiratorios1. El tipo A de la gripe procede de un reservorio salvaje solo parcialmente desvelado en aves, donde se han identificado 16 tipos distintos de hemaglutinina (H1-H16)2 y otros 2 tipos adicionales, H17 y H18, en murciélagos3 (fig. 1). Una de las características que deben tenerse en cuenta es su capacidad de salto de la barrera de especie desde el reservorio aviar hacia otros huéspedes mamíferos, incluido el hombre, en el cual produce cuadros clínicos de gripe animal con una presentación clínica diferente a la clásica del síndrome gripal por virus gripales humanos4,5.
En cambio, el tipo de gripe B es exclusivamente humano, con casos esporádicos de infecciones a otros huéspedes animales6,7. Por tanto, desde un punto de vista meramente epidemiológico, es un virus que podría llegar a ser erradicado si no aparece otro huésped animal desconocido y se dispone, en un futuro, de una vacuna de efectividad superior a la actual.
A pesar de los años transcurridos desde el descubrimiento del virus de la gripe, de la existencia de una vacuna desde hace más de 60 años y de una red importante de laboratorios a nivel mundial incluidos en el Sistema de Vigilancia Internacional y de Respuesta frente a la Gripe (GISRS, Global Influenza Surveillance and Response System)8, existen todavía numerosos retos diagnósticos que precisan respuesta.
En términos generales, conviene recordar que a lo largo de la vida asistencial y laboral de un profesional sanitario, entre el 22 y el 25% de las gripes clínicas estarán causadas por el virus B. Esto nos vuelve a situar ante el título de este artículo y nos obliga a formularnos otra pregunta: ¿tenemos los recursos necesarios para un correcto diagnóstico, control, tratamiento, vigilancia epidemiológica y prevención que necesitan aplicarse para cualquier caso de gripe? La respuesta, evidentemente, es no.
La gripe A se presenta anualmente con una estacionalidad muy marcada en los meses de invierno, excepto en los países de clima tropical9, y en forma pandémica con un intervalo temporal desconocido cada vez que un salto de la barrera de especie o un reordenamiento genético permite la emergencia de un subtipo viral, frente al cual una gran parte de la población no tiene experiencia infecciosa previa. La gripe B, aunque solo produce epidemias estacionales, generalmente acompaña a subtipos A, principales responsables de las epidemias estacionales; puede liderar y predominar algunas temporadas y sustituir al protagonismo epidémico del tipo A.
Desde el punto de vista taxonómico y virológico hay que recordar que desde 1977 han cocirculado en seres humanos 2 subtipos humanos de gripe: el subtipo H3N2 desde su aparición en la pandemia de gripe de Hong Kong y el subtipo H1N1pdm que emergió en 2009 y reemplazó rápidamente al subtipo H1N1, reemergido en 197710. El virus B no tiene subtipos, pero desde finales de los ochenta del siglo pasado se observó una divergencia filogenética importante, según la cual se diferenciaron 2 linajes, B/Victoria y B/Yamagata11,12, con suficiente distancia antigénica para que la respuesta inmunológica frente a uno de ellos no proteja adecuadamente frente al otro13.
Los retos diagnósticos son diferentes si el objetivo del diagnóstico de la gripe es clínico-virológico o se encuadra dentro de la vigilancia epidemiológica (tabla 1). En ambos casos, este diagnóstico puede requerirse en casos clínicos atípicos, tanto en medicina ambulatoria como hospitalaria, en brotes en instituciones cerradas y, finalmente, en casos graves que requieran ingreso hospitalario o en unidades de críticos (UCI)14,15. Además, los virus de la gripe de huéspedes animales pueden producir casos en seres humanos, en los cuales los retos diagnósticos son diferentes16,17.
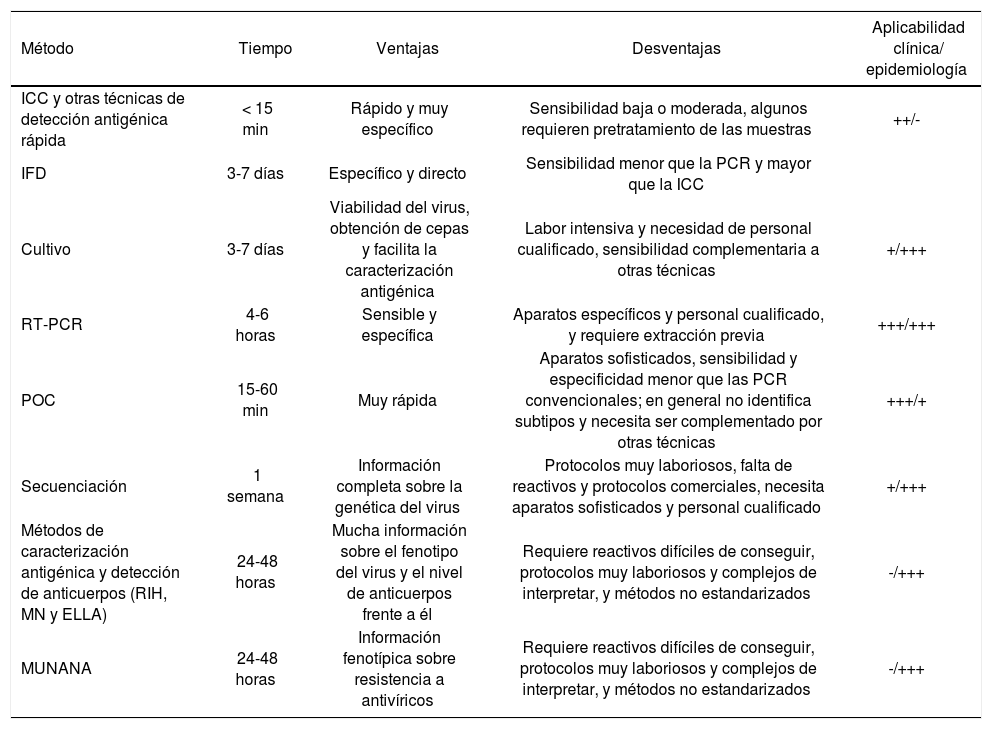
Ventajas, desventajas y aplicabilidad de las técnicas diagnósticas de gripe en la práctica clínica y la vigilancia epidemiológica
| Método | Tiempo | Ventajas | Desventajas | Aplicabilidad clínica/ epidemiología |
|---|---|---|---|---|
| ICC y otras técnicas de detección antigénica rápida | < 15 min | Rápido y muy específico | Sensibilidad baja o moderada, algunos requieren pretratamiento de las muestras | ++/- |
| IFD | 3-7 días | Específico y directo | Sensibilidad menor que la PCR y mayor que la ICC | |
| Cultivo | 3-7 días | Viabilidad del virus, obtención de cepas y facilita la caracterización antigénica | Labor intensiva y necesidad de personal cualificado, sensibilidad complementaria a otras técnicas | +/+++ |
| RT-PCR | 4-6 horas | Sensible y específica | Aparatos específicos y personal cualificado, y requiere extracción previa | +++/+++ |
| POC | 15-60 min | Muy rápida | Aparatos sofisticados, sensibilidad y especificidad menor que las PCR convencionales; en general no identifica subtipos y necesita ser complementado por otras técnicas | +++/+ |
| Secuenciación | 1 semana | Información completa sobre la genética del virus | Protocolos muy laboriosos, falta de reactivos y protocolos comerciales, necesita aparatos sofisticados y personal cualificado | +/+++ |
| Métodos de caracterización antigénica y detección de anticuerpos (RIH, MN y ELLA) | 24-48 horas | Mucha información sobre el fenotipo del virus y el nivel de anticuerpos frente a él | Requiere reactivos difíciles de conseguir, protocolos muy laboriosos y complejos de interpretar, y métodos no estandarizados | -/+++ |
| MUNANA | 24-48 horas | Información fenotípica sobre resistencia a antivíricos | Requiere reactivos difíciles de conseguir, protocolos muy laboriosos y complejos de interpretar, y métodos no estandarizados | -/+++ |
ELLA: enzyme-lynked lectin assay; ICC: inmunocromatografía; IFD: inmunofluorescencia directa; MN: microneutralización; MUNANA: ácido 4-metilumbeliferil-N-acetilneuramínico; POC: point of care; RIH: reacción de inhibición de la hemaglutinación; RT-PCR: reacción en cadena de la polimerasa en tiempo real.
Desde un punto de vista meramente conceptual, el diagnóstico de gripe de casos individuales debe, a partir de muestras individualizadas, permitir el diagnóstico, tratamiento y control del paciente concreto, mientras que en el caso de brotes institucionales o de la vigilancia epidemiológica reglada estas muestras obedecerán a criterios de definición epidemiológica de caso, casos inusuales, casos graves o en huéspedes peculiares (inmunodeprimidos, obesos, enfermos reumáticos, etc.) sin que su objetivo final sea exclusivamente el diagnóstico clínico. Por tanto, los retos diagnósticos no son iguales en el período de vigilancia epidemiológica de la gripe que fuera de él, ni en la práctica clínica habitual que en la vigilancia epidemiológica.
La vigilancia epidémica de gripe abarca desde la semana 40 de un año a la semana 20 del siguiente; en España existe, además, un período de vigilancia ampliada por parte del Sistema de Vigilancia de la Gripe en España (SVGE) que abarca el período interepidémico18. Des-de el punto de vista epidemiológico, los retos diagnósticos de la gripe no son iguales en períodos interpandémicos que en el período pandémico, en el cual la aparición de un subtipo nuevo plantea un escenario diagnóstico distinto, en el cual la mayor parte de los reactivos existentes en forma comercial no han podido ser debidamente ensayados y comprobados.
A partir de la emergencia pandémica del año 2009 han proliferado numerosos sistemas diagnósticos, basados principalmente en la metodología molecular, que han sido objeto de revisión reciente en Enfermedades Infecciosas y Microbiología Clínica19. Sin embargo, ninguno de ellos cumple con todos los requerimientos diagnósticos para una enfermedad tan compleja desde la aproximación virológica y epidemiológica. En otro orden de cosas, el diagnóstico de tipos pandémicos emergentes puede plantear problemas adicionales de seguridad en el laboratorio, así como en la gestión de los enfermos en el hospital.
Por todo lo expuesto, los retos diagnósticos de la gripe son variados y abarcan desde la asistencia ambulatoria pediátrica y de adultos hasta la de hospitalizados. Si se tiene presente la existencia de unidades especiales de hospitalización (hematológicos, inmunodeprimidos, trasplantados, etc.) en los complejos sistemas sanitarios actuales, estos servicios precisan un diagnóstico etiológico de gripe para reducir la difusión intrahospitalaria de esta enfermedad y optimizar los cuidados de sus pacientes.
La gripe es una enfermedad de declaración obligatoria en el sistema sanitario español y es objeto de especial vigilancia a nivel nacional por el SVGE y los laboratorios asociados con este dispositivo sanitario. A nivel internacional es la única enfermedad que tiene una cobertura mundial asegurada por una red de 144 laboratorios nacionales de gripe (NIC, National Influenza Center) repartidos por todo el mundo, los cuales forman la columna dorsal del GISRS8. Los retos diagnósticos a este nivel epidemiológico son diferentes a los requeridos en otros apartados del diagnóstico microbiológico de gripe y su implementación no es, de momento, una prioridad para los centros hospitalarios ordinarios (tabla 1).
En conjunto, todos los diagnósticos de gripe, virológico clínico, epidemiológico y de vigilancia mundial, así como otros que sean implantados, deberán coordinarse y deberá compartirse la información generada, procedimiento absolutamente necesario en una enfermedad infecciosa de relevancia sanitaria, que sobrecarga anual y habitualmente los sistemas asistenciales de todos los países y con periodicidad imprevisible en la presentación pandémica, lo que representa un desafío mundial.
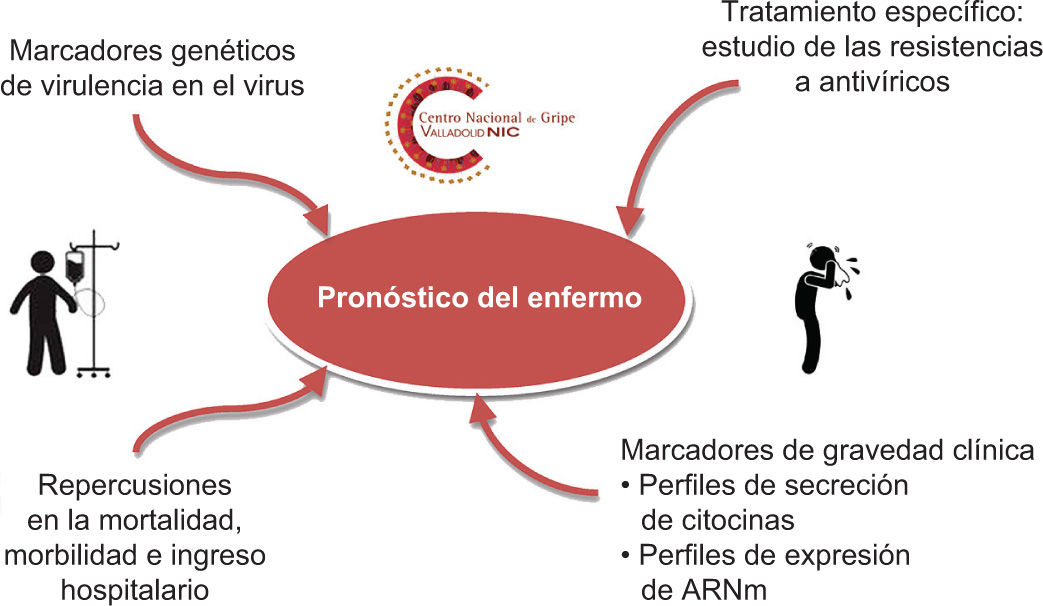
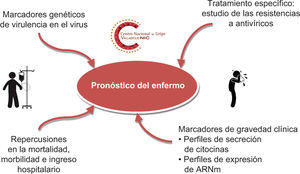
Retos en el diagnóstico virológico de casos clínicos de gripeLa gripe es una enfermedad que, en su presentación clínica típica en el período estacional, no requiere necesariamente diagnóstico virológico. Sin embargo, el diagnóstico es necesario siempre en casos que requieran atención médica hospitalaria o en personas con patologias de base13 (fig. 2). Este diagnóstico es importante en casos fuera del período estacional y deberá llevarse a cabo en casos clínicos en que haya una patología infecciosa sin filiar cuando las redes de vigilancia hayan notificado la circulación del virus.
Los síntomas no permiten diferenciar el tipo responsable de virus, así que para el diagnóstico virológico los métodos empleados deberán tener las mismas características operacionales para el tipo A que para el B. Uno de los principales retos diagnósticos de la gripe es que el síndrome clínico puede estar ocasionado por otros virus respiratorios, además de la gripe20. Aparte de ello, los casos pueden ser de distinta gravedad y en huéspedes de muy diferente condición clínica. La infección gripal puede adoptar distintas formas clínicas de presentación que van desde las asintomáticas o paucisintomáticas hasta las que requieren cuidados intensivos o causan la muerte a personas con patología subyacente o, con menos frecuencia, a otras previamente sanas21. La urgencia del diagnóstico en cada uno de esos supuestos es completamente distinta. Por ello, la base del control clínico de la enfermedad gripal pasa por ofrecer un diagnóstico microbiológico rápido y específico mediante técnicas de laboratorio con características operacionales (sensibilidad, especificidad, etc.) aceptables, y disponible y adaptable a los distintos entornos clínicos22.
Como queda reflejado en la revisión de los controles de calidad de la Sociedad Española de Enfermedades Infecciosas y Microbiología Clínica (SEIMC) de los últimos años23,24, el diagnóstico microbiológico del virus gripal, desde el punto de vista técnico, ha avanzado de manera exponencial en los últimos años, sobre todo en las técnicas basadas en ensayos moleculares. La participación de los laboratorios españoles ha experimentado un notable incremento a expensas de la paulatina adopción de técnicas rápidas basadas en tecnología molecular, y muy raramente por la implementación en los laboratorios de microbiología de otras técnicas clásicas basadas en el cultivo celular del virus o en la detección antigénica. Así, en el año 2006, el sistema de control de calidad remitió a 70 participantes un portaobjetos para realizar inmunofluorescencia directa (IFD) de virus respiratorios25. El portaobjetos contenía material celular fijado, procedente de un lavado nasofaríngeo de un bebé de 4 meses para que se realizara IFD frente a antígenos virales de VRS, virus de la gripe A, virus de la gripe B y virus parainfluenza 1, 2, 3 y 4. Es importante destacar que el material remitido no fue idéntico para todos los laboratorios, y fue, aproximadamente, el 50% positivo en el caso del virus de la gripe A y el 50% restante en el caso del virus de la gripe B. Solamente el 58%, 40 laboratorios de los participantes, respondieron y la inmunofluorescencia para VRS fue la más utilizada y, la que menos, la del virus parainfluenza 425. Hubo menor concordancia entre los participantes que recibieron el portaobjetos con virus de gripe A (87,9%), comparada con la obtenida entre quienes recibieron virus de la gripe B (93,9%). Casi la mitad de los participantes sustituyeron la técnica IFD para realizar procedimientos moleculares o técnicas rápidas inmunocromatográficas.
Tras la aparición del nuevo virus A (H1N1)pdm09 en el año 2009, los laboratorios intentaron abastecerse de técnicas de diagnóstico rápidas basadas en reacción en cadena de la polimerasa (PCR, polymerase chain reaction) a tiempo real, que con una escasa manipulación evitan en gran medida el error humano. De esta manera se intentó paliar la poca respuesta y la incertidumbre diagnóstica que surgió en el año 2009 tras la aparición del nuevo subtipo.
En los años 2014, 2015 y 2016, la SEIMC volvió a enviar controles de calidad para la detección del virus de la gripe. El del año 2014 se trataba de una muestra respiratoria de exudado nasofaríngeo resuspendida en medio de transporte de virus y el objetivo solicitado era detectar mediante diagnóstico molecular el virus de la gripe. El número de participantes se vio incrementado de manera notable respecto al control de calidad de 2006, pues participaron 91 laboratorios y una concordancia con el resultado de referencia del 92,5%. Sin embargo, el 16,6% no comunicó el subtipo de virus, que era una gripe A(H1N1)pdm09. En este control, 3 laboratorios utilizaron, además, el cultivo celular en células Madin-Darby de riñón de perro (MDCK, Madin-Darby canine kidney).
En el año 2015 se envió otro control de calidad similar al año previo en cuanto a tipo de muestra y solicitud diagnóstica, y en aquel control el resultado de referencia fue un virus de la gripe A subtipo H3. Entonces participaron 93 laboratorios, 2 más respecto al año anterior, y en esta ocasión solo el 29% comunicaron el subtipo H3. Es importante destacar que en este control 8 laboratorios participantes ya realizaron técnicas combinadas de detección de otros virus respiratorios, comunicaron codetección en la muestra remitida y abrieron la puerta a otro reto diagnóstico sobre el significado clínico cuando se encuentran simultáneamente gripe y otros virus respiratorios en la misma muestra23.
A partir de 2015, muchas de las técnicas para el diagnóstico del virus de la gripe han incluido otras dianas de virus respiratorios debido a la importancia de estos en la patología respiratoria y más concretamente en la neumonía adquirida en la comunidad26. Actualmente, existen paneles sindrómicos que, además de virus, permiten detectar bacterias responsables de neumonía atípica, como Legionella pneumophila, Mycoplasma pneumoniae y Chlamydophila pneumoniae, a las cuales se añadió posteriormente Bordetella pertussis.
En el control de calidad del año 2016, siguiendo la tendencia ascendente, participaron 98 laboratorios, a los cuales se les requirió una vez más un diagnóstico basado en técnicas rápidas moleculares de un virus A H1N1pdm. En general, hubo un alto porcentaje de concordancia con el diagnóstico de referencia y no se informó en el informe de control de calidad sobre el subtipado. La posibilidad de infecciones dobles o consecutivas por distintos tipos o subtipos de virus de la gripe obliga a disponer, al menos, de métodos que permitan el tipado del virus (A o B) y, en otros casos, de subtipado aunque clínicamente es menos importante determinar el linaje de la gripe B.
La evolución ascendente en el número de laboratorios participantes en la identificación de virus respiratorios, entre ellos la gripe, muestra que el uso de métodos moleculares basados en diferentes principios se ha ido modificando a lo largo de los años y no tienen ningún uso mayoritario un tipo de tecnología ni una sola marca comercial. El manejo sencillo y la escasa manipulación que requieren han convertido a algunas de ellas en métodos point of care (POC). Cada laboratorio elige entre la amplia oferta existente la técnica que mejor se adapta a sus necesidades. En general, se observa una tendencia a informar más frecuentemente el subtipo cuando este es H1 y menor cuando es H3 debido a la menor presencia de la diana H3 en las pruebas moleculares.
El avance en los hospitales de los Programas de Optimización de Uso de los Antibióticos (PROA) y el interés por realizar una adecuada política antimicrobiana es otro pilar importante en la implantación de equipos POC para el diagnóstico tanto del virus gripal como de otros virus responsables de patología respiratoria, que limitan el consumo de antibióticos al presentar un diagnóstico virológico confirmado por el laboratorio. Las nuevas técnicas ofrecen un diagnóstico en 3060 minutos y realizan el tipado y subtipado dentro de la misma reacción27. Aunque en los POC se corre el riesgo de perder sensibilidad y obtener resultados falsos negativos en muestras con escasa carga viral y con algunos equipos frente a subtipos A(H3N2). En este sentido, hay que prestar especial atención al posible descenso de rentabilidad de las técnicas moleculares asociado con mutaciones genéticas en regiones consideradas muy conservadas, como la proteína M, en la cual se basan numerosas técnicas moleculares. Este descenso de sensibilidad puede deberse al predominio de clones circulantes o a la emergencia del virus de la gripe de origen zoonótico que no coincide exactamente con las dianas que detecta la técnica molecular empleada29.
En el entorno hospitalario deberían elegirse plataformas que detecten gripe en un gran número de muestras, de forma semiautomatizada, para determinar el tipo y subtipo de virus gripal. El uso de técnicas exclusivamente para diagnóstico de gripe A y B ofrece, en general, mejores resultados de sensibilidad y especificidad que cuando se utilizan técnicas combinadas, denominadas multiplex, que pueden ofrecer más de 20 dianas de patógenos respiratorios (virus y bacterias) en una ronda diagnóstica. Estas pueden tener mermas de sensibilidad debido a la variabilidad del virus en determinadas temporadas gripales27,28. Esta menor sensibilidad y especificidad es producida por la competitividad de los reactivos moleculares hacia el material genético que se quiere detectar y puede producir resultados falsos negativos29.
Cultivo celular en el diagnóstico virológico del virus de la gripeEl diagnóstico clásico basado en el cultivo celular solo está presente en laboratorios impregnados de lo que podría denominarse «cultura de diagnóstico virológico independiente», anterior a la existencia de técnicas comercializadas. Algunos laboratorios han inter-pretado que el cultivo de líneas celulares ofrece una nueva dimensión y potencialidad diagnóstica frente a nuevos virus, que debe ser combinada obligatoriamente con la seguridad del laboratorio. Aunque la realización de la técnica continúa siendo lenta, costosa y necesita mucho tiempo de manipulación, concede ventajas sobre las técnicas moleculares, como la prueba de viabilidad del virus o su utilización en otras técnicas de caracterización e identificación antigénica o genética30,31. En la actualidad existen células comercializadas MDCK, MDCKsiat1 y A549, tanto en frascos para su tripsinización, posterior reparto en tubos o viales, y mantenimiento, así como ya listas en shell-vial, que hacen más asequible la implementación de esta metodología para el uso hospitalario.
Un requisito determinante para el diagnóstico mediante cultivo celular es que el virus sea viable y ello requiere exigencias adicionales de transporte y conservación de la muestra, superiores a las requeridas para el diagnóstico molecular. El cultivo celular es el único que permite obtener virus vivo y establece el diagnóstico cuando el genoma del virus sufre variaciones respecto a las sondas en que están basadas las técnicas de PCR, de especial importancia en el diagnóstico de casos de gripe animal o zoonótica, o en la emergencia de variantes mayores pandémicas. En 2009 se diagnosticó de forma fortuita (serendípica) la emergencia pandémica del subtipo A(H1N1)pdm09, ante la imposibilidad de subtipado con las PCR existentes, de una muestra clínica positiva para gripe A y varios falsos negativos moleculares solo detectables mediante cultivo. Esta falta de sensibilidad de algunos equipos de detección genómica se ha hecho evidente frente a variantes del subtipo A(H3N2) mucho más variable que el A(H1N1)pdm0927,28,32. Por ello, en determinados casos, el cultivo celular puede servir como técnica de rescate en el diagnóstico clínico y, aunque con menor sensibilidad que el diagnóstico molecular, continúa siendo uno de los métodos diagnósticos de referencia.
Detección de mutaciones asociadas con resistencia y carga viralOtros retos pendientes de estandarización y solución global en el diagnóstico virológico clínico de la gripe son la correlación y medida de la carga viral en determinados procesos gripales graves o la detección de mutaciones asociadas con resistencia a los antivíricos. También son necesarios otros métodos estandarizados y marcados para cuantificar y seguir virológicamente la evolución de la infección. Desde la pandemia de 2009 se demostró la importancia de la «tormenta de citocinas», así como sus perfiles de expresión de ácido ribonucleico mensajero (ARNm) y la activación celular, que no han tenido continuidad en su aplicación clínica33-35. Finalmente, es necesaria una metodología asequible para los laboratorios de microbiología hospitalarios que permita el control de la resistencia a antivíricos específicos36. Estos aspectos permitirán una gestión óptima de la asistencia clínica moderna a los enfermos de gripe.
Actualmente, solo la detección de la mutación H275Y del subtipo A(H1N1)pdm09 podría ser implementada a nivel hospitalario de forma generalizada. Otras mutaciones diferentes del mismo subtipo, así como las que pueden aparecer en el subtipo A(H3N2), solo pueden abordarse mediante técnicas como la secuenciación o snips (single nucleotide poymorphism), que no están adecuadamente homologadas. La gran variabilidad del virus de la gripe A hizo que en 1997 se produjera un reemplazo del subtipo salvaje sensible a adamantanos por un subtipo mutado resistente que sustituyó en 2 años al salvaje. La aparición en 2009 del subtipo A(H1N1)pdm09 genéticamente resistente a estos fármacos hizo que solo dispusiéramos de la familia de inhibidores de la neuraminidasa. Sin embargo, nadie puede asumir que no pueda volver a reemerger un subtipo A que pueda ser sensible a las aminas cíclicas (amantadina y rimantadina); antivíricos que volverían a estar otra vez vigentes como opciones terapéuticas frente al virus gripal. Aparte de ello, debe citarse la autorización en algunos países (Estados Unidos y Japón) de baloxavir marboxil, un inhibidor de la endonucleasa que solo requiere una dosis en las pri-meras 48 horas de aparición de los síntomas; aunque hace que sea más difícil la selección de resistencias por tratamientos incompletos, se han descrito ya variantes resistentes en un porcentaje pequeño, hecho que incide en la necesidad de que los laboratorios de microbiología clínica se doten de tecnología para afrontar las resistencias a los antivíricos frente a la gripe, al igual que frente a las bacterias.
Retos en el diagnóstico de gripe en la vigilancia epidemiológicaLos objetivos del diagnóstico de gripe en la vigilancia epidemiológica son distintos a los del diagnóstico virológico clínico. El diagnóstico epidemiológico busca confirmar la circulación y difusión del virus, el virus predominante en brotes y epidemias y otras características que se exponen en la tabla 2.
Principales objetivos del diagnóstico de gripe en la vigilancia epidemiológica
| Detección precoz de la circulación de virus de la gripe y caracterización de tipo y subtipo |
| Confirmación virológica de la aparición o emergencia en seres humanos de virus de gripe con potencial pandémico |
| Informar en tiempo real a la OMS (GISRS) y SVGE sobre el tipo y subtipo de gripe circulante para la selección de cepas vacunales |
| Analizar la concordancia de las cepas de gripe circulantes con las vacunales |
| Detectar la aparición de variantes asociadas con resistencia a antivíricos o mutaciones de gravedad |
| Analizar la eficacia de la vacuna en términos de producción de anticuerpos protectores |
GISRS: Sistema de Vigilancia Internacional y de Respuesta frente a la Gripe; OMS: Organización Mundial de la Salud; SVGE: Sistema de Vigilancia de la Gripe en España.
Los sistemas de vigilancia epidemiológica de gripe requieren datos virológicos precisos, en los cuales primen la calidad y profundidad diagnósticas más que la urgencia del diagnóstico virológico asistencial. Esta información detallada permite que el GISRS cumpla su función y esté preparado para detectar cualquier amenaza gripal lo antes posible. A este respecto, conviene recordar que en el año 2008 el SVGE fue capaz de detectar un caso de infección por un virus de gripe porcina del subtipo Asw(H1N1), linaje clásico de cerdos, en una mujer de Aragón que tenía contacto con cerdos16,17. Este caso demostró cómo la red de Aragón del SVGE, coordinada con los servicios asistenciales, permitió detectar a una persona con gripe porcina, vigilada por los médicos centinela. Aun así, el sistema de vigilancia necesita refinarse continuamente para afrontar los retos que el diagnóstico de gripe requiere en este campo.
Técnicas moleculares en la vigilancia epidemiológicaEl diagnóstico directo en la vigilancia epidemiológica busca la detección de virus de la gripe y su caracterización genética y antigénica. Al igual que en el diagnostico virológico asistencial, la primera línea diagnóstica para la vigilancia epidemiológica moderna es la PCR, la herramienta más útil, rápida, sensible y específica disponible en la actualidad37,38. Un reto para el diagnóstico epidemiológico es disponer de una técnica automatizada molecular con el 100% de sensibilidad que permitiera la eliminación rápida de casos negativos de los 18 subtipos de gripe A existentes. De esta forma, el laboratorio de virología se concentraría de forma preferente en aquellas muestras con resultados positivos en este cribado diagnóstico. Aunque una gran parte de las técnicas comercializadas tienen como dianas el gen de la proteína M o NP, muy conservados entre todos los subtipos A, pueden producirse variaciones importantes de sensibilidad debido a la deriva genética del virus32,39,40 que ocurre con relativa frecuencia27,28.
Otro de los grandes retos para el diagnóstico con fines de vigilancia es disponer de técnicas de PCR para subtipos no humanos más asequibles ya que las distribuidas a través de organismos internacionales como IRR (International Reagent Resource) o ATCC (American Type Culture Collection) exigen trámites complejos, incluidos los aduaneros. Se facilitaría así la detección de los subtipos de gripe aviares no estacionales más importantes (de H5 a H16), especialmente los que causan casos humanos (H5, H7 y H9), y los del cerdo (H1sw y H3sw). A estos paneles sería necesario añadir otros con dianas moleculares que permitieran identificar mutaciones de resistencia a antivíricos en todos los virus estacionales humanos.
Cultivo del virus en vigilancia epidemiológicaEl cultivo de los virus de la gripe es un método complementario que continúa conservando un enorme valor diagnóstico añadido. Uno de los retos del diagnóstico en vigilancia epidemiológica que ayuda a solventar el cultivo de virus es la posibilidad de recuperar falsos negativos en pruebas moleculares de casos de gripe de origen animal o composiciones genéticas «exóticas» que escapen a la primera línea de diagnóstico molecular. El cultivo viral es la técnica fundamental para la obtención de antígenos para realizar pruebas serológicas específicas o análisis antigénico y genético in extenso41.
Para el cultivo del virus de la gripe hay 2 sistemas universalmente reconocidos: el más clásico, realizado en huevos embrionados de gallina de 9 a 11 días de incubación, durante 48-72 horas a 33-35 °C, y el más moderno en líneas celulares continuas MDCK y MDCK/Siat1, que han sustituido a las líneas continuas de riñón de mono empleadas antiguamente42. En general, el cultivo en huevo embrionado facilita el aislamiento de los virus de origen aviar y, por extensión, de aquellos que han franqueado la barrera de especie a seres humanos en épocas recientes. A medida que un virus de la gripe, tras el salto de la barrera de especie, continúa infectando a huéspedes humanos, cambia su afinidad por receptores siálicos de células diana epiteliales: Neu-α2,3Gal (receptor aviar) presente en órganos profundos por la de Neu-α2,6Gal (receptor humano) presente en epitelios superficiales, como la faringe humana, y crece más difícilmente en huevo43. A este respecto, conviene recordar que la distribución de esos receptores siálicos es distinta en la cavidad alantoidea (mayoría de receptores Neu-α2,3Gal) que en la membrana amniótica que presentan ambos receptores44.
Debido al cultivo del virus en células durante su propagación, se seleccionan mutaciones cercanas a los epítopos inmunodominantes de la hemaglutinina (HA) que pueden traducirse en variaciones biológicas con consecuencias relevantes. Así, se ha demostrado con vacunas producidas en huevo embrionado y menor efectividad por cambios próximos al receptor de la HA45 o incapacidad para hemaglutinar (subtipo H3N2 propagado en células MDCK)46. Las líneas celulares más frecuentemente empleadas por orden de antigüedad son las MDCK, una variante de ellas, MDCK/Siat1, y recientemente A549 para los virus del subtipo A(H3N2). Estas líneas celulares expresan los 2 receptores: Neu-α2,3Gal y α2,6Gal44.
Entre los virus A que cocirculan en la actualidad, los que mejor crecen en huevo embrionado son las variantes del subtipo A(H1N1)pdm09 y las que peor crecen, incluso en células, son las variantes de A(H3N2), sobre todo los clados 3c.2a y 3c.3a aparecidos en la temporada 2014201547. Los virus B crecen preferentemente en células MDCK48.
El reto del cultivo de virus gripal es lograr líneas celulares que faciliten su propagación modificando lo menos posible el virus, aspecto fundamental para la composición de las vacunas, ya que la efectividad de la vacuna antigripal depende de su fidelidad con las cepas circulantes. La dificultad de crecimiento y correcta caracterización de ciertos clados del subtipo A(H3N2) disminuyen la representación de esas cepas algunos años, lo que provoca un descenso de la efectividad vacunal49,50. Este fenómeno se observó en las campañas vacunales de las temporadas 2014-2015 y 2016-2017, en las cuales la efectividad vacunal fue baja (19-30%) debido a las diferencias antigénicas entre la cepa vacunal y la cepa salvaje por las modificaciones antigénicas sufridas por la primera durante su propagación en huevo embrionado50-52.
Retos diagnósticos en la caracterización antigénica y genéticaLos objetivos del diagnóstico de la gripe perseguidos por la vigilancia epidemiológica obligan a determinar con exactitud el subtipo de gripe A o el linaje de B, en su caso, y llegar hasta el nivel de cepa o clado genético cuando sea posible. Esta caracterización precisa protocolos antigénicos y genéticos de forma separada. Los métodos de caracterización antigénica se realizan mediante la reacción de inhibición de hemaglutinación (RIH), en la cual se enfrenta el virus, previamente cultivado, a anticuerpos específicos frente a cepas de gripe, obtenidos en animales hiperinmunizados, generalmente ovejas, gallinas, conejos o hurones. La RIH utiliza hematíes de diferentes especies animales (gallina, pavo o cobaya), difíciles de obtener en ocasiones.
Uno de los principales retos es la caracterización antigénica de los virus de la gripe del subtipo A(H3N2). Desde hace varios años, este virus se propaga difícilmente en huevo embrionado o en líneas celulares, y ha sufrido modificaciones en su actividad enzimática y en los receptores de la HA tras su cultivo en células MDCK46,53. El virus de este subtipo cultivado en MDCK selecciona ciertas mutaciones en la posición aminoacídica 150 de la neuraminidasa, cerca del sitio catalítico, por lo que esta proteína se comporta con actividad hemaglutinante53. Estas mutaciones pueden afectar a la infecciosidad y a la capacidad de aglutinar eritrocitos, por lo que se hace indispensable utilizar oseltamivir en los ensayos de hemaglutinación y caracterización antigénica de los A(H3N2) ya que la actividad hemaglutinante de la neuraminidasa altera los resultados de la RIH para la caracterización antigénica46.
La caracterización genética es una herramienta indispensable para la vigilancia epidemiológica ya que ofrece toda la información sobre la composición genética del virus, sobre todo en aquellos casos en que resulta impracticable la caracterización antigénica. Mientras que todos los cambios antigénicos responden a cambios en la genética del virus, no todos los cambios genéticos que se producen en la secuencia de alguno de los 8 genes de la gripe se traducen finalmente en variaciones antigénicas de la proteína que codifican. Hay mutaciones silenciosas o sinónimas, y sustituciones de aminoácidos que no se traducen en cambios de la proteína. Por ello tienen especial interés tanto la caracterización antigénica como genética y la información obtenida por ambas metodologías se complementa.
La secuenciación identifica con total fiabilidad la cepa de gripe y su relación con otras, por lo que permite construir árboles filogenéticos y evaluar la evolución y deriva genética del virus54. Asimismo, sirve para identificar la existencia de mutaciones de resistencia a fármacos antivíricos y otras mutaciones compensatorias en la HA y la neuraminidasa (NA) del virus55,56. También ayuda a identificar mutaciones relacionadas con la patogenicidad, como la D222G, que permite al virus infectar las vías respiratorias bajas, o evaluar mutaciones relacionadas con la especificidad del hospedador57. Los métodos de secuenciación se realizan principalmente por Sanger o técnicas de NGS (next generation sequencing).
Un reto diagnóstico de este apartado consiste en el hecho de que la gestión de la información genética obtenida es difícil y laboriosa con las herramientas informáticas disponibles, lo que implica especialización del personal que maneje este tipo de técnicas, y limita su uso en ciertos laboratorios.
Diagnóstico indirectoLos métodos de diagnóstico indirecto tienen una aplicación mucho más epidemiológica que clínica. Tanto la RIH como la microneutralización (MN) y otras técnicas ya mucho menos utilizadas, como la hemólisis radial simple, son técnicas para evaluar la eficacia serológica vacunal frente a la gripe y estudios de seroepidemiología. Estas técnicas, realizadas por un número muy escaso de laboratorios, sirven para determinar el nivel de anticuerpos basales en una población, la eficacia serológica de una vacuna antigripal, la persistencia de los anticuerpos, diferenciar poblaciones de pacientes que no han estado expuestas previamente al patógeno y aquellas que lo han estado, o evaluar la protección heterotípica frente a variantes de gripe pandémica58,59.
Uno de los grandes problemas de los métodos de diagnóstico indirecto es su escasa estandarización, su nula automatización y el difícil acceso a la compra y adquisición de los reactivos. La estandarización de estos métodos diagnósticos es más difícil que la de los utilizados en el diagnóstico microbiológico clínico, que utiliza productos aprobados por la FDA o con marcado CE con certificados IVD (in vitro diagnostic) listos para su uso en cualquier laboratorio.
Muchas de estas técnicas han sufrido adaptaciones a lo largo del tiempo, sin coordinación entre los diferentes laboratorios. Por ello, no hay consenso sobre estos protocolos, lo que dificulta la comparación de los resultados entre laboratorios. Para solucionar este reto, han aparecido consorcios internacionales, como CONSISE (Consortium for the Standarization of Influenza Seroepidemiology), cuyo objetivo es la estandarización de los protocolos utilizados para la seroepidemiología de gripe60.
El reto que intentan solucionar estos consorcios es la adecuación de los correlatos de protección serológica a los nuevos conocimientos científicos. Por ejemplo, en RIH un título > 1:40 se acepta internacionalmente como protector61. Sin embargo, bibliografía reciente sugiere que este correlato no es del todo correcto para personas menores de 14 años o para personas mayores de 65 años, por lo que es necesario adecuarlo a esas poblaciones. Además, el estudio de los anticuerpos frente a otras proteínas del virus, como la neuraminidasa, obliga a determinar si los correlatos de protección son para cada antígeno, o por el contrario, pueden usarse los mismos para todos.
Los requerimientos y retos de futuro para el diagnóstico de la gripe en la vigilancia epidemiológica son, fundamentalmente, universalizar y estandarizar las técnicas existentes al simplificarlas y volverlas más accesibles a los laboratorios. Sin embargo, las nuevas aproximaciones a las futuras vacunas antigripales obligan a buscar nuevas técnicas de diagnóstico indirecto que sean capaces de detectar anticuerpos frente a zonas conservadas de proteínas víricas, frente a las cuales no se dispone de ningún ensayo estandarizado. La medida de anticuerpos frente al tallo (región 2 de la HA)62, 63 y no solo frente a la cabeza de la HA (dominio HA1), o frente a otras proteínas conservadas internas o externas, como la M2 o la NP62, es una necesidad futura. A este respecto, existen técnicas para medir anticuerpos frente a otras proteínas distintas de la hemaglutinina, como la técnica ELLA (enzyme-linked lectin assay), que detecta anticuerpos frente a la neuraminidasa64. Finalmente, otro de los aspectos o retos pendientes de diagnóstico es el del análisis de parámetros que evalúen la inmunidad celular frente a la gripe (medida de CD4 y CD8 específicos) para establecer correlatos de protección.
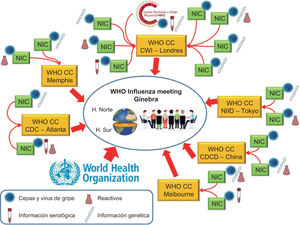
Retos diagnósticos para el GISRSHacer más eficiente el trabajo del GISRS es el reto diagnóstico más importante de la vigilancia epidemiológica en gripe. Dadas las necesidades y objetivos de esta red internacional41, los NIC deben disponer de la mejor tecnología y los recursos necesarios para llevar a cabo todos los ensayos que requiere el tipado e identificación de la gripe y liberar a los 6 Centros Colaboradores de la OMS (WHO CC) de una parte importante de su trabajo. De esta forma, los CC podrían recibir cepas más identificadas, caracterizadas más detalladamente y más procesadas, y podrían destinar más recursos y tiempo a la comparación entre ellas y a su relación geográfica.
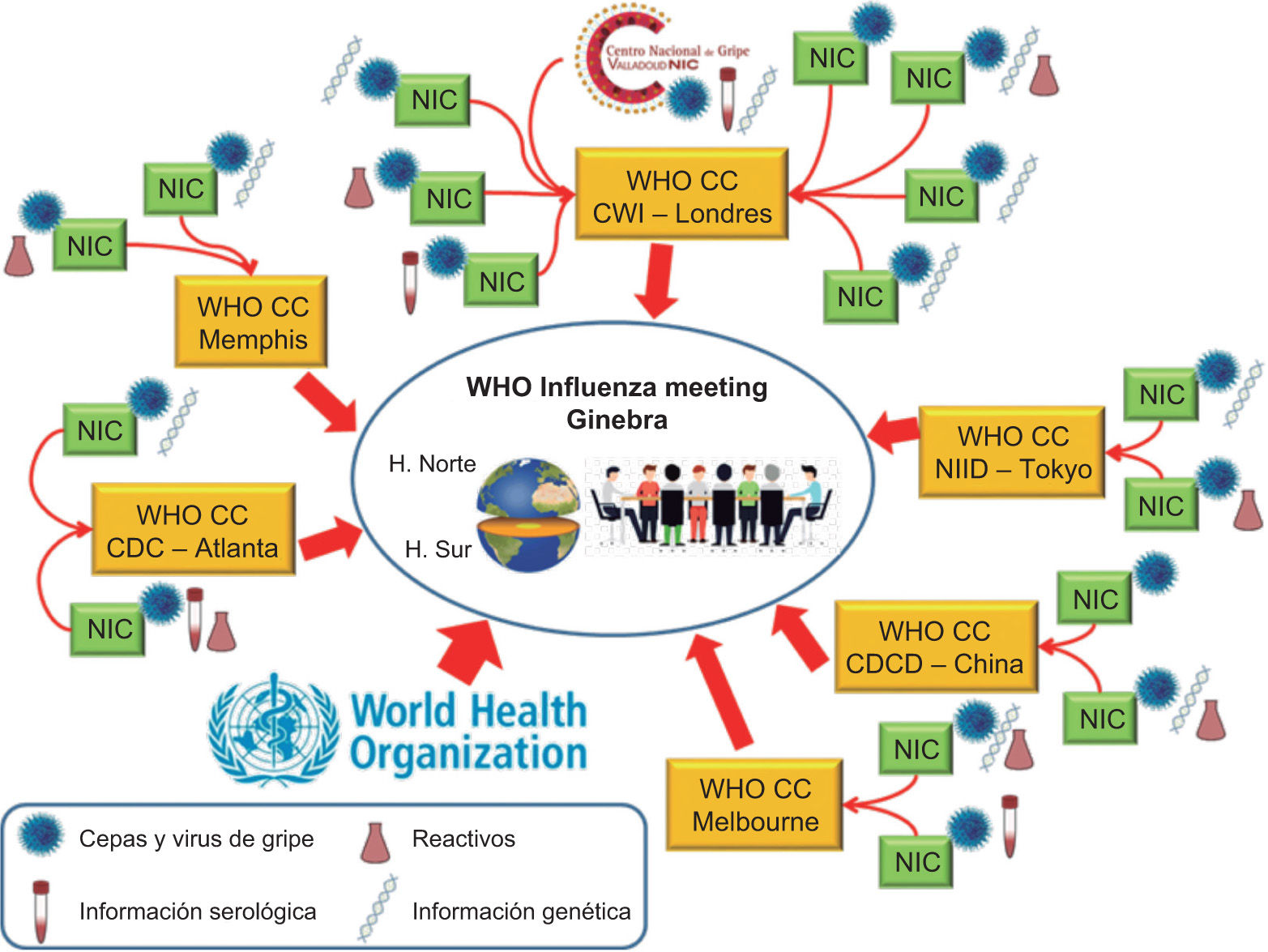
El objetivo principal del GISRS es informar en tiempo real de la actividad gripal a nivel mundial y seleccionar las cepas que deben ser incluidas en la vacuna antigripal de la temporada siguiente. El funcionamiento y flujo de trabajo y de la información del GISRS para la selección de cepas de la vacuna antigripal de la siguiente temporada se muestra en la figura 3.
Flujo de trabajo y de la información del diagnóstico epidemiológico y de vigilancia por parte de los integrantes del GISRS para la selección de cepas de la vacuna antigripalde la siguiente temporada. CDC: Center for Disease Control and Prevention; CDCD: Chinese Center for Disease Control and Prevention; CWI: Crick Worldwide Institute; NIC: National Influenza Centre; NIID: National Institute of Infectious Diseases of Japan; WHO-CC: WHO Collaboration Center.
Dado que la gripe es una antropozoonosis global emergente, el reto del GISRS es extender las habilidades de vigilancia diagnóstica a otras zonas geográficas y ayudar a implementar laboratorios en países de recursos bajos y medios, consolidando las zonas donde ya cuenta con una buena cobertura.
ConclusionesEl diagnóstico de la gripe por una técnica suficientemente sensible no está disponible en todos los centros hospitalarios españoles. La primera línea diagnóstica son los métodos POC y, en segundo lugar, los diagnósticos moleculares frente a distintas dianas que subtipen H1 y H3. La mayor parte de hospitales carece de cultivo celular y algunos lo han abandonado ante las prestaciones del diagnóstico molecular. Faltan recursos para la puesta a punto de métodos asequibles y completos para la detección de resistencias a antivíricos, marcadores de virulencia, incluida la medida de carga viral, y otros marcadores complementarios de alteración del sistema inmunitario del paciente (citocinas, perfiles de expresión de ARNm, etc.). Finalmente, para el diagnóstico virológico de uso epidemiológico se precisa un acceso más fácil a reactivos y técnicas de detección de subtipos, así como la estandarización de algunos métodos de caracterización antigénica y genética.
Conflicto de interesesLos autores declaran no tener ningún conflicto de interés en relación con el tema objeto de este artículo.
Información sobre el suplementoEste artículo forma parte del suplemento titulado «Programa de Control de Calidad Externo SEIMC. Año 2016», que ha sido patrocinado por Roche, Vircell Microbiologists, Abbott Molecular y Francisco Soria Melguizo, S.A.