La mayor parte de los casos de fiebre de duración intermedia (FDI) en España corresponden a enfermedades infecciosas (principalmente fiebre Q y rickettsiosis). En la práctica clínica el diagnóstico causal de estas entidades se basa en el inmunodiagnóstico, con una escasa utilidad en fases precoces. Por ello, el objetivo de este trabajo fue la evaluación de la utilidad de técnicas moleculares en el diagnóstico precoz de fiebre Q y rickettsiosis en pacientes con FDI. Se estudió mediante PCR la presencia de material genético de Coxiella burnetii y Rickettsia spp. en muestras sanguíneas de 271 pacientes con FDI. La especificidad de ambas técnicas es elevada, permitiendo el diagnóstico en casos no diagnosticados mediante detección de anticuerpos específicos. Estos datos sugieren que el empleo de técnicas moleculares, con una adecuada selección de la muestra de estudio y el empleo de cebadores adecuados, es un elemento útil en el diagnóstico precoz de las principales causas de FDI, principalmente si la serología es negativa o no es concluyente.
Most cases of fever of intermediate duration (FDI) in Spain are associated with infectious diseases (mainly Q fever and rickettsia infections). In clinical practice, the causal diagnosis of these entities is based on immunodiagnostic techniques, which are of little help in the early stages. Therefore, the aim of this study was to evaluate the usefulness of molecular techniques for the early diagnosis of Q fever and rickettsia diseases in patients with FDI. A PCR method was used to detect the presence of genetic material of Coxiella burnetii and Rickettsia spp. in blood specimens from 271 patients with FDI. The specificity of both techniques is high, allowing diagnosis in cases undiagnosed by specific antibodies detection. These data suggest that the use of molecular techniques, with proper selection of the study specimen, and using appropriate primers is a useful tool in the early diagnosis of the main causes of FDI, especially if serology is negative or inconclusive.
Se denomina fiebre de duración intermedia (FDI) a la situación clínica en la que se detecta una temperatura axilar superior a 38°C, sin foco aparente, con una duración superior a una semana e inferior a 3 semanas, que se presenta en pacientes no inmunodeprimidos, sin ingreso hospitalario previo y en los que tras una evaluación inicial se desconoce su etiología1. La mayor parte de los casos corresponden a enfermedades infecciosas (principalmente ocasionadas por Coxiella burnetii, Rickettsia spp., Brucella spp., citomegalovirus [CMV] y virus de Epstein-Barr [VEB]2). En la práctica clínica, el diagnóstico causal de estas entidades se basa en el inmunodiagnóstico, lo que requiere al menos 15-21 días desde el comienzo de la infección hasta que aparecen los anticuerpos. Por ello, las técnicas serológicas son poco útiles en el diagnóstico precoz de estas enfermedades. Sin embargo, la identificación de los principales agentes causales en las fases iniciales tiene gran interés por varias razones: a)frecuentemente son enfermedades con una gran afectación del estado general que se benefician del tratamiento etiológico, si existe; b)en ocasiones se presentan como cuadros clínicos graves o complicados que requieren hospitalización3; c)la duración del tratamiento empírico recomendado difiere entre las 2 formas principales encontradas (fiebre Q y tifus murino), y d)en algunos casos de fiebre Q, a pesar del tratamiento antimicrobiano adecuado, persisten las manifestaciones clínicas, lo que indica la necesidad de añadir corticoides4.
Por ello, el objetivo de este trabajo fue la evaluación de la utilidad de técnicas moleculares (polymerase chain reaction [PCR]) en el diagnóstico precoz de fiebre Q y rickettsiosis en pacientes con FDI.
Pacientes y métodosLa población total de estudio estaba constituida por 271 pacientes con criterios de FDI1, evaluados durante un periodo de 6 años (2004-2009) en las consultas externas de la Unidad de Enfermedades Infecciosas y Medicina Tropical (UEIMT). En todos ellos se recogieron de forma protocolizada los datos epidemiológicos, clínicos, estudio analítico elemental en sangre y orina, radiografía de tórax, hemocultivos, y se realizaron pruebas para la detección de anticuerpos frente a C.burnetii, Rickettsia typhi, CMV y VEB. Se prestó un especial interés en la determinación del intervalo desde el inicio del cuadro clínico y el estudio serológico y el empleo previo de antimicrobianos.
El diagnóstico serológico de la infección por C.burnetii o R.typhi se realizó por inmunofluorescencia indirecta (IFI) (Coxiella burnetii-Spot IF, Rickettsia mooseri Spot IF, BioMérieux, Marcy l’Étoile, Francia). Los criterios empleados para el diagnóstico fueron: a)un título único de IgM≥1/80 e IgG≥1/320 frente a antígenos de C.burnetii en faseii o un título único de IgM≥1/40 e IgG≥1/160 frente a R.typhi; b)seroconversión (desde títulos iniciales negativos a positivos a las 2-3 semanas), o c)un incremento de 4 veces el título de IgG ente el suero de la fase aguda y la fase de convalecencia.
Los pacientes se clasificaron en varios grupos: a)«diagnóstico definido de fiebre Q o de rickettsiosis», que fueron subdivididos atendiendo a si la serología inicial era negativa o positiva; b)«otros diagnósticos definidos» (infección por VEB y CMV, empleando los criterios usuales de diagnóstico); c)«diagnóstico “incompleto”», en los que no pudo establecerse un diagnóstico definido debido a que el paciente no presentaba criterios iniciales diagnósticos y no acudió para su seguimiento, y d)«no diagnóstico», si tras todos los estudios no se llegó a un diagnóstico definido.
Se recogió de cada paciente una muestra de plasma con EDTA en el momento que acudió a la consulta. Se mantuvieron congeladas y posteriormente se procesaron 200μl para la extracción de ADN (QIAamp® DNA Mini kit, Qiagen, Alemania) siguiendo las indicaciones del fabricante. El diagnóstico molecular se realizó mediante técnicas de amplificación y detección del ADN bacteriano mediante la reacción en cadena de la polimerasa (PCR) para C.burnetii y Rickettsia spp. utilizando en el primer caso cebadores que amplifican el segmento de inserción IS11115 y para Rickettsia spp. utilizando como diana el espacio intergénico 23S-5S rRNA6.
ResultadosDe los 271 pacientes con FDI se realizó el diagnóstico de fiebre Q en 86 casos (31,7%), de tifus murino en 66 (24,3%), de infección por CMV en 16 (5,9%) y de infección por VEB en 13 (4,3%). En 64 pacientes no se pudo llegar al diagnóstico final, y 57 presentaron un «diagnóstico incompleto». Por ello, la mitad de los pacientes con FDI fueron diagnosticados serológicamente de fiebre Q o tifus murino.
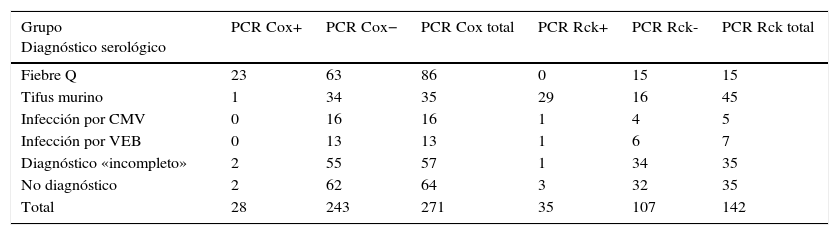
Se estudió mediante PCR la presencia de material genético de C.burnetii en 271 pacientes con diferentes diagnósticos serológicos (tabla 1). Existía una asociación significativa desde un punto de vista estadístico entre la positividad de la prueba (detección molecular mediante PCR de C.burnetii) y el diagnóstico serológico de fiebre Q (test de Fisher, p<0,05). Las características de la prueba fueron las siguientes: a)sensibilidad: 26,7% (IC95%: 18,5-36,9%); b)especificidad: 97,9% (IC95%: 95,8-99,8%); c)valor predictivo positivo: 82,1% (IC95%: 64,4-92,2%), y d)valor predictivo negativo: 74,1% (IC95%: 68,2-79,2%).
Resultados de ambas PCR en los grupos de estudio
| Grupo Diagnóstico serológico | PCR Cox+ | PCR Cox− | PCR Cox total | PCR Rck+ | PCR Rck- | PCR Rck total |
|---|---|---|---|---|---|---|
| Fiebre Q | 23 | 63 | 86 | 0 | 15 | 15 |
| Tifus murino | 1 | 34 | 35 | 29 | 16 | 45 |
| Infección por CMV | 0 | 16 | 16 | 1 | 4 | 5 |
| Infección por VEB | 0 | 13 | 13 | 1 | 6 | 7 |
| Diagnóstico «incompleto» | 2 | 55 | 57 | 1 | 34 | 35 |
| No diagnóstico | 2 | 62 | 64 | 3 | 32 | 35 |
| Total | 28 | 243 | 271 | 35 | 107 | 142 |
CMV: citomegalovirus; PCR Cox: PCR para Coxiella burnetii; PCR Rck: PCR para Rickettsia; VEB: virus de Epstein-Barr.
Con el objetivo de identificar la utilidad de la detección molecular mediante PCR de C.burnetii en pacientes con fiebre Q, reevaluamos los resultados atendiendo a los resultados de la serología. Los datos se expresan en la tabla 2a. Empleando el test de Fisher, se observó que existía una asociación significativa (p<0,001) entre la presencia de una serología inicial positiva y una PCR de C.burnetii negativa.
Relación entre los datos serológicos y los resultados de la PCR
| Serología inicial negativa | Serología inicial positiva | Total | |
|---|---|---|---|
| PCR Cox+ | 20 | 1 | 21 |
| PCR Cox− | 20 | 43 | 63 |
| Total | 40 | 44 | 84a |
PCR Cox: PCR para Coxiella burnetii.
Este hecho se relaciona con el tiempo de evolución de la enfermedad, que era significativamente menor en los pacientes con PCR positiva (7 días [5-12]) con respecto a los que la PCR era negativa (14 días [4-90]). Por otro lado, un 56% de los pacientes con fiebre Q en los que la PCR de C.burnetii fue negativa habían recibido algún tratamiento antimicrobiano (doxiciclina en 2 casos, quinolonas en 6 casos y betalactámicos en 27 casos).
El estudio mediante PCR para detectar genoma de Rickettsia spp. se realizó en 142 pacientes con diferentes diagnósticos serológicos (tabla 1). Existía una asociación significativa desde un punto de vista estadístico entre la positividad de la prueba (detección molecular mediante PCR de Rickettsia spp.) y el diagnóstico de tifus murino (test de Fisher, p<0,0001). Las características de la prueba fueron las siguientes: a)sensibilidad: 82,9% (IC95%: 67,3-91,9%); b)especificidad: 85,0% (IC95%: 77,1-90,6%); c)valor predictivo positivo: 64,4% (IC95%: 49,8-76,8%), y d)valor predictivo negativo: 93,8% (IC95%: 82,7-97,1%).
Con el objetivo de identificar la utilidad de la detección molecular mediante PCR de Rickettsia spp. en pacientes con tifus murino, reevaluamos los resultados atendiendo a los resultados de la serología. Los datos se expresan en la tabla 2b. No existía asociación entre la presencia de serología negativa frente a R.typhi y una PCR positiva genérica para Rickettsia spp. (test de χ2, p=0,80). No encontramos diferencias en el tiempo de evolución de la enfermedad según los resultados de la PCR, siendo en ambos casos entre 4 y 15 días.
DiscusiónExisten escasos estudios en humanos acerca de la utilidad de la PCR de C.burnetii en muestras sanguíneas para el diagnóstico de la fiebre Q7-10. Nuestro estudio presenta similitudes, pero también algunas diferencias con ellos. Así, como hemos comprobado, la rentabilidad de la PCR es mayor cuando se realiza en fases precoces de la enfermedad7,8. Los resultados también son diferentes cuando se emplean cebadores que amplifican secuencias diferentes (p.ej., htp AB o ompA) o técnicas diferentes (PCR clásica, nested PCR o PCR a tiempo real)7-10. Por otro lado, nuestros resultados son similares a los de otros autores al obtener una mayor rentabilidad diagnóstica cuando se emplean muestras de plasma frente a muestras de suero9. De cualquier forma, en nuestro trabajo se observa que esta técnica presenta una elevada especificidad en el diagnóstico de la fiebre Q aguda en las fases precoces de la enfermedad. La negatividad de la prueba en fases precoces puede relacionarse con el elevado empleo de antimicrobianos (incluidos los betalactámicos), lo que refuerza la actitud de evitar su empleo en casos de FDI. En este sentido, tiene interés señalar que C.burnetii posee peptidoglucano en su pared celular11 y es sensible in vitro a ampicilina12. Por ello, es posible que los betalactámicos sean eficaces en la eliminación de C.burnetii en sangre y por ello capaces de negativizar la PCR. Sin embargo, su dificultad para penetrar en el interior de las células condiciona su ineficacia en la práctica. Por otro lado, la presencia de material genético de C.burnetii en pacientes en los que la serología es positiva puede sugerir la posibilidad de desarrollo de una forma crónica7,8.
El empleo de la PCR para el diagnóstico precoz de infecciones por el género Rickettsia empleando muestras sanguíneas en la literatura es aún menor que para la fiebre Q, y en estos trabajos el número de pacientes estudiados es muy escaso13-15. En general, los datos de la literatura demuestran una baja sensibilidad, aunque también se observan diferencias atendiendo al momento evolutivo de la enfermedad. En nuestro trabajo, el empleo de cebadores genéricos permite detectar no solo R.typhi sino también otras rickettsias que pueden estar presentes en nuestro medio (p.ej., R.felis o R.massiliae)16,17, por lo que no sorprende la detección de casos positivos mediante PCR en pacientes con serología negativa frente a R.typhi.
El uso combinado de estas 2 PCR nos permitió llegar al diagnóstico en 8 pacientes (4 casos de fiebre Q y 4 rickettsiosis) en los cuales la serología fue negativa.
En resumen, nuestros datos sugieren que el empleo de técnicas moleculares, con una adecuada selección de la muestra de estudio y el empleo de cebadores adecuados, es un elemento útil en el diagnóstico precoz de las principales causas de FDI, principalmente si la serología es negativa o no es concluyente. El empleo de otras técnicas moleculares como LAMP, que no necesita del uso de termociclador y se realiza en 60min, podría aportar ventajas adicionales18-20.
FinanciaciónEste trabajo ha sido financiado en parte por el INIA, FAU2006-00002-C04-01.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.