Los carbapenémicos son los antibióticos betalactámicos con mayor espectro de actividad en el tratamiento de infecciones por Pseudomonas aeruginosa. El objetivo de este trabajo fue caracterizar molecularmente una colección de aislados de P. aeruginosa resistentes a carbapenémicos (PARC).
MétodosSe recogieron 85aislados PARC de 60pacientes en el Hospital San Pedro, Logroño (período 2008-2011). La relación clonal se determinó por electroforesis en gel de campo pulsado (PFGE), la sensibilidad a 15antipseudomónicos por método de difusión en disco y las alteraciones en oprD, la caracterización de integrones y la tipificación molecular (MLST) por PCR y secuenciación.
ResultadosLas 85PARC se clasificaron en 35perfiles diferentes de PFGE. Se seleccionaron 61cepas de los 60pacientes y se observó que eran multirresistentes, aunque ninguna mostró fenotipo carbapenemasa. Se detectó un gran polimorfismo de OprD, destacando que el 59% de las cepas presentaban un codón de finalización prematuro. ISPa1328 e ISPsp4 truncaban el gen oprD en 2 cepas (GenBank KF517097 y KF517098). El 67% de las cepas presentó integrones de clase 1 con genes codificantes de enzimas modificantes de aminoglucósidos, 2 de las cuales portaban un nuevo integrón: aac(3)-Ia+aadA1h (nombrado In272, GenBank GQ144317). Se detectaron 4 secuencias tipo (ST) (número de cepas): ST175 (35), ST308 (3), ST235 (2) y ST639 (1).
ConclusiónLa multirresistencia, el alto polimorfismo de oprD, el alto porcentaje de integrones, la moderada relación clonal de las cepas y la elevada diseminación epidémica de clones de alto riesgo son aspectos de gran preocupación clínica para erradicar la diseminación de PARC.
Carbapenems are the beta-lactam antibiotics with the best spectrum of activity in the treatment of Pseudomonas aeruginosa infections. The objective of this study was to molecularly characterise a collection of carbapenem-resistant P. aeruginosa isolates (PARC).
MethodsA total of 85 PARC isolates were recovered from 60patients in the Hospital San Pedro, Logroño (period 2008-2011). Clonal relationship was determined using pulsed-field gel electrophoresis (PFGE), susceptibility testing to 15anti-pseudomonal agents was performed using the disk diffusion method, and alterations in oprD, characterisation of integrons and molecular typing (MLST) using PCR and sequencing.
ResultsThe 85 PARC were classified into 35 different PFGE profiles. Of the 61selected strains from 60patients all of them were multiresistant, although none of them showed a carbapenemase phenotype. High polymorphism was detected in OprD, emphasising that 59% of the strains had a premature stop codon. ISPa1328 and ISPsp4 insertion sequences truncated oprD gene into 2 strains (GenBank KF517097 and KF517098). Two-thirds (67%) of the strains showed class 1 integrons with genes encoding aminoglycoside modifying enzymes, and 2 of them carried a new integron: aac(3)-Ia+aadA1h, named In272, GenBank GQ144317. Four sequence types were detected (Strain Nos.): ST175 (35), ST308 (3), ST235 (2), and ST639 (1).
ConclusionMultidrug resistance, high polymorphism in oprD, a high percentage of integrons, moderate clonal relationship of strains, and the high epidemic dissemination of high-risk clones are clinical aspects of great concern in order to eradicate the spread of PARC.
La especie Pseudomonas aeruginosa es uno de los patógenos oportunistas y nosocomiales de mayor relevancia clínica, no solo por su gran adaptabilidad, diseminación y persistencia (principalmente en ambientes húmedos), sino también por su resistencia intrínseca a diversos agentes antimicrobianos y su facilidad para adquirir nuevos mecanismos de resistencia, lo que dificulta el tratamiento exitoso en los hospitales cuando dicha bacteria se encuentra implicada en procesos infecciosos. Además, este patógeno causa infecciones graves en pacientes que sufren enfermedades respiratorias crónicas o en pacientes en condiciones de inmunodepresión, principalmente en el ámbito hospitalario, en unidades de cuidados intensivos (UCI) y en unidades de críticos oncohematológicos1,2.
Los carbapenémicos son los antibióticos betalactámicos con mayor espectro de actividad y son frecuentemente usados para el tratamiento de infecciones causadas por P. aeruginosa3. Para ejercer su acción, los carbapenémicos deben atravesar la pared celular, lo que sucede a través de las porinas de la membrana externa en las bacterias gramnegativas. En concreto, OprD es una porina de membrana externa sustrato-específica de P. aeruginosa, que permite la difusión de carbapenémicos, aminoácidos básicos y péptidos pequeños3. Sin embargo, en los últimos años se ha observado un incremento de cepas de P. aeruginosa resistentes a carbapenémicos (PARC)1,2. Los principales mecanismos de resistencia a los carbapenémicos en P. aeruginosa son: 1) alteraciones o pérdida de la porina OprD; 2) sobreexpresión de bombas de expulsión activa; 3) hiperproducción de la betalactamasa cromosómica AmpC, y 4) producción de enzimas carbapenemasas. Se han descrito carbapenemasas de clase A, de clase B o metalo-betalactamasas (MBL), y de clase D, oxacilinasas4. Aunque la prevalencia de PARC productoras de MBL está aumentando a nivel mundial, los mecanismos intrínsecos de resistencia a carbapenémicos como la presencia de alteraciones en OprD son los mecanismos más frecuentes5-7.
El objetivo de este trabajo fue caracterizar molecularmente una colección de aislados PARC recogidos en el Hospital San Pedro de Logroño. Se realizó el estudio fenotípico de resistencia a agentes antipseudomónicos, se analizaron los mecanismos de resistencia a carbapenémicos implicados, y se detectó la presencia de integrones, así como la tipificación molecular de los aislados seleccionados.
Material y métodosAislados clínicosSe recogieron 85aislados de P. aeruginosa con resistencia al menos a uno de los antibióticos carbapenémicos testados (imipenem, meropenem y/o doripenem), procedentes de muestras clínicas del Hospital San Pedro de Logroño durante el período de septiembre 2008 a diciembre 2011.
Los aislados se obtuvieron de diferentes tipos de muestras: herida (25), orina (16), úlcera (16), frotis rectal (11), frotis faríngeo (6), esputo (3), exudado conjuntival (2) y otros (6: frotis inguinal, exudado nasal, aspirado, punta de catéter central, hemocultivo y lavado broncoalveolar). Recogidas de 60pacientes hospitalizados (46 hombres y 14 mujeres), cuya media de edad era 69años (rango de edad entre 16 y 99años) y procedentes mayoritariamente de los servicios de neumología (15%), cirugía (15%), medicina general (8%), medicina interna (8%), hematología (7%), nefrología (5%), UCI o medicina intensiva (5%) o enfermedades infecciosas (5%).
Tipificación molecularLa relación clonal entre los aislados se estudió por electroforesis en gel de campo pulsado (PFGE). Se realizaron los insertos de ADN bacteriano en agarosa según el procedimiento descrito por Rojo-Bezares et al.8; y tras digestión con la enzima SpeI, los fragmentos se separaron por PFGE bajo las siguientes condiciones: 2 rampas lineales pulsadas de 5-15s y 15-45s, respectivamente, durante 10h cada una, manteniendo un gradiente de voltaje de 6V/cm y a 14°C. Los perfiles de bandas obtenidos fueron analizados con las recomendaciones de Tenover et al.9 y, posteriormente, por el programa informático BioNumerics 2.0 (Applied Maths, Bélgica).
La secuencia tipo (ST) se determinó por la técnica de multilocus sequence typing (MLST), basada en la amplificación y secuenciación de 7 genes housekeeping y comparación de estos alelos con la base de datos http://pubmlst.org/paeruginosa/.
Estudios de sensibilidad a antibióticosSe estudió la sensibilidad a 15agentes antipseudomónicos (ticarcilina, piperacilina, piperacilina-tazobactam [TZP], ceftazidima [CAZ], cefepima, aztreonam, imipenem [IPM], meropenem [MEM], doripenem [DOR], gentamicina, tobramicina, amikacina, ciprofloxacino, norfloxacino y colistina) mediante el método de difusión por disco, siguiendo las recomendaciones del Clinical and Laboratory Standards Institute (CLSI)10. Se determinó la concentración mínima inhibitoria (CMI) de los antibióticos IPM y MEM por el método de dilución en agar10, y de los antibióticos TZP y CAZ mediante tiras de test de CMI (Liofilchem, Italia; bioMérieux, Francia).
Los fenotipos betalactamasa de espectro extendido (BLEE), metalo-betalactamasa (MBL) y carbapenemasa de clase A fueron analizados por la técnica del test sinérgico de doble disco. Los discos empleados en cada caso fueron los siguientes: 1) cefepima-amoxicilina/ácido clavulánico-ceftazidima; 2) IPM-EDTA (0,5 M, pH8)-MEM, y 3) IPM-ácido 3-aminofenilborónico (300μg)-MEM. La hiperproducción de la betalactamasa AmpC se investigó en las cepas CAZ resistentes usando placas en presencia y ausencia de cloxacilina (250μg/ml) con tiras de E-test de CAZ (bioMérieux, Francia). Las cepas PAO1 y PAOΔdB se usaron como controles negativo y positivo, respectivamente. Se definió el aislado hiperproductor de AmpC cuando se observaba una diferencia de, al menos, una dilución doble entre la CMI de CAZ y CAZ con cloxacilina.
Análisis de los genes determinantes de metalo-betalactamasas y carbapenemasas de clase ALa presencia de genes codificantes de MBL (IMP, VIM, GIM, SIM y SPM) o de carbapenemasas de clase A (KPC, SME, IMI y GES) se llevó a cabo por PCR múltiples8.
Caracterización de la porina OprDLos cambios aminoacídicos en la proteína OprD se analizaron por PCR11,12, secuenciación y posterior comparación con la secuencia de la cepa de P. aeruginosa PAO1 (número de acceso en GenBank: AE004091) empleada como cepa de referencia. Se diseñaron nuevos cebadores de secuenciación para los aislados Ps92 y W64 en los que diferentes secuencias de inserción truncaban el gen oprD (tabla 1).
Cebadores empleados en el análisis del gen oprD
| Nombre del cebador | Secuencia 5′→ 3′ | Referencias |
|---|---|---|
| OprD-F | CGCCGACAAGAAGAACTAGC | 12 |
| OprD-R | GTCGATTACAGGATCGACAG | 12 |
| OprD-seq | GCCGACCACCGTCAAATCG | 12 |
| OprDlong-F | CTACGCAGATGCGACATGC | 11 |
| OprDlong-R | CCTTTATAGGCGCGTTGCC | 11 |
| ISPs92-Fa | TCTTGCTGATGTTGCGTAGC | En este estudio |
| ISPs92-Ra | CGCTGAGGTTATCGGTGATT | En este estudio |
| ISW64-Fa | GGCAGATTCCAGAGATGGTC | En este estudio |
| ISW64-Ra | AGCGGGATTGTGTAGTTGGT | En este estudio |
La detección de los genes codificantes de las integrasas de tipo 1, 2 o 3, así como la presencia del segmento 3’ conservado qacEΔ1+sul1 (característico de los integrones de clase 1) fueron estudiados por PCR. El polimorfismo de los promotores Pc y P2, y la caracterización de las regiones variables de los integrones de clase 1 se analizaron por PCR y secuenciación13.
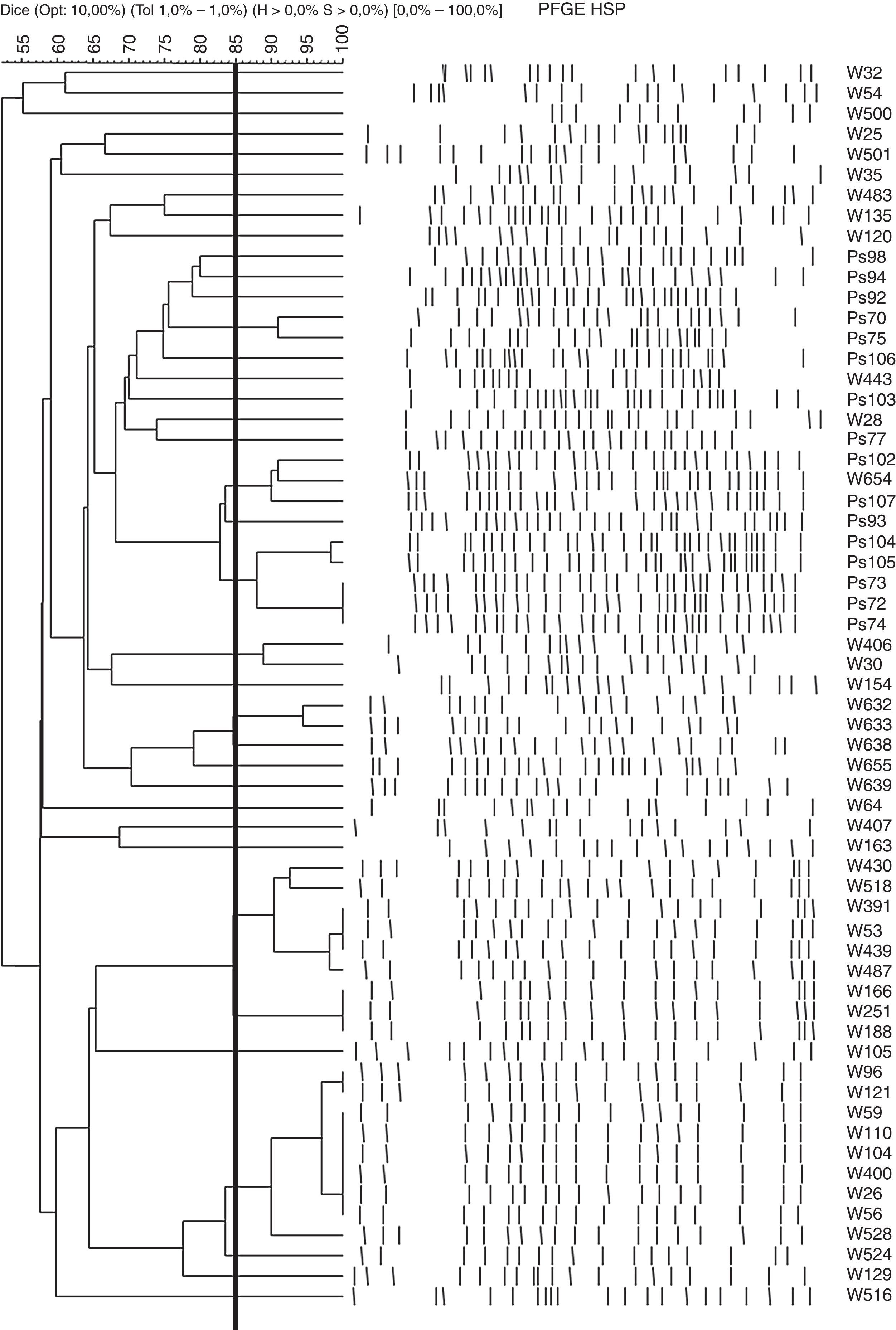
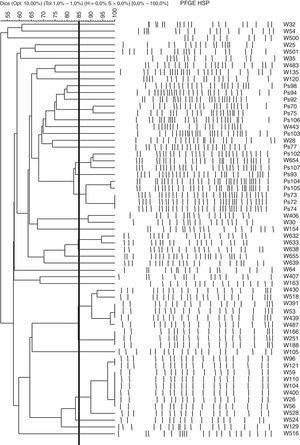
ResultadosRelación clonal de los aisladosSe estudió mediante PFGE la relación clonal de los 85PARC aislados de 60pacientes. Se observó que las cepas provenientes de un mismo paciente mostraban perfiles de PFGE indistinguibles, excepto en un paciente en el que las 2 cepas aisladas presentaron patrones diferentes. Para continuar el estudio, el criterio de selección fue caracterizar una cepa PARC por paciente (salvo la inclusión de las 2 cepas con distinto PFGE aisladas de un mismo paciente). Se seleccionaron 61cepas de los 60pacientes, entre las que se observaron 35patrones de bandas de PFGE diferentes. En la figura 1 se puede observar el dendrograma de PFGE obtenido con estas 61cepas.
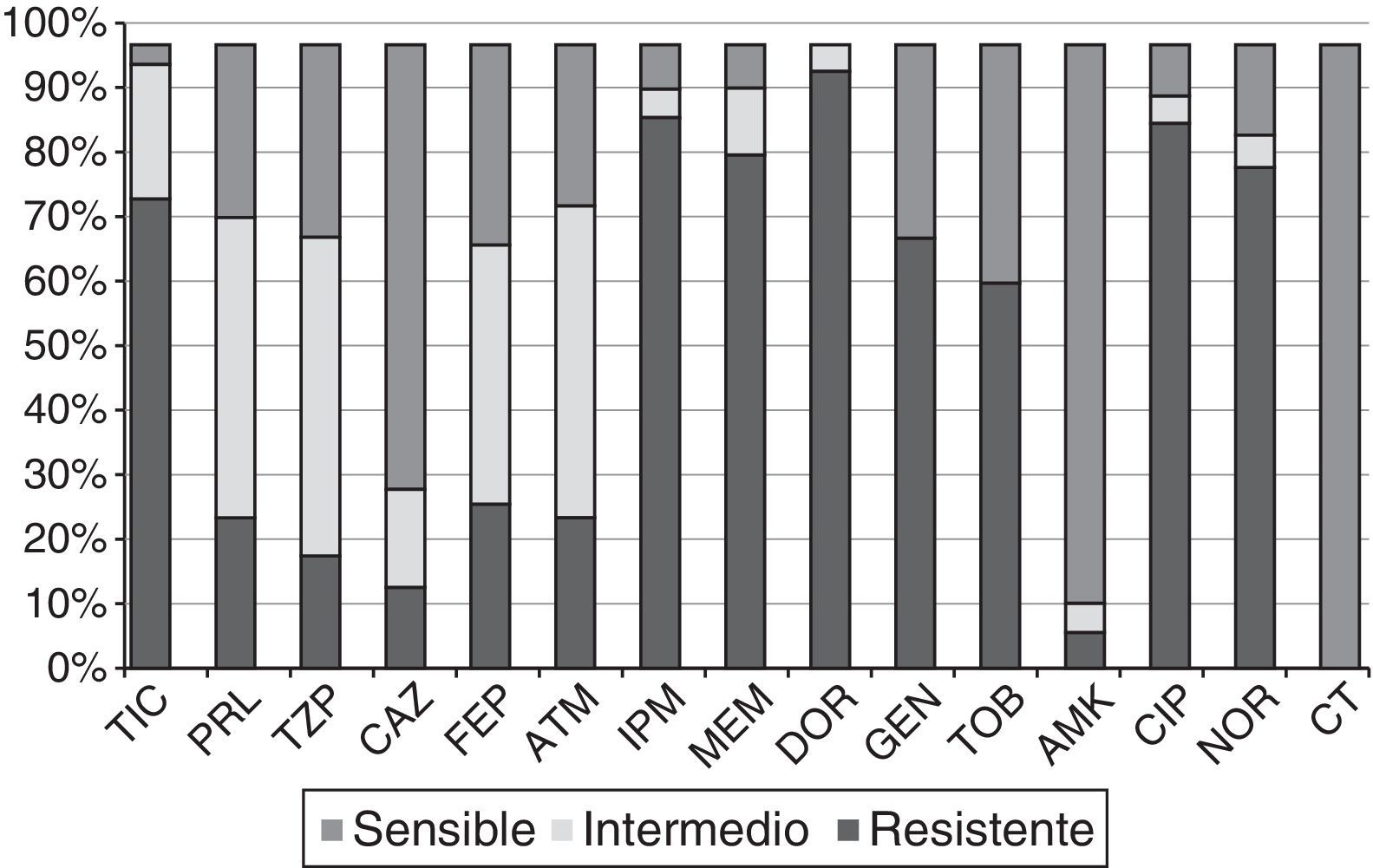
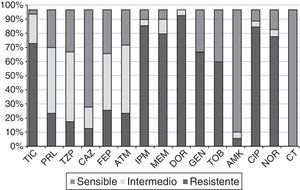
Perfiles de sensibilidad a antibióticosLa figura 2 muestra los porcentajes de resistencia a los 15antibióticos testados en las 61cepas seleccionadas. Todas mostraban un fenotipo de multirresistencia, según el criterio de Magiorakos et al.14. Los porcentajes de resistencia a IPM, MEM, CAZ y TZP fueron 88, 82, 15 y 20%, respectivamente, observándose unos rangos de resistencia según CMI de 8 a >64μg/ml para IMP; 8 a 128μg/ml para MEM; 32 a >256μg/ml para CAZ, y >256μg/ml para TZP entre las cepas resistentes a dichos antibióticos. Ninguna de las cepas presentó el fenotipo BLEE, MBL o carbapenemasas de clase A, ni fue resistente a colistina, y solo un único aislado resistente a CAZ (Ps75) mostró un fenotipo de hiperproducción de AmpC. Se observó corresistencia de los carbapenémicos (nuestro criterio de selección) con los aminoglucósidos y las fluoroquinolonas.
Porcentajes de resistencia de las 61cepas PARC del HSP frente a los 15antibióticos testados.
AMK: amikacina; ATM: aztreonam; CAZ: ceftazidima; CIP: ciprofloxacino; CT: colistina; DOR: doripenem; FEP: cefepime; GEN: gentamicina; HSP: Hospital San Pedro; IPM: imipenem; MEM: meropenem; NOR: norfloxacino; PRL: piperacilina; TIC: ticarcilina; TZP: piperacilina-tazobactam; TOB: tobramicina.
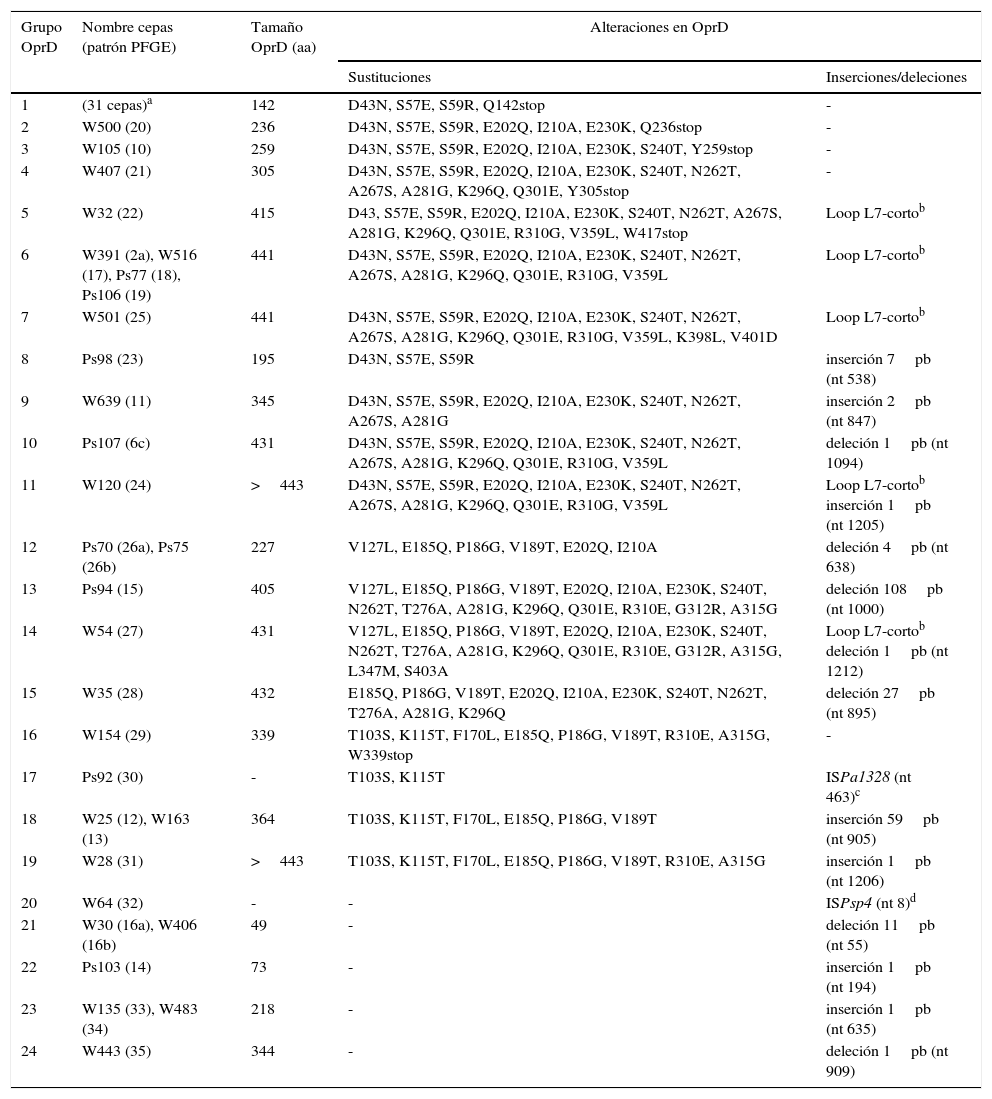
En la tabla 2 se muestran las alteraciones de la porina OprD encontradas en las 61PARC seleccionadas, observando que en todas ellas se detectaron una gran variedad de cambios aminoacídicos (sustituciones, presencia de inserciones, deleciones y codones de finalización prematuros). Cabe destacar que el 59% (36/61) de las cepas estudiadas presentaban un codón de finalización prematuro, y 31 de las cuales (pertenecientes a 9 patrones de PFGE diferentes) lo mostraban en el aminoácido 142 (sustitución Q142stop).
Alteraciones encontradas en la porina OprD de las 61cepas PARC seleccionadas
| Grupo OprD | Nombre cepas (patrón PFGE) | Tamaño OprD (aa) | Alteraciones en OprD | |
|---|---|---|---|---|
| Sustituciones | Inserciones/deleciones | |||
| 1 | (31 cepas)a | 142 | D43N, S57E, S59R, Q142stop | - |
| 2 | W500 (20) | 236 | D43N, S57E, S59R, E202Q, I210A, E230K, Q236stop | - |
| 3 | W105 (10) | 259 | D43N, S57E, S59R, E202Q, I210A, E230K, S240T, Y259stop | - |
| 4 | W407 (21) | 305 | D43N, S57E, S59R, E202Q, I210A, E230K, S240T, N262T, A267S, A281G, K296Q, Q301E, Y305stop | - |
| 5 | W32 (22) | 415 | D43, S57E, S59R, E202Q, I210A, E230K, S240T, N262T, A267S, A281G, K296Q, Q301E, R310G, V359L, W417stop | Loop L7-cortob |
| 6 | W391 (2a), W516 (17), Ps77 (18), Ps106 (19) | 441 | D43N, S57E, S59R, E202Q, I210A, E230K, S240T, N262T, A267S, A281G, K296Q, Q301E, R310G, V359L | Loop L7-cortob |
| 7 | W501 (25) | 441 | D43N, S57E, S59R, E202Q, I210A, E230K, S240T, N262T, A267S, A281G, K296Q, Q301E, R310G, V359L, K398L, V401D | Loop L7-cortob |
| 8 | Ps98 (23) | 195 | D43N, S57E, S59R | inserción 7pb (nt 538) |
| 9 | W639 (11) | 345 | D43N, S57E, S59R, E202Q, I210A, E230K, S240T, N262T, A267S, A281G | inserción 2pb (nt 847) |
| 10 | Ps107 (6c) | 431 | D43N, S57E, S59R, E202Q, I210A, E230K, S240T, N262T, A267S, A281G, K296Q, Q301E, R310G, V359L | deleción 1pb (nt 1094) |
| 11 | W120 (24) | >443 | D43N, S57E, S59R, E202Q, I210A, E230K, S240T, N262T, A267S, A281G, K296Q, Q301E, R310G, V359L | Loop L7-cortob inserción 1pb (nt 1205) |
| 12 | Ps70 (26a), Ps75 (26b) | 227 | V127L, E185Q, P186G, V189T, E202Q, I210A | deleción 4pb (nt 638) |
| 13 | Ps94 (15) | 405 | V127L, E185Q, P186G, V189T, E202Q, I210A, E230K, S240T, N262T, T276A, A281G, K296Q, Q301E, R310E, G312R, A315G | deleción 108pb (nt 1000) |
| 14 | W54 (27) | 431 | V127L, E185Q, P186G, V189T, E202Q, I210A, E230K, S240T, N262T, T276A, A281G, K296Q, Q301E, R310E, G312R, A315G, L347M, S403A | Loop L7-cortob deleción 1pb (nt 1212) |
| 15 | W35 (28) | 432 | E185Q, P186G, V189T, E202Q, I210A, E230K, S240T, N262T, T276A, A281G, K296Q | deleción 27pb (nt 895) |
| 16 | W154 (29) | 339 | T103S, K115T, F170L, E185Q, P186G, V189T, R310E, A315G, W339stop | - |
| 17 | Ps92 (30) | - | T103S, K115T | ISPa1328 (nt 463)c |
| 18 | W25 (12), W163 (13) | 364 | T103S, K115T, F170L, E185Q, P186G, V189T | inserción 59pb (nt 905) |
| 19 | W28 (31) | >443 | T103S, K115T, F170L, E185Q, P186G, V189T, R310E, A315G | inserción 1pb (nt 1206) |
| 20 | W64 (32) | - | - | ISPsp4 (nt 8)d |
| 21 | W30 (16a), W406 (16b) | 49 | - | deleción 11pb (nt 55) |
| 22 | Ps103 (14) | 73 | - | inserción 1pb (nt 194) |
| 23 | W135 (33), W483 (34) | 218 | - | inserción 1pb (nt 635) |
| 24 | W443 (35) | 344 | - | deleción 1pb (nt 909) |
aa: aminoácidos; nt: nucleótido; PARC: P. aeruginosa resistentes a carbapenémicos; PFGE: electroforesis en gel de campo pulsado.
Cepas (patrón PFGE): W26(1a), W56(1a), W59(1a), W104(1a), W110(1a), W400(1a), W96(1b), W121(1b), W528(1c), W524(1d), W53(2a), W439(2a), W487(2b), W166(2c), W188(2c), W251(2c), W518(2d), W430(2e), Ps104(3a), Ps105(3b), W632(4a), W633(4b), W638(4c), Ps72(5), Ps73(5), Ps74(5), W654(6a), Ps102(6b), W129(7), W655(8), Ps93(9). Todas estas cepas pertenecen a la línea clonal ST175.
Del mismo modo, se observaron diferentes inserciones/deleciones, tanto en tamaño (desde 1pb hasta >1.000pb) como en posición (desde el nucleótido 8 hasta el nucleótido 1212), destacando las cepas Ps92 y W64 que presentaban el gen oprD interrumpido por las secuencias de inserción ISPa1328 e ISPsp4, respectivamente. ISPa1328 pertenece a la familia de IS256 e ISPsp4 a la familia de IS30. Las secuencias del gen oprD en las cepas Ps92 y W64 fueron nuevas y por ello se introdujeron en la base de genes GenBank, cuyos números de acceso fueron KF517097 y KF517098, respectivamente.
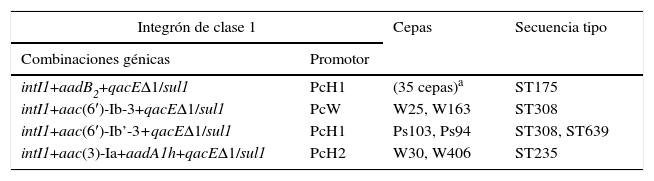
Caracterización de integrones y secuencias tipoEl 67% (41/61) de las cepas estudiadas amplificaron el gen codificante de la integrasa de tipo 1 (intI1) y el segmento 3′ conservado (qacEΔ1+sul1), mientras que todas ellas fueron negativas para las integrasas de tipo 2 y 3. Todas las cepas intI1-positivas incluían genes codificantes de enzimas modificantes de aminoglucósidos en sus regiones variables. Es importante resaltar que ninguna de nuestras cepas presentó 2 o más integrones simultáneamente (tabla 3).
Integrones de clase 1 y secuencias tipo detectadas en las 41 cepas intI1-positivas
| Integrón de clase 1 | Cepas | Secuencia tipo | |
|---|---|---|---|
| Combinaciones génicas | Promotor | ||
| intI1+aadB2+qacEΔ1/sul1 | PcH1 | (35 cepas)a | ST175 |
| intI1+aac(6′)-Ib-3+qacEΔ1/sul1 | PcW | W25, W163 | ST308 |
| intI1+aac(6′)-Ib’-3+qacEΔ1/sul1 | PcH1 | Ps103, Ps94 | ST308, ST639 |
| intI1+aac(3)-Ia+aadA1h+qacEΔ1/sul1 | PcH2 | W30, W406 | ST235 |
El casete génico más prevalente fue aadB2 regulado por un promotor híbrido tipo 1 (PcH1), presente en 35 de las cepas intI1-positivas (85%). Este casete génico es idéntico al descrito en la base de datos GenBank con el número de acceso AJ871915; ambos presentan 4mutaciones aminoacídicas respecto al casete génico de aadB (número de acceso GenBank: L06418): K11Q, I60M, M69T y V142M.
Además, se encontraron otras estructuras génicas (número de cepas): aac(6′)-Ib-3 (2), aac(6′)-Ib’-3 (2) y la combinación aac(3)-Ia+aadA1h (2). La diferencia existente entre aac(6′)-Ib-3 y aac(6′)-Ib’-3 es una mutación en la posición 102 (L102S), mientras que ambos genes presentaban la mutación N5T respecto del gen aac(6′)-Ib (número de acceso GenBank: AF034958). Los promotores asociados en estos casos fueron promotor débil (PcW) para el casete génico aac(6′)-Ib-3, mientras que en el caso de aac(6′)-Ib’-3 se observaba el promotor PcH1. Por último, la combinación aac(3)-Ia+aadA1h estaba asociada a un promotor híbrido de tipo 2 (PcH2). Este integrón, descrito por primera vez en este estudio y encontrado en 2 cepas (W30 y W406) de diferentes pacientes y aisladas en 2 años distintos (2008 y 2009, respectivamente), fue depositado en la base de genes GenBank con el número de acceso GQ144317 y fue denominado como In272 en la base de datos de INTEGRALL (http://integrall.bio.ua.pt/).
Por otro lado, se analizó la ST en todas las cepas portadoras de integrón. Entre las 41cepas se detectaron únicamente 4 ST diferentes (número de cepas): ST175 (35), ST308 (3), ST235 (2) y ST639 (1). En la tabla 3 se puede ver la asociación de la ST con el integrón detectado en las cepas portadoras de estos. Todas las cepas portadoras del integrón con el casete génico aadB2 (11 patrones de PFGE diferentes) pertenecieron a la ST175, destacando además que en este grupo se incluyen las 31cepas que presentaban la mutación Q142stop en la porina OprD. Mientras que la ST308 se encontró asociada a cepas portadoras de diferentes tipos de integrones —aac(6′)-Ib-3 y aac(6′)-Ib’-3 e, incluso, uno de ellos relacionado también con la ST639. ST308 y ST639 no están relacionados, puesto que la combinación alélica de ambos ST es totalmente diferente.
DiscusiónP. aeruginosa es uno de los patógenos más problemáticos en el ámbito clínico y el aumento en la resistencia a carbapenémicos en esta especie es de gran preocupación a nivel mundial debido a las limitaciones que supone en su tratamiento clínico. Existen múltiples publicaciones sobre la presencia de PARC, incluso en forma de brotes epidémicos, en las UCI de diferentes hospitales15,16. Sin embargo, en nuestro trabajo solo el 5% procedían de UCI o medicina intensiva. Por otro lado, a pesar de que las 61PARC seleccionadas mostraban un fenotipo de multirresistencia, ninguna de ellas mostraba el fenotipo BLEE, MBL o carbapenemasas de clase A. Existen multitud de artículos alertando en los últimos años del problema emergente de la diseminación de PARC portadoras de carbapenemasas7,17. Sin embargo, la prevalencia de PARC productoras de MBL en España es todavía relativamente baja comparada con la de otros países como Italia o Brasil5-8,18. De hecho, al igual que se observa en nuestro trabajo, la presencia de alteraciones en OprD es el mecanismo más frecuentemente implicado en la resistencia a imipenem6,16,19. Entre la gran cantidad de alteraciones en OprD de las 61cepas analizadas, la presencia de codones de finalización prematuros encontradas en 36 de las cepas estudiadas o el truncamiento de oprD por la inclusión de secuencias o un elemento de inserción, inactivan OprD relacionándolo directamente con la resistencia a imipenem8,12,19. La alteración en OprD más frecuentemente detectada entre nuestras cepas es Q142stop, asociada con cepas adscritas a la línea clonal ST175. Esta mutación en OprD se ha observado en cepas multirresistentes de la línea clonal ST175 diseminadas en otros hospitales españoles20. Dos cepas (Ps92 y W64) tenían truncado el gen oprD por las secuencias de inserción ISPa1328 e ISPsp4, respectivamente. Wolter et al.11 describieron por primera vez la inactivación de la expresión del gen oprD por la presencia de secuencias de inserción (ISPa1328 e ISPa1635), lo que permitía a sus cepas un incremento de la resistencia a los carbapenémicos. En los últimos años, se ha incrementado el número de trabajos que describen la inclusión de secuencias de inserción (ISPa27, ISPa45, ISPa46, ISPa47, ISPa133, ISPa1328, ISPa1635, ISPst12 o ISPpu21, entre otras) impidiendo la funcionalidad de OprD, aunque no existe una posición concreta donde se introducen estas secuencias de inserción interrumpiendo la lectura del gen, así como tampoco su direccionalidad o la familia de IS a la que pertenecen8,12,19,21-23.
Por otra parte, no todas las alteraciones encontradas en nuestras cepas clínicas están implicadas en la manifestación de resistencia a imipenem y/o meropenem. Existen cambios aminoacídicos (como los observados en los patrones de OprD 6 y 7 en este trabajo), que se han descrito tanto en cepas resistentes como en cepas sensibles a carbapenémicos8,22,24. Del mismo modo, el acortamiento del Loop-7 en 2 aminoácidos es una alteración que no implica directamente la resistencia a carbapenémicos porque la porina sigue mostrando una apertura suficiente que no interrumpe la penetración óptima del antibiótico25. Otros mecanismos cromosómicos como la hiperproducción de bombas de expulsión activa o de AmpC podrían estar implicados en la resistencia a carbapenémicos1,6.
Como ocurre en nuestro trabajo, en cepas clínicas de P. aeruginosa se ha observado una asociación de resistencia entre betalactámicos, aminoglucósidos y fluoroquinolonas2,26, lo que dificulta la elección adecuada del tratamiento antibiótico para erradicar esta especie en el ámbito hospitalario1,27. En cuanto a los valores de resistencia a otros antibióticos no betalactámicos, se debe destacar la alta resistencia a aminoglucósidos encontrada entre nuestras cepas (>60% para gentamicina y tobramicina). La mayoría de las cepas resistentes a aminoglucósidos portaban integrones de clase 1, que a su vez albergaban en su región variable algún gen codificante de enzimas modificantes de aminoglucósidos. También se ha descrito en otros trabajos que en P. aeruginosa los principales casetes génicos incluidos en integrones de clase 1 confieren resistencia a betalactámicos y/o a aminoglucósidos8,28.
La variante del casete génico aadB (aadB2) se detectó en el 85% de las cepas integrón-positivas y todas ellas adscritas al clon ST175. Esta asociación ya había sido descrita por Nemec et al.29 en la República Checa y por trabajos previos de nuestro grupo de investigación8. En los últimos años, la diseminación de clones de alto riesgo en la especie P. aeruginosa ha desempeñado un papel primordial en la expansión de la resistencia a nivel mundial27. Entre los clones de alto riesgo diseminados por los hospitales de todo el mundo, las secuencias tipo ST235, ST111 y ST175 son las más frecuentes, normalmente asociadas con cepas multirresistentes, portadoras de integrones de clase 1, resistentes a carbapenémicos y/o productoras de MBL6,8,20,30,31.
En resumen, no se han detectado carbapenemasas entre las cepas de PARC aisladas de muestras clínicas en el hospital de estudio. Sin embargo, el alto polimorfismo observado en el gen oprD, así como la presencia de elementos de inserción truncándolo, inactivarían la porina OprD relacionándose directamente con la resistencia a carbapenémicos entre nuestras cepas. Asimismo, se han detectado cepas multirresistentes que, albergando integrones de clase 1 con casetes génicos implicados en la resistencia a aminoglucósidos, pertenecían a clones epidémicos de alto riesgo (ST175 y ST235). El fenotipo de multirresistencia, el alto polimorfismo de oprD, el alto porcentaje de cepas portadoras de integrones, la moderada relación clonal de las cepas y la elevada diseminación epidémica de clones de alto riesgo son aspectos de gran preocupación que requieren un control y seguimiento que eviten la diseminación de estas PARC.
FinanciaciónEste trabajo ha sido financiado en parte por el Instituto de Salud Carlos III (proyecto FIS PI12/01276). Vanesa Estepa disfrutó de una beca predoctoral de la Universidad de La Rioja (España) durante la realización del estudio experimental de este trabajo.
Conflicto de interesesLos autores declaran no tener conflictos de intereses.
Parte de este trabajo fue presentado en el XIV Congreso de la Sociedad Española de Enfermedades Infecciosas y Microbiología Clínica (abstract 720, Barcelona, España, 19-22 mayo 2010).