La espectrometría de masas Matrix-Assisted Laser Desorption-Ionization Time-of-Flight (MALDI-TOF) permite la identificación rápida de los microorganismos causantes de bacteriemia. Se requieren métodos fiables y rápidos que permitan acortar el tiempo necesario hasta disponer de los resultados de sensibilidad a antibióticos de los aislados de hemocultivos.
MétodosSe evalúa la fiabilidad de un método que combina la identificación con MALDI-TOF y el estudio de sensibilidad en paneles de microdilución inoculados a partir de un subcultivo incubado durante solo 4h.
ResultadosLa concordancia de los resultados de sensibilidad a antibióticos de la técnica evaluada frente a la técnica de referencia fue del 99,3%, sin que se observaran errores máximos.
ConclusiónLa inoculación de paneles de microdilución a partir de un subcultivo de solo 4h de incubación es un método fiable y fácil de realizar que permite acortar el tiempo de informe de hemocultivos positivos.
Mass spectrometry Matrix-Assisted Laser Desorption-Ionization Time-of-Flight (MALDI-TOF) helps in the rapid identification of microorganisms causing blood stream infection. Rapid and reliable methods are required to decrease the turnaround time for reporting antimicrobial susceptibility results from blood culture isolates.
MethodsAn evaluation was performed on the reliability of a method for antimicrobial susceptibility testing of positive blood culture isolates from briefly incubated solid medium cultures.
ResultsThe agreement between the evaluated and standard methods was 99.3%. The major and minor error rates were 0.4% and 0.3%, respectively, and no very major errors were observed.
ConclusionThe inoculation of briefly incubated solid medium cultures into antimicrobial susceptibility testing panels is an easy and reliable technique, and helps to decrease the turnaround time for reporting antimicrobial susceptibility results of positive blood cultures.
La infección del torrente sanguíneo es una causa importante de morbimortalidad1. El hemocultivo continúa siendo la técnica de referencia para el diagnóstico etiológico de bacteriemia, permitiendo además el estudio de sensibilidad de los aislados2. La rápida información de estos resultados permite la instauración precoz de un tratamiento antibiótico adecuado que mejora el pronóstico del paciente séptico3,4.
La espectrometría de masas Matrix-Assisted Laser Desorption-Ionization Time-of-Flight (MALDI-TOF) es una técnica rápida y fiable para la identificación bacteriana5,6. Su realización directamente a partir de hemocultivos positivos reduce considerablemente el tiempo de obtención de resultados7,8. Se han utilizado estrategias que combinan la identificación con MALDI-TOF y la inoculación de paneles de antibiograma directamente del frasco del hemocultivo positivo; sin embargo, los protocolos publicados incluyen diferentes pasos de centrifugación y lavado que resultan laboriosos para ser implementados en la rutina del laboratorio8-11. Se han comunicado buenos resultados combinando la identificación con MALDI-TOF con la inoculación de paneles de microdilución a partir de subcultivos incubados durante cortos periodos de tiempo12,13.
Con el objetivo de disminuir el tiempo de informe de los hemocultivos positivos, en el presente trabajo evaluamos la fiabilidad de los resultados de sensibilidad obtenidos con paneles del sistema WIDER (Francisco Soria Melguizo, Madrid, España) inoculados a partir de un subcultivo de 4h de incubación.
MétodosDurante un periodo de 3 meses (enero-marzo 2015) se han estudiado 138 hemocultivos consecutivos positivos correspondientes a 138 pacientes diferentes. Los hemocultivos fueron procesados con el sistema BD BACTEC™ FX (Becton Dickinson, Maryland. EE.UU.). A los frascos positivos se le realizó tinción de Gram y subcultivo en agar chocolate (37°C, 5%CO2).
Después de 4h de incubación, se realizó un barrido del crecimiento en agar chocolate con una espátula de madera y se realizó la identificación con el sistema MALDI-TOF (Bruker, MALDI Biotyper 3.0) mediante la técnica de trasferencia directa a placa con ácido fórmico6,8,12. Se consideró aceptable un score >1,7.
Cuando la identificación tuvo un score aceptable, se determinó la concentración mínima inhibitoria (CMI) por técnica de microdilución en paneles WIDER MIC/IDGP y MIC/IDGN para bacterias grampositivas y gramnegativas, respectivamente. Para la inoculación de los paneles se preparó una suspensión bacteriana a partir del subcultivo de 4h. Se utilizó el sistema de inoculación Prompt™ Inoculation System Wands, siguiendo instrucciones del fabricante. Para comprobar la pureza del inóculo se realizó siembra en agar sangre de 10μl de la suspensión bacteriana. Los paneles fueron incubados a 37°C, realizándose la lectura después de 14-18h de incubación.
En paralelo, tras 24h de incubación, a partir de las colonias aisladas en el medio agar chocolate se realizó la inoculación estandarizada de los aislados en los mismos paneles WIDER. Los resultados obtenidos con esta técnica se han considerado como referencia para evaluar los obtenidos a partir del subcultivo de 4h.
Para comparar los resultados, las CMI obtenidas se han transformado en categorías clínicas (sensible, intermedio, o resistente) de acuerdo con el sistema experto de WIDER que aplica los criterios del Clinical Laboratory Standard Institute (CLSI)14. Los resultados obtenidos con la técnica evaluada con respecto a la técnica estándar se han clasificado como concordantes (interpretación idéntica en ambos métodos), errores máximos (falsa sensibilidad), errores mayores (falsa resistencia) y errores menores (sensible/resistente vs. intermedio).
Los paneles WIDER empleados permiten la identificación del aislado; sin embargo, la fiabilidad de MALDI-TOF como técnica de identificación ha sido suficientemente evaluada y nuestro objetivo fue únicamente evaluar la fiabilidad de los resultados de sensibilidad obtenidos a partir del subcultivo de 4h de incubación. En este sentido, no se han evaluado discrepancias en la identificación entre los sistemas MALDI-TOF y WIDER. Cuando se encontró alguna discrepancia en la identificación, se introdujo manualmente en el sistema la identificación obtenida con MALDI-TOF.
ResultadosDe los 138 hemocultivos estudiados, 19 fueron excluidos. En 9 aislados (2 Bacteroidesspp., un Streptococcus pneumoniae, 3 estafilococos coagulasa negativa, un Escherichia coli y 2 cultivos polimicrobianos) no se consiguió la identificación con MALDI-TOF (score <1,7) después de 4h de incubación. Otros 6 aislados (3 S.pneumoniae, un Streptococcus agalactiae, un Streptococcus pyogenes y un Listeria monocytogenes), aunque fueron correctamente identificados, fueron excluidos porque en la rutina del laboratorio no se realiza estudio de sensibilidad por técnica de microdilución a estos patógenos. Otros 4 casos en los que se observaron levaduras en la tinción de Gram también fueron excluidos.
En 119 hemocultivos se obtuvo una identificación aceptable (score >1,7) con MALDI-TOF y se realizó CMI a partir del subcultivo incubado durante 4h: 98 (83%) fueron bacilos gramnegativos (BGN) y 21 (17%) cocos grampositivos (CGP) (tabla 1).
Agentes causales de bacteriemia identificados mediante MALDI-TOF (score>1.7)
| Etiología | Microrganismos, n (%) |
|---|---|
| Cocos grampositivos | |
| Staphylococcus aureus | 9 (42,8) |
| Staphylococcus epidermidis | 5 (23,8) |
| Enterococcus faecalis | 5 (23,8) |
| Enterococcus faecium | 2 (9,5) |
| Total | 21 (100) |
| Bacilos gramnegativos | |
| Escherichia coli | 51 (52,0) |
| Klebsiella pneumoniae | 14 (14,3) |
| Pseudomonas aeruginosa | 8 (8,1) |
| Enterobacter aerogenes | 6 (6,1) |
| Enterobacter cloacae | 5 (5,1) |
| Klebsiella oxytoca | 4 (4,0) |
| Proteus mirabilis | 3 (3,0) |
| Acinetobacter baumannii | 1 (1,0) |
| Aeromonas veronii | 1 (1,0) |
| Citrobacter freundii | 1 (1,0) |
| Citrobacter koseri | 1 (1,0) |
| Proteus vulgaris | 1 (1,0) |
| Kluyvera ascorbata | 1 (1,0) |
| Serratia marcescens | 1 (1,0) |
| Total | 98 (100) |
Solo en 3 casos (un Citrobacter koseri, un Enterobacter cloacae y un Enterobacter sakazaki) se observaron discrepancias en la identificación de especie entre los 2 sistemas empleados.
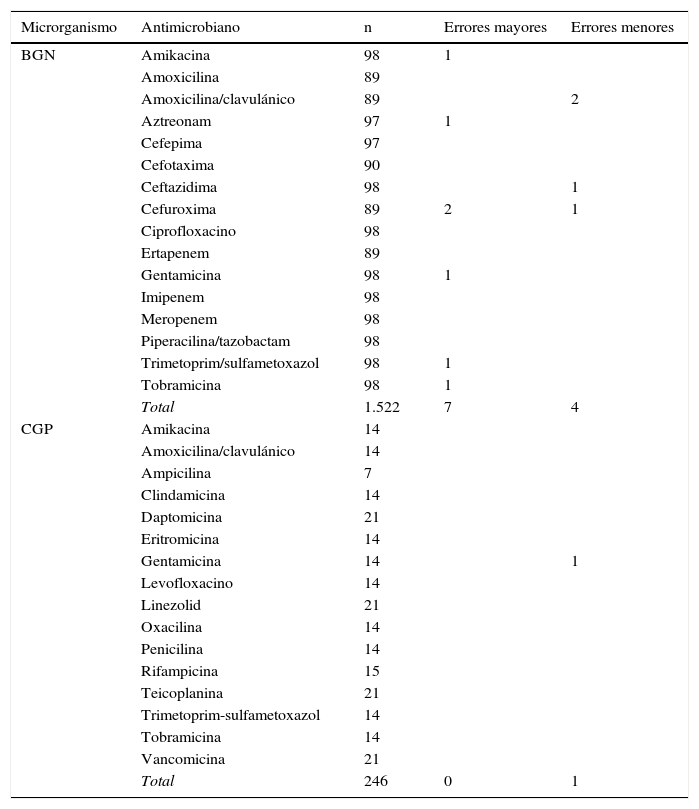
Para BGN se han evaluado un total de 1.522 combinaciones de antibióticos/microorganismos. Para CGP, el total de combinaciones estudiadas ha sido de 246. La concordancia entre los resultados de sensibilidad obtenidos con la técnica estándar y la técnica evaluada fue del 99,3%; la tasa de errores mayores y menores fue del 0,4 y del 0,3%, respectivamente, sin que se observaran errores máximos (tabla 2). No se observaron diferencias en las tasas de error entre las diferentes especies bacterianas incluidas. No se detectó ninguna cepa productora de carbapenemasa, y entre 3 cepas de E.coli productoras de betalactamasa de espectro extendido y una cepa de S.aureus resistente a meticilina hubo una concordancia total con el método de referencia.
Correlación entre los resultados del estudio de sensibilidad obtenidos con la técnica estándar y la técnica evaluada
| Microrganismo | Antimicrobiano | n | Errores mayores | Errores menores |
|---|---|---|---|---|
| BGN | Amikacina | 98 | 1 | |
| Amoxicilina | 89 | |||
| Amoxicilina/clavulánico | 89 | 2 | ||
| Aztreonam | 97 | 1 | ||
| Cefepima | 97 | |||
| Cefotaxima | 90 | |||
| Ceftazidima | 98 | 1 | ||
| Cefuroxima | 89 | 2 | 1 | |
| Ciprofloxacino | 98 | |||
| Ertapenem | 89 | |||
| Gentamicina | 98 | 1 | ||
| Imipenem | 98 | |||
| Meropenem | 98 | |||
| Piperacilina/tazobactam | 98 | |||
| Trimetoprim/sulfametoxazol | 98 | 1 | ||
| Tobramicina | 98 | 1 | ||
| Total | 1.522 | 7 | 4 | |
| CGP | Amikacina | 14 | ||
| Amoxicilina/clavulánico | 14 | |||
| Ampicilina | 7 | |||
| Clindamicina | 14 | |||
| Daptomicina | 21 | |||
| Eritromicina | 14 | |||
| Gentamicina | 14 | 1 | ||
| Levofloxacino | 14 | |||
| Linezolid | 21 | |||
| Oxacilina | 14 | |||
| Penicilina | 14 | |||
| Rifampicina | 15 | |||
| Teicoplanina | 21 | |||
| Trimetoprim-sulfametoxazol | 14 | |||
| Tobramicina | 14 | |||
| Vancomicina | 21 | |||
| Total | 246 | 0 | 1 |
BGN: bacilos gramnegativos; CGP: cocos grampositivos; n: número de combinaciones de antibiótico/microorganismo probadas.
El informe de identificación y sensibilidad se emitió en los 119 casos estudiados en menos de 24h desde la detección del hemocultivo positivo.
DiscusiónLa técnica de identificación directa con MALDI-TOF a partir de hemocultivos positivos ha demostrado ser eficaz y fiable; sin embargo, el procesamiento previo de la muestra resulta bastante laborioso. Aun en detrimento de la rapidez, para organizar mejor el flujo de trabajo, algunos autores recomiendan agrupar los hemocultivos positivos y realizar la identificación en bloques 2 veces al día15. En este sentido, la identificación a partir de un subcultivo de 4h puede obtenerse en un tiempo similar con la ventaja de no necesitar un procesamiento previo de la muestra. En nuestra serie, solo en 9 (6%) de los 138 hemocultivos incluidos inicialmente en el estudio no se consiguió una identificación aceptable. En la serie de Hoyos-Mallecot et al.8 realizando identificación directa desde el hemocultivo, con un score de 1,7, el 4% de los BGN y el 26% de bacterias grampositivas no fueron identificados. Es posible que la muestra obtenida a partir de un subcultivo contenga mayor cantidad de proteínas que permiten obtener perfiles más fiables, aumentando la sensibilidad de la técnica.
Las estrategias que combinan la identificación con MALDI-TOF y la inoculación de paneles de CMI directamente del frasco de hemocultivo permiten acelerar el tiempo de emisión de informes, y en general han demostrado buenos resultados; sin embargo, la mayoría de los métodos publicados requieren un pretratamiento de la muestra. Machen et al.10, realizando un procesamiento previo de lisis-filtración, obtienen, con el sistema Vitek-2, una tasa global de error del 5,5%, con un 1,3% de errores máximos. Wimmer et al.11, después de un primer paso de centrifugación de la muestra, con el sistema Phoenix, encuentran una tasa de error próxima al 2%, con un 0,26% de errores máximos.
Trabajos previos han demostrado que la identificación obtenida con MALDI-TOF a partir del barrido de un subcultivo de muy pocas horas de incubación es concordante con la obtenida con la técnica de identificación recomendada a partir de subcultivos de 18-24h11,12. Idelevich et al.12, en una serie de 104 aislados de hemocultivos, han evaluado la inoculación de paneles de CMI del sistema Vitek a partir del crecimiento en subcultivo de 2,5 a 7h, y encuentran que el tiempo medio de incubación para CGP era de 3,8h y para BGN, de 2,4h. En base a estos resultados nosotros establecimos el tiempo de incubación en 4h, y empleando por primera vez paneles de CMI del sistema WIDER conseguimos identificar el 93,5% de los aislados estudiados. Para los 119 aislados a los que se realizó CMI tras 4h de incubación obtuvimos una concordancia con la técnica de referencia del 99,3%, sin observar errores máximos. Además, el método que evaluamos tiene la ventaja de no requerir ningún procesamiento previo de la muestra, por lo que no conlleva costes adicionales y puede adaptarse fácilmente al flujo de trabajo del laboratorio.
Entre las limitaciones de la técnica se encuentran las bacteriemias por anaerobios, en las que no se consigue crecimiento suficiente al cabo de 4h de incubación, y las bacteriemias polimicrobianas. Otra limitación del estudio sería el bajo número de grampositivos incluidos en esta serie.
En conclusión, la inoculación de los paneles de CMI a partir de un subcultivo incubado durante 4h, previa identificación con MALDI-TOF, es un método fiable comparado con el sistema convencional de inoculación tras 24h de incubación y permite acortar el tiempo de informe de hemocultivos positivos.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.