Con el objetivo de estudiar la evolución del brote por Enterococcus faecalis ST6 genotipo vanB2 descrito en 2009-2010 en 3 hospitales de Zaragoza, se caracterizaron todos los aislados clínicos E.faecalis resistentes a vancomicina obtenidos entre 2011 y 2013 en dichos hospitales.
MétodosCaracterización molecular de los aislados y estudio de su relación clonal por electroforesis en campos pulsados. Revisión de las historias clínicas de los pacientes.
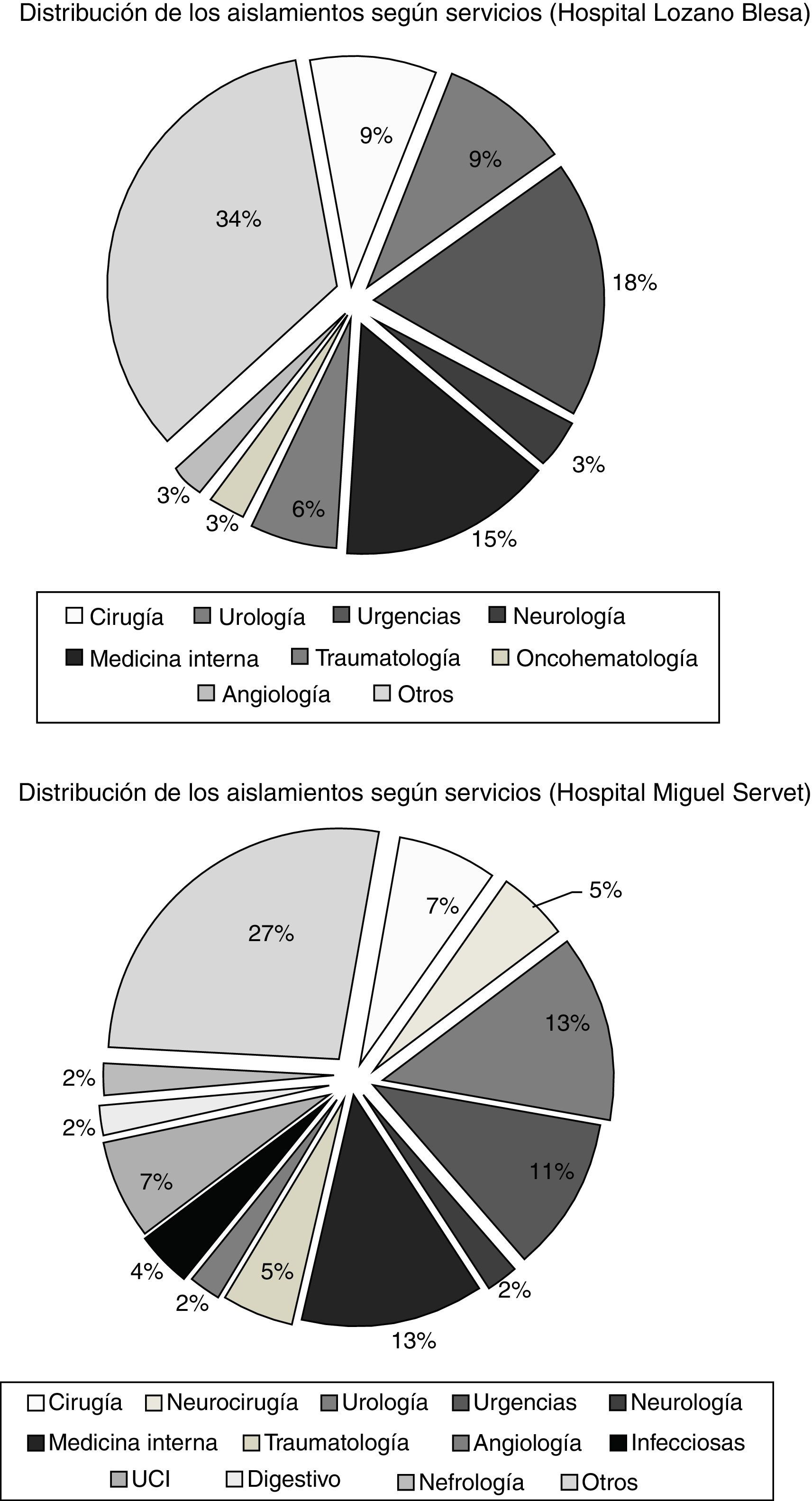
ResultadosSe detectaron 79 aislados E.faecalis genotipo vanB2 de 73 pacientes de 2 de los 3 hospitales analizados, la mayoría de origen urinario. El 46,5% de los casos fueron nosocomiales. La distribución según servicios hospitalarios mostró gran variabilidad, no pudiéndose identificar una fuente de infección común. Todas las cepas fueron multirresistentes (vancomicina, eritromicina, tetraciclina, ciprofloxacino, estreptomicina, gentamicina, kanamicina) y pertenecieron al clon ST6. El 93,7% eran indistinguibles al clon del inicio del brote o subtipos estrechamente relacionados.
ConclusiónEl brote se mantiene constante en los 3 años posteriores a su descripción, lo que señala la necesidad de mantener un control activo que limite la emergencia y diseminación de clones resistentes a vancomicina.
In order to study the evolution of the outbreak that occurred between 2009 and 2010 in 3 hospitals in Zaragoza, all vancomycin-resistant clinical Enterococcus faecalis isolates identified between 2011 and 2013 at these hospitals were characterised.
MethodsMolecular characterisation of the isolates and analysis of their clonal relationships was performed using pulsed field electrophoresis, along with a retrospective review of the patient records.
ResultsA total of 79 vancomycin-resistant E.faecalis isolates with genotype vanB2 of 73 patients were recovered in 2 of the 3 hospitals, most of them from urine specimens. About 46% of the cases were nosocomial. Distribution of the isolates among hospital services demonstrated high variability, making it difficult to predict a common source of infection. All the strains were multiresistant (vancomycin, erythromycin, tetracycline, ciprofloxacin, streptomycin, gentamicin, kanamycin) and belonged to lineage ST6. Seventy-four isolates (93.7%) were identical or closely related to the dominant one in the origin of the outbreak.
ConclusionThe outbreak remains constant over three years after being initially described, indicating the need to implement an active control in order to limit the emergence and spread of vancomycin-resistant clones.
Enterococcus faecalis y Enterococcus faecium constituyen la tercera-cuarta etiología más frecuente de infección nosocomial1. En los años ochenta se describieron en Europa las primeras cepas de Enterococcus resistentes a vancomicina (EVR), y desde entonces los brotes hospitalarios fundamentalmente asociados a E.faecium han ido en aumento. El aislamiento de E.faecalis resistente a vancomicina (EFRV) continúa siendo infrecuente. La resistencia adquirida a glucopéptidos está mediada por diversos mecanismos, entre los cuales vanA y vanB son los más frecuentes. La adquisición de estas resistencias dificulta el tratamiento de las infecciones por EVR, ya que las alternativas terapéuticas están aprobadas para indicaciones clínicas específicas y, frente a ellas, también se han descrito resistencias2.
España es uno de los países europeos con menor tasa de EVR, si bien se han publicado brotes nosocomiales esporádicos3-5, la mayoría asociados a E.faecium ST17. El único brote por E.faecalis vanB2 publicado hasta la fecha en España se detectó entre 2009 y 2010 en 3 hospitales de Zaragoza6. Posteriormente, entre 2011 y 2013 se identificaron en 2 de dichos hospitales 79 aislados con similares características. Nuestro objetivo fue caracterizar estos aislados para determinar si pertenecían al mismo brote y analizar las características y la evolución del mismo.
MétodosDescripción del brote y aislamientos bacterianosTras el inicio del brote por EFRV con genotipo vanB2 en noviembre de 2009 y su exhaustivo estudio hasta 20106, se llevó a cabo un seguimiento posterior mediante selección y caracterización de todos los aislados EFRV resistentes a vancomicina obtenidos en el Hospital Universitario Miguel Servet (HUMS), el Hospital Clínico Universitario Lozano Blesa (HCULB) y el Hospital Royo Villanova (HRV) entre enero de 2011 y diciembre de 2013. Los aislados procedían de muestras clínicas recibidas en el Laboratorio de Microbiología para el diagnóstico de la infección.
Se revisaron las historias clínicas de los pacientes, analizándose variables microbiológicas, demográficas y clínicas: edad, sexo, origen de la infección, patología de base, tratamiento antibiótico previo, tratamiento del proceso, evolución del paciente. En el caso de cepas del mismo paciente, se incluyeron solo las procedentes de muestras de distinto origen o diferente patrón de electroforesis en campo pulsado (ECP).
Identificación y antibiogramaEl fenotipo de resistencia se determinó por métodos de difusión en disco y microdilución en caldo (MicroScan, Siemens). Se estudió la sensibilidad frente a ampicilina, clindamicina, eritromicina, cloranfenicol, ciprofloxacino, teicoplanina, vancomicina, tetraciclina, quinupristina-dalfopristina, trimetoprim-sulfametoxazol, gentamicina, estreptomicina, kanamicina, linezolid y daptomicina siguiendo los criterios EUCAST7. La producción de betalactamasa no fue evaluada. La concentración mínima inhibitoria de vancomicina, teicoplanina y daptomicina se determinó por el método del Etest (Biomérieux). Los genes de resistencia a vancomicina (vanA, vanB, vanC-1, vanC-2/3 y vanD) se analizaron por PCR en todos los aislamientos y el gen vanB2 fue secuenciado en una cepa seleccionada por cada perfil de ECP6,8.
Tipificación molecularLa relación clonal de los EFRV vanB2 se analizó mediante digestión del ADN cromosómico (SmaI) y posterior separación de los fragmentos mediante ECP. Los perfiles genéticos se compararon siguiendo los criterios de Tenover9.
Se seleccionó un aislamiento por cada patrón de ECP, y estos fueron estudiados por multilocus sequence typing (MLST), amplificando y secuenciando los fragmentos de los genes gdh, gyd, pstS, gki, aroE, xpt e yqiL (http://efaecalis.mlst.net).
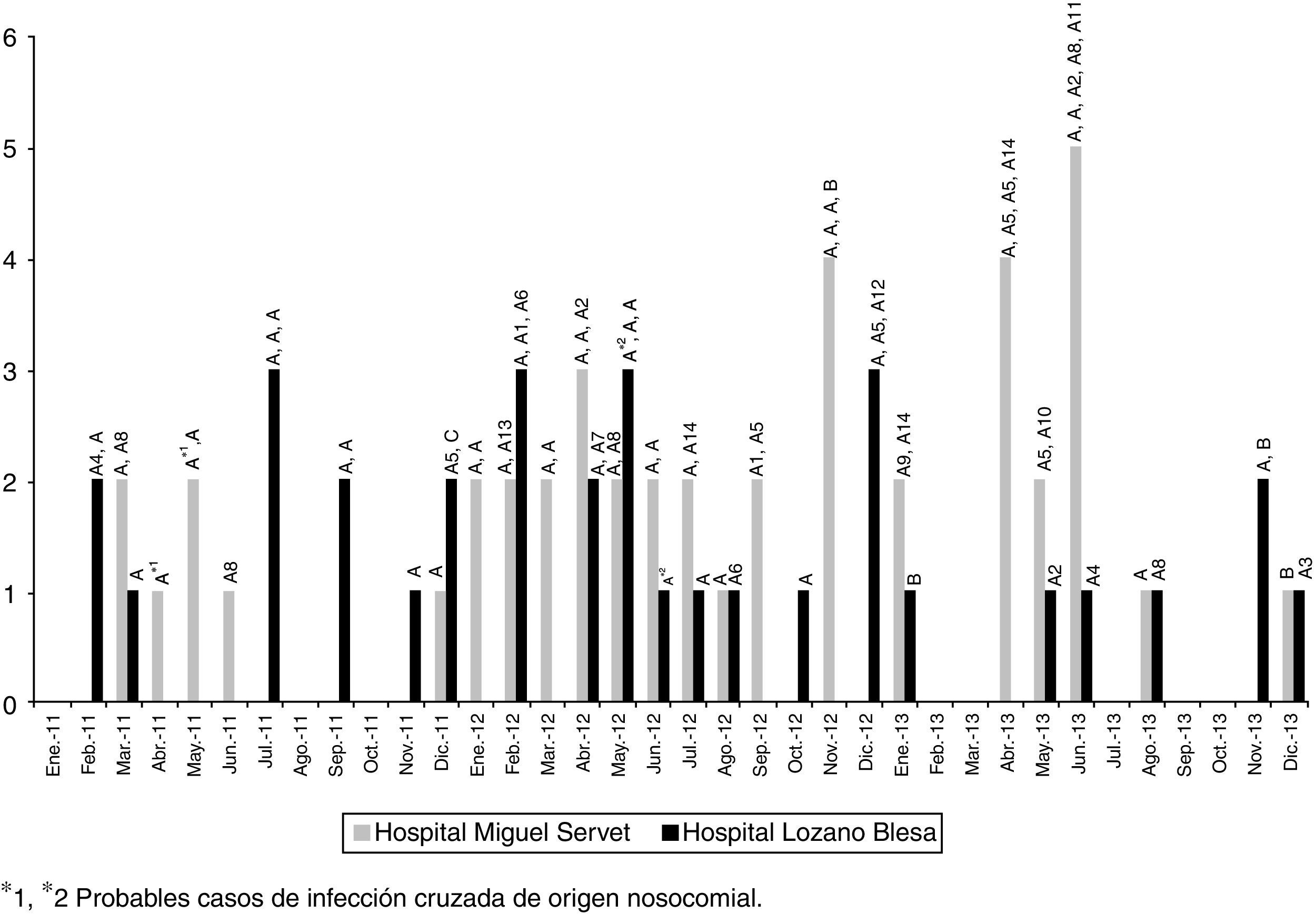
ResultadosEn el periodo de estudio (2011-2013) se detectaron 79 aislados E.faecalis resistentes a vancomicina con genotipo vanB2 (46 en HUMS, 33 en HCULB), procedentes de 73 pacientes (45,5% varones; edad media: 73años). En el HRV no se detectó ningún EFRV en el periodo en estudio. La mayoría de los aislados (62; 78,5%) se obtuvieron de muestras urinarias. Ninguno de los pacientes con datos disponibles sobre terapia antibiótica previa había recibido vancomicina, pero sí betalactámicos (14/32) y/o fluoroquinolonas (20/32). En 54 pacientes, E.faecalis vanB2 se confirmó como el agente causal del cuadro infeccioso. De los 71 casos en los que pudo estudiarse el origen de la infección, 32 (45,1%) fueron comunitarios, 33 (46,5%) nosocomiales y 6 (8,4%) asociados a cuidados sanitarios. La distribución de los aislamientos según servicios hospitalarios mostró una gran dispersión (fig. 1), lo que sugiere la existencia de diferentes fuentes de infección o la transmisión de estos clones a través del personal sanitario o el traslado de pacientes. La infección cruzada de origen nosocomial parece especialmente probable en 2 casos; uno en el servicio de neurocirugía del HUMS en abril-mayo de 2011 y otro en el servicio de traumatología del HCULB en mayo-junio de 2012. En ambos casos se aislaron EFRV vanB2 con igual pulsotipo de distintos pacientes que coincidieron en el mismo servicio y periodo de tiempo durante su estancia hospitalaria.
En cuanto al estudio genotípico, todas las cepas portaban el gen vanB2, y en todas se detectó una mutación puntual (G34T) que implicaba un cambio aminoacídico (Met11Ile) en dicho alelo. Los aislamientos presentaron un fenotipo multirresistente, que incluía, además de resistencia a vancomicina, también a eritromicina, tetraciclina, ciprofloxacino y alto nivel de resistencia a estreptomicina, gentamicina y kanamicina.
La ECP reveló la existencia de 3 perfiles genéticos (patrón A y sus subtipos, patrón B y patrón C). La mayoría de las cepas (n=74; 93,7%) pertenecían a un clon idéntico al del inicio del brote (A, n=43) o a algún subtipo (A1-A14, n=31) estrechamente relacionado (1-3 bandas de diferencia). Cuatro cepas, aisladas en ambos hospitales y pertenecientes a distintos pacientes, fueron identificadas como clon B y una como clon C (>6 bandas de diferencia). El tipado por MLST realizado en 16 aislados representativos de los diferentes tipos y subtipos de ECP demostró que todos ellos pertenecían a la secuencia tipo ST6. La figura 2 muestra que la frecuencia en la detección de los aislamientos EFRV vanB2-ST6 durante los 3 años de seguimiento del brote se mantiene estable.
DiscusiónA diferencia de Estados Unidos, donde se han descrito brotes por EVR (mayoritariamente E.faecium) con relativa frecuencia, en Europa la prevalencia ha sido menor, aunque en los últimos años se ha evidenciado un incremento en algunos países1. Durante años, la detección de E.faecium con genotipo vanA ha sido frecuente en animales de granja y alimentos, probablemente en relación con el amplio uso de avoparcina como promotor de crecimiento en animales hasta 1997, año en que fue prohibido este uso en la Unión Europea. Si bien E.faecalis es el principal responsable de las infecciones enterocócicas, E.faecium presenta mayores tasas de resistencia adquirida a los antimicrobianos. Las infecciones por EFRV siguen siendo infrecuentes. Así, debido a las especiales características del primer brote por E.faecalis vanB2 descrito en España, en este trabajo realizamos el seguimiento y la caracterización de los aislamientos clínicos de los años siguientes. Cabe señalar que la relación clonal de los aislados se estableció mediante ECP, indicado preferentemente para cepas aisladas en hospitales durante periodos inferiores a 6 meses10. Sin embargo, la falta de metodologías alternativas en términos de coste, reproducibilidad y poder discriminatorio ha favorecido la extensión de su uso en estudios de evolución de brotes. Por otra parte, la adquisición de elementos de transferencia horizontal puede dar lugar a cambios en más de 6 bandas, lo que podría repercutir en una subestimación del total de clones relacionados.
En cuanto al origen de las cepas, la mayoría provenían de muestras urinarias, como también se describe en anteriores trabajos11. Ninguno de los pacientes de los que pudo obtenerse información acerca del tratamiento antibiótico previo había recibido vancomicina, por lo que, a diferencia de otros estudios3, no se observó asociación entre consumo previo de vancomicina y emergencia de EFRV vanB2. Sin embargo, la selección de estos clones como consecuencia de la administración previa de antibióticos de otras familias frecuentemente relacionadas con adquisición de EVR3,12 (como fluoroquinolonas, frente a las cuales mostraron resistencia los clones estudiados, o cefalosporinas), no puede descartarse.
La secuencia tipo ST6 a la que pertenecieron los aislamientos de E.faecalis es, junto con la ST17 (en E.faecium), una de las líneas genéticas mejor adaptadas al ambiente hospitalario en España13. En 2004 se describió la diseminación en 3 hospitales portugueses de un clon de E.faecalis perteneciente a este linaje pero portador del gen vanA14. Estos hechos respaldan los estudios que sugieren que ST6 es particularmente apto para captar genes exógenos (como vanA o vanB) por recombinación15. Cepas de E.faecalis pertenecientes a este linaje también han sido detectadas en cerdos16 y en una rata silvestre17, mostrando características muy similares a las cepas clínicas de Portugal y España. Cabe destacar que la mutación detectada en el alelo vanB2 (G34T), que conlleva un cambio aminoacídico (Met11Ile), también se observó en el aislado proveniente de la rata. Serán necesarios futuros estudios que determinen la posible implicación biológica de esta mutación presente en el alelo vanB2 de cepas de enterococo distribuidas en entornos tan variados.
La tipificación molecular nos permitió observar la circulación de 3 clones presentes en ambos hospitales y con una distribución temporal sin una tendencia definida. A diferencia de lo que se observa en los brotes causados por E.faecium, usualmente policlonales y en los que el clon predominante al inicio va dejando paso a otras variantes6,7,11, en este brote tanto el clon predominante (A) como otros clones (B, C) e incluso sus subtipos (A1-A14) se detectaron a los largo de los 3años, sin una asociación clara de una variante a un determinado hospital. Esto, junto con la elevada tasa de casos comunitarios y la gran variabilidad en la distribución de los aislados por servicio hospitalario, plantea la posibilidad de que se trate de un clon que esté ampliamente distribuido en esta área. Es necesario, por tanto, la implementación de estrategias de control para reducir la persistencia de estos EFRV, evitar la diseminación y emergencia de nuevos clones, y la posible transferencia horizontal de genes vanB a otras cepas para preservar la utilidad clínica de la vancomicina.
FinanciaciónEste trabajo ha sido financiado en parte por el proyecto SAF2012-35474 del Ministerio de Economía y Competitividad (MINECO) y el Fondo Europeo de Desarrollo Regional (FEDER). C.A. Alonso tiene una beca FPI predoctoral del MINECO adscrita al proyecto SAF2012-35474.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.