La mayoría de laboratorios de microbiología utilizan diferentes técnicas para el diagnóstico de las infecciones gastrointestinales. Algunas requieren hasta 72 h para obtener resultados definitivos.
Material y métodosEl panel gastrointestinal de Luminex (xTAG-GPP, Luminex Molecular Diagnostics, Toronto, Canadá) se trata de un ensayo cualitativo multiplex rápido y sensible capaz de detectar e identificar simultáneamente los 15 patógenos más frecuentes causantes de gastroenteritis. Nuestro objetivo ha sido evaluar este panel multiplex comparándolo con los métodos habituales empleados en nuestro laboratorio.
ResultadosSe analizaron 225 muestras de heces. A través de los métodos convencionales fueron 74 las muestras positivas (32,9%). A través de Luminex fueron 137 las muestras positivas (60,9%).
ConclusiónEl uso del panel gastrointestinal de Luminex puede mejorar el diagnóstico de las infecciones gastrointestinales principalmente porque proporciona resultados en menos de 8 h. Determinados microorganismos deben interpretarse con precaución y basándose en datos clínicos y epidemiológicos del paciente.
Most Microbiology laboratories use different techniques for the diagnosis of gastrointestinal infections. Some of which require at least 72 hours to obtain final results.
Material and MethodsThe gastrointestinal panel Luminex (xTAG-GPP, Luminex Molecular Diagnostics, Toronto, Canada) is a qualitative multiplex fast and sensitive assay able to detect and to identify the 15 most common pathogens causing gastrointestinal infection simultaneously. We evaluated this multiplex panel comparing it with conventional methods used in our laboratory.
ResultsWe analyzed 225 samples of feces. Through the conventional methods were positive 74 samples (32.9%). Through the Luminex method were positive 137 samples (60.9%).
ConclusionsThe use of the xTAG® GPP system in Clinical Microbiology can improve the diagnosis of gastrointestinal infectious because it provides results in less than 8 hours. Some pathogens should be applied with caution and should be interpreted based on the patient¿s clinical data.
Las infecciones gastrointestinales causan una elevada morbilidad en nuestro medio. Un amplio espectro de microorganismos pueden ser los responsables y presentar las mismas manifestaciones clínicas. Por tanto, la identificación del agente etiológico es importante para su diagnóstico y tratamiento. La mayoría de laboratorios de Microbiología utilizan diferentes técnicas para su diagnóstico y algunas, como es el cultivo, necesitan hasta 72h para obtener resultados definitivos. Como alternativa, ciertos laboratorios disponen de métodos moleculares que son utilizados en situaciones concretas, pero estos detectan un número limitado de microorganismos en un mismo análisis.
El Panel Gastrointestinal xTAG-GPP se trata de un ensayo multiplex cualitativo de PCR capaz de detectar e identificar de forma simultánea, y en menos de 8h, los 15 patógenos más comunes causantes de gastroenteritis. Los patógenos incluidos son: a)3virus: Adenovirus 40/41, Rotavirus A, Norovirus GI/GII; b)9bacterias: Salmonella spp., Shigella spp., Vibrio cholerae, Yersinia enterocolitica, Campylobacter jejuni, Escherichia coli O 157 y toxinas como la toxina A/B de Clostridium difficile, Escherichia coli enterotoxigénico (ETEC): enterotoxina lábil (LT)/enterotoxina termoestable (ST), Escherichia coli O 157, E.coli (STEC) productor de toxina shiga (stx1/stx2), y c)3parásitos: Crypstosporidium spp., Entamoeba histolytica y Giardia lamblia.
Nuestro principal objetivo ha sido comparar los resultados obtenidos a través de los métodos convencionales de diagnóstico utilizados en nuestro laboratorio con los resultados obtenidos mediante el panel molecular de Luminex.
Material y métodosSe analizaron 225 muestras de heces frescas remitidas en envase estéril de tapón de rosca, de pacientes con diarrea aguda y de cualquier edad. Las muestras se enviaron al Servicio de Microbiología del Consorcio Hospital General Universitario de Valencia durante el mes de julio y hasta noviembre de 2013, siguiendo las normas de envío y conservación adecuadas.
Inicialmente, las muestras fueron procesadas de forma rutinaria. El cultivo se utilizó para la detección de Salmonella spp., Shigella spp., Yersinia enterocolitica, Campylobacter spp., Aeromonas spp. y Vibrio spp. Para ello se utilizaron medios poco selectivos, como el agar MacConkey y el agar sangre, medios de cultivo con selectividad media como el agar Hecktoen y medios líquidos de enriquecimiento como el caldo de selenito. Todos ellos se incubaron 24h a 37°C, excepto el caldo selenito, que se incubó 48h. Para el aislamiento de Campylobacter spp. se utilizó un medio selectivo y se incubó 48h en atmósfera microaerófila a una temperatura de 42°C. Para la identificación, las colonias sospechosas se analizaron a través del sistema MALDI-TOF, y para el estudio de sensibilidad se utilizó el panel NC69 de MicroScan (Siemens). Clostridium difficile toxigénico se detectó por enzimoinmunoanálisis (C.difficile Quik Chek Complete, Alere). Este test detecta el antígeno glutamato deshidrogenasa y las toxinas A/B directamente de la muestras de heces. Los resultados toxina A/B positivos se confirmaron mediante PCR, tal y como indican los protocolos. Para la detección de Adenovirus y Rotavirus se utilizó un test rápido de detección de antígenos en heces (inmunocromatografía, Rota-Adeno Letitest). En nuestro hospital no se estudia de forma rutinaria Norovirus. La presencia de parásitos se realizó mediante técnicas microscópicas.
Inmediatamente después de su procesamiento habitual, las heces se almacenaron a −80°C hasta su posterior análisis mediante el sistema Luminex. Esta técnica incluye varios pasos: pretratamiento de la muestra, extracción del ácido nucleico, RT-PCR múltiple, reacción de hibridación con microesferas y fase de detección e interpretación mediante el instrumento Luminex (Luminex Molecular Diagnostics, Toronto, Canadá) a través del programa TDAS GPP 1.11 software.
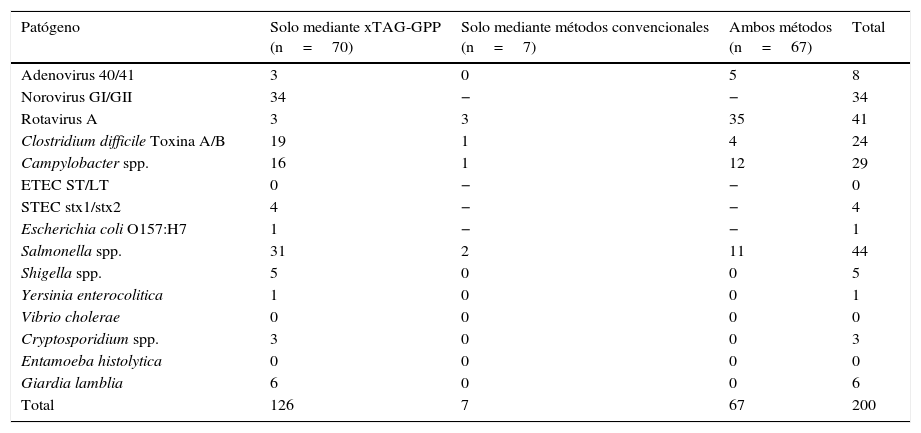
ResultadosSe analizaron 225 muestras de heces, y en 144 (64%) se detectaron uno o más patógenos (tabla 1). Las muestras positivas coincidentes en resultados por ambos métodos fueron 67/225 (29,7%). Rotavirus A fue el patógeno más frecuente.
Patógenos detectados según el método empleado (n=144)
| Patógeno | Solo mediante xTAG-GPP (n=70) | Solo mediante métodos convencionales (n=7) | Ambos métodos (n=67) | Total |
|---|---|---|---|---|
| Adenovirus 40/41 | 3 | 0 | 5 | 8 |
| Norovirus GI/GII | 34 | − | − | 34 |
| Rotavirus A | 3 | 3 | 35 | 41 |
| Clostridium difficile Toxina A/B | 19 | 1 | 4 | 24 |
| Campylobacter spp. | 16 | 1 | 12 | 29 |
| ETEC ST/LT | 0 | − | − | 0 |
| STEC stx1/stx2 | 4 | − | − | 4 |
| Escherichia coli O157:H7 | 1 | − | − | 1 |
| Salmonella spp. | 31 | 2 | 11 | 44 |
| Shigella spp. | 5 | 0 | 0 | 5 |
| Yersinia enterocolitica | 1 | 0 | 0 | 1 |
| Vibrio cholerae | 0 | 0 | 0 | 0 |
| Cryptosporidium spp. | 3 | 0 | 0 | 3 |
| Entamoeba histolytica | 0 | 0 | 0 | 0 |
| Giardia lamblia | 6 | 0 | 0 | 6 |
| Total | 126 | 7 | 67 | 200 |
Por el método Luminex, las muestras positivas fueron 137/225 (60,9%), pero las positivas únicamente por este método fueron 70 de 225 (31,1%). Los patógenos «extra» detectados fueron: Norovirus (n=34), Adenovirus y Rotavirus (n=3), Salmonella spp. (n=31), seguido de C.difficile toxigénico (n=19) y Campylobacter spp. (n=16). Shigella spp estuvo presente en 5 muestras y Yersinia spp., en una. Cuatro muestras fueron positivas para STEC y una para E.coli serotipo O 157. Vibrio cholerae, ETEC y E.histolytica no se encontraron en ninguna muestra. Cryptosporidium spp. y Giardia lamblia estuvieron presentes en 3 y 6 muestras, respectivamente.
Por medio de los métodos convencionales fueron positivas 74/225 (32,9%), pero únicamente fueron 7 las muestras positivas por este método exclusivamente (3,1%). Los microorganismos implicados fueron: Rotavirus A (n=3), Salmonella spp. (n=2), Campylobacter spp. y C.difficile toxigénico (n=1).
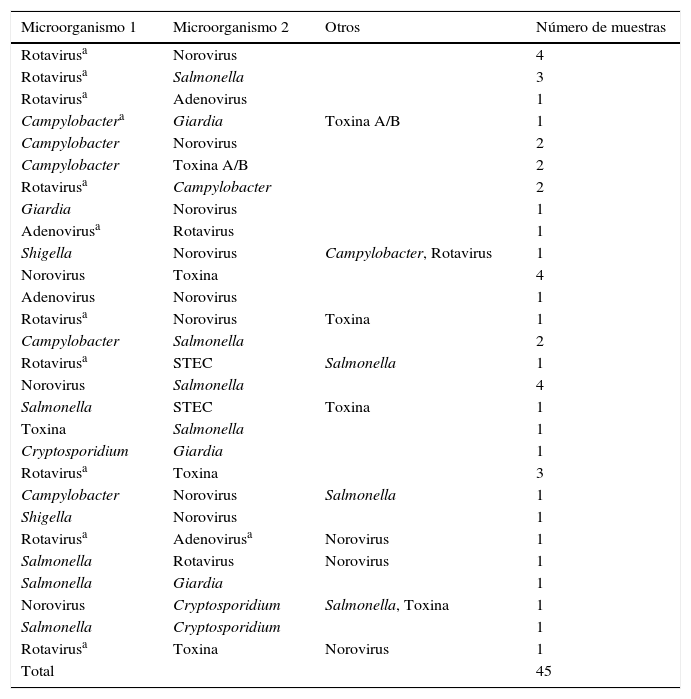
En 45 muestras (20%) se detectó más de un patógeno mediante Luminex. Los microorganismos y combinaciones halladas se detallan en la tabla 2.
Combinaciones detectadas (n=45)
| Microorganismo 1 | Microorganismo 2 | Otros | Número de muestras |
|---|---|---|---|
| Rotavirusa | Norovirus | 4 | |
| Rotavirusa | Salmonella | 3 | |
| Rotavirusa | Adenovirus | 1 | |
| Campylobactera | Giardia | Toxina A/B | 1 |
| Campylobacter | Norovirus | 2 | |
| Campylobacter | Toxina A/B | 2 | |
| Rotavirusa | Campylobacter | 2 | |
| Giardia | Norovirus | 1 | |
| Adenovirusa | Rotavirus | 1 | |
| Shigella | Norovirus | Campylobacter, Rotavirus | 1 |
| Norovirus | Toxina | 4 | |
| Adenovirus | Norovirus | 1 | |
| Rotavirusa | Norovirus | Toxina | 1 |
| Campylobacter | Salmonella | 2 | |
| Rotavirusa | STEC | Salmonella | 1 |
| Norovirus | Salmonella | 4 | |
| Salmonella | STEC | Toxina | 1 |
| Toxina | Salmonella | 1 | |
| Cryptosporidium | Giardia | 1 | |
| Rotavirusa | Toxina | 3 | |
| Campylobacter | Norovirus | Salmonella | 1 |
| Shigella | Norovirus | 1 | |
| Rotavirusa | Adenovirusa | Norovirus | 1 |
| Salmonella | Rotavirus | Norovirus | 1 |
| Salmonella | Giardia | 1 | |
| Norovirus | Cryptosporidium | Salmonella, Toxina | 1 |
| Salmonella | Cryptosporidium | 1 | |
| Rotavirusa | Toxina | Norovirus | 1 |
| Total | 45 |
El panel de Luminex presenta diversas ventajas con respecto a los métodos convencionales. La más resaltada es la detección simultánea de múltiples patógenos y la disminución del tiempo de diagnóstico a 6-8h1-6. Además, permite la detección de microorganismos de los cuales no disponemos métodos de diagnóstico rutinario, y de infecciones mixtas.
En este estudio, en total se detectaron 126 patógenos «extras»: Salmonella spp. y Norovirus fueron los mayoritarios, seguidos de la toxina de Clostridium difficile y Campylobacter spp. En el caso de Salmonella, en nuestro medio es muy importante un diagnóstico preciso. Su alta e inesperada detección mediante el panel de Luminex también ha sido destacada en otros artículos1-3,7. La misma situación se describe con E.histolytica en regiones donde este parásito es muy prevalente1,7,8. En nuestro estudio, sin embargo, no se detectó en ninguna muestra.
En la detección de C.difficile, el 69,5% (16/23) pertenecieron a muestras de heces de niños menores de 5años. Es decir, se detectó en un grupo de población donde el test rápido no había sido solicitado por el clínico. En la población pediátrica continúa siendo un tema en debate debido a la alta posibilidad de portadores asintomáticos. Aun así, como indican Khare et al.9, la alta detección de la toxina de C.difficile en niños representa una importante área de estudio.
Norovirus también fue detectado en gran número de muestras (n=34). Muchos laboratorios no estudian este virus de forma rutinaria debido a la escasa sensibilidad de los métodos de EIA, por lo que se sospecha que es el causante de gran parte de los casos sin diagnosticar. En este trabajo no se realizó la detección de antígeno, y por tanto carecemos de los datos para poder compararlos. No obstante, resultados inesperados, tanto de Norovirus como de Campylobacter, han sido descritos por otros autores3,4.
En nuestro estudio, la detección de Rotavirus mediante Luminex, al compararlos con los métodos inmunológicos de detección de antígeno, observamos que en el 85% (35/41) de los casos los resultados fueron coincidentes.
Patógenos menos frecuentes, como Shigella spp., Yersinia enterocolitica, STEC, Cryptosporidium spp. y Giardia lamblia, Luminex los detectó en diferentes muestras, a diferencia de los métodos habituales. Nosotros no disponemos de resultados con un segundo método molecular, por lo que no podemos descartar la posibilidad de falsos positivos. En publicaciones de otros autores, como Beckmann et al.4, la mitad de los casos en los que se detectó Giardia lamblia no se confirmaron. En el caso de Shigella fueron 5 los pacientes implicados, y sí los comprobamos con una PCR simple. Se confirmaron 2 casos, de los cuales tuvimos acceso a la historia clínica, y ambos habían viajado recientemente a la India.
En una minoría de muestras (n=7), las discrepancias se debieron a que fueron positivas por los métodos de rutina pero negativas por Luminex. Una posible causa sería la degradación de los ácidos nucleicos pese a la conservación óptima de la muestra a −80°C.
Las infecciones mixtas no son frecuentes. En este estudio se detectó más de un patógeno en 45 muestras (20%). Las combinaciones cambian de unos estudios a otros2,4,9-12, probablemente porque varían el área geográfica y tipo de población.
La alta sensibilidad de la técnica y la amplia detección de microorganismos en un mismo análisis proporcionan mucha información, a veces difícil de interpretar. Algunos estudios han indicado la necesidad de cuantificar los enteropatógenos, sobre todo en infecciones mixtas, y diferenciar entre patógenos con replicación activa o colonizadores para poder conocer la implicación de cada uno de ellos12, ya que se sabe que determinados microorganismos pueden estar presentes en pacientes asintomáticos o un largo periodo de tiempo después tras la resolución de la infección.
Con respecto a otros paneles moleculares comercializados y aprobados para su uso en clínica13, Luminex presenta un alto rendimiento porque tiene capacidad para analizar hasta 24 muestras en un único panel, además de ser de los paneles con más número de patógenos detectados por análisis. Pero es una técnica laboriosa y compleja que para su manejo requiere personal cualificado y con experiencia en técnicas de biología molecular. Por otro lado, existe un alto riesgo de contaminaciones, al tratarse de un sistema abierto. Y es imprescindible un mantenimiento y una limpieza meticulosos diarios, tanto del instrumento como del lugar de trabajo. Todo esto dificulta su implantación como método rutinario en la mayoría de laboratorios de Microbiología, además de que no reemplazaría al cultivo, ya que en el caso de muestras positivas sería necesaria su confirmación o disponer de la cepa para estudios de sensibilidad. Por último, cabe mencionar su coste más elevado con respecto a otros métodos. Para obtener una relación coste/beneficio óptima es necesario analizar un número muy elevado de muestras, y esto no es factible en muchos laboratorios. No obstante, sería necesario realizar un estudio en profundidad con tales fines.
Como conclusión, el panel multiplex gastrointestinal de Luminex proporciona grandes ventajas en el diagnóstico de la gastroenteritis aguda por ser rápido e identificar en un mismo análisis los patógenos causantes del 80% de los casos de gastroenteritis infecciosa. Resultados positivos para determinados patógenos deben ser interpretados con precaución y dentro del contexto clínico del paciente. Nosotros recomendamos su uso principalmente en brotes, en estudios epidemiológicos, en casos de diarrea del viajero y en pacientes graves con alta sospecha de patología infecciosa.
Conflicto de interesesLos autores no declaran ningún conflicto de intereses.
Los autores agradecen a Luminex Molecular Diagnostic el haberles proporcionado el instrumento y los reactivos utilizados en este estudio.