El objetivo del diagnóstico molecular es la identificación de cambios en la secuencia o en la expresión de genes críticos para el desarrollo de cáncer, mediante técnicas de biología molecular. Estas técnicas se caracterizan por una alta sensibilidad, lo que permite realizar un diagnóstico preciso.
Los hallazgos moleculares al diagnóstico proporcionan información acerca de factores predictivos de respuesta terapéutica y en el pronóstico en muchos casos. Asimismo, permiten detectar la presencia de enfermedad mínima residual en pacientes en remisión clínica. También permiten el análisis de mutaciones en genes supresores y oncogenes, útil en el diagnóstico y en algunos casos para predecir la respuesta de las células tumorales a los fármacos antineoplásicos. Los avances en el desarrollo y la aplicación de nuevas tecnologías para el análisis molecular han contribuido de forma significativa al entendimiento de la genética y patogénesis molecular del cáncer pediátrico, y su relación con el pronóstico de la enfermedad.
En el futuro la utilización de estas herramientas permitirá la identificación de nuevos biomarcadores útiles en el diagnóstico, definición del pronóstico y seguimiento de las distintas neoplasias, además del reconocimiento de nuevos blancos moleculares para el desarrollo de fármacos antineoplásicos con mayor actividad antitumoral, mayor selectividad y con menores efectos adversos.
The aim of molecular diagnosis is the identification of changes in the sequence or expression of critical genes for cancer development, through molecular biology techniques. These techniques are characterized by a high sensitivity, which allows an accurate diagnosis. Molecular findings at diagnosis provide information about predictors of treatment response and prognosis in many cases. Similarly, allow the detection of minimal residual disease in patients with clinical remission. They also enable mutational analysis of suppressor genes and oncogenes, useful in diagnosis and in some cases, to predict response of tumor cells to antineoplasic agents.
Advances in the development and application of new technologies for molecular analysis have contributed significantly to the understanding of genetics and molecular pathogenesis of pediatric cancer and their relationship with the prognosis of the disease.
In the future the use of these tools will allow the identification of new biomarkers useful in diagnosis, prognosis definition and monitoring of different neoplasms and the recognition of new molecular targets for the development of anticancer drugs with increased antitumor activity and higher selectivity with fewer adverse effects.
Introducción
En la actualidad el cáncer es un problema muy importante de salud pública, siendo la segunda causa de mortalidad infantil en los niños mayores de 4 años de edad, sólo superada por los accidentes. En México, el cáncer pediátrico pasó del décimo tercer lugar como causa de muerte, al segundo lugar entre la población de 1 a 14 años a partir del año 2000.
Esta enfermedad se caracteriza por la acumulación de alteraciones genéticas en oncogenes, genes supresores y genes reparadores de ADN; que producen alteraciones en la expresión de los mismos o en su función. Es por esto que el comportamiento de la célula cancerosa es distinto al de la célula normal, ya que las células malignas tienen un crecimiento potencialmente indefinido, son capaces de invadir tejidos circundantes y tienen la capacidad de diseminación a distancia a otros sitios del organismo.
La investigación de las alteraciones genéticas de las enfermedades neoplásicas ha permitido conocer la existencia y localización de una serie de genes críticos, en el desarrollo de cáncer. Lo que ha llevado no sólo a un mejor conocimiento de los mecanismos moleculares de los diversos tipos de cáncer, sino que tiene también implicaciones directas en la práctica clínica.
Este conocimiento ha permitido la identificación de portadores de mutaciones germinales que predisponen al desarrollo de algunos tipos de cáncer hereditario, ha sido útil en la identificación de marcadores moleculares de utilidad para el diagnóstico, la caracterización de anomalías genéticas relacionadas con el pronóstico de la enfermedad. El avance en la investigación genética y molecular es imprescindible para el desarrollo de nuevos tratamientos y métodos, para prevenir y disminuir los efectos secundarios de la quimioterapia.
El diagnóstico molecular tiene el objetivo de identificar las alteraciones que por diferentes mecanismos afectaron la estructura o secuencia de estos genes críticos, para el desarrollo de cáncer. Este diagnóstico se realiza mediante técnicas de biología molecular que se caracterizan por una alta sensibilidad y especificidad, lo que permite realizar un diagnóstico preciso, incluso antes de que el cáncer se manifieste clínicamente, permiten además determinar la presencia de enfermedad mínima residual en pacientes con remisión clínica.
El análisis de mutaciones en genes que promueven la proliferación celular, incluyendo factores de crecimiento y sus receptores, transductores de señales, factores de transcripción o inhibidores de la apoptosis (oncogenes), o de genes que regulan negativamente la división celular (genes supresores de tumores), es útil en el diagnóstico y en la estratificación pronóstica de diversas neoplasias.
Por otra parte, la identificación de variantes en los genes que metabolizan o transportan fármacos, puede ser de utilidad en la predicción de la respuesta a fármacos antineoplásicos y en la identificación de pacientes con mayor riesgo de toxicidad.
Por estas razones, los estudios de genética molecular en cáncer han adquirido una relevancia trascendental. En este artículo, se analizan algunas de las principales técnicas moleculares y sus aplicaciones en diversos tipos de cáncer pediátrico.
Reacción en Cadena de la Polimerasa (PCR)
La PCR, descrita por Karry Mullis en 1985, amplifica secuencias específicas de ADN obtenido de cualquier tipo de tejido o muestra biológica (ADN molde). La reacción se realiza mezclando en un amortiguador con pH neutro, el ADN molde con los iniciadores (primers) específicos del gen que se quiere analizar, los desoxinucleótidos trifosfatos (dATP, dCTP, dTTP y dGTP), iones Mg2+ y la enzima Taq polimerasa.
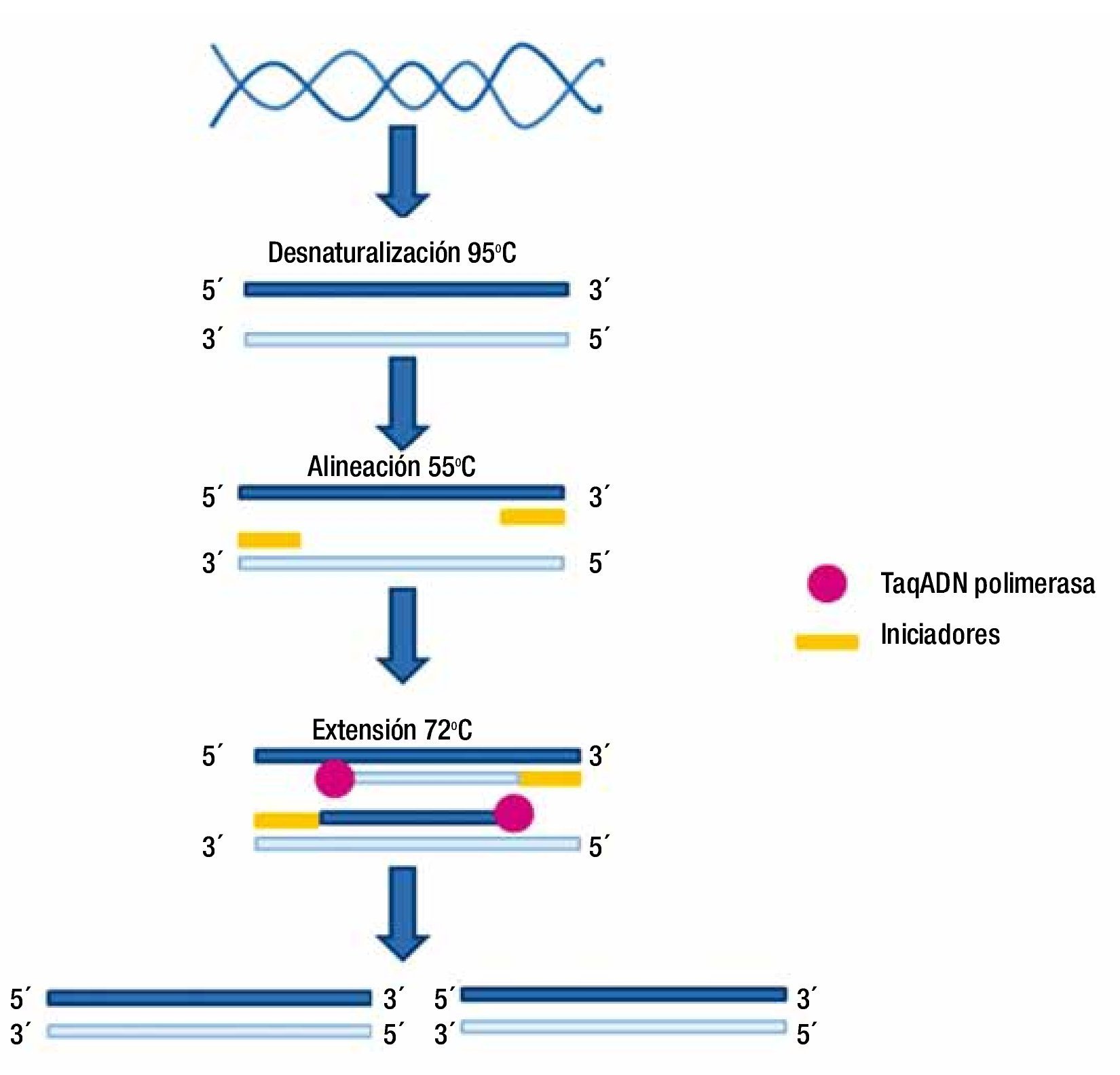
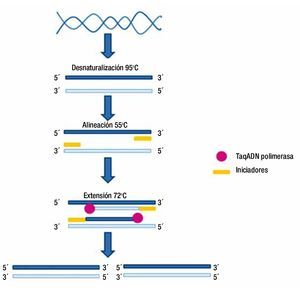
El proceso incluye 3 pasos principales, el primero es la desnaturalización del ADN, para lo cual se requiere una temperatura de 94°C o 95°C, lo que favorece el rompimiento de los puentes de hidrógeno entre las bases del ADN, para separar las 2 cadenas que forman la doble hebra de la molécula; el segundo paso es la alineación de los iniciadores, para lo cual se disminuye la temperatura entre 50°C y 70°C; y el tercero corresponde a la extensión o elongación (de la cadena complementaria), lo cual se efectúa a una temperatura de 72°C, a la cual actúa la enzima polimerasa, que copia la secuencia del gen seleccionado. Estos pasos se repiten por varios ciclos, generalmente entre 20 y 30, y al final de la reacción las cadenas nuevas vuelven a ser duplicadas por la polimerasa, lo que resulta en la producción exponencial de millones de copias del gen o segmento de ADN específico sometido al proceso (fig. 1)1-3.
Figura 1. Esquema general de las etapas de la reacción en cadena de la polimerasa (PCR).
La técnica de PCR es muy versátil, ya que puede adecuarse a ciertas necesidades, dependiendo del proceso y objetivo del análisis. Actualmente, se emplean diferentes modalidades de esta técnica para analizar el material genético con el fin de estudiar y diagnosticar diversas patologías.
RT-PCR (Reacción en Cadena de Polimerasa -Transcripción Reversa)
La técnica de RT-PCR se utiliza para la detección y amplificación de ARN. Permite estudiar la expresión de determinados genes.
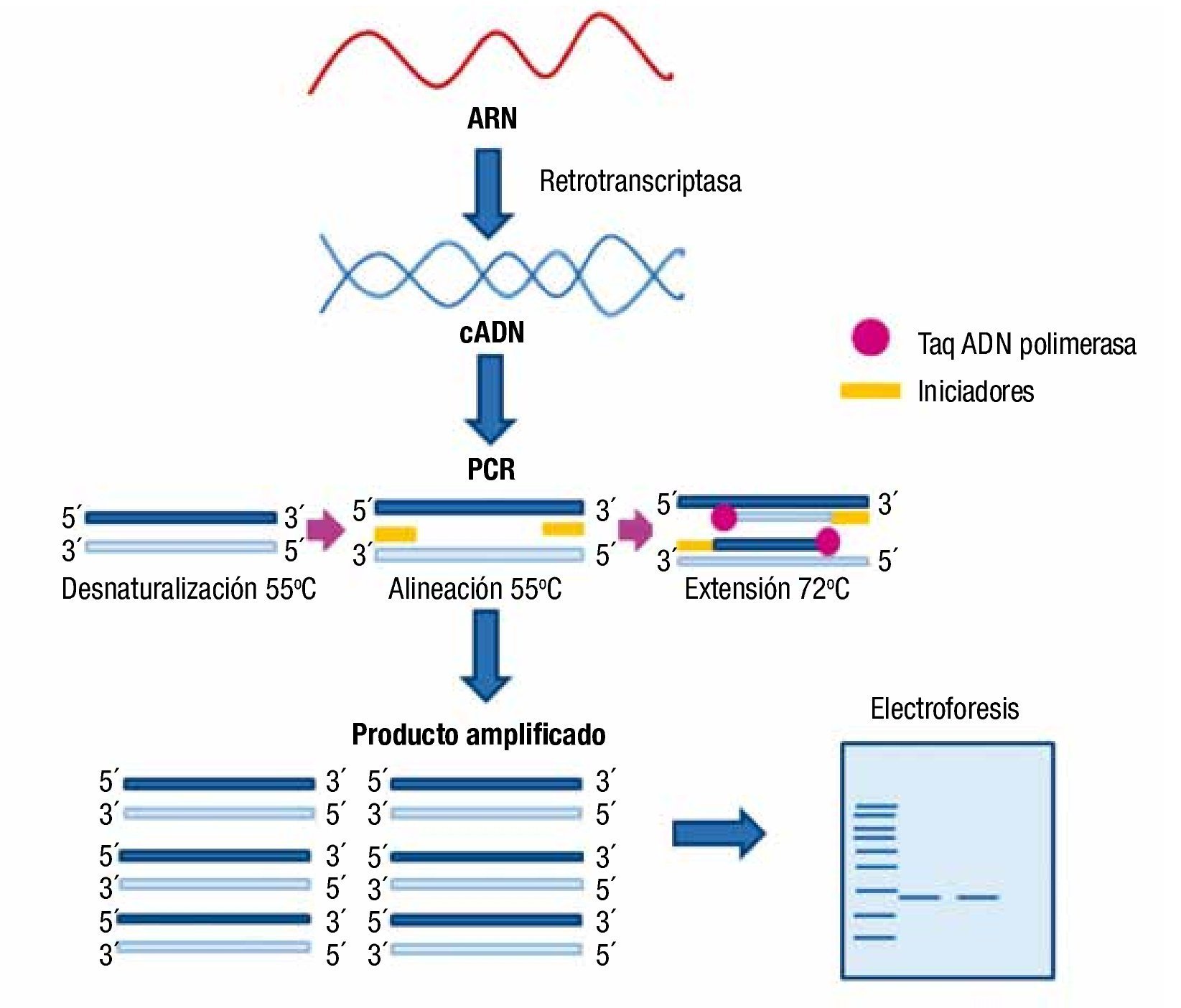
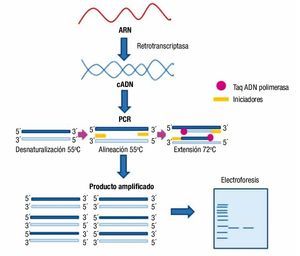
En primer lugar se extrae el ARN de las células en estudio, el cual es posteriormente transcrito a ADN mediante una transcriptasa inversa. Por cada molécula de ARN, se sintetiza una molécula de ADN complementario (cADN) (fig. 2). A partir de aquí se aplica la técnica de PCR, lo que permite una detección de la expresión de ARN mucho más sensible que con Northern blot o con hibridación in situ3,4.
Figura 2. Esquema general de la técnica RT-PCR. Se obtiene cADN por medio de una transcriptasa inversa, que utiliza ARN como molde para la obtención de la molécula de cADN, a partir de la cual se amplifica la secuencia de interés con una reacción de PCR convencional, y el fragmento es finalmente visualizado en un gel de electroforesis.
Una alteración o cambio en la información genética se conoce como mutación, y puede resultar en el desarrollo de una enfermedad. Cuando estas alteraciones afectan la estructura de uno o varios cromosomas se denominan mutaciones cromosómicas, y éstas a su vez pueden ser numéricas y estructurales. Las alteraciones cromosómicas estructurales pueden ser deleciones, duplicaciones, inversiones o traslocaciones.
En las leucemias de la edad pediátrica, tanto linfoblásticas como mieloides, al igual que en diversos tumores sólidos, se han identificado alteraciones cromosómicas que se repiten de manera consistente, muchas de las cuales tienen un significado pronóstico y determinan el tipo de tratamiento que recibirá el paciente5,6. La mayoría de estas alteraciones cromosómicas corresponden a traslocaciones. La técnica de RT-PCR ha sido particularmente aplicable en la detección de los transcritos de fusión resultantes de estas traslocaciones.
Un ejemplo es la t(9;22), también llamada cromosoma Filadelfia (Ph), del que deriva un oncogén conocido como BCR-ABL. En la leucemia linfoblástica aguda (LLA) de la edad pediátrica, el cromosoma Ph+ está presente en 2% al 4% de los casos y se considera un factor de mal pronóstico, debido a que las proteínas de fusión BCR-ABL presentan actividad aumentada de tirosina cinasa y confieren resistencia a la quimioterapia6-8.
La translocación t(12;21) (p12;q22) que ocasiona la fusión de los genes TEL y AML1, se presenta con una frecuencia de aproximadamente 25% en pacientes pediátricos con LLA, y se asocia con un buen pronóstico y una supervivencia libre de enfermedad de 85% a 90%9. Las traslocaciones t(1;19), t(9;22) y t(4;11) son alteraciones de mal pronóstico, que además se asocian con falla terapéutica. Las últimas 2 se consideran factores de mal pronóstico, independientemente de la edad y cuenta de leucocitos al diagnóstico. La t(9;22) y la t(4;11) están presentes en 10% de todas las LLA de alto riesgo10. La traslocación t(4;11)(q21;23), produce la fusión de los genes MLL y AF4 y los lactantes menores de un año que presentan esta alteración, tienen una supervivencia del 10% a 30% cuando son tratados sólo con quimioterapia11. Debido a lo anterior, en los pacientes menores de un año con t(4;11) está indicado el trasplante de progenitores hematopoyéticos en primera remisión.
El 85% de los pacientes pediátricos con leucemia mieloblástica aguda (LMA) presenta alteraciones cromosómicas, que originan genes de fusión. La presencia de algunas de ellas se asocia a determinados tipos de leucemias mieloblásticas, y en diversos estudios su presencia se ha relacionado con el pronóstico de la enfermedad12.
En el subtipo M2 se encuentra la translocación (8;21) (q22;q22) y la translocación (15;17) (q22;q12), es característica del subtipo M3. Los pacientes con la t(15;17) presentan una buena respuesta al tratamiento con ácido transretinoico (ATRA) y este conocimiento modificó radicalmente el pronóstico de las leucemias promielocíticas, que antes de la introducción del ATRA se asociaban a alta mortalidad por coagulopatía y que actualmente, se consideran de buen pronóstico cuando se maneja en combinación de ATRA y quimioterapia13.
La técnica RT-PCR que tiene una sensibilidad 100 veces superior a la citogenética, por eso se ha utilizado para detectar la enfermedad mínima residual (EMR) en numerosos pacientes en remisión, a través de la identificación de traslocaciones específicas de células leucémicas, de rearreglos del gen de inmunoglobulinas de cadena pesada o genes del receptor de células T (TCR)6. La EMR consiste en la persistencia del clon anormal, aún en niveles bajos, durante y después de terminar el tratamiento, además de tener un significado pronóstico. Por lo tanto, dentro de las posibles aplicaciones de esta prueba es predecir la recaída inminente, siempre y cuando se efectúe la detección en forma seriada durante el curso de tratamiento. Otros han señalado que también sirve para estratificar grupos de riesgo14,15.
La introducción de estas técnicas ha conducido a una nueva definición de remisión en la LLA, según la cual se considera remisión molecular cuando el nivel de EMR es menor de 1x10-4 (0.01%)16. Diversos trabajos reportan que los pacientes con EMR ≥ 1x10-2 al finalizar la inducción, tienen un riesgo de recaída superior al 60% en el curso de los 3 años posteriores, en contraste con menos del 15% en los que no se detecta EMR17,18. Con niveles ≥ 1x10-3 después de la consolidación, se tiene un riesgo de recaída de aproximadamente el 70%18. Es importante saber que una prueba aislada con niveles altos no indica necesariamente falla terapéutica, pero si persistencia de células leucémicas residuales y puede ser una indicación para intensificar el tratamiento. La persistencia de EMR por más de 6 meses después de la inducción, o la reaparición de EMR se asocian con recaída19. Es por esto que la determinación de la EMR puede predecir el curso clínico de la enfermedad, y le permite al médico tomar las decisiones en cuanto al tratamiento.
La determinación de ciertas translocaciones en algunos tumores sólidos ha tenido implicaciones en la clasificación del tumor y en la elección del tratamiento. En el tumor de Ewing, la t(11;22), produce la fusión de los genes EWS (22q12) y FLI-1 (11q24), además diversas combinaciones de los genes EWS y FLI-1 y está asociada con un peor pronóstico de la enfermedad20.
Los avances recientes en biología molecular han aumentado la comprensión de la patogénesis de los tumores cerebrales, particularmente en niños. El uso de las herramientas de diagnóstico molecular se está convirtiendo en componente fundamental del diagnóstico y clasificación de los tumores cerebrales en los niños, además de proporcionar una visión que conduce a la estratificación del tratamiento y al pronóstico de la enfermedad. Nuevos protocolos de tratamiento para algunos tumores cerebrales en los niños, incluyen el uso de biomarcadores para la identificación de subtipos moleculares y posibles estrategias de intervención21.
En neuroblastoma, la amplificación del oncogén N-MYC, puede ser determinada con RT-PCR cuantitativa, está asociada con tumores agresivos y pobre respuesta al tratamiento, sugiriendo el uso de tratamientos más intensos para estos pacientes22.
PCR-aninada o NESTED-PCR
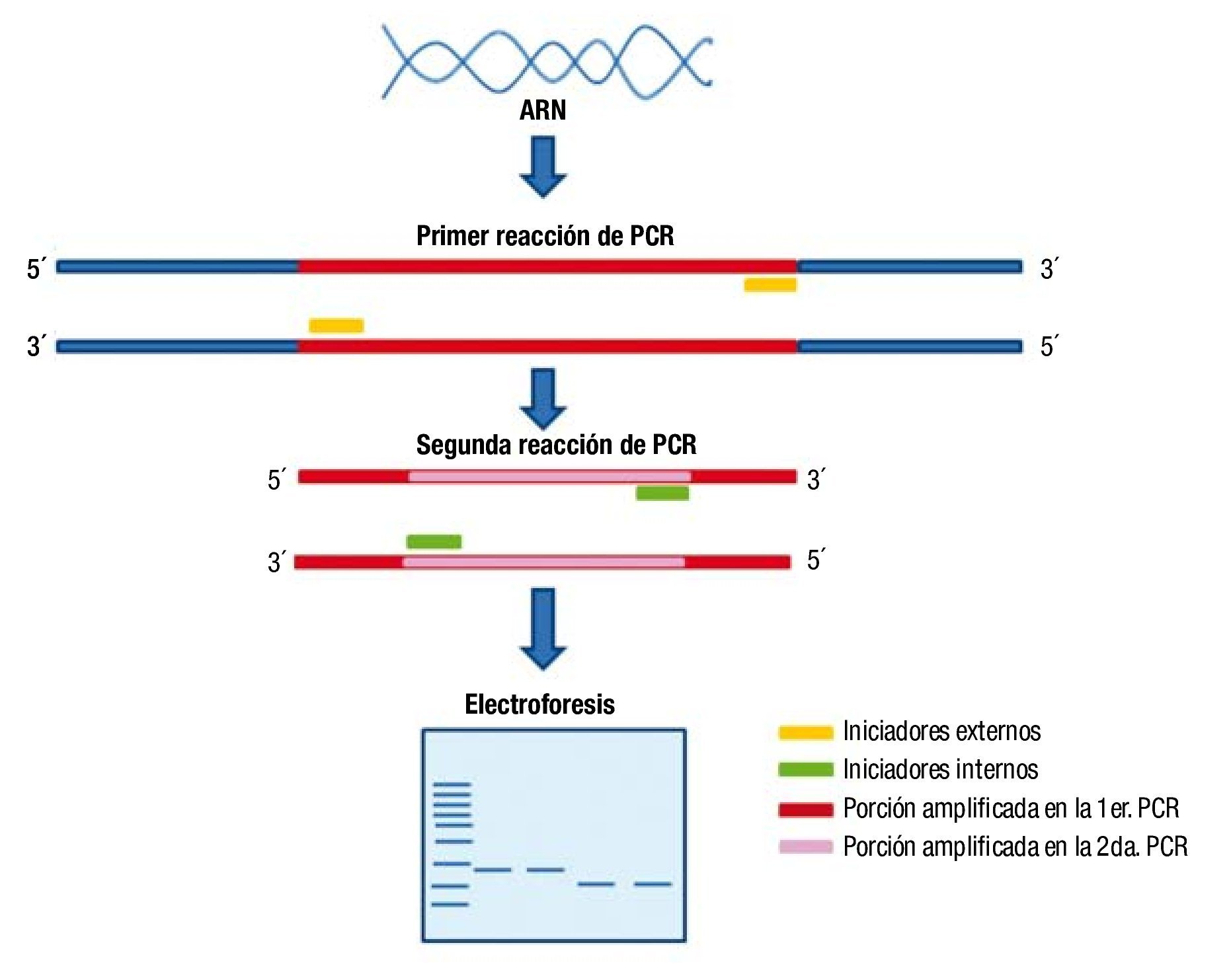
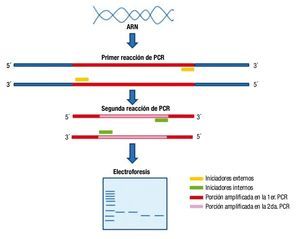
Es una técnica de PCR muy sensible, en la que el producto de una amplificación es utilizado como molde para realizar una segunda amplificación con iniciadores que se ubican dentro de la primera secuencia amplificada, es decir, el primer producto de PCR se une a los cebadores y se hace de nuevo una amplificación dentro del amplicón inicial (fig. 3). Este tipo de PCR tiene la ventaja de brindar alta sensibilidad y especificidad. La especificidad aumenta porque se amplifica un amplicón obtenido previamente, los iniciadores sólo van a unirse en un sitio dentro de la molécula y el resultado será una única banda. Así se evitan posibles alineaciones inespecíficas de los iniciadores, con lo que aumenta la sensibilidad de la prueba3,23.
Figura 3. Esquema general de NESTED-PCR o PCR anidada. A partir de ADN se realiza una primera PCR con un par de iniciadores que flanquean la región de ADN a analizar y posteriormente, se realiza una segunda PCR, utilizando como molde el producto de PCR obtenido en la primera reacción y con un par de iniciadores de una región más pequeña incluida dentro de la primera región amplificada.
Uno de los principales usos de esta técnica es la detección de infección por citomegalovirus (CMV), en pacientes sometidos a trasplante de células progenitoras hematopoyética. Después de una infección primaria por CMV, el virus permanece en forma latente en las células progenitoras hematopoyéticas CD34+ y en los monocitos de sangre periférica, sin causar enfermedad en pacientes inmunocompetentes. La reactivación viral ocurre en un estado de inmunodeficiencia; este proceso se ha estudiado principalmente en pacientes con trasplantes de células hematopoyéticas y de órganos sólidos24. Un receptor de trasplante, que es seropositivo para CMV tiene un 70% de riesgo de reactivación. La infección por CMV puede presentarse 1-4 meses después del trasplante, ser asintomática u ocasionar falla del injerto. Se ha reportado la reactivación por CMV humano como causa de morbilidad y mortalidad, en pacientes oncológicos en tratamiento con quimioterapia25.
Es importante la implementación de técnicas que permitan detectar tempranamente la infección activa e iniciar medidas terapéuticas, que eviten complicaciones potencialmente mortales en los pacientes. Los métodos de biología molecular, como la PCR-anidada, ofrecen alta sensibilidad (95%) para el diagnóstico de infecciones por CMV, además permiten detectar la infección activa en casos de concentraciones leucocitarias muy bajas como ocurre en pacientes inmunodeprimidos26,27. La detección por PCR de la infección activa es un indicador confiable, que anticipa riesgo elevado de padecer la enfermedad por CMV. La detección temprana del virus en plasma y orina, permite estimar el nivel de carga viral a la que el paciente está expuesto28. La técnica de PCR-anidada presenta muchas ventajas en comparación con la técnica de antigenemia, la cual requiere cuentas leucocitarias altas, lo que con frecuencia no es posible en pacientes inmunodeprimidos29.
Reacción en cadena de la polimerasa -polimorfismo conformacional de cadena sencilla (PCR-SSCP)
La PCR-SSCP se utiliza para la búsqueda de cierto tipo mutaciones, que incluyen sustituciones de bases, pequeñas inserciones, deleciones y reordenamientos4,30,31.
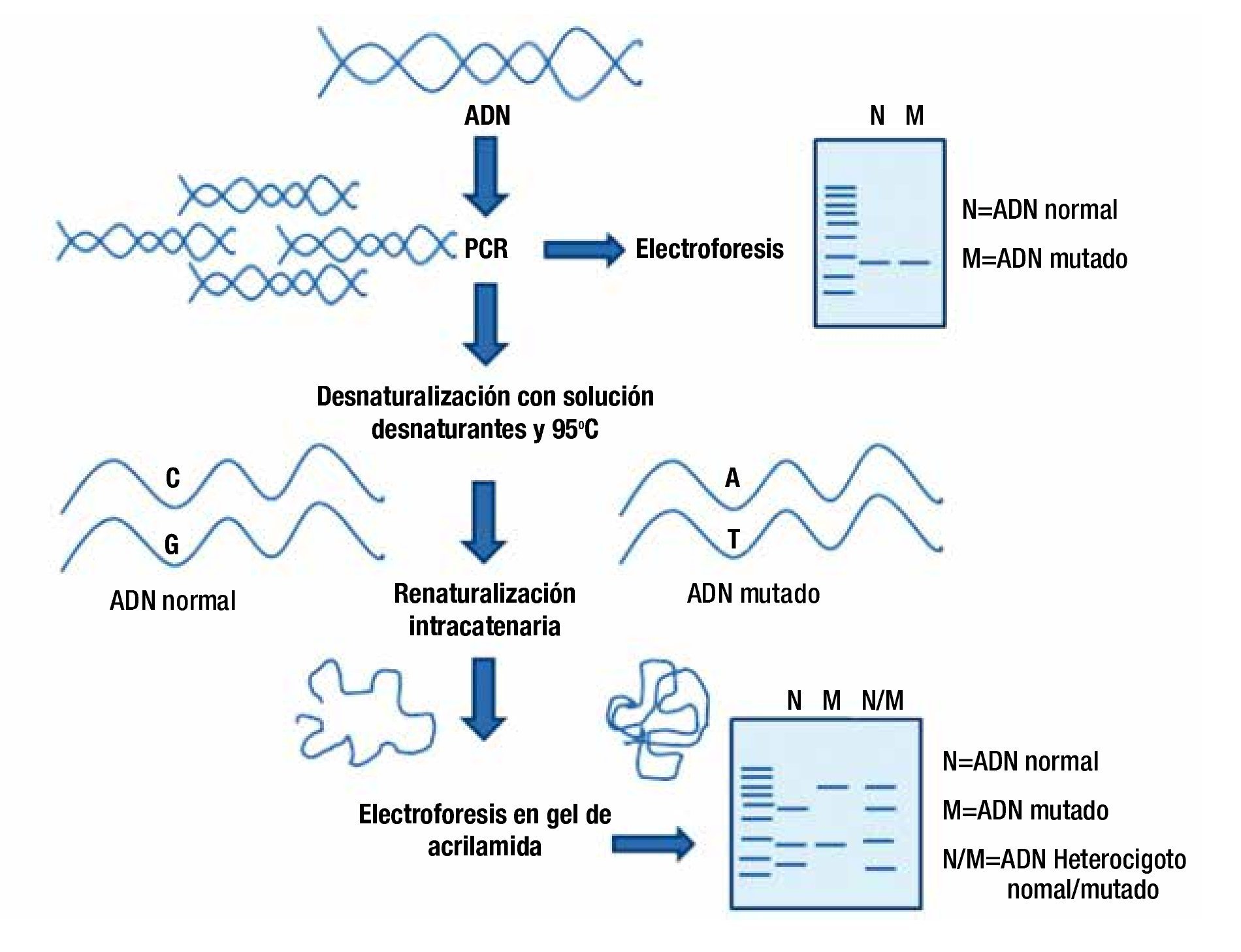
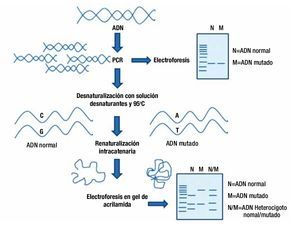
Es un proceso donde los productos de PCR son desnaturalizados en cadena simples de ADN, luego renaturalizados para favorecer los apareamientos intracatenarios y finalmente, analizados en un gel no desnaturalizante de poliacrilamida. De esta manera, la estructura de cada hebra de ADN de un amplicón adoptará una estructura secundaria que depende de su secuencia nucleotídica. Debido a que las mutaciones puntuales modifican la conformación y alteran la movilidad electroforética, la migración en el gel del ADN silvestre será distinta a la del ADN que contiene la mutación (fig. 4). Así, 2 productos de PCR con diferencias puntuales en su secuencias, presentarán distintos patrones electroforéticos de los fragmentos de ADN monocatenarios.
Figura 4. Esquema general de la técnica de PCR-SSCP (polimorfismo conformacional de una sola cadena). En este método se amplifica la región de ADN a analizar, las dobles cadenas se desnaturalizan y posteriormente se favorece una renaturalización intracatenaria, las diferencias en la secuencia de cada cadena le dan una conformación distinta a cada una de ellas, modificando el patrón de electroforesis.
Para que esta técnica sea eficiente y específica, es esencial la optimización de las condiciones. Los factores críticos incluyen las condiciones del corrimiento electroforético, especificidad de los iniciadores para la PCR, controles positivos y negativos.
Una de las principales aplicaciones de esta técnica en oncología es la identificación de mutaciones en el gen TP53, que codifica para una proteína nuclear involucrada en la regulación del ciclo celular, reparación de ADN y apoptosis (la proteína p53). Cuando el ADN se daña, p53 bloquea la división celular en la fase G1, antes de entrar a la fase de síntesis, para que no se duplique el material genético afectado. Si el daño al ADN no puede repararse, p53 activa mecanismos que conducen a la célula hacia apoptosis. Además, la proteína p53 actúa como un factor de transcripción que se une a más de 300 diferentes elementos promotores en el genoma32-34.
La mutación germinal en el gen supresor de tumores TP53 causa el síndrome de Li-Fraumeni, un síndrome de cáncer familiar que predispone al desarrollo de varios tipos de cáncer, incluyendo leucemia, sarcomas óseos y de tejidos blandos, cáncer de mama, carcinoma suprarrenal y tumores cerebrales. Su identificación permite brindar consejo genético a familias, que presentan esta afectación35.
Debido a que el gen TP53 está implicado en diversos tipos de cáncer, su detección se ha propuesto como un marcador temprano en la evolución de diferentes neoplasias, además de que el análisis de mutaciones de TP53 puede ser útil en el seguimiento después del tratamiento35,36.
Análisis de los fragmentos de restricción (RFLP, Restriction Fragment Lenght Polymorphims)
Esta modalidad, basada también en la técnica de PCR, tiene diversas aplicaciones, que incluyen el análisis de huellas genéticas y pruebas de paternidad. Es muy utilizada en la detección de una gran variedad de polimorfismos genéticos, principalmente SNP (polimorfismos de un solo nucleótido), asociados muchos de ellos a la susceptibilidad a padecer diferentes enfermedades y que pueden ser biomarcadores de riesgo, pronóstico, y respuesta a determinado tratamiento.
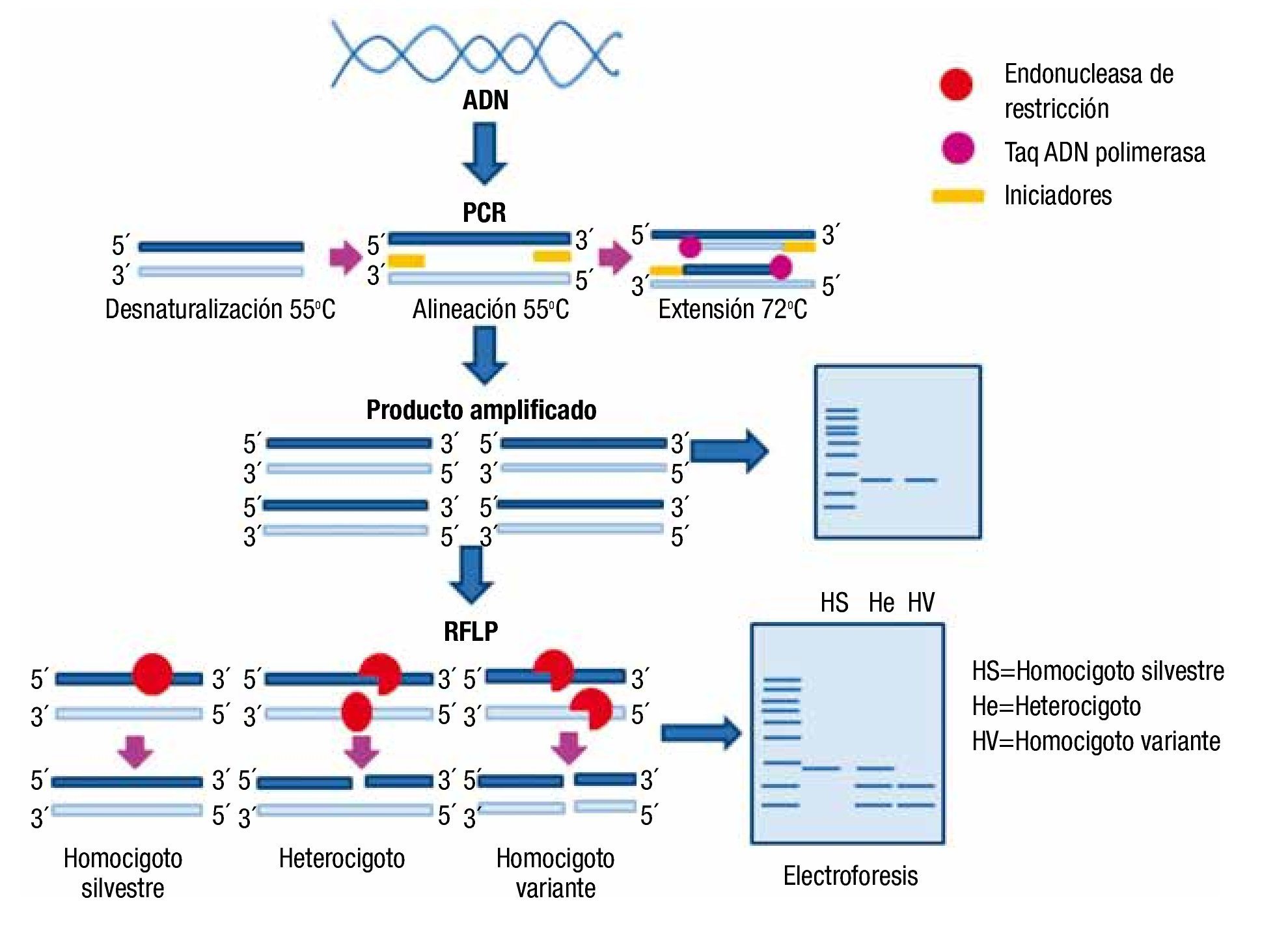
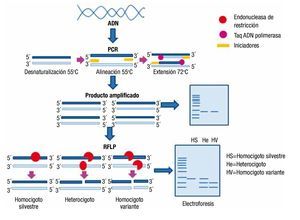
Mediante esta técnica es posible amplificar millones de veces una secuencia específica, en donde esté contenido un sitio de restricción determinado. Una vez amplificada la región, el producto de PCR se digiere con la enzima de restricción adecuada, posteriormente los productos de restricción (fragmentos) se visualizan directamente mediante electro-foresis en gel (fig. 5). Si 2 moléculas de ADN son cortadas con la misma enzima de restricción y difieren en un nucleótido, reconocido o no por esta enzima, la molécula resulta en fragmentos de diferente tamaño para cada individuo, esta diferencia en la longitud de los fragmentos de restricción es lo que se conoce como RFLP. Para emplear esta modalidad de PCR es necesario que la variante genética que se pretende identificar, genere o suprima un sitio de restricción para alguna endonucleasa conocida3,37.
Figura 5. Análisis de polimorfismos por medio de PCR-RFLP. Se realiza la amplificación de un fragmento de ADN, posteriormente el producto de PCR es digerido con una enzima de restricción adecuada. En este caso la enzima reconoce el alelo variante generando 2 fragmentos, en el alelo silvestre se suprime el sitio de restricción (corte), por lo que el amplificado permanece sin digerir. Posteriormente, se separan los fragmentos por medio de electroforesis, obteniendo un patrón de bandas específico para cada genotipo.
Como ejemplo de la utilidad de esta técnica en Oncología, está la determinación de las variantes genéticas del gen TPMT (Tiopurina S-Metil Transferasa), ya que la proteína para la que codifica este gen es una enzima que participa en el metabolismo de las tiopurinas como es la 6-Mercatopurina, medicamento utilizado en el tratamiento de pacientes con diversas neoplasias como es la LLA. Diversos SNP en este gen producen variantes con una baja actividad enzimática. Individuos portadores de estas variantes genéticas pueden presentar mielosupresión grave o fatal con dosis estándar de 6-Mercaptopurina. La identificación prospectiva de este grupo de pacientes puede prevenir complicaciones graves38-41.
Esta técnica tiene la ventaja de requerir menor tiempo y cantidad de ADN, en comparación con el análisis de Southern. Es fácil de diseñar y no requiere instrumentos muy sofisticados. Además, implica menor costo en comparación con tecnologías como la secuenciación de ADN, sin disminuir con esto la confiabilidad de los resultados.
Análisis de quimerismo molecular
Una de las aplicaciones de la técnica de PCR en Oncología es el análisis de quimerismo, en pacientes sometidos a trasplante alogénico de células progenitoras hematopoyéticas, siendo una herramienta importante en el monitoreo de estos pacientes. El estado de quimera es uno de los factores que determinan el curso del periodo postrasplante, y la posibilidad de rechazo o recaída. En el pasado no era posible establecer el tipo de quimerismo, ya que se utilizaban técnicas con poca sensibilidad y marcadores con bajo grado de polimorfismo, que requerían que el donante y el paciente fueran de diferente sexo. El análisis de secuencias polimórficas del ADN constituye un método informativo y versátil para poder distinguir entre las células del donante y del receptor, su aplicación es independiente de la identidad del sexo.
En la actualidad, el análisis de quimerismo se realiza mediante la amplificación por PCR en el paciente y donador, de pequeñas zonas del ADN altamente polimórficas, como los VNTR (número variable de repeticiones en tándem) o STR (repeticiones cortas en tándem), con el objetivo de analizar varios de ellos y encontrar alguno diferente al que se le denomina marcador informativo, y que es útil para el estudio de quimerismo. Dentro de los marcadores más utilizados, se encuentran los genes D1S80, HUMARA, TH01, D17S30,D1S111 y APO-B42.
Una de las ventajas de la técnica de PCR en el análisis de quimerismo es la alta sensibilidad, que permite detectar pequeñas poblaciones de células del donante o del receptor. Con el uso de esta técnica, es posible tener evidencias de un injerto establecido antes que las evidencias morfológicas aparezcan43-45.
Además, la utilización de esta técnica ha permitido realizar estudios de la evolución de la quimera y relacionar el grado de quimerismo, establecido con el comportamiento del injerto y de la enfermedad de injerto contra hospedero (EICH) en los diferentes regímenes de acondicionamiento. También ha posibilitado la detección precoz de la recaída en los pacientes trasplantados, y la administración oportuna de inmunoterapia adicional46-48.
FISH (Hibridación fluorescente in situ)
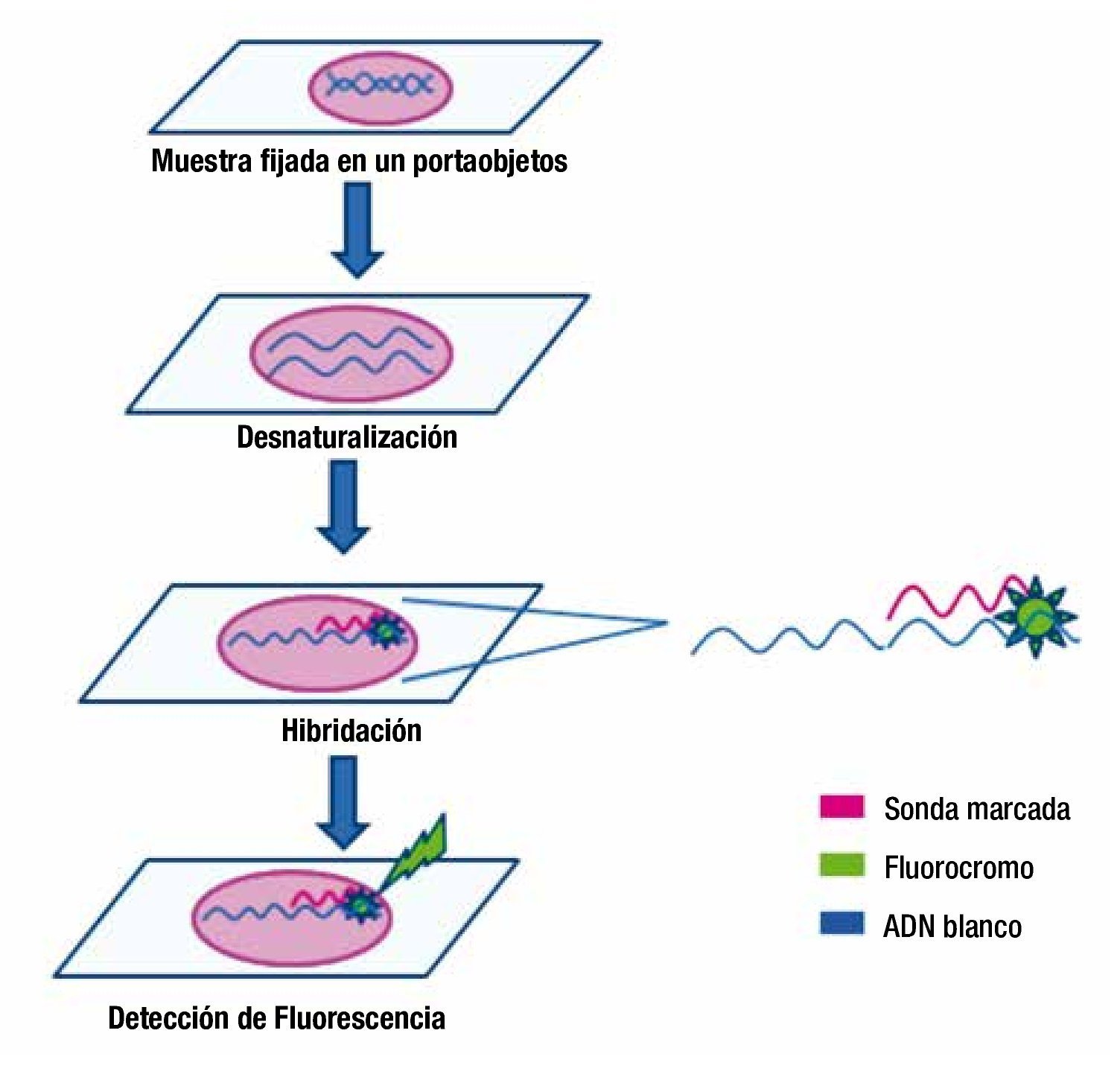
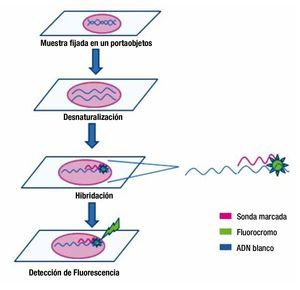
La hibridación fluorescente in situ (FISH) es un método molecular mediante el cual se detecta la cantidad, el tamaño y la ubicación de los segmentos de ADN y ARN, dentro de las células individuales en una muestra de tejido. Se basa en la capacidad del ADN de cadena simple para hibridar con ADN complementario. En esta técnica la muestra de ADN (cromosomas metafásicos o núcleos en interfase), se somete a un proceso de desnaturalización para separar las cadenas de la molécula, y una vez desnaturalizada se adiciona una sonda de ADN marcada con un fluoróforo, la cual se une al sitio específico del ADN. La luz emitida por la sonda se observa mediante un microscopio de fluorescencia (fig. 6). De esta manera se confirman o descartan anormalidades génicas o cromosómicas4,49.
Figura 6. Hibridación fluorescente in situ (FISH). Empleando una muestra de tejido fijada en un portaobjetos se desnaturaliza el ADN dentro de las células del tejido a analizar, se adiciona la sonda marcada con un fluorocromo, la cual se unirá a su secuencia complementaria en el ADN blanco cuando esté presente la alteración genética o cromosómica buscada. La luz emitida por la sonda se observa mediante un microscopio de fluorescencia.
En comparación con la citogenética, la prueba de FISH tiene la ventaja de poder identificar los cambios genéticos que son demasiado pequeños para ser vistos con un microscopio. Otra ventaja es que no tiene que ser efectuada en las células que se están dividiendo activamente. Otras pruebas sólo pueden realizarse hasta que las células hayan crecido en cultivo durante unas semanas y el proceso suele ser muy prolongado. Con la técnica de FISH los resultados pueden obtenerse en unos días. Además, la técnica de FISH es más sensible y confiable que la citogenética convencional, y es posible utilizar varios fluorocromos para discriminar blancos diferentes simultáneamente 6,50-52.
La prueba de FISH sirve para detectar anomalías genéticas relacionadas con el cáncer, es útil para el diagnóstico de algunos tipos de la enfermedad. En algunos casos, cuando el tipo de cáncer ha sido previamente diagnosticado, una prueba FISH también puede proporcionar información para ayudar a predecir el resultado de un paciente y si es probable que respondan a los fármacos de quimioterapia. La técnica de FISH en oncología pediátrica puede identificar anomalías cromosómicas en leucemias, tales como t(9;22), t(4;11), t(12;21), entre otras, algunas de las cuales están asociadas con formas agresivas de la enfermedad. También puede detectar microdeleciones, duplicación cromosómica y amplificación de genes. La amplificación del oncogén NMYC, en el neuroblastoma, puede ser determinada por PCR o FISH, se asocia con tumores agresivos y pobre respuesta al tratamiento, independiente de la edad y del estadio de la enfermedad, lo que ha llevado a usar protocolos terapéuticos más intensivos en este grupo de pacientes6,22,53,54.
La incorporación de esta técnica molecular al tratamiento clínico de pacientes pediátricos con leucemias, linfomas y tumores sólidos tiene la ventaja de identificar translocaciones críticas y reordenamientos cromosómicos complejos, y definir la procedencia de segmentos cromosómicos involucrados en gran número de reordenamientos. Su combinación con otras técnicas como la RT-PCR ayuda en el análisis de la actividad de genes supresores de tumores y oncogenes en núcleos interfásicos de tejidos tumorales, proporcionando grandes ventajas diagnósticas y terapéuticas55.
Cariotipo espectral (SKY)
El análisis espectral de cariotipos (o SKY) es una herramienta de la citogenética molecular, que permite la visualización y estudio de los 23 pares de cromosomas humanos simultáneamente. En esta técnica cada par de cromosomas se marca con diferentes colores fluorescentes, mediante sondas de ADN. Cada sonda es marcada con una molécula fluorescente que corresponde con el cromosoma, del que es complementario. La discriminación por el color se hace mediante el análisis espectral de las señales fluorescentes. Mediante el uso de esta metodología se observa claramente cuando un fragmento de determinado cromosoma, se marca con un color distinto. Esta técnica es útil para identificar cambios en la morfología de los cromosomas, incluso alteraciones cromosómicas complejas, detectando con mayor facilidad y precisión translocaciones, inversiones, deleciones, etc., en comparación con el cariotipo y citogenética convencional56,57.
El análisis espectral de los cariotipos ha mejorado el diagnóstico y detección de alteraciones cromosómicas en células cancerosas, y ha facilitado la identificación de los loci de genes implicados en la carcinogénesis. Lo que aporta información importante con valor diagnóstico y pronóstico en neoplasias hematológicas y tumores sólidos58,59.
El conocimiento de las alteraciones cromosómicas asociadas a una determinada neoplasia permite disponer de marcadores útiles en la definición del pronóstico, en la evaluación de la respuesta a tratamiento y en el seguimiento de la enfermedad. La detección y cuantificación de la enfermedad mínima residual (EMR) en pacientes con LLA, es un ejemplo de cómo los marcadores moleculares pueden ser utilizados para todos estos propósitos60,61.
Microarreglos de ADN
Los microarreglos de ADN son una herramienta utilizada para el estudio de expresión de genes, identificación de marcadores genéticos y análisis de mutaciones en diversas enfermedades. Un microarreglo se compone de 2 partes, el material biológico o sintético denominado prueba y el soporte sólido, que puede ser plástico, vidrio, gel, silicón, membranas porosas y oro, en el cual se inmoviliza el material biológico. El trabajo con microarreglos inicia con la selección del tipo de microarreglo y de las secuencias que se colocarán en el soporte, después viene la obtención de la muestra biológica y purificación del ADN o ARN. El material genético extraído se amplifica por medio de la técnica de PCR, para posteriormente realizar el marcaje de la muestra y la hibridación del microarreglo, captura de la señal, interpretación y análisis del resultado62.
Los microarreglos de ADN tienen la ventaja de que permiten analizar un gran número de secuencias, aproximadamente 48,000 genes de manera simultánea en cada ensayo. Este análisis simultáneo de la expresión génica de miles de transcripciones, ha acelerado significativamente el avance en la comprensión de la base molecular de diversas neoplasias.
Durante los últimos años, el análisis de muestras de LLA con matrices de ADN ha facilitado el reconocimiento de grupos de leucemia molecularmente distintos. El avance en el conocimiento de los mecanismos de la sensibilidad y resistencia a la quimioterapia, ha generado nuevas herramientas para la predicción del pronóstico y ha permitido la identificación de nuevos blancos moleculares para el desarrollo de agentes antineoplásicos. La implementación de microarreglos de ADN ha acelerado el progreso en el diagnóstico y tratamiento de LLA, que eventualmente se traducirá en el desarrollo de nuevos tratamientos, más eficaces y menos tóxicos para esta enfermedad62,63.
También ha contribuido a identificar y caracterizar algunas neoplasias que muestran dificultades diagnósticas particulares, tal es el caso de algunos linfomas de linfocitos B64.
En otros tipos de cáncer, el perfil de expresión génica ha permitido diseñar mejores esquemas de tratamiento y hacer predicciones de la respuesta a quimioterapia.
El poder de los microarreglos de ADN para predecir el pronóstico ha aumentado, con el uso simultáneo de los microarreglos de tejidos y microarreglos de proteínas62.
Secuenciación de ADN
Actualmente, la secuenciación automática de ADN se basa en la detección de fluorescencia específica para cada uno de los nucleótidos trifosfatos que forman el ADN (adenina, guanina, citosina y timina), los cuales pueden ser identificados al ser marcados con un fluorocromo distinto que emite luz de diferente longitud de onda, al excitarse con un rayo láser. Dicha fluorescencia es recibida por un detector que interpreta la señal generando un registro de los perfiles de color, que combinados se interpretan como una secuencia y los datos resultantes se dan como cromatogramas, que son gráficas que registran los picos de fluorescencia65. Este es el método que actualmente se utiliza para analizar cualquier tipo de variación en secuencias de ADN, únicamente se amplifica el fragmento deseado, se purifica para eliminar los restos de desoxinucleótidos trifosfatos, iniciadores, etc. y se somete al proceso de secuenciación. Es una técnica muy sensible y rápida, que requiere poca cantidad de muestra.
En el área de la Oncología, la secuenciación ha permitido analizar una gran cantidad de genes involucrados en procesos de carcinogénesis y en el comportamiento biológico de distintas neoplasias, como la resistencia a fármacos anti-neoplásicos.
Sin embargo, en nuestro país esta tecnología no se ha implementado aún como parte de los recursos diagnósticos de rutina y su uso prácticamente se limita a la investigación.
Espectrometría de masas aplicada a la proteómica
En la actualidad se han desarrollado técnicas de análisis múltiple de proteínas (proteómica), mediante espectrometría de masas (EM). Esta tecnología, permite el estudio simultáneo del conjunto de proteínas (proteoma) de una muestra biológica. Al igual que ocurre en el caso de la genómica, la proteómica aplicada al cáncer se ha centrado fundamentalmente en la búsqueda de marcadores proteicos diferencialmente expresados en tumores y nuevas dianas terapéuticas66.
El primer paso en cualquier estudio proteómico es la separación de proteínas de la muestra, mediante técnicas del tipo de la electroforesis bidimensional y/o la cromatografía en tándem. Esto con el fin de elegir las proteínas que, se encuentren aumentadas o disminuidas al comparar 2 muestras de interés, como pueden ser muestras de pacientes sanos y enfermos. El análisis por EM se basa en la digestión de las proteínas separadas en gel y el análisis de los péptidos, para determinar con gran precisión su masa y/o obtener su secuencia de aminoácidos67. Aunque la proteómica se está estableciendo como una técnica fundamental para la investigación del cáncer, no está aún clara su implementación a nivel clínico. La complejidad tecnológica, el alto costo del equipamiento necesario, su difícil automatización, junto a la poca disponibilidad de personal capacitado dificultan el traslado de proteómica a la práctica clínica rutinaria68.
Sin embargo y a pesar de todas las limitaciones mencionadas, algunos estudios presentados indican que el análisis proteómico del suero mediante EM, es una potente herramienta de investigación dirigida a la identificación de nuevos marcadores biológicos aplicables al diagnóstico clínico69.
Conclusiones
Los avances en el desarrollo y aplicación de nuevas tecnologías para el análisis molecular, han contribuido de forma significativa al entendimiento de la genética y patogénesis molecular del cáncer. Es así como el estudio y determinación de mutaciones y diversas variantes genéticas, nos permite identificar biomarcadores de riesgo para el desarrollo de un tipo específico de cáncer, con lo que se podría seleccionar población de riesgo y tomar medidas de prevención. También hace posible la identificación de marcadores genéticos de pronóstico, que han contribuido a la estratificación del tratamiento. Asimismo, el reconocimiento de marcadores de sensibilidad a un fármaco en particular, ha permitido al oncólogo ajustar la dosis de algunos agentes antineoplásicos, a fin de evitar efectos adversos graves, brindando al paciente una terapia individualizada.
En el futuro, la utilización de estas herramientas permitirá la identificación de nuevos biomarcadores útiles en el diagnóstico, definición del pronóstico y seguimiento de las distintas neoplasias, además del reconocimiento de nuevos blancos moleculares para el desarrollo de fármacos anti-neoplásicos con mayor actividad antitumoral, mayor selectividad y con menores efectos adversos.
Financiamiento
No se recibió ningún patrocinio para la realización de este artículo.
Conflicto de intereses
Los autores declaran no tener conflicto de intereses.
* Autor para correspondencia:
Laboratorio de Oncología, Hospital Infantil de México Federico Gómez.
Dr. Márquez N° 162, Colonia Doctores, Delegación Cuauhtémoc, C.P. 06720, México D.F., México.
Teléfono: 5228 9917, ext. 2357.
Correo electrónico: artur_tauro@yahoo.com.mx (Arturo Ramírez-Pacheco).