Diariamente los seres humanos están en interacción con objetos de uso continuo, como el papel moneda, sin el conocimiento de que estos almacenan microorganismos y de que nos exponemos al contacto con potenciales patógenos. La composición de la comunidad bacteriana en un billete colombiano fue determinada mediante el secuenciamiento profundo de librerías de amplicones 16S. Se encontraron 233 géneros bacterianos; 12 de estos géneros corresponden a especies con potencial patogénico. El género más abundante fue Propionibacterium, seguido de Streptococcus, Staphylococcus y Pseudomonas.
Este es el primer reporte de la diversidad bacteriana que puede ser alojada en este objeto de alta circulación en Colombia. Pocos estudios en el mundo han mostrado este nivel de detalle de la microbiota en billetes de circulación y ofrece un panorama mucho más amplio de la exposición diaria a microorganismos al utilizar papel moneda en las condiciones en las que se utiliza en Colombia.
Commonly used objects such as currency paper can be colonised by bacteria and can serve as carriers of microbes. This colonisation might expose us to unnoticed pathogenic bacteria. In this study, the researchers obtained a detailed panorama of the microbes that can be carried on currency notes in Colombia by using 454 next-generation deep sequencing of 16S amplicón libraries. A total of 233 bacterial genera were detected and classified, 12 of which are potential human pathogens. The most abundant genera were Propionibacterium, Streptococcus, Staphylococcus and Pseudomonas. To date, this is the first in-depth analysis of the microbiota carried by circulating banknotes in our continent and it offers insights into daily exposure to microbes when using banknotes in Colombia.
Desde que Luis Pasteur en 1859 refutó la teoría de la generación espontánea, fue evidente que los microorganismos se encuentran, establecen y desarrollan en diferentes hábitats. Actualmente, es claro que la diversidad bacteriana es amplia y está determinada por su potencial genético, que permite una versatilidad metabólica de estos organismos, que a lo largo del tiempo, les ha permitido establecer una serie diversa de nichos. De esta diversidad microbiana se conoce muy poco debido a que aproximadamente el 1% ha sido cultivado en las condiciones de laboratorio dada la limitación en las metodologías de cultivo1. Se estima que en un gramo de suelo es posible encontrar una abundancia de 106-109 células bacterianas en sitios prístinos, es decir entre 2.000 y 8.300.000 especies bacterianas2,3.
Se estima que el número total de células de un ser humano está alrededor de un billón, mientras que 10 billones corresponden a las bacterias, que se desarrollan en el intestino o en la piel4. El proceso de adquisición de la microbiota oral, intestinal y cutánea se encuentra bien documentado y es la microbiota materna la que inicialmente coloniza los tejidos del recién nacido durante el nacimiento, la lactancia y la interacción diaria en los cuidados rutinarios del neonato5. Los niños cuyo nacimiento se produce atravesando el tracto vaginal adquieren poblaciones de Lactobacillus spp., Prevotella spp., o Sneathia spp., microbiota normal de la vagina de la madre, y cuando el nacimiento se realiza mediante cesárea los niños adquieren especies que se encuentran comúnmente en la piel de la madre, específicamente, Corynebacterium spp., Propionibacterium spp. y Staphylococcus spp.6. Asi mismo, posteriores modificaciones de esta se han asociado sobre todo a la dieta y a la exposición a microorganismos presentes en los alimentos7. En el caso de la piel, la microbiota se modifica según las condiciones abióticas de cada zona del cuerpo (axilas, conductos, pliegues) de acuerdo con variables como temperatura, concentración de sal, etc. Igualmente es influida por el contacto constante con diferentes objetos, animales u otros humanos4. Uno de los elementos que continuamente pasa de mano en mano es el papel moneda, el cual es inoculado con la microbiota de las manos de quienes lo manipulan. En Colombia el papel moneda está compuesto por algodón 100%, siendo así una matriz absorbente y favorable para la colonización y mantenimiento de microorganismos. Las condiciones ambientales propias del trópico donde se encuentra localizado el país, la sudoración del usuario y las variadas condiciones de almacenamiento también permiten que el billete sea hábitat de bacterias y hongos. Estudios previos desarrollados en la ciudad de Medellín en billetes de diferente denominación, utilizando métodos de cultivo, detectaron 9 géneros bacterianos dentro de los que se cuentan: Bacillus, Staphylococcus, Enterococcus, Streptococcus, Pantoea, Escherichia, Acinetobacter, Enterobacter y Klebsiella8. Un estudio desarrollado en Nigeria en 2011, mostró que el 50% de los billetes estudiados estaban colonizados con E. coli9. En un estudio similar realizado en México se reportó que el 69% de los billetes presentaron bacterias patógenas o potencialmente patógenas10.
El objetivo de este estudio fue conocer la estructura de la comunidad microbiana residente en un billete colombiano de baja denominación utilizando métodos independientes de cultivo mediante el secuenciamiento profundo y determinar la presencia de poblaciones potencialmente patógenas para los seres humanos.
Material y métodoPapel monedaSe seleccionaron 2 billetes deteriorados de 1.000 pesos colombianos ($ 1.000) los cuales tenían como últimos números de su serie 908 (acuñado el 15 agosto de 2007) y 115 (acuñado el 28 agosto del 2008). La revisión del estado físico del billete mostró evidencias de decoloración y pérdida de la rigidez. Los billetes fueron obtenidos en 2 cafeterías universitarias de la ciudad de Medellín.
Extracción de ADNEl procedimiento para la extracción de ADN total se desarrolló empleando el kit de extracción para bacterias retenidas en papel de filtro de MoBio (PowerWater DNA Isolation Kit CAT# 14900). A partir de la parte central de cada billete se recortó un fragmento de 27mm de diámetro aproximadamente y se siguieron las indicaciones del kit para la obtención del ADN.
Amplificación y purificación de las regiones variables V1-V3 y V6-V9 y pirosecuenciamientoPara la amplificación por PCR se utilizaron oligonucleótidos descritos más abajo, con los adaptadores recomendados por Roche 454 para librería tipo «L» y códigos de barras de 10 bases (recomendados por Roche).
Iniciador (secuencia 5′- 3′)Oligo V1-V3, forward, adaptador A: CCA TCT CAT CCC TGC GTG TCT CCG ACT CAG AGA CTC GAC GTA TTA CCG CGG CTG CTG G.
Oligo V1-V3, reverse, adaptador B: CCT ATC CCC TGT GTG CCT TGG CAG TCT CAG AGA GTT TGA TCC TGG CTC AG.
Oligo V6-V9, forward, adaptador A: CCA TCT CAT CCC TGC GTG TCT CCG ACT CAG AGT GTA GTA GTA AAC TYA AAK GAA TTG ACG G.
Oligo V6-V9, reverse, adaptador A: CCT ATC CCC TGT GTG CCT TGG CAG TCT CAG ACG GGC GGT GTG TRC.
La reacción se realizó durante 35 ciclos, así: 95°C un minuto, 55°C 30 s y 72°C 45 s. La amplificación fue verificada por electroforesis en gel de agarosa y los amplicones fueron purificados con el kit Ampure XT (Bechman Coulter). Los productos purificados fueron cuantificados por fluorimetría con Picogreen y la pureza fue evaluada por electroforesis capilar en el equipo Bioanalyzer 2100 (Agilent) con el kit High Sensitivity DNA.
Los amplicones purificados fueron titulados y posteriormente secuenciados en un GS FLX 454 (Roche) con la tecnología Titanium en un octavo de plato aproximadamente. Este procedimiento se realizó en el Centro Nacional de Secuenciación Genómica (CNSG) de la Universidad de Antioquia (Medellín, Colombia).
Procesamiento de las lecturas (reads)El tratamiento de las lecturas para eliminar el ruido introducido durante la reacción de PCR y durante la pirosecuenciación se realizó empleando el paquete Amplicon Noise versión 1.27. Para este proceso el software fue alimentado con los archivos con extensión .sff emitidos como resultado del software del 454. Posteriormente, se aplicó el software Amplicon Noise11 que selecciona las lecturas que no presenten mutación en el código de barras o en el oligonucleótido usado para la amplificación. Finalmente, se eliminaron los reads con tamaños inferiores a 250 bases. Para los análisis de asignación taxonómica se emplearon las secuencias depuradas que estuvieron soportadas por un mínimo de 5 lecturas limpias.
Clasificación taxonómica de las secuenciasPara la asignación taxonómica de cada una de las secuencias depuradas se utilizó el programa Classiffier de la plataforma Ribosomal Database Project (RDP), de la Universidad de Michigan (http://rdp.cme.msu.edu) con un valor mínimo de confianza de 0,8 y un bootstrap ≥ 0,8. Las tablas de clasificación obtenidas fueron procesadas utilizando los programas de Python y Shell desarrollados en el CNSG para resumir y tabular los resultados en las categorías taxonómicas de phylum, clase, orden, familia y género.
ResultadosProcesamiento de las lecturas obtenidasSe obtuvo un total 108.936 y 56.846 lecturas brutas del rDNA 16S para las 2 regiones estudiadas, V1-V3 y V6-V9, respectivamente. La corrección de errores introducidos por la reacción de PCR o por el pirosecuenciamiento se llevó a cabo en el paquete Amplicon Noise v. 1.27. El resumen final de este procesamiento computacional reportó un total de 3.767 secuencias depuradas para la región V1-V3, que representaron un total de 40.340 lecturas limpias. Se detectaron 1.248 quimeras que fueron excluidas del grupo de datos. Para la región V6-V9 se obtuvo un total de 3.546 secuencias depuradas, que representan un total de 32.109 lecturas limpias y se removieron del conjunto de datos 1.414 quimeras. Para brindar mayor soporte a los datos en los pasos analíticos posteriores, se eliminaron las secuencias depuradas que estuvieran soportadas por menos de 5 lecturas limpias. Las secuencias que superaron ese filtro se emplearon para el análisis de clasificación taxonómica: 419 secuencias para la región V1-V3 y 477 para la región V6-V9.
Clasificación taxonómicaPara la clasificación taxonómica se utilizó el programa Classifier de la plataforma RDP. Los resultados obtenidos fueron formateados y tabulados según la frecuencia de cada grupo y el número de lecturas limpias asignadas en cada categoría taxonómica.
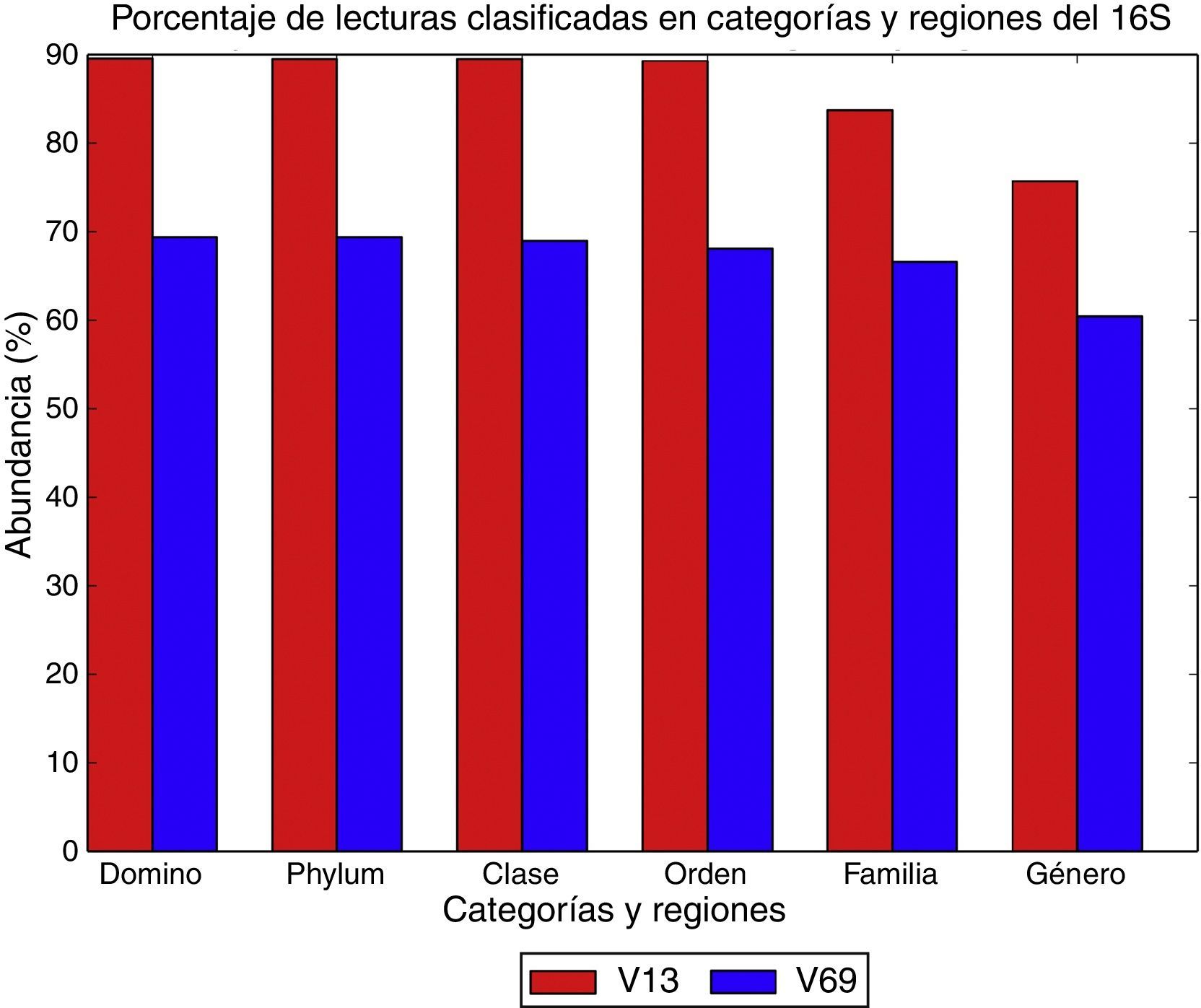
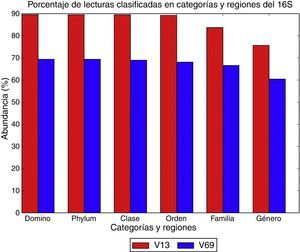
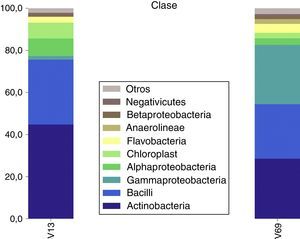
Con el amplicón de la región V1-V3 se clasificaron 36.130 lecturas limpias (89,6%) a nivel del dominio bacteria, de las que 36.107 (89,5%) fueron asignadas a un phylum. El umbral mínimo de valor de bootstrap fue de 0,8. La proporción de secuencias clasificadas se mantuvo en porcentajes similares hasta la categoría de familia y género, donde disminuyó al 83,7 y al 75,6%, respectivamente (fig. 1). Empleando los amplicones de la región V6-V9, se obtuvieron resultados considerablemente diferentes. De las 32.109 lecturas limpias, 22.268 fueron clasificadas a nivel de dominio (bootstrap ≥ 0,8), es decir el 69,4%. Todas las lecturas correspondieron al dominio bacteria y fueron asignadas a nivel de phylum. A nivel de familia, las secuencias clasificadas se redujeron al 66,5 y 60% para género. Probablemente este fenómeno se puede presentar debido a que con este amplicón se tuvieron una cantidad importante de lecturas limpias que fueron asignadas al dominio Archea (6.204 lecturas, 19,3%), pero ninguna de estas superó el umbral de corte de bootstrap en la asignación taxonómica (fig. 1).
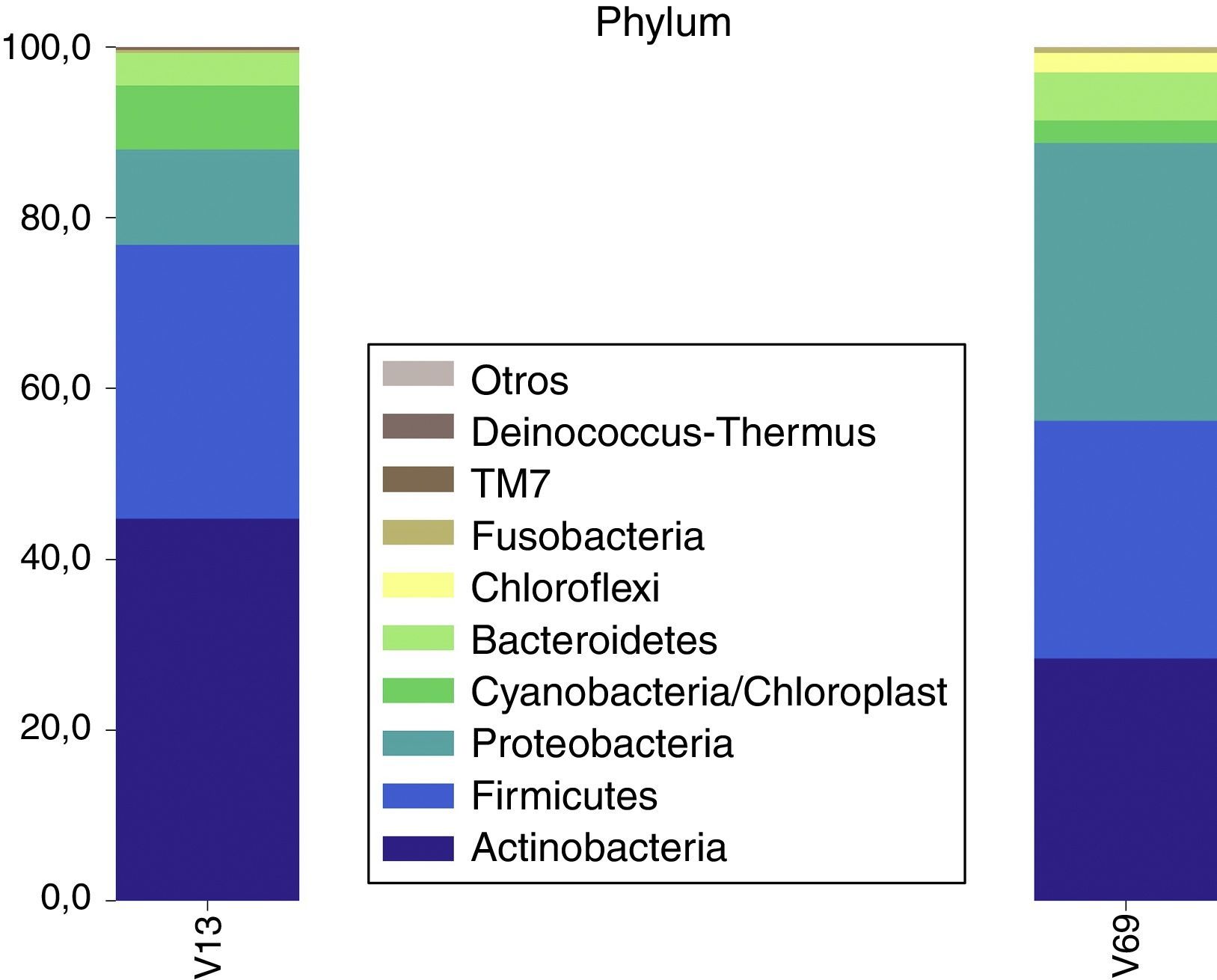
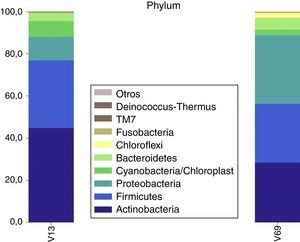
A partir de la región V1-V3 a nivel de phylum se determinó que los más abundantes fueron Actinobacteria y Firmicutes, seguidos de Proteobacteria, Cyanobacteria/Cloroplasto y Bacteroidetes. Los phyla detectados con menor representación fueron Fusobacteria, TM7, Deinococcus-Thermus y SR1. Con los reads de la región V6-V9, los 3 phyla más abundantes coincidieron con la región V1-V3, aunque en diferente orden de abundancia, Proteobacteria, Actinobacteria y Firmicutes, los phyla menos abundantes fueron los mismos Bacteriodetes, Cyanobacteria/Cloroplasto y Chloroflexi y también en menor proporción Fusobacteria y Synergistetes (fig. 2). Los oligonucleótidos utilizados para esta región (V6-V9) permitieron amplificar la región correspondiente del gen ribosomal 16S en Archea pero con valores inferiores de bootstrap (>0,8), por lo cual fueron eliminadas del estudio.
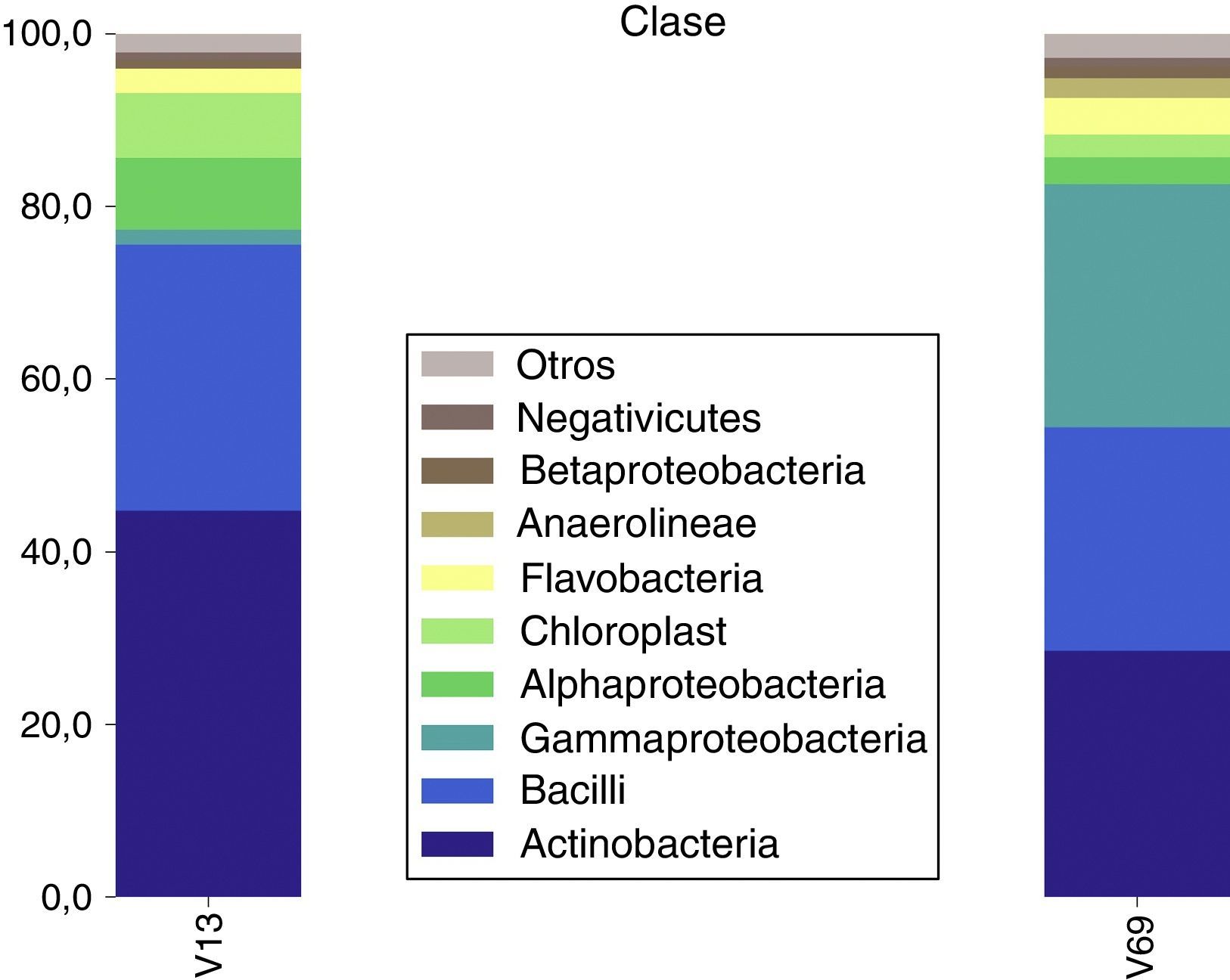
Para la categoría taxonómica clase se observó con ambos amplicones que el grupo dominante fue Actinobacteria con 16.170 y 6.317 lecturas limpias asignadas para las regiones V1-V3 y V6-V9, respectivamente. Todas las lecturas asignadas al phylum Actinobacteria correspondieron a la clase del mismo nombre. El phylum Proteobacteria estuvo representado por las clases Alfaproteobacteria, Gammaproteobacetria y Betaproteobacteria.
A partir del amplicón de la región V1-V3 se determinaron lecturas de la clase Epsilonproteobacteria. La clase Bacilli, dominante del phylum Firmicutes, representó la segunda abundancia para la región V1-V3 y la tercera para V6-V9. Otra clase que se determinó y que pertenece a este phylum correspondió a Clostridia, reportada dentro de las clases con menor abundancia (fig. 3).
Con ambas regiones estudiadas, las clases Flavobacteria, Bacteroidia y Sphingobacteria presentaron las mayores abundancias del phylum Bacteriodetes (fig. 3). Sin embargo, la mayoría de las lecturas asignadas a este phylum no fueron clasificadas con el punto de corte establecido (0,8), lo que no permite hacer un análisis detallado de este grupo.
Dentro del grupo clasificado por la RDP como Cyanobacteria/Chloroplast, se encontró que la mayoría de los reads se clasificaron como pertenecientes a cloroplasto en ambas regiones estudiadas. Solamente para el amplicón de la región V1-V3 se detectaron 9 lecturas asignadas al grupo de las Cyanobacteria.
La clase Fusobacteria es la única representante de su respectivo phylum y con una abundancia menor en ambas regiones. El amplicón de la región V6-V9 detectó bacterias de la clase Anaerolineae, las cuales constituían el 100% de las lecturas asignadas para su phylum. Siguiendo este mismo grupo en las categorías taxonómicas más bajas, se observó que todas estas se clasificaron en la familia Anaerolineaceae (figs. 3-5).
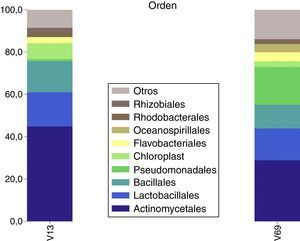
A nivel de clase, ambos amplicones coincidieron en mostrar a los grupos Actinomycetales, Lactobacillales, Bacillales y Flavobacteriales como los grupos más abundantes. Sin embargo, con el amplicón V6-V9, la clase Pseudomonadales se ubicó segunda en el ranking, en discordancia con el amplicón V1-V3 que la ubicó en la décima posición (fig. 3).
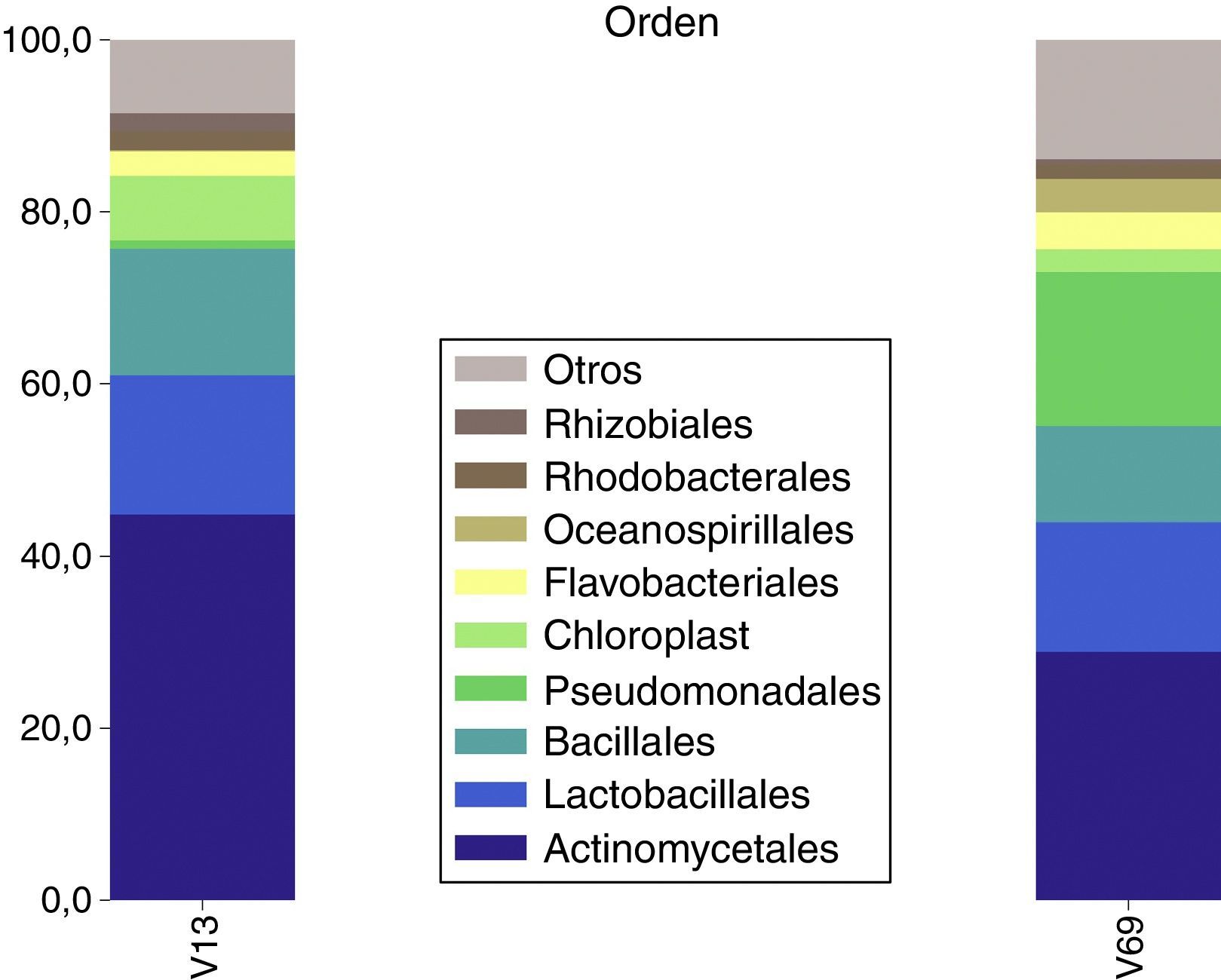
Los órdenes más abundantes detectados por ambos amplicones fueron Actinomycetales, Lactobacillales, Bacillales y Flavobacteriales. Con la excepción de que el orden Pseudomonadales subió a la segunda posición del ranking en el amplicón V6-V9 (fig. 4).
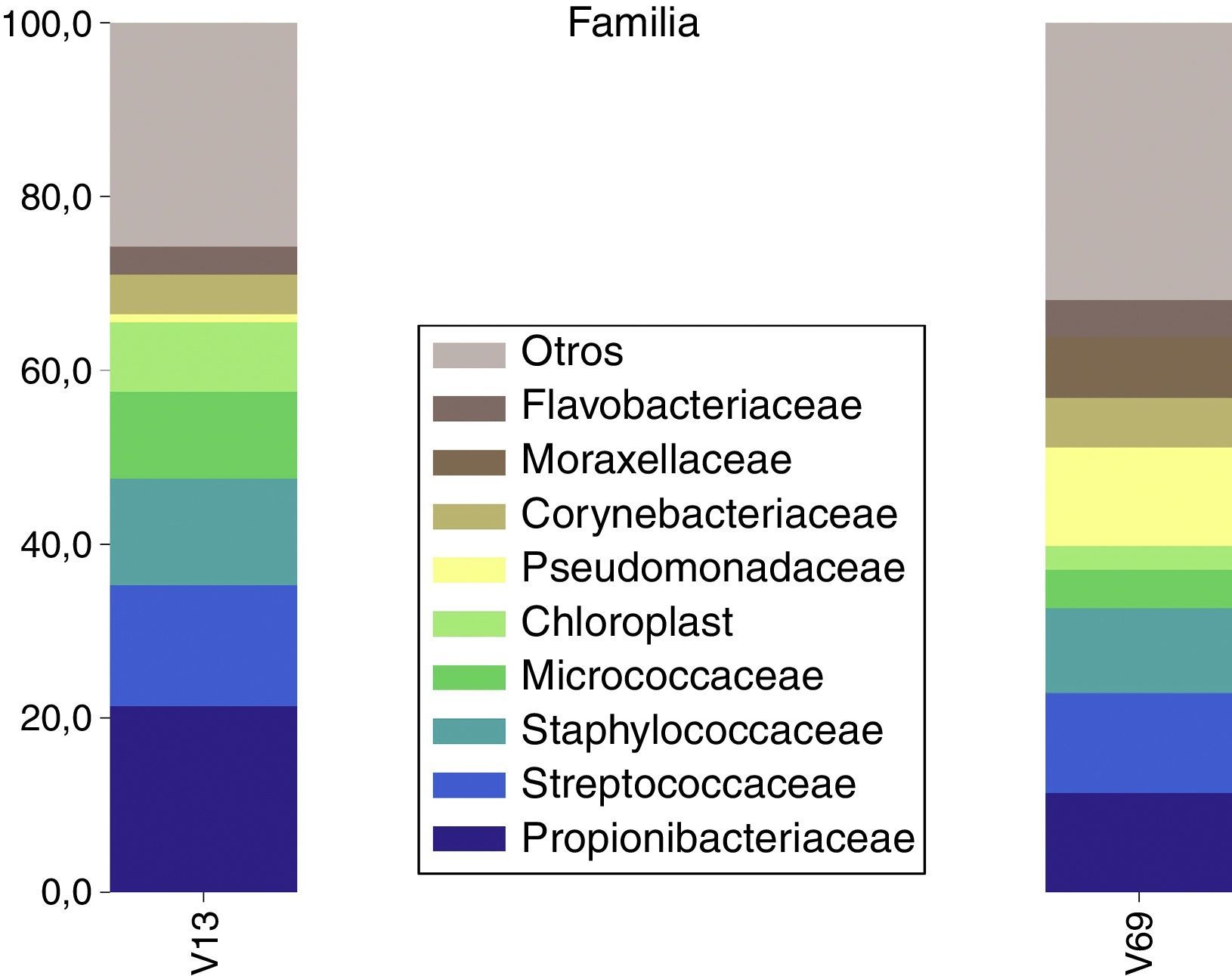
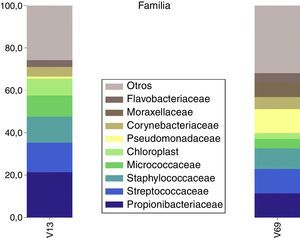
Para la categoría familia igualmente se observó una buena concordancia, ya que de manera similar se detectaron con ambos amplicones que los grupos más abundantes fueron Propionibacteriaceae, Streptococcaceae, Staphylococcaceae, Micrococcaceae y Corynebacteriaceae (fig. 5). Estas familias representaron, respectivamente, el 62 y el 43% de las lecturas limpias de la región V1-V3 y V6-V9. Nuevamente, el grupo Pseudomonadaceae no coincidió en el ranking de ambos amplicones, ya que para el V6-V9 es el 2.° en abundancia mientras que para el V1-V3 está en la posición 18. Igualmente ocurrió para la familia Moraxellaceae, la cual fue 5.ª en el amplicón V6-V9 y la 44.ª con la región V1-V3.
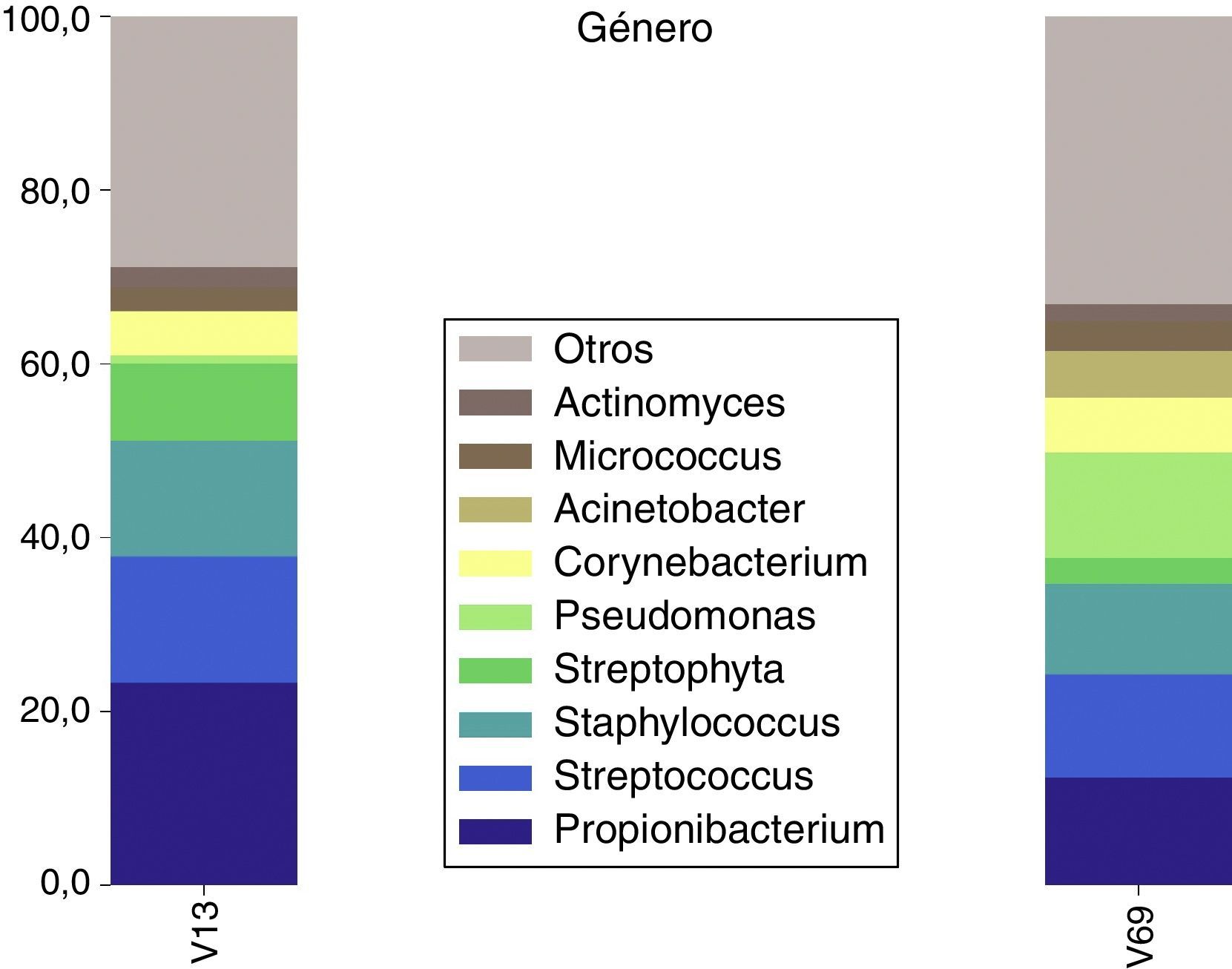
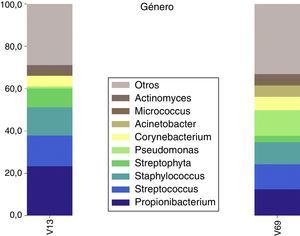
El análisis de los amplicones derivados en la región V1-V3 logró detectar 127 géneros, mientras que en la región V6-V9 logró detectar 109. De estos, se detectaron entre ambas un total de 233 en común. Con ambas regiones del rDNA 16S analizadas, se observó que el organismo más abundante es Propionibacterium, el cual acumula el 23% de las lecturas limpias de la región V1-V3 y el 12% de la región V6-V9. Asimismo, fueron comúnmente detectados con ambas regiones del rDNA 16S los géneros Streptococcus, Staphylococcus, Corynebacterium y Micrococcus. Estos acumularon el 36% de las lecturas V1-V3 y el 32% de las V6-V9. El género Pseudomonas fue detectado con ambos amplicones aunque, siguiendo en la misma línea de los resultados ya mostrados, la región V6-V9 tuvo un enriquecimiento preferencial para este grupo, que fue el 2.° en abundacia de lecturas, mientras que para la región V1-V3 fue el 17.° (fig. 6).
Géneros potencialmente patógenosEmpleando ambas regiones del rRNA 16S, fue posible detectar los mismos 10 géneros más abundantes que contienen especies patógenas: Streptococcus, Staphylococcus, Pseudomonas, Corynebacterium, Neisseria, Haemophilus, Stenotrophomonas, Mycobacterium, Bacillus y Enterococcus. Adicionalmente, los análisis con la región V6-V9 mostraron la presencia de bacterias de los géneros Vibrio y Escherichia/Shigella, aunque en muy baja representación, 20 y 6 lecturas, respectivamente. Los géneros Streptococcus, Staphylococcus y Pseudomonas fueron los potencialmente patógenos más abundantes y, a su vez, unos de los grupos más abundantes en general de la microbiota presente en el billete. Estos 3 géneros acumularon el 28,9% de las lecturas del amplicón V1-V3 y el 34,5% de las de la región V6-V9.
DiscusiónLas tecnologías de secuenciamiento de 2.ª generación (NGS, next generation sequencing) han revolucionado muchos aspectos de las investigaciones en ciencias biomédicas, al permitir ver con gran detalle las características genéticas de los seres vivos y las mezclas complejas de estos en los ambientes naturales o artificiales12,13. Estudios basados en el análisis de librerías de amplicones, como el 16S, han extendido profundamente nuestra frontera de visión del mundo microbiano14. Aplicamos estas tecnologías a un billete en circulación en Colombia, uno de los artículos más usados en el día a día de los colombianos, y encontramos una gran riqueza de especies allí alojadas y no observadas hasta el día de hoy. Ningún estudio previo publicado en una revista científca había detectado la presencia del género Propionibacterium en billetes15, y con este estudio demostramos que es la bacteria más abundante en este objeto.
La clasificación usando los amplicones V1-V3 y V6-V9 mostró diferencias en los grupos detectados y en sus abundancias. Esta diferencia se debe, primero, al sesgo introducido por las regiones de hibridación de los cebadores. Esto explica los perfiles diferenciales de grupos amplificados entre ambos amplicones, lo cual ya ha sido reportado en otros trabajos16. Segundo, la limitación que tiene la clasificación del programa del RDP en función de las diferentes regiones variables del gen 16S estudiadas17. A pesar de esto, la mayoría de los grupos analizados en este trabajo se detectaron simultáneamente con ambas regiones estudiadas e incluso se observó coincidencia en las abundancias relativas de la mayoría de los grupos clasificados, lo que respalda adecuadamente los resultados, a pesar de las diferencias. La menor proporción de secuencias clasificadas con el amplicón V6-V9 se debe primordialmente al alto número de secuencias de Archaea detectadas con esta región, las cuales fueron descartadas de este trabajo, dado el bajo poder de clasificación observado por programa Classifier (bootstrap ≥ 0,8).
Los billetes y monedas pueden ser colonizadas por microorganismos cuya fuente es dependiente del contacto continuo con las manos de diferentes individuos, las costumbres de almacenamiento y las condiciones higiénicas de los usuarios. Los estudios de este tipo de materiales se ha relacionado siempre con el potencial de transportar patógenos que pueden afectar la salud del usuario. Varios estudios de estos elementos se han realizado empleando metodologías dependientes de cultivo y reportan aislamientos frecuentes de bacterias grampositivas y bacilos gramnegativos dentro de las cuales fueron frecuentes Bacillus, Staphylococcus, Streptococcus, Pseudomonas, Enterobacter, Acinetobacter, Escherichia y Klebsiella15.
Los resultados de esta investigación muestran por primera vez la gran diversidad de especies bacterianas presentes en el papel moneda y contribuyen exponencialmente a su conocimiento ya que, debido a la aplicación de tecnologías de secuenciamiento profundo, se incrementó en más de 200 el número de géneros detectados sobre las decenas que se han determinado empleando métodos dependientes de cultivo.
Los géneros bacterianos más abundantes encontrados en el billete de 1.000 pesos colombianos corresponden a los mismos ya reportados en las manos de humanos: Propionibacterium, Streptococcus y Staphylococcus18. Lo que es esperado dado que el inóculo y sustrato de crecimiento (sudor) provienen en su mayoría de esta parte del cuerpo. Además los géneros Streptococcus y Staphylococcus han sido caracterizados por presentar la capacidad de sobrevivir en superficies inertes durante meses19. Un estudio metagenómico similar en billetes de la India, pero aplicando una metodología de whole genome shotgun, reportó la presencia de ADN de bacterias patógenas de los géneros Mycobacterium, Staphylococcus, Corynebacterium, Pseudomonas, Bacillus, Enterococcus, coincidiendo con nuestros resultados. Sin embargo, en esta publicación se concentran solo en las especies patógenas y que degradan celulosa, y no mencionan tampoco las abundancias relativas de secuencias de cada especie encontrada20.
Propionibacterium ha sido reportado como el género más abundante en la superficie de la mano18. Morfológicamente se caracterizan como bacilos grampositivos no formadores de endosporas, de los cuales se encuentran reportadas 14 especies en la base de datos del NCBI. Recientemente, Fitz-Gibbon et al. (2013)21 indicaron que la especie dominante de los folículos sebáceos de la piel es Propionibacterium acnes (P. acnes), que corresponde al 90% de la microbiota. El nombre se debe a la habilidad de este grupo en fermentar carbohidrados a ácido propiónico, sustancia química que presenta actividad antimicrobiana21. Este organismo es considerado comensal y un agente patogénico del desarrollo del acné. Sin embargo, en este estudio se evidenció la presencia en la piel de cepas de la especie con características patogénicas y otras que se establecen pero no provocan la enfermedad. Recientemente los mismos autores reportaron que bacteriófagos de P. acnes también se encuentran en la piel y que estos, al infectar selectivamente las cepas, influyen la estructura de la población de P. acnes presentes en la piel22. Además de esto, P. acnes, mediante la producción de ácido propiónico, puede inhibir el crecimiento de cepas de S. aureus resistentes a la meticilina y puede influir la composición de la comunidad bacteriana en la piel y actuar como barrera protectora frente a estos patógenos23. Otra especie que también ha sido reportada asociada a humanos, y específicamente a caries, es P. acidifaciens. Otras especies como P. olivae y P. damnosum, P. freudenreichii, P. acidipropionici, P. jensenii y P. thoenii han sido aisladas de leche y productos lácteos, vegetales y ensilajes y, al tener contacto directo con las manos de humanos, podrían establecerse en los billetes. Sin embargo, estas especies no han sido reportadas como patógenas, sino que algunos de sus metabolitos confieren características organolépticas deseables a los productos o, en algunos casos, provocan su deterioro24.
El género Streptococcus tiene reportadas actualmente 110 especies en la base de datos BACTERIO.NET (http://www.bacterio.net/s/streptococcus.html). De las cuales algunas pueden causar infecciones en humanos o animales, o ser beneficiosas y actualmente tiene usos industriales. El hallazgo en este estudio de este género coincide con otros trabajos donde se hallaron frecuentemente estos cocos grampositivos en billetes de varios países15.
Las especies de Staphylococcus comúnmente reportadas en piel son S. aureus, S. saprophyticus, S. epidermidis, S. haemolyticys. Estos 2 últimos también reportados como causantes de enfermedades nosocomiales25. La especie S. aureus y otros estafilococos coagulasa positiva colonizan la piel y la membrana mucosa y pueden causar enfermedades en la piel o infecciones de tejidos blandos en animales y humanos. Dentro de los blancos de estudio actualmente más relevantes en salud pública se encuentran las cepas de esta especie que presentan resistencia a la meticilina (SAMR)26. Debido a la capacidad de los billetes de Colombia de alojar Staphylococcus, es muy posible que pueda alojar cepas con resistencias a antibióticos, por lo tanto, es prioritario determinar en estudios posteriores la capacidad de los billetes en Colombia para alojar y transmitir estas especies patógenas.
Corynebacterium es un género perteneciente al phylum Actinobacteria con aproximadamente 112 especies según información del NCBI. Algunas especies, como C. glutamicum, son utilizadas en procesos industriales para la producción de aminoácidos o en la producción de proteínas recombinantes27. Algunas especies son comensales y otras patógenas. En humanos han sido aisladas del tracto respiratorio, de la placa dental o de lesiones ulcerativas en la piel. Corynebacterium diphteriae es el agente causal de la difteria debido a la producción de una toxina. Como comensales se reportó que la presencia de Corynebacterium spp. en el tracto respiratorio superior de niños reduce en un 49% la probabilidad de que presenten otitis media28. De tal manera que es necesario alertar a las poblaciones expuestas de los riesgos de no lavarse las manos al manipular estos objetos, ya que es posible que se favorezca la aparición de enfermedades por malos hábitos higiénicos.
Los géneros potencialmente patógenos de mayor relevancia detectados en los billetes pueden ser Streptococcus, Staphylococcus y Pseudomonas, ya que estos no solo presentan potencial virulento, sino que pueden presentar resistencia a muchos de los antibióticos de uso rutinario en humanos. Además, estos 3 grupos representaron, en promedio, una tercera parte de las lecturas procesadas de ambos amplicones, indicando una preponderante abundancia en el billete. Sin embargo, estudios posteriores con mayor cantidad de muestras y mayor resolución taxonómica son necesarios para determinar las especies alojadas en los billetes, así como la posible carga de genes de resistencia a antibióticos.
Responsabilidades éticasProtección de personas y animalesLos autores declaran que para esta investigación no se han realizado experimentos en seres humanos ni en animales.
Confidencialidad de los datosLos autores declaran que en este artículo no aparecen datos de pacientes.
Derecho a la privacidad y consentimiento informadoLos autores declaran que en este artículo no aparecen datos de pacientes.
FinanciaciónEsta investigación fue financiado por el Comité para el Desarrollo de la Investigación (CODI) (Estrategia de Sostenibilidad 2014-2015), Vicerrectoría de Investigación, Universidad de Antioquia y Colciencias.
Conflicto de interesesLos autores declaran no tener conflicto de intereses.