Se han comparado las frecuencias alélicas de HLA de amerindios con las de otros primeros habitantes americanos y también con otras poblaciones del mundo, con el objetivo de esclarecer el discutido poblamiento de América y el origen de los amerindios. Se han utilizado todos los datos disponibles de HLA de las primeras poblaciones nativas americanas. Se utilizaron métodos para medir distancias genéticas y dendrogramas Neighbour-Joining (NJ). Los resultados y su discusión han originado las siguientes conclusiones: 1) los atabascos del noroeste canadiense muestran flujo génico con poblaciones vecinas, con amerindios, con habitantes de las islas del Pacífico, incluyendo australianos orientales, y con siberianos, ya que comparten haplotipos DRB1-DQB1 con estas poblaciones (por ejemplo: DRB1*14:01-DQB1*05:03, DRB1*09:01-DQB1*03:03); 2) la entrada de los amerindios en América pudo haber sido diferente a la de atabascos, aleutianos y esquimales; los amerindios pudieron haber llegado al continente mucho antes que los atabascos y esquimales ya que presentan un conjunto completamente diferente de frecuencias alélicas HLA-DRB1; 3) los amerindios muestran muy pocos alelos estrictamente particulares (DRB1*04:11, DRB1*04:17), pero presentan haplotipos extendidos únicos (por ejemplo: A*02-B*35-DRB1*04:07-DQB1*03:02, A*02-B*35-DRB1*08:02-DQB1*04:02); 4) nuestros resultados no apoyan el modelo clásico de poblamiento del continente de las tres oleadas migratorias, sino otro en el que la entrada pudo ser también por la costa pacífica. La llegada de gentes por el Océano Pacífico ha podido contribuir al perfil genético HLA americano. La migración inversa (de América a Asia) de gentes en diferentes épocas no se puede descartar.
HLA allele frequencies were compared with those of other First American Natives and also those of other worldwide populations in order to clarify the still unclear peopling of the Americas and the origins of Amerindians. All possible HLA data already obtained on early Native American populations are used. Genetic distances and N-J dendrogram methods are applied. Results and discussion have led to the following conclusions: 1) North West Canadian Athabaskans have had gene flow with close neighbouring populations, Amerindians, Pacific Islanders, including East Australians, and Siberians, since they share DRB1-DQB1 haplotypes with these populations (i.e.: DRB1*14:01-DQB1*05:03, DRB1*09:01-DQB1*03:03); 2) Amerindians entrance to America may have been different to that of Athabaskans, Aleuts and Eskimos; Amerindians may have been in their lands long before Athabaskans and Eskimos as they present an altogether different set of HLA-DRB1 allele frequencies; 3) Amerindians show very few “particular” single-locus alleles (i.e.: DRB1*04:11, DRB1*04:17), but have unique extended haplotypes (i.e.: A*02-B*35-DRB1*04:07-DQB1*03:02, A*02-B*35-DRB1*08:02-DQB1*04:02); 4) Our results do not support the three-wave model of American peopling but another model, where the Pacific Coast is also an entrance point. Pacific Ocean sea voyages may have contributed to the HLA genetic American profile. Reverse migration (America to Asia) is not discarded, and different movements of people in either direction in different times are supported by the Athabaskan population admixture with Asian-Pacific population and with Amerindians.
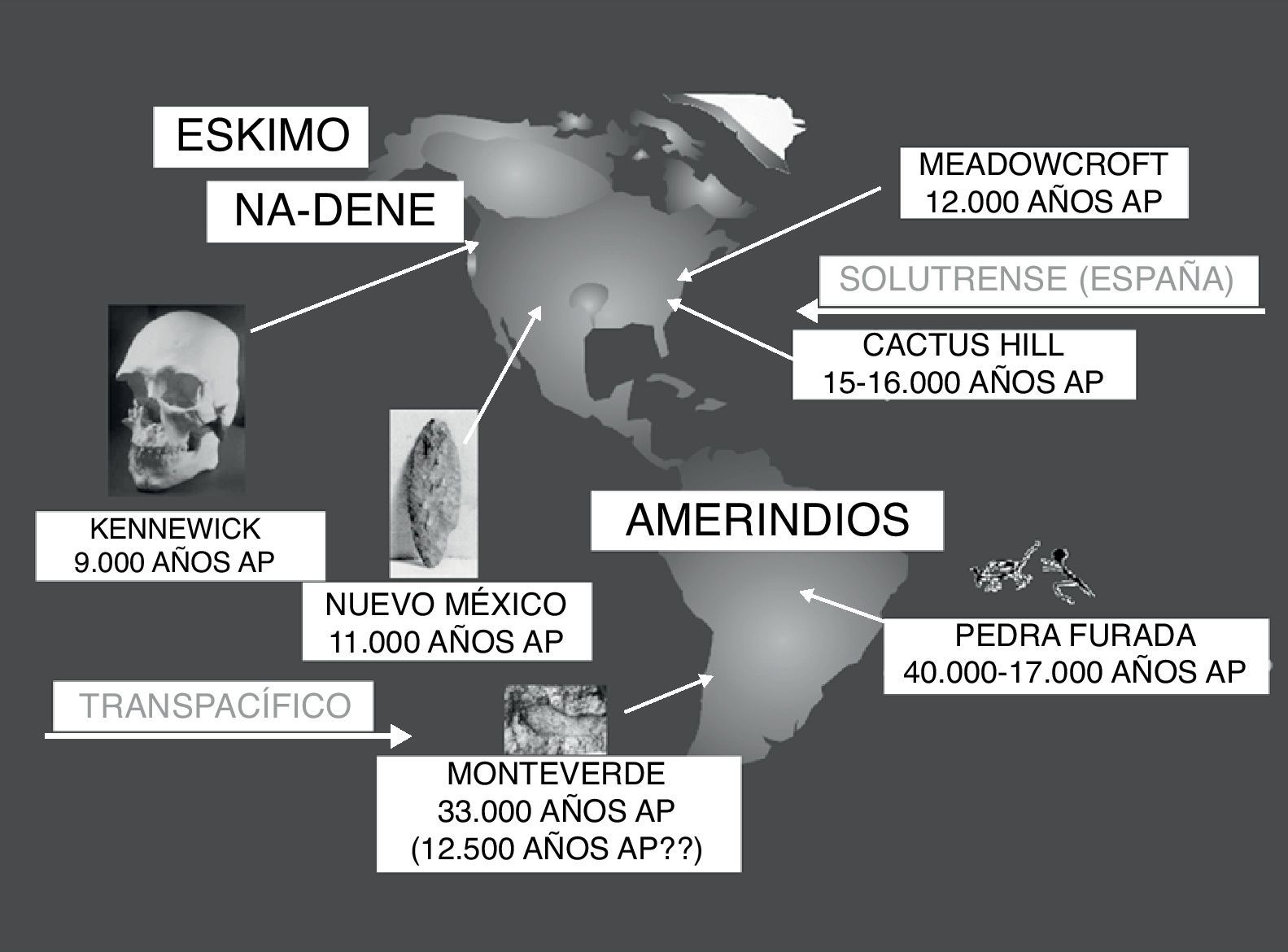
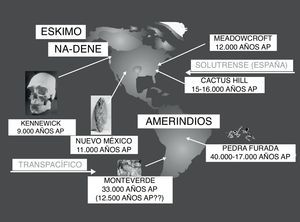
Se ha propuesto que los primeros amerindios proceden de Asia y entraron a través del Estrecho de Bering, entre 30.000 y 10.000 años antes del presente (AP). Estas conclusiones se basan en similitudes culturales, morfológicas y genéticas entre las poblaciones de América y Asia. Tanto Siberia1 como Mongolia2,3 en Asia, se han sugerido como los lugares más probables del origen asiático. Greenberg fue el primero en postular la teoría de la triple migración para explicar el poblamiento de América4: amerindios (12.000 años AP), na-dene–atabascos, navajo y apaches- (8.000 años AP) y eskimo-aleutianos (6.000 años AP) (fig. 1); otros autores sugieren que hubo una única oleada migratoria de los ancestros de los primeros nativos americanos y habría tenido lugar desde Mongolia/Norte de China2,3.
Representa la teoría más popular del poblamiento de este continente desde Asia a través del Estrecho de Bering4. Gris: amerindios (30.000-12.000 años AP); gris claro: Na-Dene (8.000 años AP), atabascos de Canadá, grupos aislados de indios de California y Navajo y Apache del sur del Estados Unidos; gris oscuro: Eskimo (6.000 anos AP). Los aleutianos de las islas Aleutianas en el Estrecho de Bering están separados de los Eskimo en cuanto a la lengua y otros parámetros antropológicos y estaban presentes en las islas antes de que los Eskimo alcanzaran Norteamérica; además, el perfil HLA aleutiano es diferente del perfil de los Eskimo30. Otras teorías del poblamiento de América (flechas grandes): ruta Transpacífica (de Australia e islas del Pacífico), y del pueblo Solutrense de la Península Ibérica6,12. También están representados los descubrimientos arqueológicos más relevantes6. El hombre de Kennewick (Estado de Washington, EE. UU.); Meadowcroft (Pensylvania, EE. UU.); Cactus Hill (Virginia, EE. UU.); Pedra Furada (Brasil); Monte Verde (Chile).
Los marcadores de ADN mitocondrial y cromosoma Y se han utilizado para estudiar los orígenes, tiempo y lugar de entrada de amerindios, atabascos y esquimales5 en América. También, han sido contrastados hallazgos arqueológicos con los datos genéticos1,5,6. Finalmente, las conclusiones son diversas, y no existe consenso sobre el origen y el emparentamiento de los amerindios2,3,5,7. La cuestión más importante es dilucidar si los inmigrantes (amerindios) estaban ya diferenciados (en Asia) en cada uno de los grupos étnicos cuyos descendientes permanecen todavía en Asia. El cómo y el cuándo cruzaron el Estrecho de Bering serían preguntas secundarias8.
En lo que a esto respecta, los datos de HLA pueden ser más informativos que los de cromosoma Y o ADN mitocondrial8, ya que se estudia, a la vez, los linajes maternos y paternos y tanto las frecuencias (es decir, distancias genéticas, dendrogramas y análisis de correspondencia) como las genealogías (alelos y haplotipos HLA específicos) pueden ser estudiados para comparar poblaciones. La mejor evidencia de que HLA es un buen marcador genético para el estudio del emparentamiento entre poblaciones es que normalmente se correlaciona con la geografía.
La investigación de inserciones Alu ofrece resultados que tampoco concuerdan con la hipótesis de varias oleadas migratorias9. Estos resultados y otros procedentes de las investigaciones de las cepas de virus HTLV-110, inducen a sugerir la existencia de una ruta transpacífica (adicional, al menos) para el poblamiento de América desde Asia o la Polinesia, que podría haber introducido algunos alelos HLA6,11. Finalmente, tanto las evidencias genéticas12 como las arqueológicas6 sugieren que una ruta transatlántica en ambos sentidos existió antes de que Colón descubriera América6 (fig. 1).
También, estas discrepancias e incertidumbres sobre el origen de los amerindios pueden deberse a errores metodológicos. Por ejemplo, marcadores genéticos menos estudiados y conocidos, como los Alu y STR se han utilizado de forma errónea pues constituyen marcadores intrónicos y exónicos no controlados en cuanto a significado cronológico y, a veces, en cuanto a situación genómica. Además, los movimientos de población deberían ser estudiados como movimientos de grupos de genes, es decir, con frecuencias génicas (distancias genéticas, dendrogramas y análisis de correspondencia), que reflejan mejor los desplazamientos poblacionales y los emparentamientos entre poblaciones, y posteriormente completados con genealogías (alelos HLA cuasi-específcicos, haplotipos HLA, ADN mitocondrial y cromosoma Y). Hoy día, los estudios genéticos con una mezcla de todos estos marcadores y de otros datos no han esclarecido todavía el poblamiento de América5,13.
En el presente estudio, se hace una revisión genealógica y de comparación de genética de poblaciones HLA para intentar esclarecer el origen de los amerindios.
Material y métodosMuestreoPor lo tanto, en el presente trabajo, hemos estudiado las frecuencias génicas HLA de amerindios de Norte, Meso y Sudamérica y las hemos comparado con las de otros indios americanos de Norteamérica y de poblaciones de todo el mundo, particularmente con la poblaciones de Asia y del Pacífico. Hemos estudiado los siguientes grupos étnicos amerindios: Seri, Mixe, Mixtecos, Zapotecos, Guaraníes14, Lakota Sioux15, Mazatecos16, Teeneks17, Mayas18, Kogi, Arsario, Arhuacos, Wayu19, Cayapa20, Lamas21, Aymaras22, Quechuas23, indios Terena24, Xavantes, Mayos25, Uros26, Nahuas27, Tarahumaras28, Toba Pilaga, Mataco Wichi, Toba orientales11, mestizos mexicanos y Jaidukama29 y también aleutianos30. Un total de 14.698 cromosomas fueron analizados.
Comparación de tipajes de HLA y comparaciones estadísticas- -
Comparaciones genealógicas: determinar los linajes alélicos altamente específicos de HLA de clase i (A y B) y de clase ii (DRB1 y DQB1) en amerindios (en lo sucesivo «alelos» para simplificar) o los haplotipos HLA específicos utilizando secuenciación de ADN y serología; en otras palabras, los alelos y haplotipos HLA más frecuentes en amerindios que no existen o existen en muy baja frecuencia en otras poblaciones.
- -
Comparaciones de grupos de genes: comparar las frecuencias alélicas de HLA en amerindios con las de otros primeros nativos americanos (na-dene, esquimales, aleutianos) y también las de otras poblaciones de todo el mundo con el fin de esclarecer el todavía nada claro poblamiento de América y el origen de los amerindios.
Los análisis estadísticos se llevaron a cabo con el programa Arlequín versión 2.0 suministrado amablemente por Excoffier y Slatkin31. En resumen, este programa calculó las frecuencias alélicas de HLA-A, -B, -DRB1 y -DQB1, el equilibrio de Hardy-Weinberg y el desequilibrio de ligamiento entre n alelos de n loci diferentes. Su nivel de significación (P) para comparaciones 2×2 fue determinado como se ha descrito previamente32,33. Además, los haplotipos completos más frecuentes fueron deducidos a partir de: (1) las frecuencias haplotípicas para 2, 3 y 4 loci32,33; (2) los haplotipos descritos previamente en otras poblaciones32,33, y (3) los haplotipos si aparecen en dos o más individuos y se definió el haplotipo alternativo32,33. Con la intención de comparar fenotipos y frecuencias haplotípicas HLA con otras poblaciones, se utilizaron las tablas de referencia del 11° y 12° International HLA Workshop34,35. Los árboles de emparentamiento (dendrogramas) se construyeron con las frecuencias alélicas a partir del método Neighbour-Joining (NJ)36 con las distancias genéticas entre poblaciones (DA)37, utilizando el programa DISPAN que comprende los programas GNKDST y TREEVIEW38,39. El análisis de correspondencias en 3 dimensiones y su representación bidimensional se llevó a cabo utilizando el programa VISTA versión 5.0540,41. El análisis de correspondencias consiste en una técnica geométrica que puede ser utilizada para presentar una visión global de las relaciones entre poblaciones de acuerdo con las frecuencias alélicas HLA (u otras). Esta metodología se basa en la variancia de las frecuencias alélicas entre poblaciones (similar a la metodología de componentes clásica) y en la visualización estadística de las diferencias.
Resultados y discusiónAlelos HLA-DRB1 y haplotipos HLA en AméricaHemos escogido los alelos DRB1 porque la mayoría de las poblaciones están tipadas para alelos de DRB1 de alta resolución y muy pocas para loci HLA de clase i u otros de clase ii. El número tan bajo de alelos de clase i encontrados puede ser un artefacto, ya que la mayoría de estos pueden no haber sido todavía detectados.
Los alelos DRB1 de amerindios de Meso y Sudamérica son casi específicos; los alelos DRB1*04:11 y DRB1*04:17 se han encontrado solamente en las poblaciones estudiadas de Meso y Sudamérica.
Por el contrario, los alelos de poblaciones de Norteamérica son compartidos claramente con otras poblaciones no amerindias (poblaciones de Asia y del Pacífico, no mostradas). Esto concuerda con la existencia de flujo génico entre amerindios y gentes de Siberia y de las islas del Pacífico, pero no necesariamente con una migración de amerindios desde áreas de Asia o del Pacífico, aunque hay signos de contacto cultural y genético con Asia o incluso con Íberos6,12 (fig. 1). DRB1*08:02 está presente en una frecuencia significativa en casi todas las poblaciones amerindias y ausente o con una frecuencia no significativa en otras poblaciones. DRB1*04:03 está presente en uno de los haplotipos más frecuentes en poblaciones sudamericanas (A*02-B*48-DRB1*04:03-DQB1*03:02) pero también se encuentra frecuentemente a lo largo de las islas del Pacífico (Samoa, Papua Nueva Guinea, Nueva Zelanda Maorí, Taiwán, Tonga, Islas Cook)42. No se puede descartar tampoco un flujo génico en el Pacífico en ambas direcciones por este enfoque genealógico.
Análisis HLA genealógico (haplotipos) vs. análisis de frecuencias de alelos HLA en poblaciones (dendogramas, correspondencia)Haplotipos HLA- -
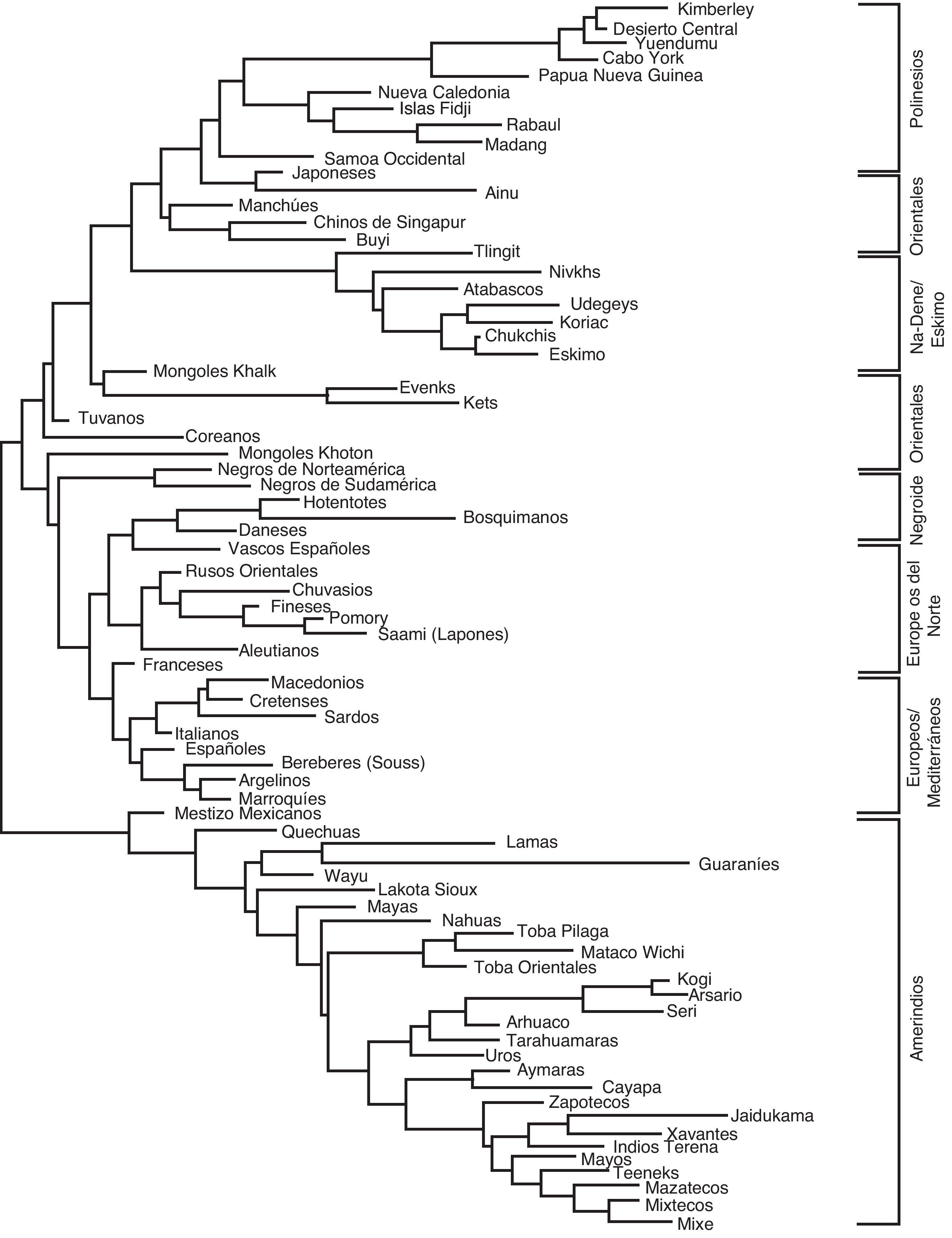
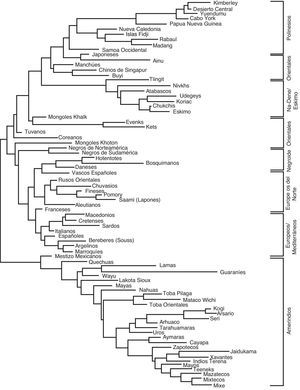
Norteamericanos. Los haplotipos HLA más frecuentes en Norteamérica son específicos de las poblaciones norteamericanas, Yupik (esquimales) y aleutianos. Uno de los haplotipos más frecuentes se ha encontrado también en poblaciones de Taiwán y de Japón (A*24-B*40-DRB1*14:01-DQB1*05:03). Esto muestra que se ha hallado un número bajo de haplotipos norteamericanos compartidos entre poblaciones de Norteamérica y de Asia y Pacífico. Sin embargo, hay un claro emparentamiento genético HLA entre poblaciones aisladas próximas a Beringia: esquimales, Udegeys, Nivkhs (en la costa noreste de Siberia), y Koryaks y Chukchi del extremo noreste de Siberia (fig. 2); y las poblaciones del noroeste de América: atabascos, esquimales de Alaska (Yupik) y Tlingit.
Estos resultados sugieren la existencia de una mezcla entre los grupos del extremo noreste de Siberia y los na-dene (atabascos y Tlingit) y esquimales (Yupik), pero no nos dicen si hubo migraciones en ambos sentidos.
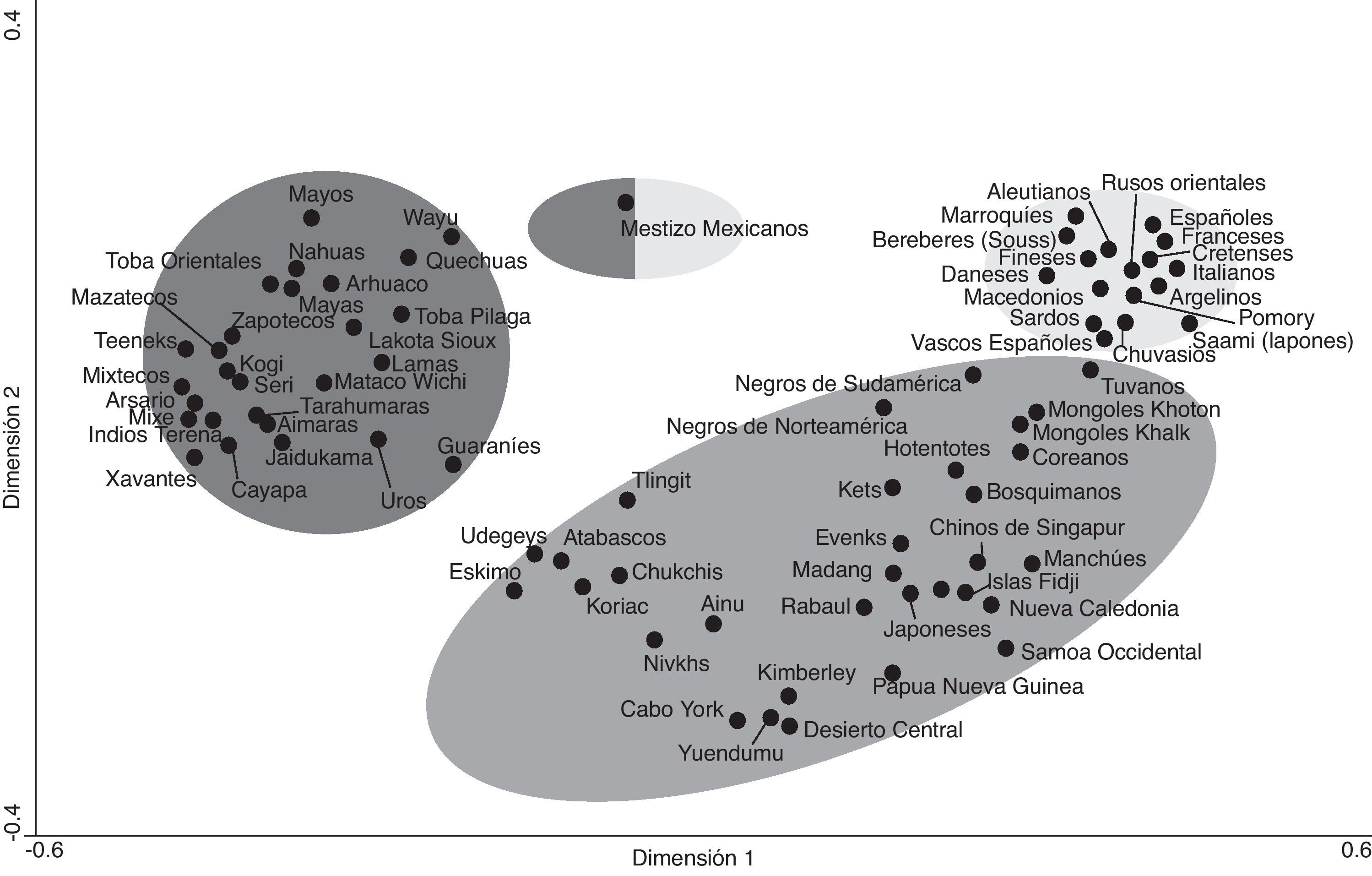
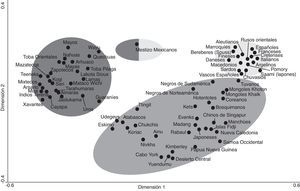
Por otro lado, las poblaciones de Asia que no se encuentran geográficamente próximas a Beringia (japoneses, Ainu, manchúes, chinos de Singapur, Buyi) no se agrupan con las de Norteamérica ni en el dendrograma NJ (fig. 2) ni en el análisis de correspondencia (fig. 3).
Figura 3.Análisis de correspondencia basado en las frecuencias alélicas de HLA-DRB1 y HLA-DQB1. El análisis muestra una visión global de las relaciones genéticas entre las poblaciones amerindias, Na-Dene, esquimales, asiáticas, negroides y europeas de acuerdo a las frecuencias alélicas de HLA-DRB1 y HLA-DQB1. Las relaciones se han calculado en n dimensiones y representado en 2. Los círculos representan un agrupamiento aproximado de poblaciones25,26,28,30.
(0.34MB).Por último, los amerindios Lakota-Sioux de los Estados Unidos de Norteamérica, no están emparentados con asiáticos o siberianos occidentales (fig. 2) pero sí con poblaciones de Meso y Sudamérica, es decir, con amerindios típicos.
Figura 2.Dendograma Neighbor-Joining obtenido utilizando las frecuencias alélicas DRB1. El emparentamiento genético entre amerindios, Na-Dene, Eskimo, asiáticos, negroides, europeos y polinesios se ha determinado calculando las distancias genéticas entre poblaciones (DA), utilizando las frecuencias HLA-DRB1. Los amerindios se agrupan juntos y separados del resto de poblaciones del mundo25,26,28,30.
(0.46MB). - -
Mesoamericanos. Los haplotipos HLA más frecuentes (no mostrados), los dendrogramas de emparentamiento (fig. 2), y los análisis de correspondencia (fig. 3) no relacionan estos amerindios con cualquiera de las poblaciones asiáticas, incluyendo los siberianos del noreste. Los haplotipos de poblaciones de Meso América son compartidos con otros amerindios y uno de ellos con los esquimales de Alaska (Yupik): A*02-B*35-*DRB1*08:02-DQB1*04:02.
- -
Sudamericanos. Estos grupos de hablantes amerindios están emparentados con otras poblaciones amerindias de Meso y Sudamérica, pero no con asiáticos (fig. 2). Los haplotipos HLA más frecuentes son compartidos con otros amerindios, pero no con asiáticos.
En resumen, los amerindios muestran poco emparentamiento con asiáticos, de acuerdo con los estudios genealógicos de haplotipos. Las poblaciones de Norteamérica muestran solamente un haplotipo (A*24-B*40-DRB1*14:01-DQB1*05:03) compartido con taiwaneses y japoneses en baja frecuencia.
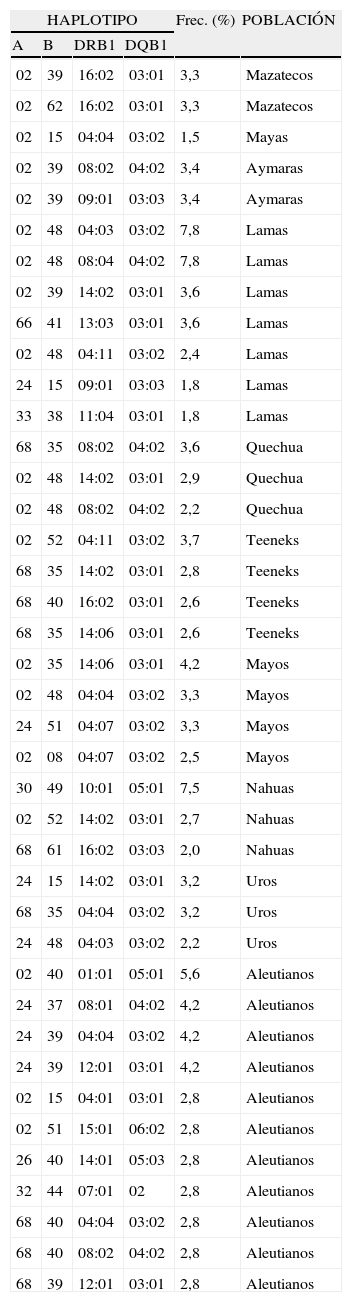
Haplotipos extendidos específicos para grupos étnicos amerindios y aleutianosAlgunos haplotipos extendidos nuevos de 4 loci han sido encontrados solamente en grupos específicos de amerindios y aleutianos y no en otras poblaciones de amerindios o de otras partes del mundo (tabla 1).
Haplotipos extendidos encontrados solamente en poblaciones amerindias
| HAPLOTIPO | Frec. (%) | POBLACIÓN | |||
| A | B | DRB1 | DQB1 | ||
| 02 | 39 | 16:02 | 03:01 | 3,3 | Mazatecos |
| 02 | 62 | 16:02 | 03:01 | 3,3 | Mazatecos |
| 02 | 15 | 04:04 | 03:02 | 1,5 | Mayas |
| 02 | 39 | 08:02 | 04:02 | 3,4 | Aymaras |
| 02 | 39 | 09:01 | 03:03 | 3,4 | Aymaras |
| 02 | 48 | 04:03 | 03:02 | 7,8 | Lamas |
| 02 | 48 | 08:04 | 04:02 | 7,8 | Lamas |
| 02 | 39 | 14:02 | 03:01 | 3,6 | Lamas |
| 66 | 41 | 13:03 | 03:01 | 3,6 | Lamas |
| 02 | 48 | 04:11 | 03:02 | 2,4 | Lamas |
| 24 | 15 | 09:01 | 03:03 | 1,8 | Lamas |
| 33 | 38 | 11:04 | 03:01 | 1,8 | Lamas |
| 68 | 35 | 08:02 | 04:02 | 3,6 | Quechua |
| 02 | 48 | 14:02 | 03:01 | 2,9 | Quechua |
| 02 | 48 | 08:02 | 04:02 | 2,2 | Quechua |
| 02 | 52 | 04:11 | 03:02 | 3,7 | Teeneks |
| 68 | 35 | 14:02 | 03:01 | 2,8 | Teeneks |
| 68 | 40 | 16:02 | 03:01 | 2,6 | Teeneks |
| 68 | 35 | 14:06 | 03:01 | 2,6 | Teeneks |
| 02 | 35 | 14:06 | 03:01 | 4,2 | Mayos |
| 02 | 48 | 04:04 | 03:02 | 3,3 | Mayos |
| 24 | 51 | 04:07 | 03:02 | 3,3 | Mayos |
| 02 | 08 | 04:07 | 03:02 | 2,5 | Mayos |
| 30 | 49 | 10:01 | 05:01 | 7,5 | Nahuas |
| 02 | 52 | 14:02 | 03:01 | 2,7 | Nahuas |
| 68 | 61 | 16:02 | 03:03 | 2,0 | Nahuas |
| 24 | 15 | 14:02 | 03:01 | 3,2 | Uros |
| 68 | 35 | 04:04 | 03:02 | 3,2 | Uros |
| 24 | 48 | 04:03 | 03:02 | 2,2 | Uros |
| 02 | 40 | 01:01 | 05:01 | 5,6 | Aleutianos |
| 24 | 37 | 08:01 | 04:02 | 4,2 | Aleutianos |
| 24 | 39 | 04:04 | 03:02 | 4,2 | Aleutianos |
| 24 | 39 | 12:01 | 03:01 | 4,2 | Aleutianos |
| 02 | 15 | 04:01 | 03:01 | 2,8 | Aleutianos |
| 02 | 51 | 15:01 | 06:02 | 2,8 | Aleutianos |
| 26 | 40 | 14:01 | 05:03 | 2,8 | Aleutianos |
| 32 | 44 | 07:01 | 02 | 2,8 | Aleutianos |
| 68 | 40 | 04:04 | 03:02 | 2,8 | Aleutianos |
| 68 | 40 | 08:02 | 04:02 | 2,8 | Aleutianos |
| 68 | 39 | 12:01 | 03:01 | 2,8 | Aleutianos |
Fuente:16–18,21–23,25–27,30.
Llama la atención que en pequeños grupos de gente aparentemente suceden recombinaciones de 4 loci HLA específicas y estas se fijan. Las fuerzas evolutivas para lograr un haplotipo extendido adecuado pueden ser ventajosas para que una población haga frente a los patógenos específicos de su ambiente43. En este caso, las fuerzas evolutivas que conducen a la aparición de novo de haplotipos HLA deben incluir los patógenos y no los genes de baja frecuencia fijados por selección. Sin embargo, ambos tipos de resultados selectivos para inducir variabilidad de genes HLA o de otros genes son matemáticamente indistinguibles44.
Nuevos alelos HLA están siendo descritos continuamente en poblaciones42,45, pero esto no significa necesariamente que se estén produciendo continuamente; podrían haber sido fijados como alelos de baja frecuencia en poblaciones durante mucho tiempo y descritos solamente en el presente gracias a los avances tecnológicos.
Nuevos haplotipos específicos se han encontrado en amerindios de Norte y Sudamérica, mientras que alelos específicos para una población particular amerindia se han encontrado raramente o no se han encontrado. Esto concuerda con el hecho de que las frecuencias haplotípicas muestran más variación entre grupos raciales que los alelos de cada locus individualmente46. Por lo tanto, la aparición de nuevos alelos debe ser un evento relativamente raro (normalmente por conversión génica)47, o bien desaparecen con la misma rapidez que aparecen48. De hecho, la evolución de la variabilidad del sistema MHC podría ser más frecuente en haplotipos que en la diversificación de alelos. La selección de la variabilidad en las poblaciones amerindias de Norte y Sudamérica actúa sobre los haplotipos más que sobre los alelos. Este descubrimiento puede ser universal para todas las poblaciones del mundo.
Las lenguas no se correlacionan con los genesAlgunos autores encuentran correlación entre genes y lenguas cuando se utilizan grupos étnicos y lenguas seleccionados pero solamente a un nivel macrogeográfico; sin embargo, el dendrograma NJ (fig. 2) muestra que el grupo etnias na-dene/esquimal/siberiano está muy próximo genéticamente al medirse por frecuencias alélicas de HLA-DRB1, y aunque sus lenguas son radicalmente diferentes. Esto lo confirma el análisis de correspondencia de HLA-DRB1 y HLA-DQB1 (fig. 3). Tanto el NJ como el análisis de correspondencia se correlacionan bastante bien con la geografía pero no con las lenguas. Esto es particularmente evidente en el grupo de los amerindios en el que sus agrupamientos genéticos (figs. 2 y 3) no se correlacionan con sus lenguas, por ejemplo: los Mayas están muy próximos a los Lakota-Sioux y a los Nahuas de México, cuyas lenguas no están relacionadas en absoluto. Los genes se heredan y las lenguas han sido impuestas a lo largo de la historia o han variado por modas u otros motivos49. También, la velocidad de evolución de genes y lenguas es diferente49.
Determinados alelos HLA en amerindios muestran flujo génico con asiáticos, australianos y habitantes del Océano PacíficoLos dendrogramas HLA o los análisis de correspondencia basados en frecuencias HLA muestran que los amerindios (en el sentido de la definición de Greenberg4) parecen estar separados de otras poblaciones de todo el mundo, incluyendo los atabascos del norte de Canadá y de los esquimales (figs. 2 y 3). Los últimos se agrupan con los siberianos (fig. 2). Esto significa que las frecuencias alélicas de HLA-DRB1 y de HLA-DQB1 son completamente diferentes en amerindios comparados con otros primeros nativos americanos u otras poblaciones del mundo.
Además, las poblaciones estudiadas muestran haplotipos HLA de 4 loci particulares para cada población (tabla 1). Esto no es el caso de los alelos de HLA-DRB1: excepto para dos alelos DRB1–DRB1*04:11 y DRB1*04:17-, los amerindios de Meso y Sudamérica comparten alelos con 1) siberianos; 2) otros primeros habitantes de América incluyendo atabascos y esquimales, pero no aleutianos30; 3) poblaciones de la costa asiática del Pacífico (Ainu, japoneses, taiwaneses) y en menor medida con gentes de Indochina, y 4) aborígenes de el este de Australia y habitantes de las islas del Pacífico, como Papua Nueva Guinea o Samoa42.
Un estudio genealógico del haplotipo DRB1-DQB130, que complementa nuestro estudio poblacional de frecuencias, mostró que los genes DRB1-DQB1 de atabascos son compartidos con: 1) sus vecinos, incluyendo esquimales de Alaska (Yupik); 2) amerindios de Norte y Sudamérica; 3) siberianos, y 4) habitantes de las islas del Pacífico, e incluso con aborígenes del este de Australia. Esto sugiere que los atabascos están compuestos de una mezcla genética HLA y que el flujo génico tuvo lugar entre estos y todas las poblaciones asiáticas y del Pacífico mencionadas anteriormente. El hipotético flujo génico HLA ocurrió en diferentes tiempos en direcciones distintas. Esto muestra que no solamente Beringia fue un paso activo de amerindios primitivos, sino que la navegación por el Pacífico también lo fue. Los resultados de los alelos HLA de las poblaciones de Norte y Sudamérica apoyan también esta interpretación.
Por qué hoy en día las poblaciones amerindias son completamente diferentes al resto de las poblaciones del mundo con respecto a las frecuencias HLA, es solamente materia de especulación: alrededor de 80 millones de los nativos americanos murieron durante el siglo xvi50, debido principalmente a la falta de una respuesta inmune apropiada a las enfermedades transmitidas por los europeos, fundamentalmente el sarampión, la gripe y la peste50. Este «cuello de botella» genético pudo haber conformado el perfil HLA de los nativos americanos al aumentar alelos raros HLA capaces de presentar nuevos patógeno a las células T44,46,51. Sin embargo, otros primeros habitantes americanos, de Norteamérica (no amerindios), también sufrieron muchas epidemias50 y no tienen un perfil HLA tan diferente al de asiáticos (por ejemplo los atabascos na-dene del Jukon, Canadá), como sí lo tienen los amerindios.
Conclusiones- 1.
Los indios atabascos de Canadá han sufrido flujo génico con a) poblaciones vecinas cercanas; b) amerindios; c) habitantes de las islas del Pacífico incluyendo australianos orientales, y d) siberianos.
- 2.
La entrada de los amerindios a América pudo haber sido distinta a la de atabascos y esquimales y los primeros pudieron haberse encontrado en tierras americanas mucho antes que estos últimos, ya que presentan un conjunto de frecuencias HLA-DRB1 completamente diferente.
- 3.
Los amerindios muestran muy pocos «alelos particulares’»; casi todos los comparten con otros amerindios, atabascos, habitantes de las islas del Pacífico (incluyendo australianos orientales) y siberianos. Sin embargo, se han encontrado haplotipos extendidos específicos en casi todos los grupos aislados estudiados (Mazatecos16, Teeneks17, Mayas18, Lamas21, Aymaras22, Quechuas23, Mayos25, Uros26, Nahuas27, Tarahumara28, Jaidukama).
- 4.
Los genes y las lenguas evolucionan de muy diferente forma y, en general, no correlacionan.
- 5.
Nuestros datos no apoyan el modelo de las 3 oleadas migratorias postuladas para el poblamiento de América4, pero sí otro modelo en el que la entrada a América no fue solamente a través de Beringia, sino también por la costa del Pacífico. Los viajes en barco a través del Océano Pacífico pudieron haber contribuido al perfil genético HLA amerindio. La migración inversa (de América a Asia) no se descarta y pudieron ocurrir diferentes movimientos de gentes en ambas direcciones en tiempos distintos; esta hipótesis se basa en la mezcla genética HLA observada de la población atabasca con poblaciones de Asia y del Pacífico y con amerindios.
Este trabajo ha sido financiado en parte por subvenciones del Ministerio de Sanidad de España (FISS PI051039 y PI080838), y tres subvenciones distintas de la Mutua Madrileña Automovilística.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Se agradece también la colaboración de Javier Alonso Rubio.