La investigación en biología ha cambiado radicalmente debido al efecto combinado de los avances en biotecnología y ciencias de la computación. En consecuencia, la biología computacional y la bioinformática son tan esenciales para la biología del siglo xxi como la biología molecular lo fue en el anterior. Sin embargo, las competencias correspondientes a razonamiento matemático y computacional en el currículo de Biología apenas han cambiado en los últimos 25 años. La formación del biólogo debería ser tan sofisticada desde el punto de vista computacional como la del físico o la del ingeniero. La incorporación de estos cambios requiere tanto de un mayor esfuerzo de integración de las asignaturas cuantitativas existentes en el ámbito de los problemas biológicos como de la contextualización de las asignaturas propias de la biología desde un punto de vista más formal y de modelización. En este trabajo se revisan algunos de los esfuerzos que en este sentido se están haciendo en el panorama internacional y se presenta también la experiencia del autor en el diseño e impartición de un curso de iniciación a la programación para biólogos usando una metodología de aprendizaje basado en problemas.
The joint effect of biotechnology and computing has changed the research in biology. Consequently, computational biology is as essential for 21st-century biologists as molecular biology was in the 20th. However, Biology curricula have little emphasis in quantitative thinking and computation. The education for biologists should become as sophisticated as the computational education of physicists and engineers. The necessary changes to reach this goal require the connection of mathematics and quantitative subjects with real biological problems and at the same time, teaching some biological subjects from a modeling and computational perspective. In the present work, some of the current international effort in this path is reviewed and additionally, the author's experience when teaching an introduction to programming for biologists is presented.
En el siglo xxi la enseñanza universitaria se encuentra ante grandes desafíos, inmersa en un contexto de gran complejidad multicultural, científica y tecnológica. Esta situación es reflejo de la nueva sociedad del conocimiento que resulta de las revoluciones de la tecnología de la información y la biotecnología. La transformación social, laboral y tecnológica y sus consecuentes demandas en los ámbitos sociales, laborales, económicos, etc., implica que la educación cobra una preponderancia antes nunca vista y aparece como un derecho, y una necesidad, permanente. Así, el aprendizaje planteado como un proceso a lo largo de la vida desempeña un doble papel tanto desde el punto de vista humanista como economicista (Dávila Heitmann, 2013). Esta transición desde la era de la información a la del conocimiento exige un gran cambio en el modelo educativo. En el ámbito de la educación superior, las universidades funcionan en un entorno cada vez más globalizado, caracterizado también por una creciente competición para captar los mejores talentos. Pero, además, están marcadas por la aparición de nuevas demandas a las que deben responder y, por ello, parece imperioso que el alumno adquiera el desarrollo de nuevas competencias, es decir, comportamientos y habilidades que permiten el desempeño de determinadas tareas (Bajo Santos, 2010). En este escenario, es innegable la necesidad de nuevos modelos de educación universitaria, donde el papel de las universidades como generadoras de conocimiento se transforme hacia el de generadoras de generadores de conocimientos (Coccoli, Guercio, Maresca y Stanganelli, 2015).
Centrémonos ahora en nuestro país, España, miembro de la Unión Europea (UE). Conviene recordar que si bien la Unión Europea es una asociación política y económica, parece cada vez más claro que su persistencia en el tiempo debe pasar por la construcción de una nueva ciudadanía, bajo un nuevo modelo educativo de calidad que aun respetando y aprovechando el rico patrimonio y acervo cultural de sus estados miembros, sea capaz de generar un ámbito común para la educación de esos mismos ciudadanos. Es en este contexto europeísta, inmerso en un mundo cada vez más globalizado, con la tecnología y el conocimiento ineludiblemente enlazados al desarrollo económico y social, donde la Declaración de Bolonia en 1999 planteaba situar la educación superior en el marco de un espacio europeo común. Esta reforma, implantada en nuestro país de forma obligatoria a partir del curso 2010-2011, ha generado intensos debates, tanto debido al modo de implementación como a las insuficientes pautas de inversión (Bajo Santos, 2010). Sin embargo, no es el objetivo del presente trabajo discutir sobre la reforma de Bolonia en general, sino que, al contrario, en el campo de la biología, se planteará la necesidad de profundizar más en este proceso y en la transformación curricular todavía necesaria. Es verdad que el paso a Bolonia obligó a un cambio de las carreras universitarias en general y de la biología en particular, aunque solo fuera por el hecho de acortar su duración en un año, además de incidir en un rol más proactivo del alumno y la adquisición de competencias. Sin embargo, si comparamos con los planes de antaño veremos que, en muchos casos, la nueva biología se ha quedado más en un tenue maquillaje de reorganización académica que en la profunda transformación en cuanto a contenidos y competencias que el papel del biólogo demanda en el mundo actual. Los motivos de esta resistencia al cambio pueden deberse tanto a causas intrínsecas al sistema universitario español y sus carencias (De la Herrán, 2010, 2011) como a que un proceso de transformación de tal calado debe necesariamente ocurrir de manera gradual. Por tanto, lo que a continuación se va a argumentar es, en general, la necesidad de transformar una enseñanza descriptiva de la biología en otra más cuantitativa y, en particular, la incorporación al currículo del biólogo de la competencia de programación informática, entendida como la capacidad de desarrollar y realizar modificaciones en programas informáticos y aplicaciones, así como de entender los principios de la algoritmia y la programación, y su utilidad en el campo de la biología.
Hacia una biología computacional y matemáticaHoy en día, dentro de las carreras científicas coexisten 2 culturas u orientaciones: una matemática y otra menos matemática y más descriptiva, enmarcándose la biología claramente dentro de esta última (Baumgartner et al., 2015; Bialek y Botstein, 2004; Chiel, McManus y Shaw, 2010). Esto es preocupante dada la gran demanda que las ciencias de la vida tienen actualmente de biólogos con conocimientos de algoritmia, estadística, modelado y programación (ver más adelante). Por diversos motivos, la manera de adquirir conocimiento en la carrera de Biología tiene una escasa orientación cuantitativa y esto afecta tanto a los estudiantes como al profesorado (Chiel et al., 2010). Además, no es raro que el estudiante en Biología tenga un fuerte sentimiento de rechazo hacia las matemáticas y conocimientos afines. Este rechazo a veces se conoce como «ansiedad matemática», y entre sus varias causas se incluyen tanto las dificultades que experimenta el alumno ante el conocimiento abstracto, ya sea este de tipo matemático o de cualquier otro ámbito, como un desconocimiento casi absoluto sobre su utilidad en biología (Lahoz-Beltrá et al., 2011). El problema de la desconexión de la biología con respecto a una formación con más razonamiento abstracto y matemático no pertenece tan solo al ámbito español, ni siquiera europeo, y su solución no consiste simplemente en añadir más asignaturas impartidas por profesorado con escasa preparación para establecer las adecuadas conexiones entre el razonamiento cuantitativo y los hechos biológicos de interés (Bialek y Botstein, 2004; Feser, Vasaly y Herrera, 2013; Karsai, Thompson y Nelson, 2015). Si queremos incorporar la Biología dentro de las ciencias cuantitativas necesitaremos cambios profundos respecto a los planes docentes tal y como se imparten hoy. Obviamente esto no es tarea fácil, pero mediante la programación informática y el modelado de problemas biológicos es posible integrar, de manera relativamente sencilla, conocimientos matemáticos, estadísticos y computacionales junto con muchas de las nociones, problemas y modelos biológicos (Eager, Peirce y Barlow, 2014; Carvajal-Rodriguez, 2012; Karsai et al., 2015; Rubinstein y Chor, 2014; Weisstein, 2011). Por ejemplo, uno de los más importantes paquetes de cálculo estadístico utilizados en biología es el llamado «R» (R Development Core Team, 2006), que permite la realización de multitud de tareas estadísticas de interés y que es, a la vez, una aplicación de cálculo estadístico y un lenguaje de programación. Por tanto, R puede utilizarse para enseñar conceptos básicos de programación, como bucles y estructuras de decisión, al mismo tiempo que métodos estadísticos de gran utilidad en biología, como la estimación de parámetros por regresión (Eager et al., 2014).
Caben pocas dudas sobre la creciente importancia de los conocimientos de algoritmia y programación en el ámbito de la biología. La biología computacional es un campo multidisciplinar que incluye el desarrollo y la aplicación de métodos de análisis de datos, el modelado matemático y el desarrollo de técnicas de simulación para el estudio de sistemas biológicos. De modo similar, según la definición de los Institutos Nacionales de Salud en Estados Unidos, el concepto de bioinformática incluye la investigación, el desarrollo y el uso de herramientas computacionales para el manejo y el aprovechamiento de datos relacionados con ciencias de la vida como Biología y Medicina. Todo ello requiere las habilidades necesarias para adquirir, almacenar, analizar, organizar, simular o visualizar dichos datos (Cresiski, 2014; Huerta, Haseltine, Liu, Downing y Seto, 2000).
La importancia de la biología computacional ha llegado a ser tal que cada vez son más las voces que sostienen que un currículo en Biología que no incluya este tipo de competencias es prácticamente irrelevante (American Association for the Advancement of Science, 2011; Bialek y Botstein, 2004; Helikar et al., 2015; Jungck y Weisstein, 2013; Maloney et al., 2010; Pevzner y Shamir, 2009; Rubinstein y Chor, 2014). De hecho, poseer la competencia de unos conocimientos básicos de programación se contempla ya, en algunos casos, como prerrequisito para una educación computacional más avanzada en los grados en ciencias de la vida (Rubinstein y Chor, 2014).
Programación informática para biologíaLos conocimientos básicos de programación tienen una utilidad inmediata para el biólogo actual, permitiéndole analizar datos –en forma numérica y gráfica–, desarrollar algoritmos, modelar sistemas y realizar simulaciones biológicas. También le permite crear sus propias aplicaciones, que, por ejemplo, integren diversos programas y distintos formatos de datos o automatizar tareas repetitivas, que consumen mucho tiempo y están sujetas a errores humanos.
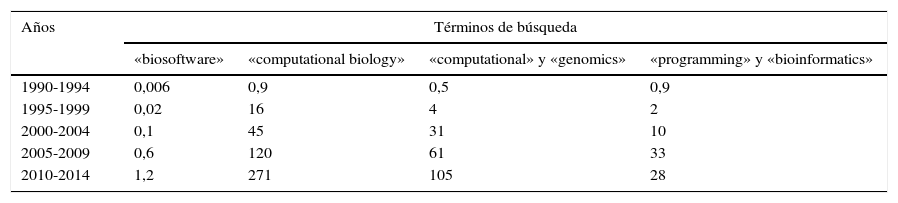
Un reflejo de la importancia creciente de este tipo de conocimientos se puede inferir del número de artículos publicados en revistas científicas internacionales que incluyan palabras (en inglés) como «biosoftware», «computational biology», etc. En la tabla 1 podemos observar los resultados (en miles de artículos), agrupados en quinquenios desde 1990 hasta el año 2014, de la búsqueda en el Google Académico de algunos de estos términos. Se aprecia que, por ejemplo, el término «biología computacional» pasó de aparecer en apenas 900 artículos entre 1990 y 1994 a hacerlo en más de un cuarto de millón de artículos entre 2010 y 2014.
Número de artículos (en miles) resultado de la búsqueda en Google Académico de diferentes términos relacionados con la biología computacional y la bioinformática
| Años | Términos de búsqueda | |||
|---|---|---|---|---|
| «biosoftware» | «computational biology» | «computational» y «genomics» | «programming» y «bioinformatics» | |
| 1990-1994 | 0,006 | 0,9 | 0,5 | 0,9 |
| 1995-1999 | 0,02 | 16 | 4 | 2 |
| 2000-2004 | 0,1 | 45 | 31 | 10 |
| 2005-2009 | 0,6 | 120 | 61 | 33 |
| 2010-2014 | 1,2 | 271 | 105 | 28 |
Fecha de la búsqueda: 14 de junio de 2015.
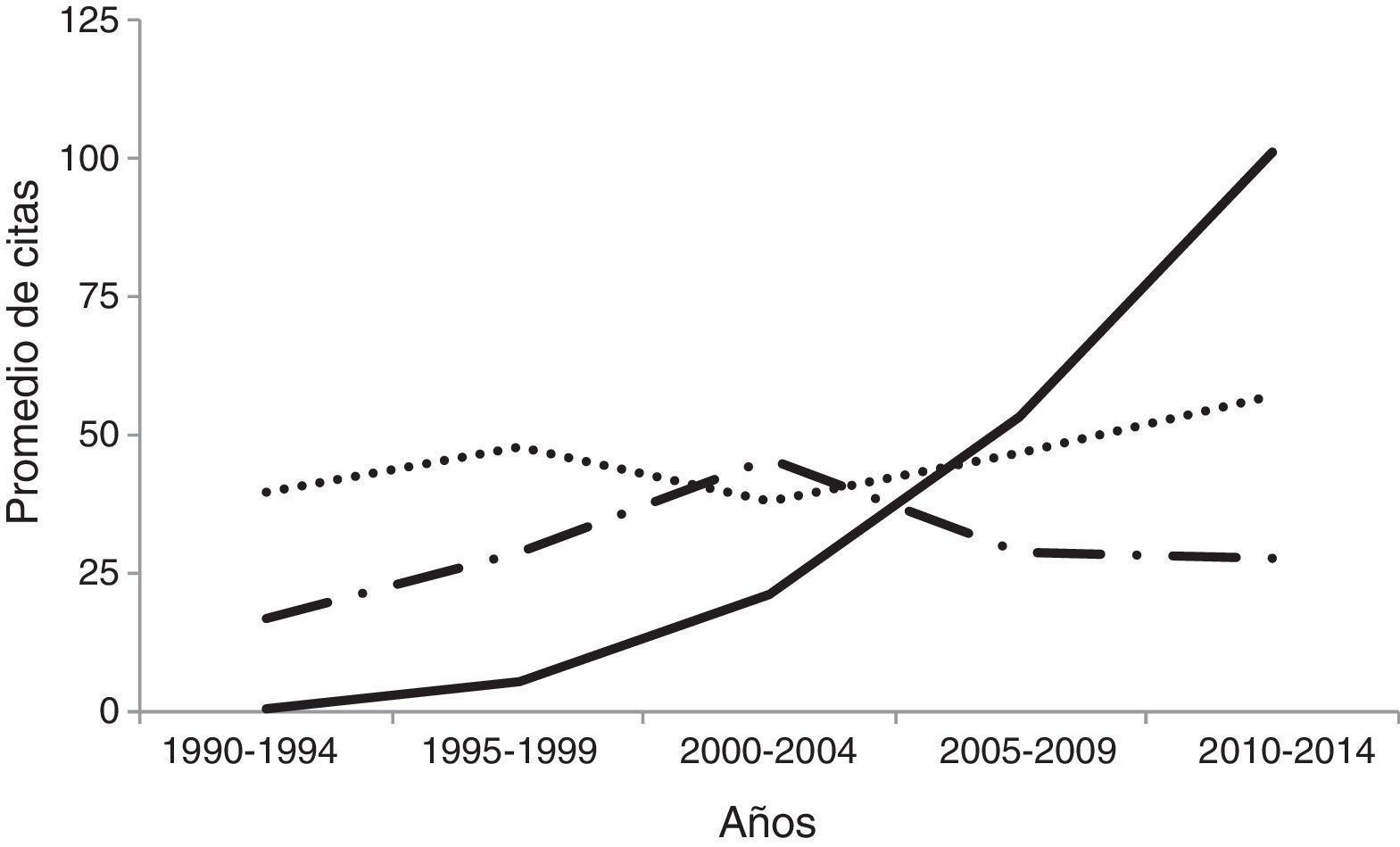
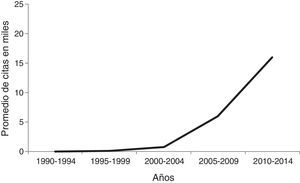
Se podría argumentar que este incremento no es específico del campo de la biología computacional, sino que se observaría un patrón similar para cualquier término, dado el incremento del número de publicaciones en general. Sin embargo, en la figura 1 se representa en trazo continuo la progresión media para los términos de la tabla 1, y en trazo discontinuo con guiones la progresión equivalente para el término «biología celular». Ambos, en miles de artículos. Queda claro que el incremento de los términos relacionados con la biología computacional no es un artefacto del número de artículos. Con trazo punteado se dan los resultados para el término «biología molecular» –en este caso las unidades son decenas de miles– y, obviamente, todavía se publican muchos más artículos con este término. Pero lo que interesa resaltar aquí es la pauta de evolución de las referencias relacionadas con el ámbito de la «biología computacional», que, como se aprecia, ha experimentado un gran incremento en los últimos años.
Progresión de las citas de artículos científicos en Google Académico. Línea continua (en miles): promedio de diferentes términos relacionados con biología computacional, según los datos de la tabla 1. Línea discontinua con guiones (en miles): término «cellular biology». Línea punteada (decenas de miles): término «molecular biology».
Parece claro que los conocimientos en bioinformática se requieren en muchos ámbitos de la biología y la medicina. De hecho, la necesidad de un cambio profundo en la formación de los biólogos para adaptarse a esta situación ha sido ya documentada por algunas de las más importantes organizaciones científicas y universidades, especialmente en Estados Unidos (American Association for the Advancement of Science, 2011). Sin embargo, la naturaleza y complejidad de los problemas que el nuevo biólogo debe abordar es multidisciplinar y diversa, de modo que las herramientas y habilidades necesarias pueden ser de muy distinta índole. En cualquier caso, un recurso fundamental y muy versátil que todo biólogo debería tener a su disposición sería el conocimiento de algún lenguaje de programación (Dudley y Butte, 2009).
Las ventajas del enfoque orientado a problemas como metodología docenteSe entiende por aprendizaje basado en problemas aquel que plantea el proceso de aprendizaje mediante la necesidad de resolver un problema real relacionado con el campo de estudio en cuestión. De este modo se ponen en juego distintas maneras de aprender (Wood, 2003; Hung, Jonassen y Liu, 2008; McLoughlin, Burns y Darvill, 2015). Durante el proceso de resolución del problema se favorece el aprendizaje significativo, y los estudiantes obtienen nuevos conocimientos y desarrollan habilidades resolutivas, así como aptitudes de autoaprendizaje (Hung et al., 2008). Este tipo de estrategia se adapta de manera esencial a la enseñanza de la bioinformática (Helikar et al., 2015; Jungck, Donovan, Weisstein, Khiripet y Everse, 2010; Jungck y Weisstein, 2013; White y Dhurjati, 2006) y, por tanto, parece adecuado para introducir la programación informática en biología.
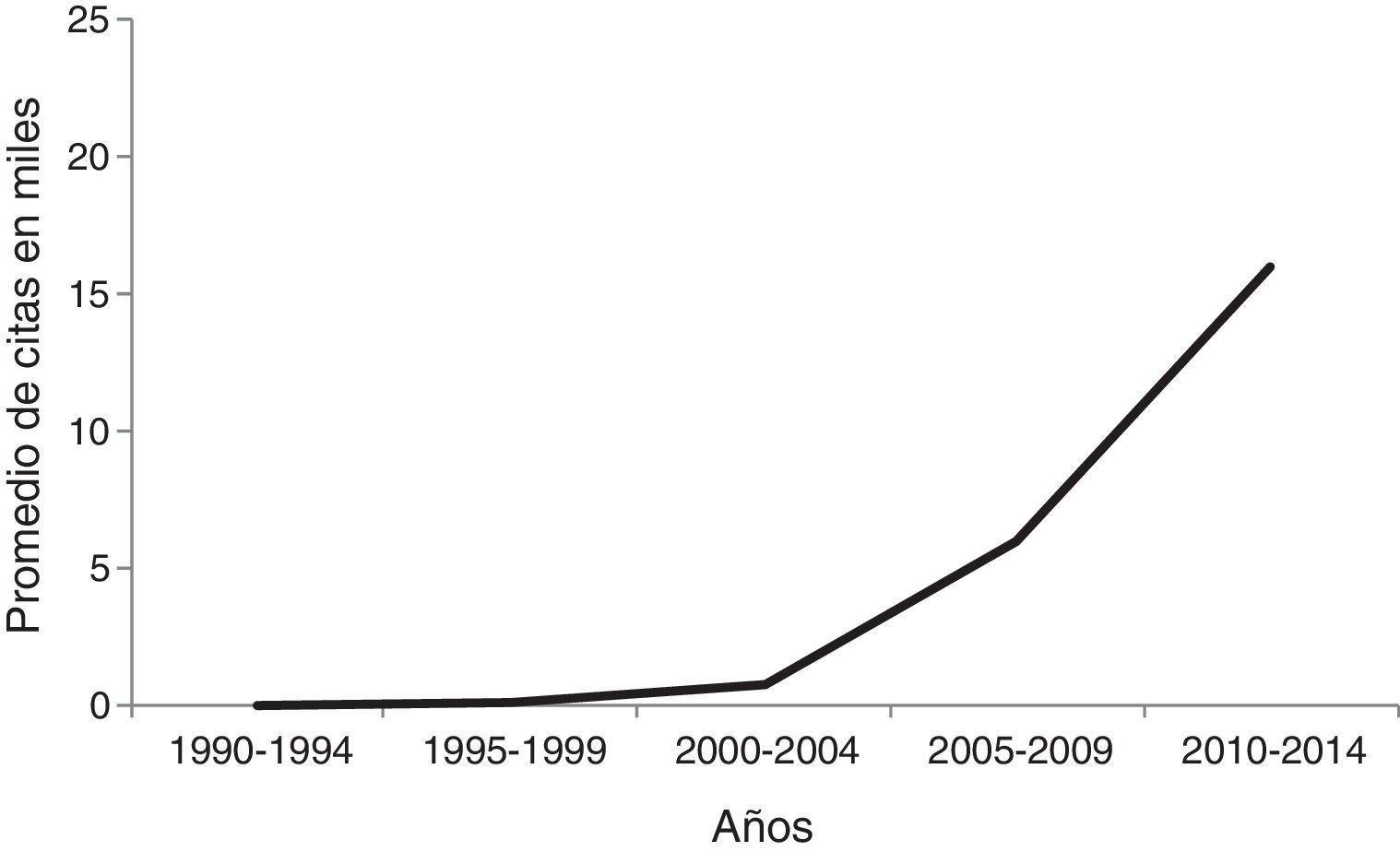
Las ventajas del lenguaje de programación Python para la docencia en biologíaEl lenguaje Python es un lenguaje gratuito de gran uso actualmente en bioinformática (Haddock y Dunn, 2011). Es un lenguaje de alto nivel y de propósito general, lo que significa que permite abordar muy diferentes tipos de tareas. Además, es portable, lo que quiere decir que funciona bajo distintos sistemas operativos. Tiene una sintaxis relativamente sencilla y hay abundantes recursos disponibles online, incluyendo un buen número de herramientas científicas con amplio soporte mediante comunidades (Perkel, 2015). Entre estos paquetes científicos se encuentran bibliotecas como Matplotlib y Pylab (para generación de gráficos), NumPy y SciPy (para aplicaciones numéricas y científicas) y Biopython (herramientas para biología computacional) (Bassi, 2007). Incluye también herramientas especializadas, como IPython, que facilitan el compartir código y trabajar de modo interactivo (Shen, 2014). Para mostrar su cada vez mayor uso en bioinformática se ha buscado en Google Académico la aparición conjunta de los términos «bioinformática» y «Python» (en inglés) en artículos científicos entre los quinquenios 1990-1994 hasta 2010-2014. En la figura 2 se puede apreciar que durante el último decenio, el número de artículos científicos incluyendo ambos términos se ha incrementado en más de un orden de magnitud.
Desde el punto de vista docente, Python tiene la ventaja extra de permitir un modo de ejecución interactivo donde los comandos y las estructuras del lenguaje pueden mostrarse paso a paso, por parte del profesor, y probarse, por parte del alumno, en tiempo real sin necesidad de haber implementado el código de un programa completo.
Diseño de un curso de introducción a la programación para biólogos usando PythonDe aquí en adelante se va a trasladar, de modo sucinto, la experiencia del autor en el diseño e impartición de un curso, nivel de posgrado, de iniciación a la programación para biólogos. Se utiliza Python como lenguaje de programación y una metodología de enseñanza-aprendizaje basada en la resolución de problemas. El curso corresponde a 3 ECT con aproximadamente 18h de docencia/trabajo presencial. Los alumnos inscritos fueron únicamente 4 y solo uno de ellos tenía ciertos conocimientos previos de programación.
Detalles técnicos: uso de una máquina virtualUna máquina virtual no es más que un programa (software) que simula un ordenador. Su uso permite ejecutar un sistema operativo a elección sobre cualquier ordenador, independientemente del sistema operativo que este tenga originalmente instalado, junto con los programas que al configurar la máquina virtual se decida instalar o que se instalen posteriormente. Ello posibilita disponer de un entorno de programación controlado e idéntico para todos los estudiantes y el profesor. La utilización de máquinas virtuales se ha extendido debido a la existencia de programas gratuitos que facilitan su creación y uso. Uno de los más conocidos es Oracle VM VirtualBox (https://www.virtualbox.org/). Por tanto, para impartir el curso se configuró una máquina virtual en la que se instaló Python junto con un entorno de desarrollo que facilita las tareas de programación (por ejemplo, Sublime Text3; http://www.sublimetext.com/3). La máquina virtual ya configurada se puso a disposición de los alumnos, a los que se envió un correo con instrucciones para su instalación, la cual es muy sencilla, realizándose en tan solo un par de pasos.
Metodología docente. Conceptos previos e introducción a los problemasComo se ha dicho, Python tiene la gran ventaja de que permite trabajar en modo interactivo, en el cual el alumno puede ir probando trozos de código y viendo los resultados de manera inmediata. Esta característica se aprovecha para que los estudiantes ensayen y prueben conceptos básicos como variables, datos de tipo numérico o tipo cadena, la función de ayuda (help) y otras funciones básicas predefinidas en el lenguaje, como len, print, etc. El tiempo dedicado a esta parte introductoria es de aproximadamente una hora. Llegados a este punto se plantea el primer problema con un diseño que se va a repetir a lo largo de los restantes, a saber, se mencionan, sin apenas o con una muy breve explicación, los conceptos de programación implicados y se plantea el enunciado. Cada problema se subdivide en varios apartados, correspondientes a subproblemas. Los problemas serán de complejidad creciente según avanza el curso y estarán siempre vinculados al ámbito de la biología. El curso se distribuye idealmente en 5 días no consecutivos. Cada día se dedica a la presentación de un problema y a trabajar en ese mismo problema o en uno anterior. Sin embargo, dada la heterogeneidad del alumnado, se permitió un ritmo muy flexible e independiente para cada alumno. Los alumnos disponen, además, de acceso a una plataforma de docencia virtual que incluye un foro donde pueden plantear y resolver cuestiones, ya sea entre ellos o con la ayuda del profesor. En la tabla 2 se muestra la temática relacionada con cada uno de los problemas utilizados en el curso. Los apuntes del curso, incluyendo los enunciados de los problemas, están disponibles en http://acraaj.webs.uvigo.es/cursos/PythonCourse.pdf
Serie de problemas que los alumnos afrontan durante el curso de introducción a la programación para biólogos
| Problema | Tipo |
|---|---|
| Lee ADN | Manejo básico de ficheros y cadenas ADN |
| Analiza ADN-1 | Análisis de formatos de secuencias y codones de ADN |
| Analiza ADN-2 | Análisis avanzado de formatos |
| Simulación Monte Carlo | Remuestreo de secuencias, estimación de parámetros |
| Creación de un módulo | Diseño del módulo ADN. Programación orientada a objetos |
Este problema se divide en 3 subproblemas, a saber: temperatura de fusión del ADN, cadena complementaria y longitud de fragmentos de restricción. Durante su desarrollo se abordan conceptos básicos como tipos de datos, variables, funciones predefinidas del lenguaje, fórmulas, la entrada de datos por teclado y la apertura y lectura de ficheros.
Problema 2. Analiza ADN-1Dividido en 2 subproblemas: maneja formatos y tabla de codones. Implican conversión entre distintos formatos de secuencias de ADN y entre codones y aminoácidos. Para ello el alumno debe manejar conceptos de programación tales como bucles, listas y diccionarios, además de la lectura y escritura de ficheros.
Problema 3. Analiza ADN-2Dos subproblemas: conversor y url. El primero es una generalización del problema maneja formatos, que permite la transformación de formato Phylip a Fasta para cualquier fichero. El segundo consiste en la utilización del módulo de Python urlib para leer el contenido de una página web, reorganizar su formato y guardarlo en un fichero. Durante la resolución de estos problemas se sigue profundizando en conceptos previamente vistos, así como la importación de módulos predefinidos del lenguaje y la definición de funciones por parte del programador.
Problema 4. Simulación Monte CarloSe introduce el módulo Random. Se divide en 2 subproblemas: dados y secuencias. El subproblema secuencias es bastante complejo, puesto que el alumno debe recapitular todo los visto hasta el momento, incluyendo el manejo de bucles, fórmulas, funciones, lectura de ficheros, etc.; deberá, además, programar un método de remuestreo sobre las secuencias leídas y calcular medias y varianzas.
Problema 5. Creación de un móduloEste problema no se llegó a realizar por falta de tiempo. Se divide en 2 subproblemas. En el primero se introducen algunos conceptos relacionados con la programación orientada a objetos; dadas unas propiedades, atributos y métodos, el alumno debe definir las clases Gen y Secuencias e incluirlas en un módulo. El segundo subproblema utilizaría el módulo recién creado para leer un fichero en formato Phylip y calcular la media y la varianza del número de purinas en las secuencias.
Evaluación del aprendizajeLa evaluación se realizó de manera continua con la entrega del código y programa de cada problema y también mediante un breve test final. Para superar la asignatura se exigió entregar al menos los 2 primeros problemas correctamente resueltos y aprobar el examen test. Respecto a la valoración del curso por parte de los alumnos, dada su heterogeneidad y bajo número, carecía de sentido realizar una encuesta anónima, que se sustituyó por charlas informales, tanto en grupo como a nivel particular. La impresión general fue de satisfacción, si bien las expectativas, al igual que los conocimientos previos, de cada uno eran muy diferentes. Al menos 2 de los alumnos continúan actualmente su formación en el campo de la biología computacional. A pesar de la heterogeneidad de los estudiantes en cuanto a la destreza en razonamiento abstracto y pensamiento computacional, el curso debería haber valido, cuando menos, para incrementar su competencia en estos aspectos.
DiscusiónEn este trabajo se ha planteado la conveniencia de transformar el currículo del biólogo. La reorientación que se propone iría desde el enfoque actual, más descriptivo, a otro más abstracto, matemático y computacional. Aun en caso de aceptarse la necesidad de este cambio, las dificultades para llevarlo a cabo son bastantes, no siendo la menor entre ellas las carencias por parte del profesorado de ciencias de la vida en los mismos aspectos que se desean incorporar, además de la complejidad inherente al enfoque multidisciplinar, los intereses más o menos corporativistas en los departamentos a la hora de diseñar la docencia en las facultades, etc. No obstante, la profunda y urgente necesidad del susodicho cambio no es solo la opinión de este autor. Hay ya en marcha una fuerte corriente a favor de este proceso transformador, especialmente en muchas universidades norteamericanas e impulsado nada menos que por la Asociación Nacional para la Ciencia en ese país (Eager et al., 2014; American Association for the Advancement of Science, 2011; Feser et al., 2013; Helikar et al., 2015; Rubinstein y Chor, 2014). Todo ello es consecuencia, al menos parcialmente, de la existencia de un nuevo mercado de trabajo para un nuevo tipo de biólogo que pueda lidiar con la ingente cantidad actual de información y datos, la genómica, la proteómica y las nuevas cuestiones y problemas que de todo ello se derivan.
Hay diversos modos de incorporar el pensamiento abstracto y computacional a un currículo. Una alternativa factible e interesante sería, entre otras, la inclusión de la enseñanza de la programación informática como asignatura base, pero también integrada en las diferentes materias. Por ejemplo, ya hemos mencionado que el lenguaje de programación R está orientado a la estadística, con lo cual es posible impartir esta asignatura al tiempo que se aprenden a programar algunas tareas sencillas en este lenguaje. Así mismo, muchos conceptos en ecología, evolución, genética y bioquímica incorporan modelos básicos que se pueden programar con relativa sencillez. En este mismo manuscrito he presentado una iniciativa mediante aprendizaje basado en problemas para impartir una introducción a la programación para biólogos. Se ha visto que es posible aprender los fundamentos de la programación utilizando el lenguaje Python, al tiempo que se resuelven diversas tareas de manejo y análisis de secuencias de ADN, cálculo de media y varianza, etc. También existen actualmente programas que permiten simular modelos de interés en ecología y otras áreas sin necesidad de que el estudiante programe todas las tareas directamente.
Adicionalmente, una de las ideas clave que es necesario resaltar es que la incorporación de un enfoque más abstracto y matemático al currículo del biólogo no necesariamente pasa por la inclusión de más asignaturas de matemáticas, estadística, etc., sino más bien por la integración de las ya existentes en el ámbito de los problemas biológicos, al mismo tiempo que se realiza una contextualización de las asignaturas propias de la biología desde un punto de vista más formal y de modelización.
Finalmente, queda indicar que, ya que hemos recordado los profundos cambios socioeconómicos que dieron lugar al proceso de Bolonia, así como algunas de las demandas concretas en la formación de los modernos biólogos, es ineludible hacer un breve apunte sobre financiación e inversión. Evidentemente, este debería ser un aspecto clave tanto en la reforma de Bolonia (Bajo Santos, 2010) como en la actualización de los currículos en ciencias de la vida en general, y en Biología en particular, para adecuarse a las demandas de la ciencia y la sociedad del siglo xxi. En este aspecto, y en opinión del autor, las administraciones y sus políticas no solo no han estado a la altura, sino que han tenido un desempeño bastante deficiente. Es verdad que hablar de financiación e inversión en ningún caso significa «café para todos» ni ausencia de valoración del mérito. Al contrario, es la tarea del alto cargo público habilitar los mecanismos que permitan diseñar políticas eficientes para costear y gestionar el gasto en investigación, educación e innovación. Lo que está claro es que sin una inversión sostenida de manera racional y eficaz no es posible generar el tipo de cambio que la educación de cualquier país desarrollado demanda en el siglo xxi. Se ha dicho muchas veces, aunque no suficientes: es fundamental desterrar todo cortoplacismo y visión partidista en temas como la educación, donde sería necesario y clave un pacto de estado.
Para concluir, y retomando el aspecto meramente docente, quiero indicar que, a nivel personal, el curso impartido ha sido una experiencia bastante satisfactoria, con el valor añadido de que este tipo de curso es fácilmente adaptable a otros ámbitos y plataformas, como, por ejemplo, los cada vez más demandados cursos online.
Conflicto de interesesEl autor declara no tener ningún conflicto de intereses.
El autor agradece a Silvina Domínguez, M. Fernández y M. Megías los comentarios y discusión sobre una versión previa del artículo.