La variabilidad de la expresión y la evolución de la COVID no se explican completamente por los factores clínicos, atribuyéndose un importante papel a los genéticos. Además, se ha planteado si los mismos componentes genéticos que participan en la susceptibilidad y gravedad de la infección influyen en su evolución hacia long COVID. Como objetivo nos propusimos revisar la literatura a fin de conocer cuáles son los factores genéticos que intervienen en la génesis de la COVID persistente.
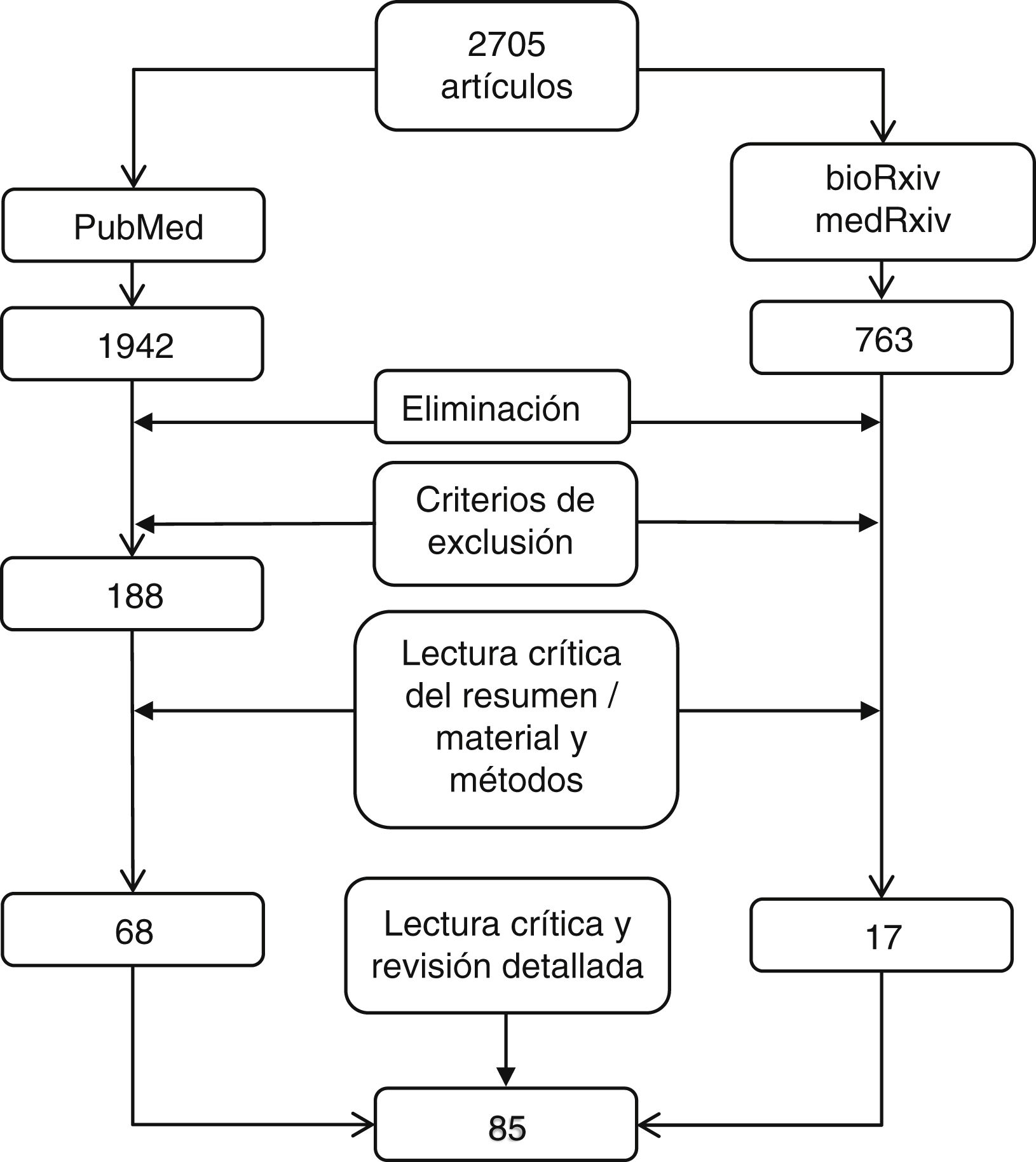
Material y métodosRevisión sistemática en PubMed y repositorios bioRxiv y medRxiv con base en los descriptores y términos Medical Subject Headings (MeSH) relacionados con COVID y factores genéticos. Fueron seleccionados 2.705 artículos. Un primer cribado, realizado de manera independiente por los autores, redujo la lista a 205 y finalmente, tras un análisis más detallado, se eligieron 85 trabajos para su lectura completa y revisión.
ResultadosLa enzima convertidora de angiotensina 2 (ACE2) y la proteasa transmembrana, serina 6 (TMPSS6) están implicadas en la susceptibilidad, sin embargo, no se ha encontrado su participación en long COVID. Sí se han hallado algunas asociaciones entre genes, que intervienen en la respuesta inflamatoria e inmune, con la gravedad de la enfermedad y el desarrollo de long COVID. La relación más importante se ha observado en el locus FOXP4.
ConclusionesAunque actualmente la información sobre long COVID es limitada, parece claro que los factores genéticos identificados hasta ahora no justifican la progresión hacia una enfermedad persistente y se debe considerar la participación de otros componentes como la acción poligénica, de genes pleiotrópicos, de la microbiota y de los cambios epigenéticos.
The variability in expression and evolution of COVID is not completely explained by clinical factors. In fact, genetic factors play an important role. Moreover, it is unknown whether the genetic factor that contribute to susceptibility and severity are also involved in the onset and evolution of long-COVID. The objective of this review is to gather information from literature to understand which genetic factors are involved in the onset of persistent COVID.
Material and methodsSystematic review in PubMed and bioRxiv and medRxiv repositories based on MeSH-descriptors and MeSH-terms related to COVID and genetic factors. Using these terms 2715 articles were pooled. An initial screening performed by authors independently, selected 205 articles of interest. A final deeper screening a total of 85 articles were chosen for complete reading and summarized in this review.
ResultsAlthough ACE2 and TMPSS6 are involved in COVID susceptibility, their involvement in long-COVID has not been found. On the other hand, the severity of the disease and the onset of long-COVID has been associated with different genes involved in the inflammatory and immune response. Particularly interesting has been the association found with the FOXP4 locus.
ConclusionsAlthough studies on long-COVID are insufficient to fully comprehend the cause, it is clear that the current identified genetic factors do not fully explain the progression and onset of long-COVID. Other factors such as polygenic action, pleiotropic genes, the microbiota and epigenetic changes must be considered and studied.
En diciembre de 2019 se identificó un nuevo coronavirus, como agente responsable de los casos de un síndrome respiratorio agudo severo que afectaba a la población de la región de Hubei en China. Desde entonces, el virus se ha extendido a lo largo del mundo. La Organización Mundial de la Salud (OMS) declaró la situación de pandemia en marzo de 2020 y el fin de la emergencia sanitaria en mayo de 2023. En este tiempo, a nivel global, se han registrado casi 800 millones de casos y unos 8 millones de muertes1, aunque informes de la Organización de las Naciones Unidas (ONU) estiman que las cifras reales pueden, al menos, duplicar estas cantidades2.
Aunque la duración de la enfermedad suele ser de tres a seis semanas, dependiendo de la gravedad3, en ocasiones, perdura durante más semanas o meses dando lugar a síntomas multisistémicos incapacitantes y persistentes sin tratamientos hasta ahora efectivos. La OMS define como COVID prolongado, persistente o long COVID cualquier síntoma que se presente posteriormente de la COVID-19 y que se mantenga después de 12 semanas4. Se estima que al menos entre 10-30% de los infectados por SARS-CoV-2 pueden desarrollar síntomas a largo plazo, que pueden ser fluctuantes y afectar de manera importante la calidad de vida5.
Se han identificado diferentes factores de riesgo que predisponen a la persistencia de la enfermedad, como ser mujer, mayor gravedad de la infección, situaciones clínicas (diabetes mellitus tipo 2, reactivación del virus de Epstein Barr o de la varicela, alteraciones del tejido conectivo, trastorno por déficit de atención con hiperactividad y urticaria crónica), así como algunos factores culturales y sociodemográficos6,7.
Sin embargo, los factores clínicos no explican completamente la variabilidad en la expresión de la enfermedad entre individuos y pronto se comenzó a valorar el papel de la genética humana como factor de riesgo8.
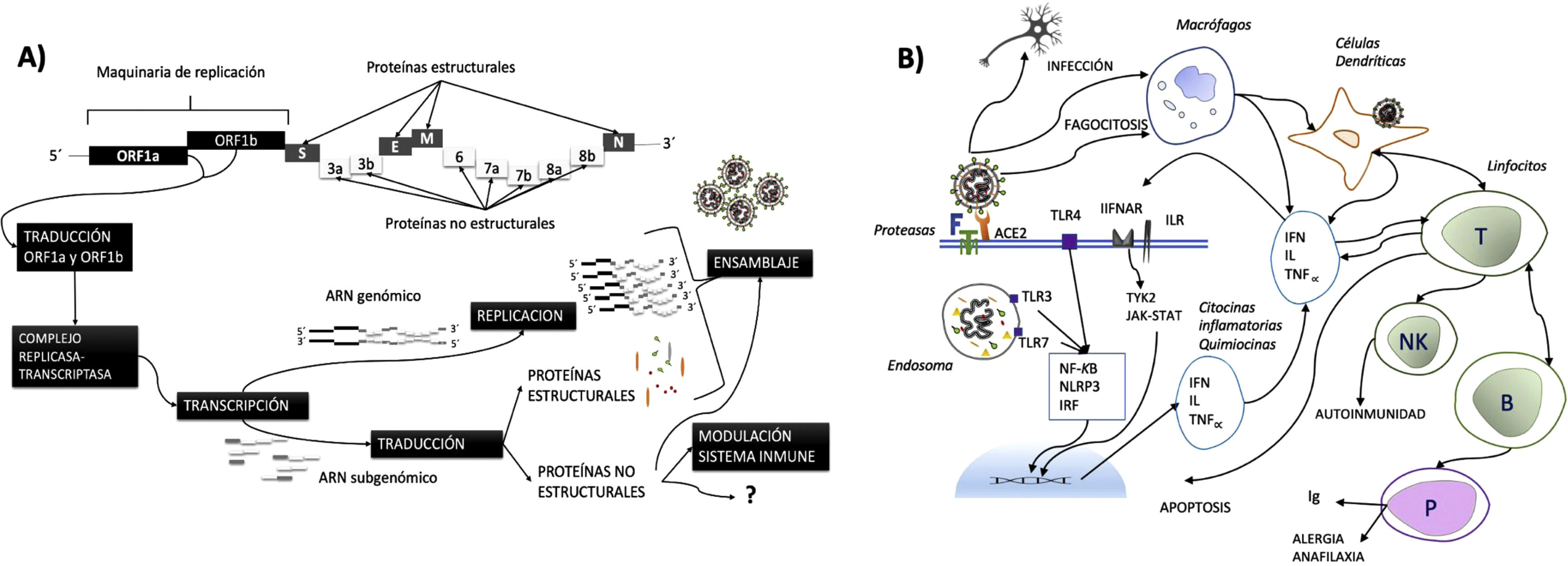
El SARS-CoV-2 contiene un ácido ribonucleico (ARN) monocatenario de polaridad positiva, de poco menos de 30 kb. Cuenta con unos 10-12 marcos de lectura abierta (ORF), seis de ellos se encuentran muy conservados y siguen un mismo orden. Estos últimos se encargan de codificar para la maquinaria de replicación y para las proteínas estructurales (N: nucleocápside, E: envoltura, M: membrana, S: spike). El resto se ocupan de la codificación para las proteínas accesorias9,10. Para su replicación necesita generar un ARN bicatenario (ARN genómico) que utiliza como molde. Sin embargo, la traducción para las distintas proteínas estructurales y accesorias se realiza mediante copias de diferentes fragmentos de ARN (ARN subgenómico)11, que muestran gran variabilidad (fig. 1).
A) Representación esquemática del genoma de SARS-CoV-2 y de los procesos de replicación, transcripción y traducción. B) Representación esquemática de la respuesta inmune e inflamatoria. En la entrada del virus en la célula interviene la enzima convertidora de la angiotensina 2 (ACE2) y los sistemas de proteasas. El ARN viral, junto con algunas partículas virales son incluidos en el endosoma, en cuya superficie se muestran receptores Toll-like (TLR) que van a activar, entre otros, factores reguladores de interferón (IRF), factor nuclear Kappa B (NF-KB) y componentes del inflamosoma (NLRP3). Todo ello activa los factores transcripcionales nucleares que conducen a la liberación de quimiocinas y citosinas inflamatorias (interferón [IFN], interleucina [IL], factor de necrosis tumoral alfa [TNF∝], etc.), que además de actuar sobre diferentes células del sistema inmune, van a generar bucles de retroalimentación positiva a través de receptores de interferón (IFNAR) de interleucinas (ILR) y TLR, ente otros y en los que participan las vías tirosínquinasa (TYK2, quinasa de Janus/transductor de señal y activador de transcripción [JAK-STAT]). La acción del virus y de las citosinas inflamatorias sobre las células defensivas generan interacciones entre ellas que conducen a la amplificación del proceso inflamatorio y a la participación multiorgánica.
ARN: ácido ribonucleico; Ig: inmunoglobulina.
El SARS-CoV-2 puede penetrar al interior de la célula mediante endocitosis microvesicular y por fusión con la pared celular. Para ello, requiere primero unirse mediante la proteína S al receptor celular de la enzima convertidora de la angiotensina 2 (ACE2)12–14; y segundo, escindirse en dos subunidades: S1 y S2 por medio de la acción de proteasas, entre las que destacan la serina proteasa celular de transmembrana tipos 2 y 4 (TMPRSS2 y TMPRSS4) y las furinas9,15.
El virus es reconocido por las células inmunitarias a través de receptores Toll-like como TLR3 y TLR7, y por otros sensores citoplasmáticos (DDX58, IFIH1, etc.) que promueven la síntesis de interferones (IFN) tipo I/III. Los IFN, mediante la unión a su receptor (IFNAR) activan vías de tirosínquinasa (TYK2, JAK1), que facilitan la expresión de genes como OAS1, OAS2 y OAS3, claves en la respuesta antiviral celular innata. El SARS-CoV-2 produce una desregulación de este sistema que resulta en una respuesta retrasada o insuficiente16,17.
Existen varias hipótesis acerca de la etiopatogenia de long COVID: la persistencia de la enfermedad en los tejidos, la respuesta inmunitaria a partir de la tormenta inflamatoria junto con patógenos subyacentes, alteraciones en la coagulación, las hormonas sexuales, cambios en la microbiota y los autoanticuerpos del mismo virus18,19. La secuenciación de ARN unicelular muestra que las células natural killer y los monocitos CD14+ se reducen, mientras que las células T reguladoras aumentan, disminuyendo todas ellas su actividad20.
Nuestro trabajo tiene por objetivo saber cuáles son los factores genéticos del huésped que intervienen en la génesis del long COVID.
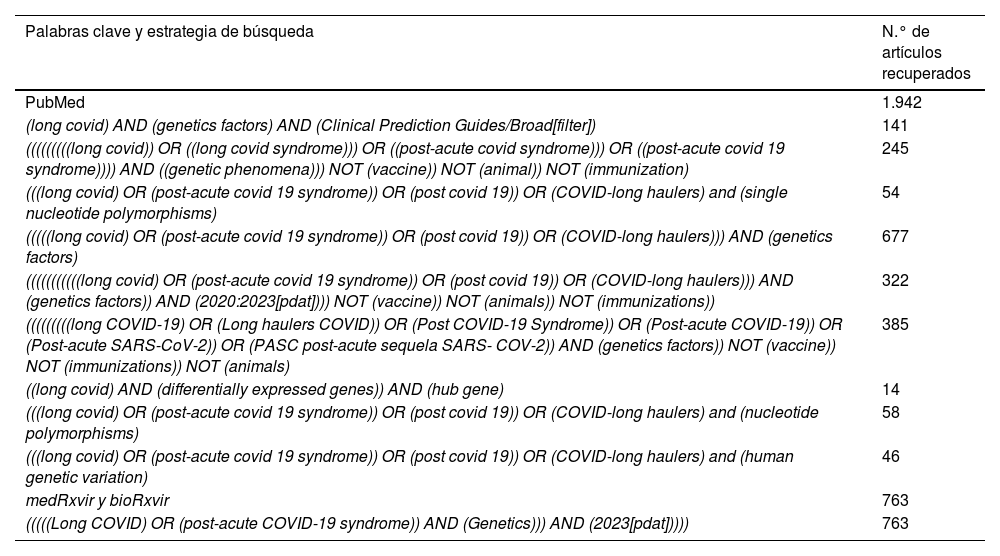
Material y métodosMétodo de búsqueda de literaturaSe realizó una primera búsqueda de artículos cardinales en PubMed relacionados con el tema de nuestro estudio. Se revisaron las principales palabras claves utilizadas en ellos. Estas fueron depuradas según el tesauro incluido en PubMed y se seleccionaron los términos Medical Subject Headings (MeSH) que las recogían.
Los términos seleccionados fueron: «post-acute COVID-19 syndrome», «long COVID», «long COVID-19», «long haulers COVID», «post-acute COVID-19», «post-acute SARS-CoV-2» «post-acute sequela SARS- COV-2», «genetics factors» «prediction guides», «genetic phenomena», «human genetic variations», «nucleotide polymorphism», «single nucleotide polymorphism». En la búsqueda se excluyeron los términos «animals», «inmunization» y «vaccine». El marco temporal fue desde enero de 2020 hasta agosto de 2023, e inicialmente se consideraron todas las publicaciones incluidas en PubMed. Con estos criterios se construyeron las estrategias de búsqueda recogidas en la tabla 1.
Estrategias de búsqueda y artículos recuperados
| Palabras clave y estrategia de búsqueda | N.° de artículos recuperados |
|---|---|
| PubMed | 1.942 |
| (long covid) AND (genetics factors) AND (Clinical Prediction Guides/Broad[filter]) | 141 |
| (((((((((long covid)) OR ((long covid syndrome))) OR ((post-acute covid syndrome))) OR ((post-acute covid 19 syndrome)))) AND ((genetic phenomena))) NOT (vaccine)) NOT (animal)) NOT (immunization) | 245 |
| (((long covid) OR (post-acute covid 19 syndrome)) OR (post covid 19)) OR (COVID-long haulers) and (single nucleotide polymorphisms) | 54 |
| (((((long covid) OR (post-acute covid 19 syndrome)) OR (post covid 19)) OR (COVID-long haulers))) AND (genetics factors) | 677 |
| (((((((((((long covid) OR (post-acute covid 19 syndrome)) OR (post covid 19)) OR (COVID-long haulers))) AND (genetics factors)) AND (2020:2023[pdat]))) NOT (vaccine)) NOT (animals)) NOT (immunizations)) | 322 |
| (((((((((long COVID-19) OR (Long haulers COVID)) OR (Post COVID-19 Syndrome)) OR (Post-acute COVID-19)) OR (Post-acute SARS-CoV-2)) OR (PASC post-acute sequela SARS- COV-2)) AND (genetics factors)) NOT (vaccine)) NOT (immunizations)) NOT (animals) | 385 |
| ((long covid) AND (differentially expressed genes)) AND (hub gene) | 14 |
| (((long covid) OR (post-acute covid 19 syndrome)) OR (post covid 19)) OR (COVID-long haulers) and (nucleotide polymorphisms) | 58 |
| (((long covid) OR (post-acute covid 19 syndrome)) OR (post covid 19)) OR (COVID-long haulers) and (human genetic variation) | 46 |
| medRxvir y bioRxvir | 763 |
| (((((Long COVID) OR (post-acute COVID-19 syndrome)) AND (Genetics))) AND (2023[pdat])))) | 763 |
- •
Trabajos no relacionados con el tema de estudio.
- •
Trabajos vinculados con la enfermedad aguda.
- •
Trabajos que no trataban los aspectos genéticos o que lo hacían exclusivamente de la genética vírica.
- •
Trabajos relacionados con otros coronavirus.
- •
Publicaciones sin revisión por pares.
- •
Editoriales, artículos de opinión, notas técnicas e informes de casos.
- •
Revisiones literarias.
Una vez retirados los trabajos duplicados, los autores, de manera independiente, revisaron los títulos y resúmenes de los artículos remanentes, excluyendo aquellos que no fueron relevantes con base en los criterios de inclusión/exclusión. Las discrepancias se resolvieron mediante discusión y acuerdo entre los investigadores. Se evaluaron 188 artículos que, tras un primer filtro, quedaron 68 para su lectura completa.
A fin de no excluir información relevante, dado lo novedoso del tema de estudio, se realizó una segunda búsqueda de literatura pendiente de publicación en los repositorios medRxvir y bioRxvir. Para ello, se utilizaron los descriptores «long COVID», «post-acute COVID-19 syndrome» y «genetics». Se revisaron todos los artículos incluidos entre enero y agosto de 2023. Esto supuso la consulta adicional de 763 trabajos de los que se seleccionaron para su lectura completa 17. Se aplicaron los mismos criterios de exclusión salvo el de revisión por pares (fig. 2).
Variables de estudioPara su evaluación, se consideraron las siguientes variables: año de publicación, tipo de estudio, población de estudio, gen, polimorfismo de un solo nucleótido (SNP), ubicación en el cromosoma y asociación del efecto.
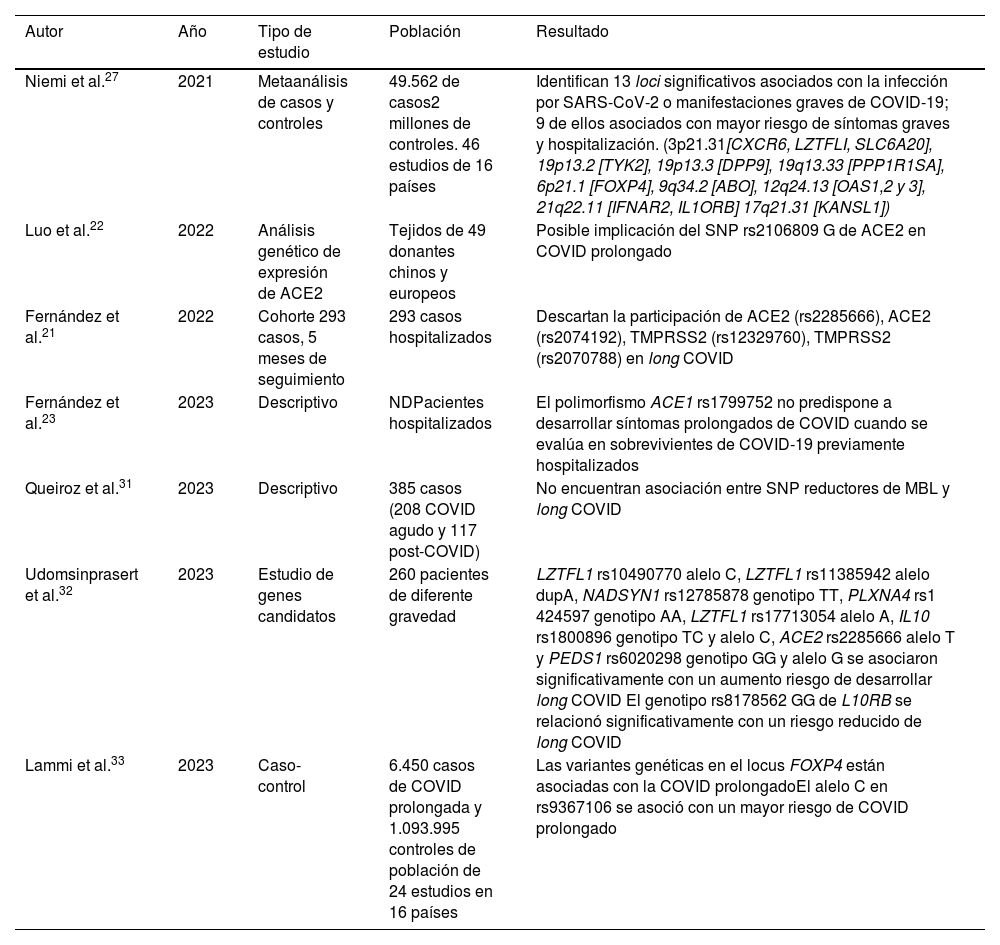
ResultadosLa mayor parte de los estudios genéticos se han centrado, por un lado, en conocer el genoma vírico y sus variantes, y por otro, en estudiar la expresión de locus de interés y genes candidatos relacionados, especialmente con los mecanismos de infectividad, la evasión del sistema inmune y con la respuesta inflamatoria disruptiva. En relación con la COVID persistente, son muy escasos los trabajos que abordan qué factores genéticos del huésped facilitan su aparición y se han enfocado en establecer si los factores genéticos de riesgo para la COVID grave lo son también para el long COVID (tabla 2).
Resumen de los principales estudios relacionados con long COVID
| Autor | Año | Tipo de estudio | Población | Resultado |
|---|---|---|---|---|
| Niemi et al.27 | 2021 | Metaanálisis de casos y controles | 49.562 de casos2 millones de controles. 46 estudios de 16 países | Identifican 13 loci significativos asociados con la infección por SARS-CoV-2 o manifestaciones graves de COVID-19; 9 de ellos asociados con mayor riesgo de síntomas graves y hospitalización. (3p21.31[CXCR6, LZTFLI, SLC6A20], 19p13.2 [TYK2], 19p13.3 [DPP9], 19q13.33 [PPP1R1SA], 6p21.1 [FOXP4], 9q34.2 [ABO], 12q24.13 [OAS1,2 y 3], 21q22.11 [IFNAR2, IL1ORB] 17q21.31 [KANSL1]) |
| Luo et al.22 | 2022 | Análisis genético de expresión de ACE2 | Tejidos de 49 donantes chinos y europeos | Posible implicación del SNP rs2106809 G de ACE2 en COVID prolongado |
| Fernández et al.21 | 2022 | Cohorte 293 casos, 5 meses de seguimiento | 293 casos hospitalizados | Descartan la participación de ACE2 (rs2285666), ACE2 (rs2074192), TMPRSS2 (rs12329760), TMPRSS2 (rs2070788) en long COVID |
| Fernández et al.23 | 2023 | Descriptivo | NDPacientes hospitalizados | El polimorfismo ACE1 rs1799752 no predispone a desarrollar síntomas prolongados de COVID cuando se evalúa en sobrevivientes de COVID-19 previamente hospitalizados |
| Queiroz et al.31 | 2023 | Descriptivo | 385 casos (208 COVID agudo y 117 post-COVID) | No encuentran asociación entre SNP reductores de MBL y long COVID |
| Udomsinprasert et al.32 | 2023 | Estudio de genes candidatos | 260 pacientes de diferente gravedad | LZTFL1 rs10490770 alelo C, LZTFL1 rs11385942 alelo dupA, NADSYN1 rs12785878 genotipo TT, PLXNA4 rs1 424597 genotipo AA, LZTFL1 rs17713054 alelo A, IL10 rs1800896 genotipo TC y alelo C, ACE2 rs2285666 alelo T y PEDS1 rs6020298 genotipo GG y alelo G se asociaron significativamente con un aumento riesgo de desarrollar long COVID El genotipo rs8178562 GG de L10RB se relacionó significativamente con un riesgo reducido de long COVID |
| Lammi et al.33 | 2023 | Caso-control | 6.450 casos de COVID prolongada y 1.093.995 controles de población de 24 estudios en 16 países | Las variantes genéticas en el locus FOXP4 están asociadas con la COVID prolongadoEl alelo C en rs9367106 se asoció con un mayor riesgo de COVID prolongado |
ACE2: enzima convertidora de la angiotensina 2; MBL: lecitina ligada a manosa; ND: no disponible; SNP: polimorfismo de un solo nucleótido; TMPRSS2: serina proteasa celular de transmembrana tipo 2.
Fernández et al.21 evaluaron cuatro SNP relacionados con el mecanismo de entrada de COVD en la célula (ACE2: rs2285666, rs2074192 y TMPRSS2: rs12329760, rs2070788) y comprobaron que, aunque se encontraban asociados a la gravedad de la enfermedad, no predisponían a desarrollar síntomas prolongados. Por otra parte, Luo et al.22 realizaron un análisis de expresión de ACE2 en tejidos de pacientes chinos y europeos. Demostraron que el alelo G en rs2106809 de ACE2 podría aumentar la actividad transcripcional de la enzima y estaría implicado en la presentación de long COVID. En un estudio posterior Fernández et al.23 informaron, de acuerdo con sus anteriores resultados, que el polimorfismo ACE1 rs1799752 no predisponía al desarrollo de long COVID.
Kousathanas et al.24 compararon los genomas de 7.491 pacientes críticos con los de 48.400 controles registrados dentro del estudio GenOMICC (https://genomicc.org). Identificaron 16 nuevas asociaciones de genes en la señalización de IFN (IL10RB y PLSCR1) y en la diferenciación de leucocitos (BCL11A), hechos que aportan evidencias sobre la señalización reducida de IFN y en la susceptibilidad a la COVID-19 grave, aunque no ofrecieron pruebas en su implicación en el long COVID. Posteriormente Pairo et al.25 llevaron a cabo un metaanálisis incluyendo pacientes del estudio GenOMICC y otras bases de datos. Encontraron 49 variantes genéticas relacionadas con COVID grave.
Angulo et al.26 en un estudio de caso/control realizado sobre 145 pacientes en Colombia evaluaron la asociación de factores no genéticos y tres polimorfismos (ACE rs4646994, ACE2 rs2285666 y LZTFL1 rs11385942) con la gravedad de la COVID y los síntomas a largo plazo. Encontraron diferencias estadísticamente significativas entre edad, sexo masculino y comorbilidades como hipertensión arterial y diabetes mellitus tipo 2. El SNP que se mostró como un importante factor de riesgo para la hospitalización fue LZTFL1 rs11385942. Sin embargo, no encontraron asociación alguna entre los polimorfismos examinados y los síntomas persistentes a largo plazo.
Niemi et al.27 llevaron a cabo un metaanálisis de 46 estudios en 19 países. Informaron de 13 loci asociados con la infección por SARS-CoV-2 y nueve de ellos estrechamente relacionados con el riesgo de presentar una COVID-19 grave, progresión de la enfermedad y peores resultados. El Severe Covid-19 GWAS Group28 había determinado que el locus 9p34.2 ligado a ABO y cinco genes ubicados en el locus 3p21.31 (SLC6A20, LZTFLI, FYCO1, CXCR6 y XCR1) se encontraban fuertemente asociados con la gravedad. Zeberg et al.29 demostraron que se debía a la existencia de un fragmento, de aproximadamente 50 kb, heredado del neandertal. Este fragmento se encontraba presente en 50% de la población del sur de Asia y en 16% de los europeos. Posteriormente30, hallaron que, con el mismo origen, un haplotipo del cromosoma 12 (chr12: 113,350,796 a 113,425,679) tenía un efecto protector.
Recientemente Queiroz et al.31 compararon, en pacientes con COVID agudo y con long COVID, la asociación de polimorfismos del gen MBL2 con los niveles plasmáticos de lecitina ligada a manosa (MBL) y citocinas inflamatorias. Si bien encontraron relación entre algunos polimorfismos de MBL2, el aumento de citocinas inflamatorias y la gravedad de COVID, no hallaron relación con long COVID; y aunque algunos de los síntomas de la enfermedad prolongada pueden estar vinculados con la respuesta inflamatoria, no parece que las alteraciones de MBL contribuyan al desarrollo de los síntomas en esa fase del padecimiento.
Estudios relacionados con riesgo de progresión a long COVIDUdomsinprasert et al.32 estudiaron la posible asociación de SNP en 37 genes candidatos implicados en la respuesta inmune e inflamatoria y encontraron que siete de ellos (LZTFL1 rs10490770 C, LZTFL1 rs11385942 dupA, LZTFL1 rs17713054 A, NADSYN1 rs12785878 TT, PLXNA4 rs1424597 AA, IL10 rs1800896 TC, ACE2 rs2285666 T y PEDS1 rs6020298 GG) se vinculaban significativamente con el aumento de riesgo para desarrollar COVID prolongado, mientras que IL10RB rs8178562 GG se relacionaba con un riesgo reducido para la progresión a long COVID.
En un estudio publicado como preimpresión en julio de 2023 por Lammi et al.33, realizado sobre 6.450 personas con long COVID en 16 países, encontraron una asociación significativa para este en el locus FOXP4, que se había relacionado previamente con la COVID grave34, la función pulmonar y diferentes tipos de tumores.
DiscusiónEl estudio de la participación de la genética humana en las enfermedades infecciosas supone un verdadero reto. Por una parte, tanto la exposición al agente infeccioso, como el manejo de esta son variables y heterogéneos, tanto entre diferentes poblaciones como dentro de la misma. Por otra parte, los resultados son difíciles de extrapolar a la población general. Las principales fuentes de información en los estudios sobre COVID son ensayos clínicos (generalmente de sujetos hospitalizados y graves), de cohortes de pacientes incluidos en biobancos (no siempre representativos de la población general), de estudios de variantes raras (que suelen presentar pocos casos, aunque fenotipos graves), de genes candidatos (que limitan el número de genes en sus análisis), y de estudios de asociación de genoma completo o GWAS (que suelen detectar variantes frecuentes, aunque de escasa repercusión en el fenotipo)35–39.
En la COVID-19 la mayor parte de los estudios se han basado fundamentalmente en identificar las variantes genómicas asociadas con la susceptibilidad a la infección (locus ABO, ACE2 y 3p21.31), con la gravedad de la enfermedad (3p21.31, genes moduladores de la respuesta inmune: TYK2, IFNAR2 y genes OAS), con la morbilidad previa y síntomas relacionados con el padecimiento (tabla 3).
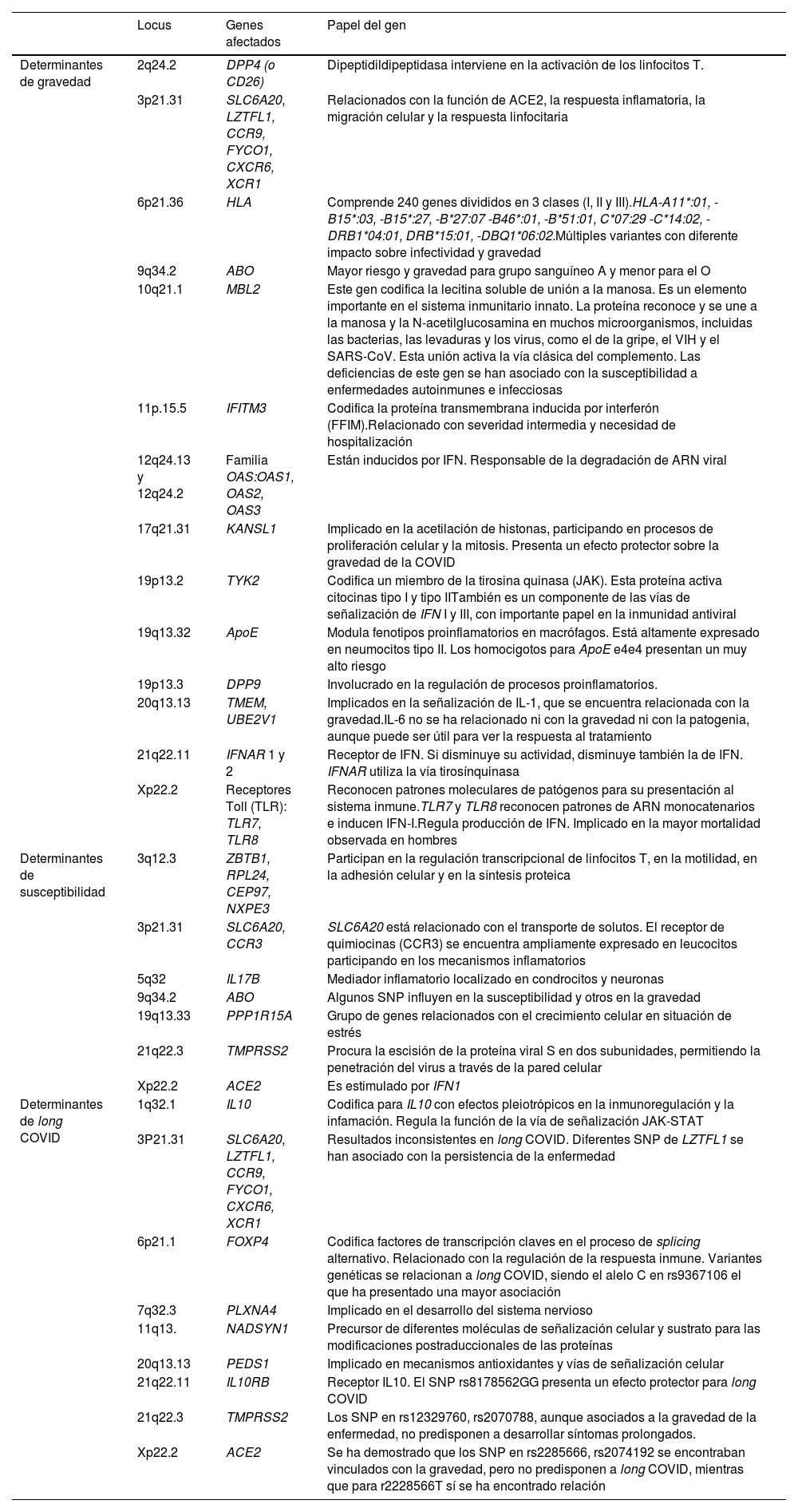
Principales loci relacionados con la susceptibilidad, gravedad y progresión de COVID-19. Algunos loci intervienen, según el polimorfismo que presenten, en varios efectos; aquí solo se recogen los más relevantes
| Locus | Genes afectados | Papel del gen | |
|---|---|---|---|
| Determinantes de gravedad | 2q24.2 | DPP4 (o CD26) | Dipeptidildipeptidasa interviene en la activación de los linfocitos T. |
| 3p21.31 | SLC6A20, LZTFL1, CCR9, FYCO1, CXCR6, XCR1 | Relacionados con la función de ACE2, la respuesta inflamatoria, la migración celular y la respuesta linfocitaria | |
| 6p21.36 | HLA | Comprende 240 genes divididos en 3 clases (I, II y III).HLA-A11*:01, -B15*:03, -B15*:27, -B*27:07 -B46*:01, -B*51:01, C*07:29 -C*14:02, -DRB1*04:01, DRB*15:01, -DBQ1*06:02.Múltiples variantes con diferente impacto sobre infectividad y gravedad | |
| 9q34.2 | ABO | Mayor riesgo y gravedad para grupo sanguíneo A y menor para el O | |
| 10q21.1 | MBL2 | Este gen codifica la lecitina soluble de unión a la manosa. Es un elemento importante en el sistema inmunitario innato. La proteína reconoce y se une a la manosa y la N-acetilglucosamina en muchos microorganismos, incluidas las bacterias, las levaduras y los virus, como el de la gripe, el VIH y el SARS-CoV. Esta unión activa la vía clásica del complemento. Las deficiencias de este gen se han asociado con la susceptibilidad a enfermedades autoinmunes e infecciosas | |
| 11p.15.5 | IFITM3 | Codifica la proteína transmembrana inducida por interferón (FFIM).Relacionado con severidad intermedia y necesidad de hospitalización | |
| 12q24.13 y 12q24.2 | Familia OAS:OAS1, OAS2, OAS3 | Están inducidos por IFN. Responsable de la degradación de ARN viral | |
| 17q21.31 | KANSL1 | Implicado en la acetilación de histonas, participando en procesos de proliferación celular y la mitosis. Presenta un efecto protector sobre la gravedad de la COVID | |
| 19p13.2 | TYK2 | Codifica un miembro de la tirosina quinasa (JAK). Esta proteína activa citocinas tipo I y tipo IITambién es un componente de las vías de señalización de IFN I y III, con importante papel en la inmunidad antiviral | |
| 19q13.32 | ApoE | Modula fenotipos proinflamatorios en macrófagos. Está altamente expresado en neumocitos tipo II. Los homocigotos para ApoE e4e4 presentan un muy alto riesgo | |
| 19p13.3 | DPP9 | Involucrado en la regulación de procesos proinflamatorios. | |
| 20q13.13 | TMEM, UBE2V1 | Implicados en la señalización de IL-1, que se encuentra relacionada con la gravedad.IL-6 no se ha relacionado ni con la gravedad ni con la patogenia, aunque puede ser útil para ver la respuesta al tratamiento | |
| 21q22.11 | IFNAR 1 y 2 | Receptor de IFN. Si disminuye su actividad, disminuye también la de IFN. IFNAR utiliza la vía tirosínquinasa | |
| Xp22.2 | Receptores Toll (TLR): TLR7, TLR8 | Reconocen patrones moleculares de patógenos para su presentación al sistema inmune.TLR7 y TLR8 reconocen patrones de ARN monocatenarios e inducen IFN-I.Regula producción de IFN. Implicado en la mayor mortalidad observada en hombres | |
| Determinantes de susceptibilidad | 3q12.3 | ZBTB1, RPL24, CEP97, NXPE3 | Participan en la regulación transcripcional de linfocitos T, en la motilidad, en la adhesión celular y en la síntesis proteica |
| 3p21.31 | SLC6A20, CCR3 | SLC6A20 está relacionado con el transporte de solutos. El receptor de quimiocinas (CCR3) se encuentra ampliamente expresado en leucocitos participando en los mecanismos inflamatorios | |
| 5q32 | IL17B | Mediador inflamatorio localizado en condrocitos y neuronas | |
| 9q34.2 | ABO | Algunos SNP influyen en la susceptibilidad y otros en la gravedad | |
| 19q13.33 | PPP1R15A | Grupo de genes relacionados con el crecimiento celular en situación de estrés | |
| 21q22.3 | TMPRSS2 | Procura la escisión de la proteína viral S en dos subunidades, permitiendo la penetración del virus a través de la pared celular | |
| Xp22.2 | ACE2 | Es estimulado por IFN1 | |
| Determinantes de long COVID | 1q32.1 | IL10 | Codifica para IL10 con efectos pleiotrópicos en la inmunoregulación y la infamación. Regula la función de la vía de señalización JAK-STAT |
| 3P21.31 | SLC6A20, LZTFL1, CCR9, FYCO1, CXCR6, XCR1 | Resultados inconsistentes en long COVID. Diferentes SNP de LZTFL1 se han asociado con la persistencia de la enfermedad | |
| 6p21.1 | FOXP4 | Codifica factores de transcripción claves en el proceso de splicing alternativo. Relacionado con la regulación de la respuesta inmune. Variantes genéticas se relacionan a long COVID, siendo el alelo C en rs9367106 el que ha presentado una mayor asociación | |
| 7q32.3 | PLXNA4 | Implicado en el desarrollo del sistema nervioso | |
| 11q13. | NADSYN1 | Precursor de diferentes moléculas de señalización celular y sustrato para las modificaciones postraduccionales de las proteínas | |
| 20q13.13 | PEDS1 | Implicado en mecanismos antioxidantes y vías de señalización celular | |
| 21q22.11 | IL10RB | Receptor IL10. El SNP rs8178562GG presenta un efecto protector para long COVID | |
| 21q22.3 | TMPRSS2 | Los SNP en rs12329760, rs2070788, aunque asociados a la gravedad de la enfermedad, no predisponen a desarrollar síntomas prolongados. | |
| Xp22.2 | ACE2 | Se ha demostrado que los SNP en rs2285666, rs2074192 se encontraban vinculados con la gravedad, pero no predisponen a long COVID, mientras que para r2228566T sí se ha encontrado relación |
ACE2: enzima convertidora de la angiotensina 2; ARN: ácido ribonucleico; IFN: interferón; IL: interleucina; JAK-STAT: quinasa de Janus/transductor de señal y activador de transcripción; SNP: polimorfismo de un solo nucleótido; VIH: virus de la inmunodeficiencia humana.
El mayor locus de susceptibilidad se encuentra en 9p34.2 ligado al sistema ABO. Existen evidencias sólidas que avalan que el grupo sanguíneo O es un factor protector mientras que el grupo A parece ser un factor de riesgo40,41.
El papel que desempeñan la ACE2 y los sistemas de proteasas relacionados (TMPRSS2, furinas, etc.) en la susceptibilidad para la infección por SARS-CoV-2 y en su gravedad, ha sido ampliamente estudiado. La angiotensina II tiene un potente efecto vasoconstrictor e inflamatorio. La ACE2 posibilita el paso de angiotensina II a angiotensina 1-7, limitando su efecto. En la infección por SARS-CoV-2 se produce una disminución de la concentración de ACE2 y angiotensina 1-742. El genotipo ACE puede afectar a los resultados clínicos de COVID-19 43. Polimorfismos de inserción (I)/deleción (D) han sido registrados en diversas patologías y su participación en la progresión de la enfermedad ha sido muy discutida44–46. Parece que los polimorfismos D, especialmente homocigotos (D/D) se relacionan con una mayor mortalidad por COVID-19, mientras que los genotipos homocigotos de inserción (I/I) se vinculan con una menor susceptibilidad y mortalidad. Estos polimorfismos presentan diferencias étnicas en su distribución y pueden justificar parte de las diferencias en la mortalidad encontradas en distintas áreas geográficas del planeta47,48.
El gen que codifica ACE2 se localiza en el cromosoma X (Xp22) y en su expresión intervienen diferentes factores genéticos y epigenéticos (edad, genero, índice de masa corporal [IMC], etc.). Así, en la explicación de las diferencias encontradas en niños y en varones, entrarían la expresión edad-dependiente de ACE2 y la participación del receptor de andrógeno en la regulación de la transcripción de ACE2 y de TMPRSS249–51.
Hay descritas decenas de polimorfismos en ACE2, con diferente distribución geográfica, étnica y de distinto impacto, tanto con resultados protectores como deletéreos. Aunque, particularmente estas variantes puedan explicar diferencias individuales, globalmente no ha sido posible establecer asociaciones significativas entre las variantes y la gravedad de la enfermedad que permitan su incorporación para la elaboración de escalas de riesgo poligénico52.
TMPRSS2 se codifica en el cromosoma 21 y es sensible a los andrógenos. Esto podría explicar la mayor gravedad y mortalidad observada en los varones53. Por otra parte, se han encontrado decenas de polimorfismos que afectan no solo a la estructura proteica sino también a un amplio rango de micro-ARA (miRNA), implicados tanto en la progresión y gravedad como en los efectos protectores54.
Aunque parece que los niveles altos de furina en plasma pueden tener que ver con los malos resultados en pacientes diabéticos55 y que se han encontrado diferentes variantes genéticas relacionadas con COVID, no se ha podido demostrar que estas tengan gran trascendencia en la enfermedad52.
Modulación de la respuesta inmune y gravedadEl locus implicado más fuertemente con la gravedad ha sido el ubicado en la región 3p21.31 y, aunque la gravedad de la enfermedad se ha relacionado con el riesgo de desarrollar long COVID, no se han encontrado evidencias directas de que los genes contenidos en este locus aumenten el peligro de ello28. En este locus se observan diferentes genes vinculados con la entrada del virus en la célula, con la señalización de linfocitos y macrófagos y con la actividad de diferentes quimiocinas.
También se han implicado en la gravedad de la infección diferentes genes relacionados con la modulación de la respuesta inmune. TYK2 parece aportar una mayor gravedad a la infección, aunque curiosamente ofrece un efecto protector para las enfermedades autoinmunes comunes56. IFNAR2, próximo a IL1ORB e IFNAR1, participan en la regulación de la respuesta de IFN I e interleucina (IL) 157–59. Los genes OAS codifican para proteínas implicadas en la eliminación viral. Se ha encontrado que niveles altos de la proteína OAS1 protegen contra la infección grave60. Igualmente, miembros de la familia OAS están implicados en el fallo cardiaco relacionado con COVID-1961.
Se han realizado diversos estudios que tratan de relacionar las manifestaciones fenotípicas de COVID con morbilidades preexistentes. Se han identificado cuatro loci asociados con las manifestaciones graves de la enfermedad y con patología pulmonar (enfermedad pulmonar intersticial, fibrosis y cáncer de pulmón). Los genes más importantes contenidos en estos loci son DPP9, FOXP4, SFTPD y MUC5B128,62. Igualmente se han encontrado genes comunes (C5AR1, MMP9, CYBB, FPR2, CSF1R, TLR2 y TLR) implicados en enfermedades cardiovasculares y COVID-1963.
Gravedad y progresión a long COVIDDiferentes estudios han aportado evidencias de que la COVID grave puede ser un factor de riesgo de su variante prolongada64–67 y se ha tratado de dilucidar si los genes implicados en la susceptibilidad o en la gravedad de la enfermedad son también un factor de riesgo para el desarrollo de long COVID. Aunque se han encontrado asociaciones, con diversa consistencia, entre algunos genes involucrados en la respuesta inflamatoria e inmune32, la relación más importante ha podido ser demostrada con el gen FOXP4. Este se ubica en el cromosoma 6 (6p21.1), en la misma área que FOXP4-AS1, LINC01276, MIR4641. FOXP4 pertenece a una familia de genes (FOX) que codifican factores de transcripción que desempeñan un importante papel en la regulación de genes específicos mediante la producción de variantes de transcripción en el splicing alternativo. Este gen se encuentra ampliamente representado en casi todos los tejidos, especialmente en cérvix, tiroides, estómago, testículos, linfocitos, pulmón y vasos sanguíneos. El alelo C de FOXP4 en rs9367106 se ha asociado con el mayor riesgo de COVID prolongado. Se encuentra irregularmente distribuido en las poblaciones, desde 1,6% en europeos hasta 36% en asiáticos orientales33. Los genes relacionados con FOXP4, corresponden a genes no codificantes. Así FOXP4-AS1 y LINC01276 codifican para un ARN no codificante de cadena larga (LnRNA), mientras que MIR4641 lo hace para miRNA. La participación de ARN no codificantes (ncARN) está ampliamente documentada en la regulación de la respuesta inflamatoria, la disfunción inmune y la trombosis inducida por SARS-CoV-268–72, así como en patologías relacionadas respiratorias, neurológicas o cardiovasculares73–75.
ConclusiónA pesar de la gran cantidad de información acumulada en los cortos años de existencia de la enfermedad, no se ha podido establecer, salvo en contadas excepciones, relaciones directas de causalidad entre las alteraciones genéticas y los desenlaces del padecimiento. Los estudios de asociación han permitido identificar numerosas variantes, aunque, por un lado, de poco impacto sobre la enfermedad, y por otro, influidas por variables demográficas, étnicas y clínicas. Cada vez parece más consistente considerar en la etiopatogenia del long COVID la contribución poligénica, la implicación de genes pleiotrópicos34, así como la participación del microbioma76–78 y de elementos epigenéticos determinantes79–81.
FinanciaciónEste trabajo no ha recibido ningún tipo de financiación.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.






![A) Representación esquemática del genoma de SARS-CoV-2 y de los procesos de replicación, transcripción y traducción. B) Representación esquemática de la respuesta inmune e inflamatoria. En la entrada del virus en la célula interviene la enzima convertidora de la angiotensina 2 (ACE2) y los sistemas de proteasas. El ARN viral, junto con algunas partículas virales son incluidos en el endosoma, en cuya superficie se muestran receptores Toll-like (TLR) que van a activar, entre otros, factores reguladores de interferón (IRF), factor nuclear Kappa B (NF-KB) y componentes del inflamosoma (NLRP3). Todo ello activa los factores transcripcionales nucleares que conducen a la liberación de quimiocinas y citosinas inflamatorias (interferón [IFN], interleucina [IL], factor de necrosis tumoral alfa [TNF∝], etc.), que además de actuar sobre diferentes células del sistema inmune, van a generar bucles de retroalimentación positiva a través de receptores de interferón (IFNAR) de interleucinas (ILR) y TLR, ente otros y en los que participan las vías tirosínquinasa (TYK2, quinasa de Janus/transductor de señal y activador de transcripción [JAK-STAT]). La acción del virus y de las citosinas inflamatorias sobre las células defensivas generan interacciones entre ellas que conducen a la amplificación del proceso inflamatorio y a la participación multiorgánica. ARN: ácido ribonucleico; Ig: inmunoglobulina. A) Representación esquemática del genoma de SARS-CoV-2 y de los procesos de replicación, transcripción y traducción. B) Representación esquemática de la respuesta inmune e inflamatoria. En la entrada del virus en la célula interviene la enzima convertidora de la angiotensina 2 (ACE2) y los sistemas de proteasas. El ARN viral, junto con algunas partículas virales son incluidos en el endosoma, en cuya superficie se muestran receptores Toll-like (TLR) que van a activar, entre otros, factores reguladores de interferón (IRF), factor nuclear Kappa B (NF-KB) y componentes del inflamosoma (NLRP3). Todo ello activa los factores transcripcionales nucleares que conducen a la liberación de quimiocinas y citosinas inflamatorias (interferón [IFN], interleucina [IL], factor de necrosis tumoral alfa [TNF∝], etc.), que además de actuar sobre diferentes células del sistema inmune, van a generar bucles de retroalimentación positiva a través de receptores de interferón (IFNAR) de interleucinas (ILR) y TLR, ente otros y en los que participan las vías tirosínquinasa (TYK2, quinasa de Janus/transductor de señal y activador de transcripción [JAK-STAT]). La acción del virus y de las citosinas inflamatorias sobre las células defensivas generan interacciones entre ellas que conducen a la amplificación del proceso inflamatorio y a la participación multiorgánica. ARN: ácido ribonucleico; Ig: inmunoglobulina.](https://static.elsevier.es/multimedia/11383593/0000005000000002/v2_202403030917/S1138359323002678/v2_202403030917/es/main.assets/thumbnail/gr1.jpeg?xkr=ue/ImdikoIMrsJoerZ+w96p5LBcBpyJTqfwgorxm+Ow=)

