Desde la última mitad del siglo xx se ha producido un espectacular progreso en el conocimiento sobre el material genético humano: se han caracterizado los cromosomas, se ha identificado la secuencia completa del genoma y se ha relacionado un gran número de alteraciones de la estructura y secuencia del ADN con todo tipo de enfermedades, tanto hereditarias como no hereditarias (especialmente asociadas a cáncer). Este desarrollo ha venido propiciado, y acompañado, por una gran cantidad de técnicas para su estudio, muchas de las cuales se han trasladado a la investigación y la caracterización de la carga genética de los seres humanos, y a su aplicación en la práctica clínica para el diagnóstico de ciertas patologías. Ello ha generado que, además de los estudios moleculares al nivel de identificación de la mutación de un gen, la citogenética convencional se haya ampliado a la detección de alteraciones extremadamente pequeñas de la estructura del cromosoma, dando lugar a una nueva área de diagnóstico denominada citogenética molecular. Esta incluye diversas técnicas que detectan alteraciones pequeñas en orden decreciente, desde las micro-deleciones y micro-duplicaciones detectables por sondas fluorescentes (FISH), hasta los arrays genómicos y los arrays basados en hibridación genómica comparada (CGH) que detectan cambios aun más pequeños. Sin embargo, debido a su alta resolución, los arrays son capaces de identificar variaciones en el ADN cuyo significado, en muchos casos, es aun incierto. Esto implica que incluso algunas de las técnicas que más se están utilizando en la actualidad, como la CGH array, precisen de una cuidadosa evaluación antes de ofrecer un diagnóstico.
Spectacular progress has been made in the knowledge of human genetic material since the middle of the last century: the chromosomes have been characterised; the complete sequence of the genome has been identified. Also, all types of diseases hereditary and non-hereditary diseases (especially cancers) have been associated to changes in DNA structure and sequence. These developments have been accompanied by a great number of techniques, which have led to the investigation and characterisation of the human genetic load, and its application in clinical practice for the diagnosis of certain diseases. This has meant that, besides studies at molecular level for identifying a gene mutation, conventional cytology has extended to the detection of extremely small changes in the structure of the chromosome, giving rise to a new area of diagnosis called molecular cytogenetics. This includes various techniques that detect small changes, in decreasing order, from microdeletions and microduplications detectable by fluorescent probes (FISH), to genomic arrays and arrays based on compared genomic (CGH) which detect even smaller changes. However, due to their high resolution, arrays are capable of identifying variations in DNA, which in many cases its importance is still uncertain. This implies that even some of the most common techniques currently used, such as CGH, require a careful evaluation before offering a diagnosis.
A mediados de los años 50 Joe Hin Tjio (que trabajaba en la estación Experimental de Aula Dei, en Zaragoza, España) y Albert Levan (en el Cancer Chromosome Laboratory, Institute of Genetic de Lund, Suecia)1 establecen el número y la estructura de los cromosomas humanos. Desde ese momento, las técnicas para la caracterización del cariotipo humano inician un continuo desarrollo en cuanto a su poder de resolución y eficacia. En 1953 Watson y Crick habían descubierto la estructura de la molécula del ADN2, dando el primer paso en el conocimiento y el posterior estudio del genoma humano. Un conocimiento que, en los últimos años de siglo pasado y los primeros de este, ha llegado a descifrar la secuencia del ADN humano y al conocimiento de su estructura3. Estos hallazgos han dado lugar a la identificación de una gran cantidad de variaciones estructurales y funcionales del genoma y a la investigación de su potencial función4.
En consecuencia, se vienen desarrollando nuevas técnicas para esos análisis que se han empezado a trasladar al diagnóstico de alteraciones estructurales de los cromosomas humanos (que no se detectan con citogenética de alta resolución) y a su correlación con ciertas patologías, incluyendo las del desarrollo embrionario y fetal.
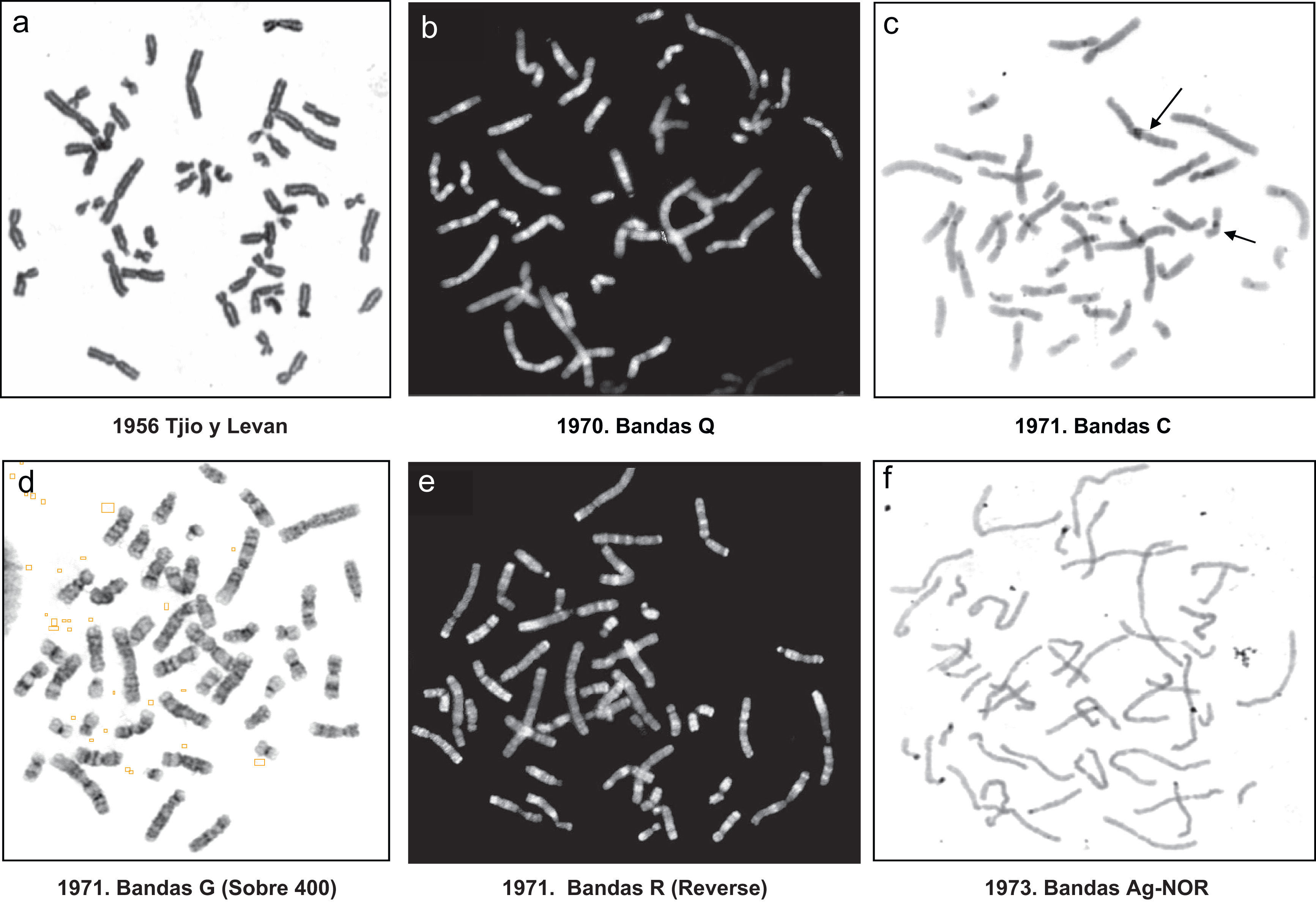
¿Cuál ha sido la evolución de las técnicas citogenéticas?Citogenética convencionalHasta el año 1970, los cromosomas se teñían de forma uniforme, por lo que las parejas de los diferentes cromosomas se ordenaban por su tamaño y parecido morfológico (fig. 1a). Más adelante, en al año 1971, se desarrollaron técnicas que teñían los cromosomas en bandas claras y oscuras (o fluorescentes y no fluorescentes)5,6. Esas bandas seguían distintos patrones que eran iguales solo en cada pareja de cromosomas paterno y materno. En conjunto se establecieron cinco tipos de análisis de los patrones de bandas citogenéticas:
- 1.
Bandas Q7,8 (fig. 1b), que aparecen como bandas fluorescentes y brillantes en contraste con otras no fluorescentes. Estas bandas, aunque fáciles de obtener, presentan problemas metodológicos y de análisis (se ven solo con luz ultravioleta, la fluorescencia dura poco tiempo…).
- 2.
Bandas C5 (fig. 1c), que tiñen específicamente la heterocromatina de las regiones centroméricas.
- 3.
Bandas G9 (fig. 1d), llamadas así porque se tiñen con giemsa en forma de bandas transversales claras y obscuras.
- 4.
Bandas R5 (fig. 1e), que en realidad constituyen la representación del patrón reverso de las bandas G. Así las G oscuras, son claras en la tinción R y viceversa.
- 5.
Bandas Ag-NOR10 (fig. 1f), que muestran las regiones organizadoras nucleolares de los cromosomas acrocéntricos (pares 13, 14, 15, 21 y 22).
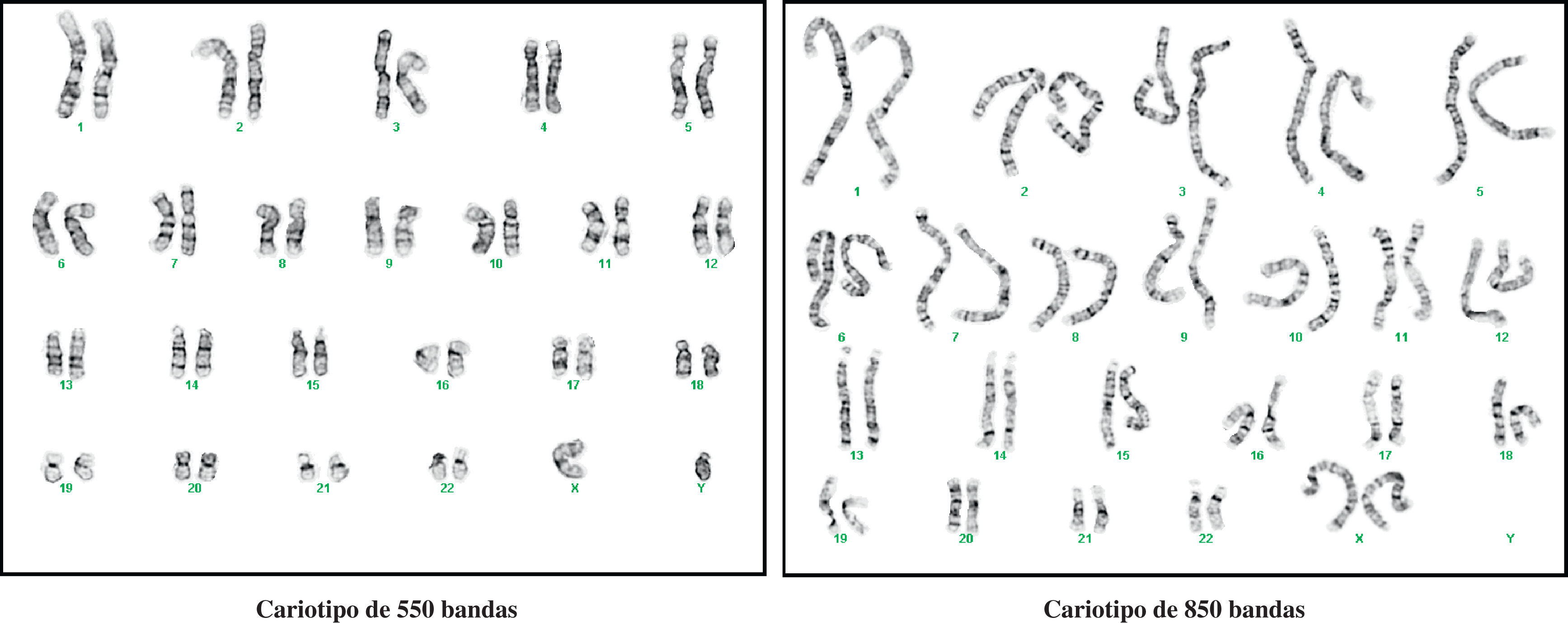
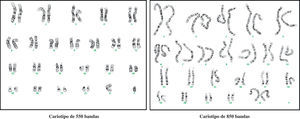
En general, la técnica más utilizada para el diagnóstico citogenético es la tinción de bandas G, quedando las C y las Ag-NOR, reservadas para el diagnóstico y/o confirmación de ciertos tipos de alteraciones cromosómicas. No obstante, a lo largo del tiempo, la técnica de bandas G ha ido modificándose, pasando a utilizar cromosomas menos metafásicos, incluso prometafásicos. Estos últimos, al estar menos condensados (durante la profase los cromosomas se condensan y acortan hasta su máximo nivel para formar la placa metafásica de la mitosis) son más largos, de tal manera que la mayoría de las bandas observadas en el cromosoma metafásico se separan en varias bandas claras y oscuras en el prometafásico10,11. En la figura 1d, se muestra un cariotipo a un nivel de resolución de bandas convencional y que se corresponde con la identificación de alrededor de 400 bandas en el conjunto de los 46 cromosomas. En la figura 2, se muestran dos cariotipos con la resolución incrementada, uno de 550 bandas y el otro de 850 bandas o de alta resolución.
Hibridación in situ con fluorescencia (FISH, de sus siglas en inglés)La FISH es una técnica basada en la estructura de doble cadena del ADN, concretamente en el hecho de que las bases nitrogenadas del ADN son complementarias: la adenina se une preferentemente con la timina y la citosina con la guanina (ver capítulo 3 de este curso). La técnica de FISH consiste en diseñar un fragmento de ADN, denominado sonda, que es complementario a una región candidata en el genoma objeto de estudio. Esta sonda se marca con una sustancia fluorescente (un fluorocromo) y se añade a la preparación de células que se quieren analizar. Mediante calor el ADN se desnaturaliza, separándose las dos cadenas, y la secuencia génica en el ADN en estudio que es complementaria a la de la sonda fluorescente se une (hibrida) con esta y queda marcada con una señal fluorescente que puede visualizarse en un microscopio de fluorescencia. La resolución de las técnicas de citogenética molecular se define como el tamaño mínimo que debe tener una alteración para ser detectada y se mide en nucleótidos o pares de bases (tabla 1).
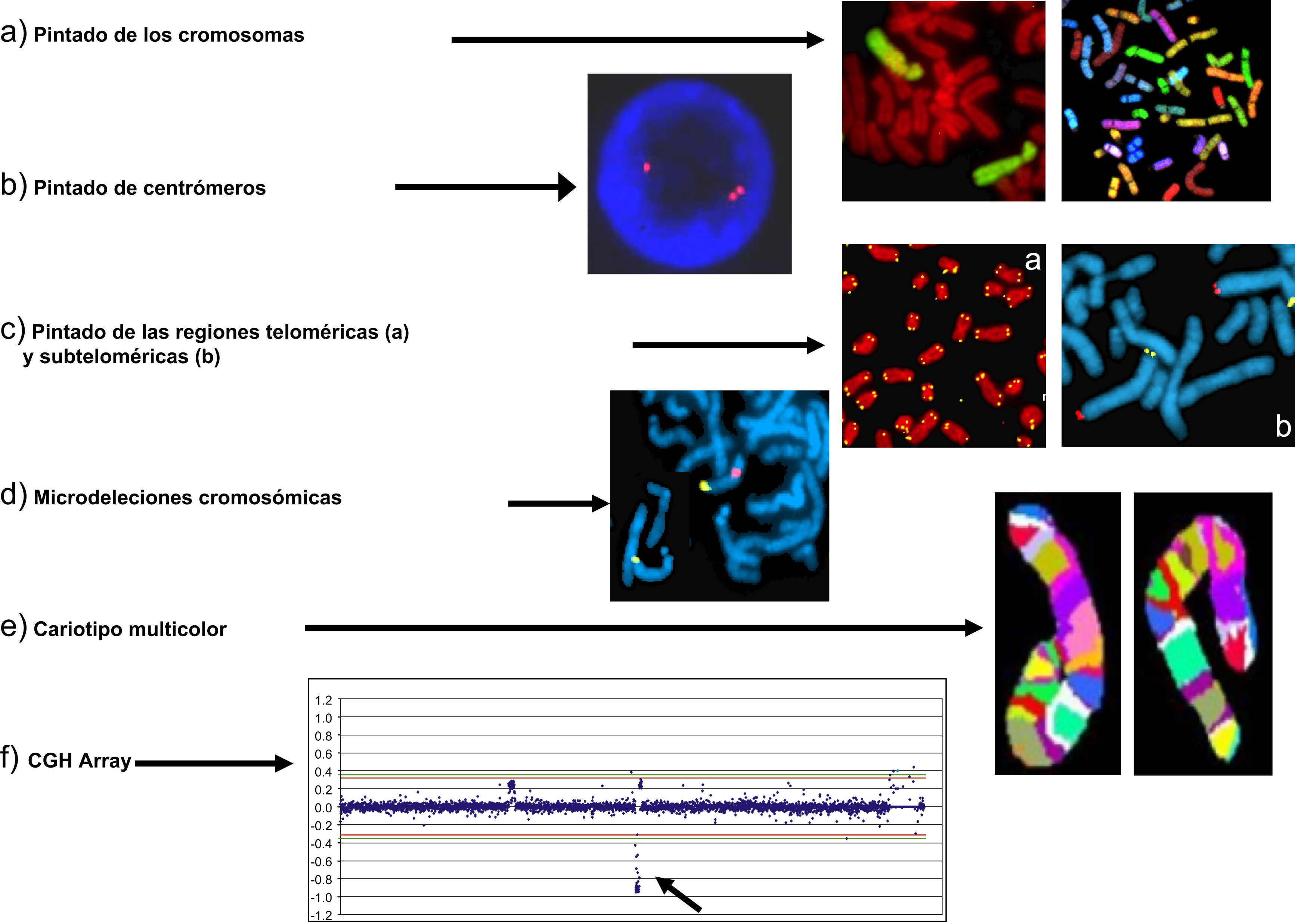
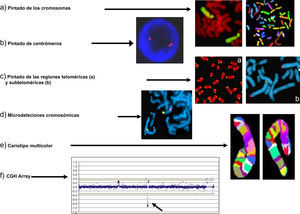
Las posibilidades que ofrece la metodología de FISH han dado lugar al desarrollo de técnicas con diferente resolución y objetivos diagnósticos. Entre ellas, podemos destacar (fig. 3):
- a)
Pintado de cromosomas completos (fig. 3a).
- b)
Localización de centrómeros (fig. 3b).
- c)
Marcado de regiones teloméricas (fig. 3c-a) y de regiones subteloméricas (fig. 3c-b)
- d)
Identificación de regiones de ADN específicas de pequeño tamaño (microdeleciones),(fig. 3d).
- e)
Cariotipo multicolor o espectral (multi-FISH o SKY-FISH), que tiñe cada cromosoma con una combinación de fluorocromos diferente (fig. 3e). En la actualidad, este tipo de tinción solo es aplicable en investigación.
Utilizando como punto de partida los fundamentos de la FISH, a principio de los años 90 se desarrolló una nueva tecnología denominada hibridación genómica comparada (CGH de sus siglas en inglés)12. La CGH se basa en marcar el ADN del propositus con un fluorocromo verde y el de un individuo normal o de referencia con un fluorocromo rojo y mezclarlos, realizando una hibridación in situ sobre cromosomas normales. En esta situación, ambos ADN (propositus y referencia) compiten por hibridar en los mismos lugares cromosómicos. Si el ADN del propositus no tiene alteraciones, el resultado va a mostrar los cromosomas con una tinción fluorescente amarilla y homogénea resultante de la mezcla, porque la cantidad de ADN rojo y verde es igual (esto es en proporción 1:1). Sin embargo, si el ADN del propositus tiene una deleción (pérdida de ADN), o una ganancia (duplicación) en una región cromosómica determinada, el patrón de fluorescencia mostrará en esa zona incremento de la señal roja en caso de deleción (ya que habrá más cantidad de ADN normal), o de la señal verde en caso de ganancia (porque habrá más ADN en el propositus). Estos resultados se representan en una gráfica similar a la de la figura 3f.
Una limitación de la CGH es que no detecta translocaciones balanceadas, en las cuales únicamente existe un cambio en la localización de las secuencias afectadas por la translocación, pero la cantidad de ADN no varía.
La alta especificidad y sensibilidad de las técnicas de FISH las han convertido en herramientas de análisis casi rutinario en los laboratorios de citogenética y, junto con la CGH, han sido incluidas dentro de un nuevo concepto denominado «citogenética molecular». No obstante, es importante destacar que, a diferencia del cariotipo de alta resolución, la aplicación de estas técnicas moleculares no es útil para realizar screening. Las técnicas moleculares (de cualquier tipo) precisan de mayor información clínica previa a su utilización, pero no sustituyen, ni excluyen al cariotipo de alta resolución, sino que lo complementan. Por ello, las técnicas de FISH son de gran utilidad en los casos en los que el cariotipo de alta resolución es normal, pero la clínica del niño hace sospechar que podría presentar alguna de las microdeleciones conocidas. También son útiles cuando los cultivos para realizar el cariotipo no son buenos y se obtienen pocas metafases o son de baja calidad.
Nuevos sistemas de análisis basados en array CGH y array de SNP (Single Nucleotide Polymorphisms) denominados Whole Genome ArrayLa CGH ha evolucionado a sistemas más sofisticados y resolutivos en los cuales las metafases han sido sustituidas por una superficie de sílice con pequeños fragmentos de ADN a la que se ha denominado «chip». El «chip» puede contener cientos de miles de pequeños fragmentos de ADN genómico de localización conocida (denominados sondas), que cubren la práctica totalidad del genoma. La técnica de hibridación comparada sobre chips se denomina CGH array y mantiene los mismos principios técnicos.
Un paso más en la tecnología de los «chips» ha sido la aparición de los arrays basados en single nucleotide polymorphisms (SNP), que son secuencias de ADN que presentan variantes en la base de un único nucleótido. Los SNP se encuentran bien caracterizados y se distribuyen a lo largo de todo el genoma humano, lo que permite diseñar sondas de muy pequeño tamaño y gran especificidad. A cada array diseñado con una distribución de sondas concreta se le denomina plataforma. Si el diseño de la plataforma incluye SNP localizados a lo largo de todos y cada uno de los cromosomas humanos, se denominan comercialmente whole genome array (WGA)
El WGA no utiliza ADN de referencia. Un escáner de alta resolución obtiene una imagen de la fluorescencia relativa en cada punto y un software con parámetros estadísticos concretos determina la intensidad y la posición de cada SNP hibridado. El resultado obtenido es igual al que se muestra en la figura 3f: una nube de puntos (las sondas) cuya fluorescencia relativa se representa sobre una escala de intensidad de fluorescencia, que se ve reducida en caso de pérdida o aumentada en caso de ganancia de número de copias en una localización concreta del genoma.
La resolución de los chips de ADN depende del número de sondas que cubren cada región génica concreta y a qué distancia se sitúan entre ellas (densidad). Estas sondas detectan alteraciones de muy reducido tamaño, que en los diseños más recientes llegan a ser de solo cientos de pares de bases. Como comparativa, en la CGH convencional o la citogenética de alta resolución, los tamaños de las alteraciones detectadas son del orden de varias megabases (tabla 1).
La aplicación de los arrays al diagnóstico de defectos congénitos, aunque aporta algunas luces, presenta aún muchas sombras. Las limitaciones radican, principalmente, en la gran variabilidad estructural del genoma humano, que ha mostrado una alta frecuencia de cambios en el número de copias. Desde hace poco más de cinco años, al estudiar series de muestras de ADN de la población general, se ha constatado que en sus genomas existe una gran cantidad de pérdidas, ganancias y secuencias repetitivas. La mayor parte de estos cambios poseen grandes diferencias en su estructura y frecuencia entre los individuos estudiados4 pero no han podido relacionarse con patología alguna. A estas variaciones se les denominan copy number variation (CNV) y se consideran polimorfismos. A pesar de que algunas de esas variaciones afectan a genes que se han relacionado con ciertas patologías, estas no se manifiestan en los individuos estudiados de la población general. No obstante, ya se han descrito casos de pacientes tanto con síndromes mendelianos como con susceptibilidad a diferentes enfermedades, que son consecuencia de modificaciones producidas por ciertas CNV que pueden alterar la expresión de genes, entre otros mecanismos, por lo que se denomina efecto de posición4,13,14.
Como conclusión, cuando aplicamos técnicas de array en un paciente con defectos congénitos es probable encontrar distintas CNV pero su significado, en muchos casos, aún no resulta de fácil interpretación15.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.