La aplicación de la inteligencia artificial y en particular de algoritmos de aprendizaje automático o «machine learning» (ML) constituye un desafío y al mismo tiempo una gran oportunidad en diversas disciplinas científicas, técnicas y clínicas. Las aplicaciones específicas en el estudio de la esclerosis múltiple (EM) no han sido una excepción mostrando un creciente interés en los últimos años.
ObjetivoRealizar una revisión sistemática de la aplicación de algoritmos de ML en la EM.
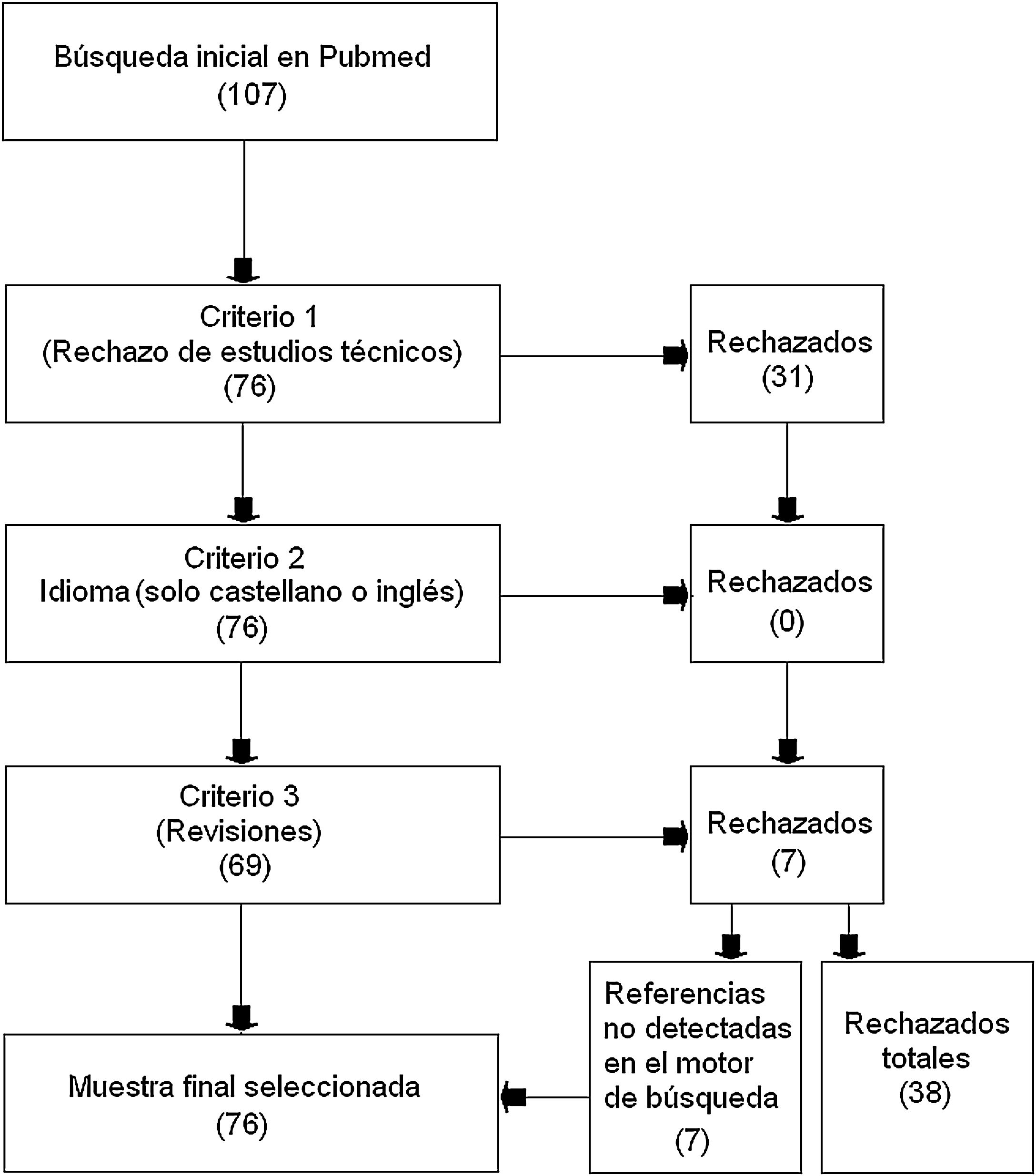
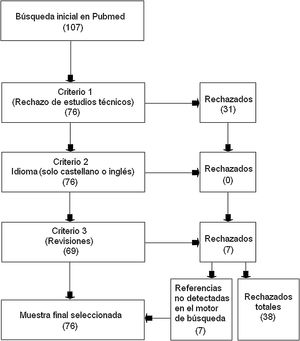
Material y métodosEmpleando el motor de búsqueda de libre acceso PubMed que accede a la base de datos MEDLINE, se seleccionaron aquellos estudios que incluyeran simultáneamente los dos siguientes conceptos de búsqueda: «machine learning» y «multiple sclerosis». Se rechazaron aquellos estudios que fueran revisiones, estuvieran en otro idioma que no fuera el castellano o el inglés, y aquellos trabajos que tuvieran un carácter técnico y no fueran aplicados para la EM. Se seleccionaron como válidos 76 artículos y fueron rechazados 38.
ConclusionesTras la revisión de los estudios seleccionados, se pudo observar que la aplicación del ML en la EM se concentró en cuatro categorías: 1) clasificación de subtipos de pacientes dentro de la enfermedad; 2) diagnóstico del paciente frente a controles sanos u otras enfermedades; 3) predicción de la evolución o de la respuesta a intervenciones terapéuticas y por último 4) otros enfoques. Los resultados hallados hasta la fecha muestran que los diferentes algoritmos de ML pueden ser un gran apoyo para el profesional sanitario tanto en la clínica como en la investigación de la EM.
The applications of artificial intelligence, and in particular automatic learning or “machine learning” (ML), constitute both a challenge and a great opportunity in numerous scientific, technical, and clinical disciplines. Specific applications in the study of multiple sclerosis (MS) have been no exception, and constitute an area of increasing interest in recent years.
ObjectiveWe present a systematic review of the application of ML algorithms in MS.
Materials and methodsWe used the PubMed search engine, which allows free access to the MEDLINE medical database, to identify studies including the keywords “machine learning” and “multiple sclerosis.” We excluded review articles, studies written in languages other than English or Spanish, and studies that were mainly technical and did not specifically apply to MS. The final selection included 76 articles, and 38 were rejected.
ConclusionsAfter the review process, we established 4 main applications of ML in MS: 1) classifying MS subtypes; 2) distinguishing patients with MS from healthy controls and individuals with other diseases; 3) predicting progression and response to therapeutic interventions; and 4) other applications. Results found to date have shown that ML algorithms may offer great support for health professionals both in clinical settings and in research into MS.
La inteligencia artificial y en particular el aprendizaje automático o «machine learning» (ML) son conceptos que están abriéndose paso en los últimos años en diversas disciplinas y la medicina no es una excepción. En la última década se ha producido un incremento significativo de procedimientos y aplicaciones del ML para el estudio de las patologías neurológicas. Esta revisión pretende analizar cuáles han sido las primeras vías de aplicación en una patología de gran impacto sanitario y social como es la esclerosis múltiple (EM)1. Antes de entrar en materia, es necesario precisar que esta revisión no pretende analizar en profundidad los métodos matemáticos que subyacen a los algoritmos de ML empleados en los diferentes estudios (véase2 para una revisión a este respecto). Sin embargo, realizaremos una pequeña introducción sobre diversos conceptos relacionados con el ML ampliamente empleados en la literatura que se analiza en esta revisión. Habitualmente es posible confundir los conceptos de inteligencia artificial y ML. ML es un campo de la inteligencia artificial que se centra en el estudio de algoritmos que aprenden de la experiencia (datos) para mejorar el rendimiento en una tarea específica. Por ejemplo, las tareas de clasificación y regresión permiten hallar patrones, en ocasiones muy complejos, del conjunto de datos de la muestra a estudio. Dentro de esta idea general, existen dos posibles alternativas dentro del ML, los procedimientos supervisados y los no supervisados. En el primer caso, se entrena al algoritmo con un conjunto de datos que han sido etiquetados por el investigador (p. ej. subtipos de pacientes en una patología (esclerosis múltiple remitente-recidivante (EMRR), esclerosis múltiple secundaria progresiva (EMSP), etc.). Este procedimiento tiene un enfoque predictivo y el algoritmo define los posibles patrones de cada etiqueta a partir de las variables o parte de las variables incluidas en el análisis. Posteriormente, y aquí está la clave del gran interés en este campo, el algoritmo permite identificar a cuál de los patrones previamente definidos (aprendidos por el algoritmo) se ajustan mejor los nuevos datos (un nuevo paciente) que se le proporcionen. En los procedimientos no supervisados, enfocados principalmente en tareas descriptivas, no se produce un etiquetado previo de los datos y el algoritmo busca similitudes entre los datos y define por sí solo, los posibles «clústeres»o grupos presentes en la muestra de datos. Existen múltiples procedimientos en el caso de los métodos supervisados que pueden ser reconocidos por el lector tales como: «neural networks», «random forest», «support vector machine» o en el caso de los métodos no supervisados: «hierarchical clustering» o «self-organizing maps»3. Una de las grandes ventajas de los algoritmos de ML es que permiten analizar conjuntamente datos cuantitativos (scores) y cualitativos (evaluaciones del profesional sanitario) de los pacientes en el proceso de aprendizaje del algoritmo. Otro aspecto clave es si el algoritmo de ML realmente está consiguiendo una eficaz clasificación de los patrones existentes entre los datos. Para ello, se emplean una serie de indicadores como son la precisión, sensibilidad o la especificidad entre otros. Sin embargo, lamentablemente no todos los estudios que se han seleccionado en este estudio muestran siempre todas estas medidas o incluso desarrollan sus propios indicadores, lo que a veces complica la comparación de resultados entre los diferentes estudios. La presente revisión pretende mostrar el panorama general de las aplicaciones del ML en el caso particular de la EM, centrada principalmente en los intereses y retos del profesional sanitario. Además, se incluirán los valores estimados de precisión de los algoritmos en su capacidad de categorización de los pacientes.
Materiales y métodosLa revisión se realizó empleando el motor de búsqueda «PubMed» de libre acceso que permite consultar la base de datos MEDLINE. La selección de estudios se realizó introduciendo como conceptos de búsqueda «machine learning» y «multiple sclerosis» unidos por el operador booleano «AND» para limitar la búsqueda a aquellos estudios que contuvieran ambos conceptos.
La búsqueda se realizó sin límite inferior en el año de publicación y con fecha de cierre del 30 de abril de 2020. Se rechazaron aquellos estudios que tuvieran un carácter técnico y sin una aportación específica a la esclerosis múltiple (criterio de exclusión 1); aquellos que estuvieran en otro idioma que no fuera el castellano o el inglés (criterio de exclusión 2); y aquellos trabajos que fueran revisiones (criterio de exclusión 3). Sin embargo, en este último caso, se rastreó entre sus referencias, para localizar estudios que trataran de la aplicación de ML en la EM y que no hubieran sido mostrados por el motor de búsqueda inicialmente.
Las características de esta investigación (criterios de selección de los estudios) establecen a priori un bajo nivel de sesgo, sin embargo algunas consideraciones se tuvieron en cuenta para minimizar esta posible amenaza. Por un lado, no se establecieron limitaciones en cuanto al tipo de revista (aspectos como la posición de cuartil o el factor de impacto) o en los años de publicación. Sí se eliminaron las revisiones como referencias elegibles (para evitar posibles duplicaciones), aunque sí se emplearon para poder rastrear referencias no halladas por el motor de búsqueda. Por último, no se han incluido aquellos trabajos enfocados a la mejora en la exploración de resonancia magnética nuclear (RMN) aunque se ha realizado un comentario específico al respecto (véase más adelante en el apartado de resultados).
ResultadosTras la búsqueda, se obtuvieron 107 estudios de los cuales: 31 fueron rechazados en atención al criterio de exclusión 1; ninguno fue retirado por el criterio de exclusión 2; y 7 fueron excluidos por el criterio de exclusión 3 (revisiones). En el caso de los estudios rechazados por el criterio 3, el rastreo posterior de sus propias referencias rindió 7 estudios no hallados en la búsqueda inicial y que por tanto fueron añadidos a la muestra seleccionada para esta revisión. El total de estudios aceptados finalmente en esta revisión fue de 76 y el número de rechazados 38 (fig. 1).
Respecto de las diversas revisiones halladas en la búsqueda, es necesario destacar que muestran ya un cierto grado de madurez del campo del ML en la disciplina de la Neurología. Sin embargo, la mayoría de estas revisiones abordan múltiples patologías neurológicas y no se centran exclusivamente sobre la esclerosis múltiple3–7. Tan solo hemos encontrado dos revisiones enfocadas principalmente en la EM. Una primera revisión que analiza posibles futuros biomarcadores de la enfermedad determinados mediante espectroscopia de resonancia magnética8 y otro estudio que describe diversas aplicaciones digitales en la EM y donde se recoge un apartado de la aplicación de algoritmos de ML en esta enfermedad9. Otra categoría que aparece bien diferenciada entre los estudios encontrados en la búsqueda es aquella en la que se investiga sobre la idoneidad de los algoritmos de ML para la realización de análisis automatizados en la exploración RMN. En este sentido, y considerando que la extensión que ocupan las otras categorías que trataremos a continuación ya está muy ajustada con los límites que la «Neurología» sugiere para un artículo de revisión, hemos decidido incluir al final de este párrafo las referencias de estos estudios para que el lector interesado en esta área pueda ampliar la información, pero no entraremos en un análisis en detalle de los mismos10–21. No obstante, podemos indicar que los estudios de ML en estos casos, pretenden cuantificar si los algoritmos pueden realizar una clasificación automática de las lesiones con un nivel adecuado de precisión. Los diversos resultados han mostrado que todavía no existen altos niveles de precisión en su aplicación y la participación del profesional sanitario es necesaria. En particular, algunos estudios apuntan a que el aspecto crítico en la aplicación del ML no depende tanto de los algoritmos empleados como de los parámetros incluidos de la RMN11. Sin duda, futuros estudios serán necesarios para obtener procedimientos válidos en la aplicación clínica del ML en la identificación de lesiones en la RMN.
Los estudios seleccionados pueden ser clasificados en las siguientes cuatro categorías: clasificación de subtipos dentro de la enfermedad; diagnóstico; predicción; y otros enfoques.
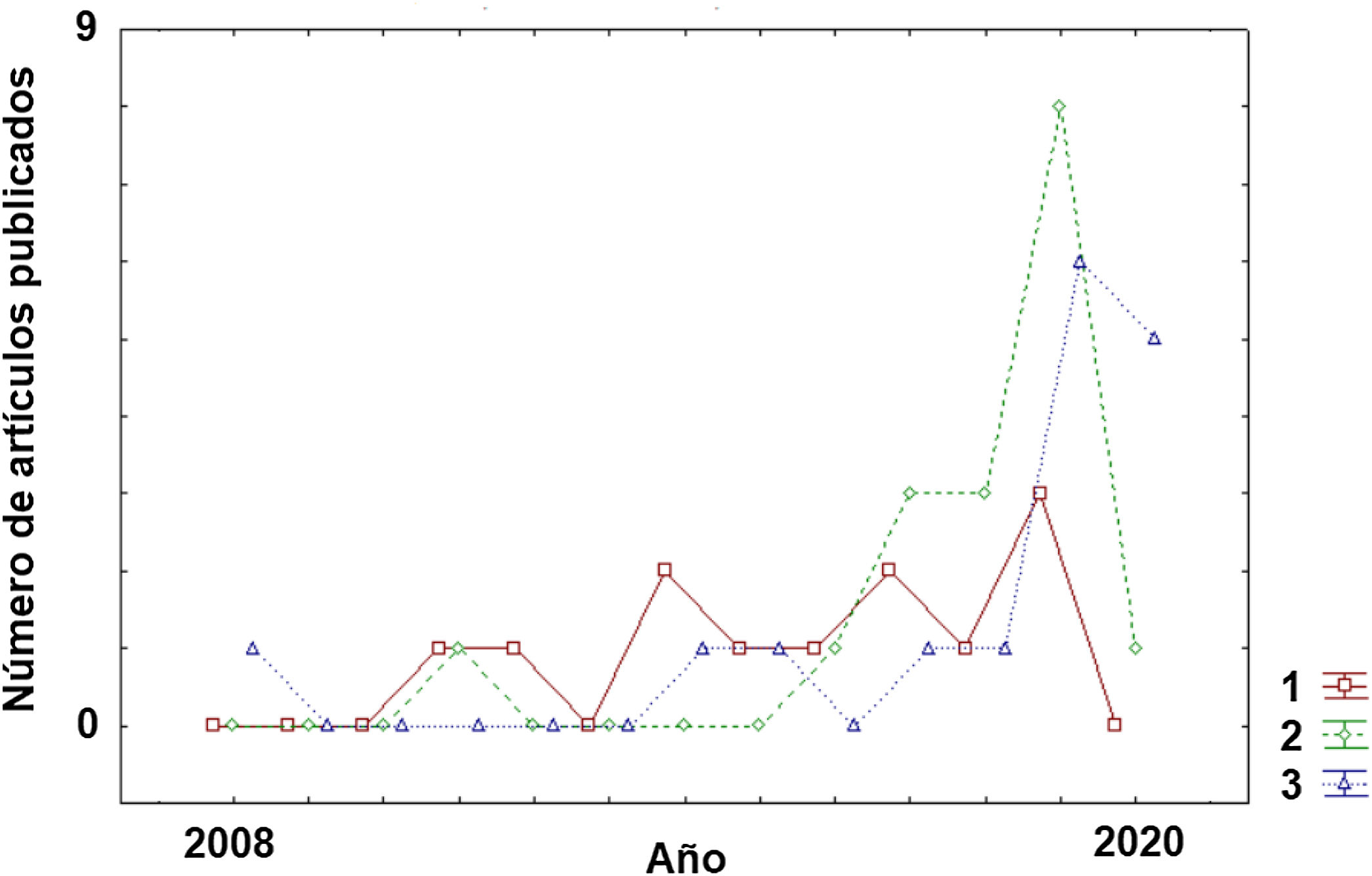
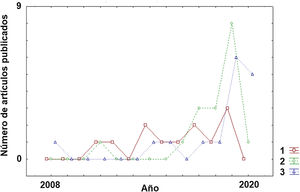
Clasificación de subtipos dentro de la enfermedadA pesar de la corta trayectoria de aplicación de los algoritmos de ML a la EM, es posible observar un claro cambio de tendencia en su breve historia sobre los objetivos perseguidos (fig. 2). En los estudios iniciales, el principal reto era la clasificación de los pacientes dentro de la enfermedad (subtipos). Como puede observarse en la tabla 1, los estudios con este objetivo22–33 se presentan de manera más homogénea a lo largo de la pasada década respecto a otras aplicaciones (diagnóstico o predicción) (tablas 2 y 3) que han aumentado considerablemente su número en los últimos años (2017-2020).
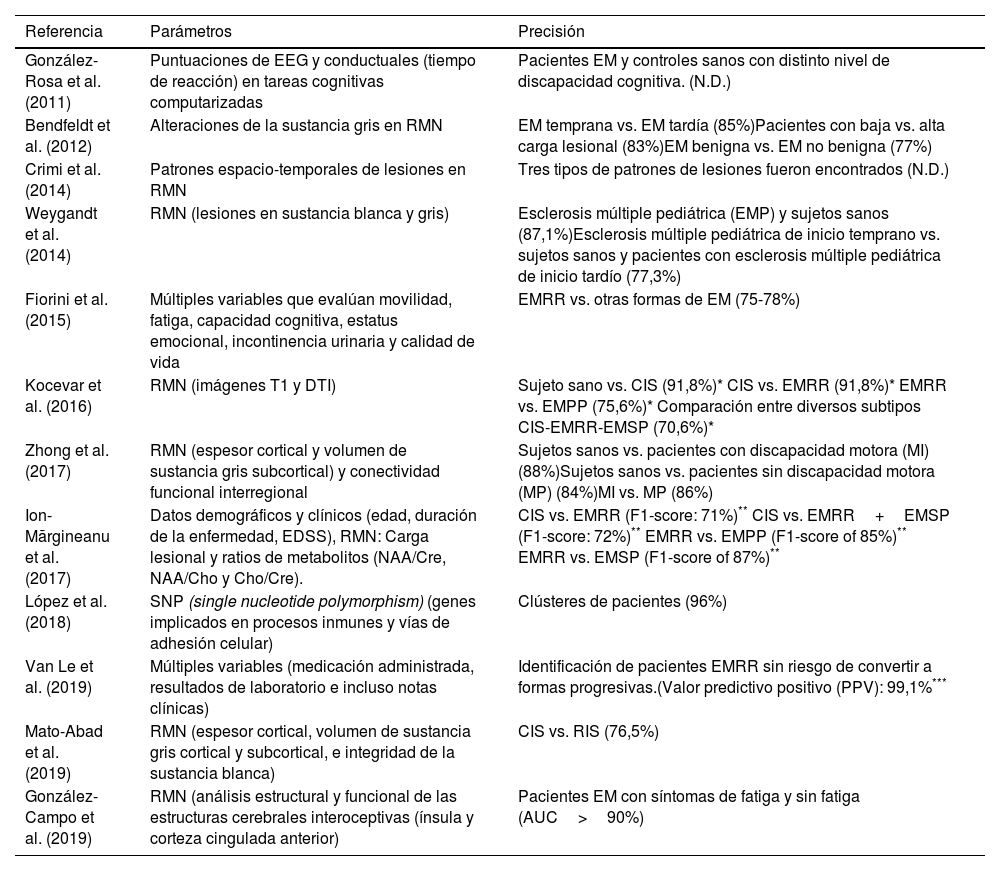
Estudios seleccionados de «machine learning» en la clasificación de subtipos en la esclerosis múltiple. Clasificación de subtipos de la enfermedad
| Referencia | Parámetros | Precisión |
|---|---|---|
| González-Rosa et al. (2011) | Puntuaciones de EEG y conductuales (tiempo de reacción) en tareas cognitivas computarizadas | Pacientes EM y controles sanos con distinto nivel de discapacidad cognitiva. (N.D.) |
| Bendfeldt et al. (2012) | Alteraciones de la sustancia gris en RMN | EM temprana vs. EM tardía (85%)Pacientes con baja vs. alta carga lesional (83%)EM benigna vs. EM no benigna (77%) |
| Crimi et al. (2014) | Patrones espacio-temporales de lesiones en RMN | Tres tipos de patrones de lesiones fueron encontrados (N.D.) |
| Weygandt et al. (2014) | RMN (lesiones en sustancia blanca y gris) | Esclerosis múltiple pediátrica (EMP) y sujetos sanos (87,1%)Esclerosis múltiple pediátrica de inicio temprano vs. sujetos sanos y pacientes con esclerosis múltiple pediátrica de inicio tardío (77,3%) |
| Fiorini et al. (2015) | Múltiples variables que evalúan movilidad, fatiga, capacidad cognitiva, estatus emocional, incontinencia urinaria y calidad de vida | EMRR vs. otras formas de EM (75-78%) |
| Kocevar et al. (2016) | RMN (imágenes T1 y DTI) | Sujeto sano vs. CIS (91,8%)* CIS vs. EMRR (91,8%)* EMRR vs. EMPP (75,6%)* Comparación entre diversos subtipos CIS-EMRR-EMSP (70,6%)* |
| Zhong et al. (2017) | RMN (espesor cortical y volumen de sustancia gris subcortical) y conectividad funcional interregional | Sujetos sanos vs. pacientes con discapacidad motora (MI) (88%)Sujetos sanos vs. pacientes sin discapacidad motora (MP) (84%)MI vs. MP (86%) |
| Ion-Mărgineanu et al. (2017) | Datos demográficos y clínicos (edad, duración de la enfermedad, EDSS), RMN: Carga lesional y ratios de metabolitos (NAA/Cre, NAA/Cho y Cho/Cre). | CIS vs. EMRR (F1-score: 71%)** CIS vs. EMRR+EMSP (F1-score: 72%)** EMRR vs. EMPP (F1-score of 85%)** EMRR vs. EMSP (F1-score of 87%)** |
| López et al. (2018) | SNP (single nucleotide polymorphism) (genes implicados en procesos inmunes y vías de adhesión celular) | Clústeres de pacientes (96%) |
| Van Le et al. (2019) | Múltiples variables (medicación administrada, resultados de laboratorio e incluso notas clínicas) | Identificación de pacientes EMRR sin riesgo de convertir a formas progresivas.(Valor predictivo positivo (PPV): 99,1%*** |
| Mato-Abad et al. (2019) | RMN (espesor cortical, volumen de sustancia gris cortical y subcortical, e integridad de la sustancia blanca) | CIS vs. RIS (76,5%) |
| González-Campo et al. (2019) | RMN (análisis estructural y funcional de las estructuras cerebrales interoceptivas (ínsula y corteza cingulada anterior) | Pacientes EM con síntomas de fatiga y sin fatiga (AUC>90%) |
AUC: área bajo la curva; CIS: síndrome clínico aislado; EEG: electroencefalografía; EM: esclerosis múltiple; EMPP: esclerosis múltiple primaria progresiva; EMRR: esclerosis múltiple remitente-recidivante; EMSP: esclerosis múltiple secundaria progresiva. N.D.: no descrito; RIS: síndrome radiológico aislado; RMN: resonancia magnética nuclear.
Todos estos valores corresponden con lo que los autores denominan «F-measure» y que incluyen la medida de precisión, pero también lo que es definido como «recall» que representa la proporción de verdaderos positivos identificados entre los falsos negativos. Véase la referencia para una definición precisa de la fórmula.
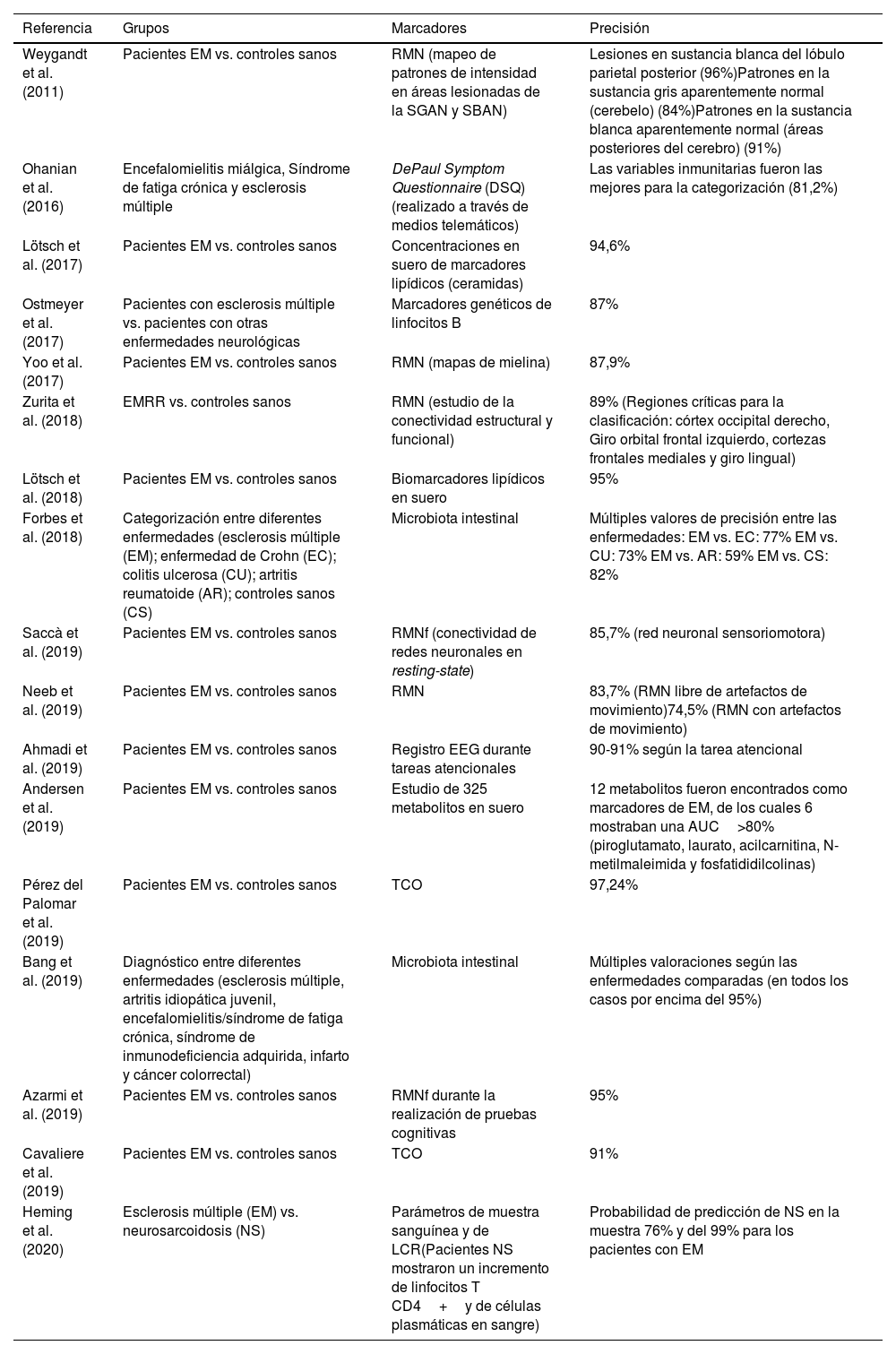
Estudios seleccionados de «machine learning» en el diagnóstico de la esclerosis múltiple. Diagnóstico
| Referencia | Grupos | Marcadores | Precisión |
|---|---|---|---|
| Weygandt et al. (2011) | Pacientes EM vs. controles sanos | RMN (mapeo de patrones de intensidad en áreas lesionadas de la SGAN y SBAN) | Lesiones en sustancia blanca del lóbulo parietal posterior (96%)Patrones en la sustancia gris aparentemente normal (cerebelo) (84%)Patrones en la sustancia blanca aparentemente normal (áreas posteriores del cerebro) (91%) |
| Ohanian et al. (2016) | Encefalomielitis miálgica, Síndrome de fatiga crónica y esclerosis múltiple | DePaul Symptom Questionnaire (DSQ) (realizado a través de medios telemáticos) | Las variables inmunitarias fueron las mejores para la categorización (81,2%) |
| Lötsch et al. (2017) | Pacientes EM vs. controles sanos | Concentraciones en suero de marcadores lipídicos (ceramidas) | 94,6% |
| Ostmeyer et al. (2017) | Pacientes con esclerosis múltiple vs. pacientes con otras enfermedades neurológicas | Marcadores genéticos de linfocitos B | 87% |
| Yoo et al. (2017) | Pacientes EM vs. controles sanos | RMN (mapas de mielina) | 87,9% |
| Zurita et al. (2018) | EMRR vs. controles sanos | RMN (estudio de la conectividad estructural y funcional) | 89% (Regiones críticas para la clasificación: córtex occipital derecho, Giro orbital frontal izquierdo, cortezas frontales mediales y giro lingual) |
| Lötsch et al. (2018) | Pacientes EM vs. controles sanos | Biomarcadores lipídicos en suero | 95% |
| Forbes et al. (2018) | Categorización entre diferentes enfermedades (esclerosis múltiple (EM); enfermedad de Crohn (EC); colitis ulcerosa (CU); artritis reumatoide (AR); controles sanos (CS) | Microbiota intestinal | Múltiples valores de precisión entre las enfermedades: EM vs. EC: 77% EM vs. CU: 73% EM vs. AR: 59% EM vs. CS: 82% |
| Saccà et al. (2019) | Pacientes EM vs. controles sanos | RMNf (conectividad de redes neuronales en resting-state) | 85,7% (red neuronal sensoriomotora) |
| Neeb et al. (2019) | Pacientes EM vs. controles sanos | RMN | 83,7% (RMN libre de artefactos de movimiento)74,5% (RMN con artefactos de movimiento) |
| Ahmadi et al. (2019) | Pacientes EM vs. controles sanos | Registro EEG durante tareas atencionales | 90-91% según la tarea atencional |
| Andersen et al. (2019) | Pacientes EM vs. controles sanos | Estudio de 325 metabolitos en suero | 12 metabolitos fueron encontrados como marcadores de EM, de los cuales 6 mostraban una AUC>80% (piroglutamato, laurato, acilcarnitina, N-metilmaleimida y fosfatididilcolinas) |
| Pérez del Palomar et al. (2019) | Pacientes EM vs. controles sanos | TCO | 97,24% |
| Bang et al. (2019) | Diagnóstico entre diferentes enfermedades (esclerosis múltiple, artritis idiopática juvenil, encefalomielitis/síndrome de fatiga crónica, síndrome de inmunodeficiencia adquirida, infarto y cáncer colorrectal) | Microbiota intestinal | Múltiples valoraciones según las enfermedades comparadas (en todos los casos por encima del 95%) |
| Azarmi et al. (2019) | Pacientes EM vs. controles sanos | RMNf durante la realización de pruebas cognitivas | 95% |
| Cavaliere et al. (2019) | Pacientes EM vs. controles sanos | TCO | 91% |
| Heming et al. (2020) | Esclerosis múltiple (EM) vs. neurosarcoidosis (NS) | Parámetros de muestra sanguínea y de LCR(Pacientes NS mostraron un incremento de linfocitos T CD4+y de células plasmáticas en sangre) | Probabilidad de predicción de NS en la muestra 76% y del 99% para los pacientes con EM |
AUC: área bajo la curva; EEG: electroencefalografía; EM: esclerosis múltiple; LCR: líquido cefalorraquídeo; RMN: resonancia magnética nuclear; RMNf: resonancia magnética nuclear funcional: SBAN: sustancia blanca aparentemente normal; SGAN: sustancia gris aparentemente normal; TCO: tomografía de coherencia óptica.
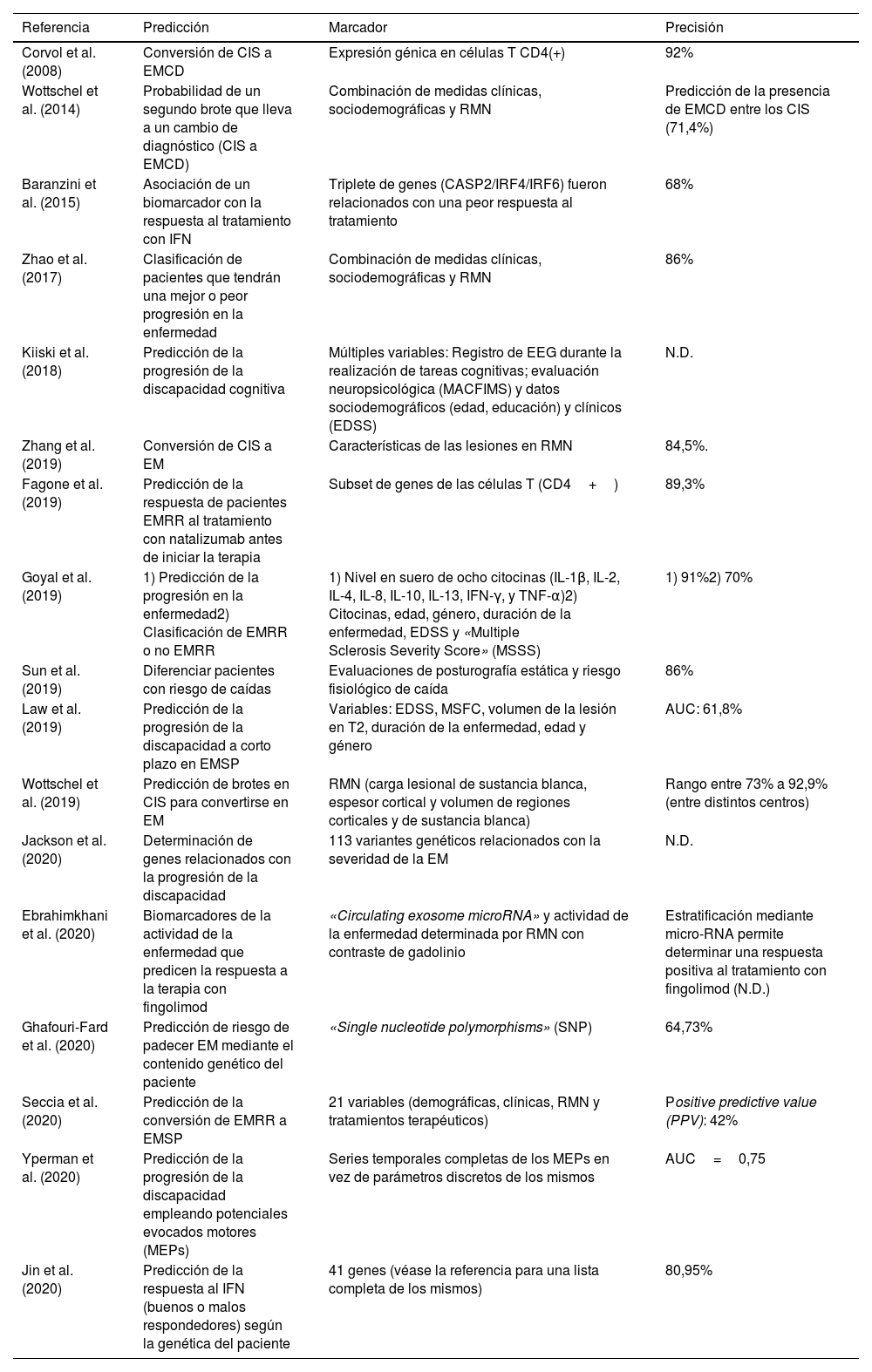
Estudios seleccionados de «machine learning» en el apartado de predicción. Predicción
| Referencia | Predicción | Marcador | Precisión |
|---|---|---|---|
| Corvol et al. (2008) | Conversión de CIS a EMCD | Expresión génica en células T CD4(+) | 92% |
| Wottschel et al. (2014) | Probabilidad de un segundo brote que lleva a un cambio de diagnóstico (CIS a EMCD) | Combinación de medidas clínicas, sociodemográficas y RMN | Predicción de la presencia de EMCD entre los CIS (71,4%) |
| Baranzini et al. (2015) | Asociación de un biomarcador con la respuesta al tratamiento con IFN | Triplete de genes (CASP2/IRF4/IRF6) fueron relacionados con una peor respuesta al tratamiento | 68% |
| Zhao et al. (2017) | Clasificación de pacientes que tendrán una mejor o peor progresión en la enfermedad | Combinación de medidas clínicas, sociodemográficas y RMN | 86% |
| Kiiski et al. (2018) | Predicción de la progresión de la discapacidad cognitiva | Múltiples variables: Registro de EEG durante la realización de tareas cognitivas; evaluación neuropsicológica (MACFIMS) y datos sociodemográficos (edad, educación) y clínicos (EDSS) | N.D. |
| Zhang et al. (2019) | Conversión de CIS a EM | Características de las lesiones en RMN | 84,5%. |
| Fagone et al. (2019) | Predicción de la respuesta de pacientes EMRR al tratamiento con natalizumab antes de iniciar la terapia | Subset de genes de las células T (CD4+) | 89,3% |
| Goyal et al. (2019) | 1) Predicción de la progresión en la enfermedad2) Clasificación de EMRR o no EMRR | 1) Nivel en suero de ocho citocinas (IL-1β, IL-2, IL-4, IL-8, IL-10, IL-13, IFN-γ, y TNF-α)2) Citocinas, edad, género, duración de la enfermedad, EDSS y «Multiple Sclerosis Severity Score» (MSSS) | 1) 91%2) 70% |
| Sun et al. (2019) | Diferenciar pacientes con riesgo de caídas | Evaluaciones de posturografía estática y riesgo fisiológico de caída | 86% |
| Law et al. (2019) | Predicción de la progresión de la discapacidad a corto plazo en EMSP | Variables: EDSS, MSFC, volumen de la lesión en T2, duración de la enfermedad, edad y género | AUC: 61,8% |
| Wottschel et al. (2019) | Predicción de brotes en CIS para convertirse en EM | RMN (carga lesional de sustancia blanca, espesor cortical y volumen de regiones corticales y de sustancia blanca) | Rango entre 73% a 92,9% (entre distintos centros) |
| Jackson et al. (2020) | Determinación de genes relacionados con la progresión de la discapacidad | 113 variantes genéticos relacionados con la severidad de la EM | N.D. |
| Ebrahimkhani et al. (2020) | Biomarcadores de la actividad de la enfermedad que predicen la respuesta a la terapia con fingolimod | «Circulating exosome microRNA» y actividad de la enfermedad determinada por RMN con contraste de gadolinio | Estratificación mediante micro-RNA permite determinar una respuesta positiva al tratamiento con fingolimod (N.D.) |
| Ghafouri-Fard et al. (2020) | Predicción de riesgo de padecer EM mediante el contenido genético del paciente | «Single nucleotide polymorphisms» (SNP) | 64,73% |
| Seccia et al. (2020) | Predicción de la conversión de EMRR a EMSP | 21 variables (demográficas, clínicas, RMN y tratamientos terapéuticos) | Positive predictive value (PPV): 42% |
| Yperman et al. (2020) | Predicción de la progresión de la discapacidad empleando potenciales evocados motores (MEPs) | Series temporales completas de los MEPs en vez de parámetros discretos de los mismos | AUC=0,75 |
| Jin et al. (2020) | Predicción de la respuesta al IFN (buenos o malos respondedores) según la genética del paciente | 41 genes (véase la referencia para una lista completa de los mismos) | 80,95% |
AUC: área bajo la curva; CIS: síndrome clínico aislado; EDSS: escala expandida de discapacidad (expanded disability status scale); EM: esclerosis múltiple; EMCD: esclerosis múltiple clínicamente definida; EMRR: esclerosis múltiple remitente-recidivante; EMSP: esclerosis múltiple secundaria progresiva; IFN: interferón; MACFIMS: Minimal Assessment of Cognitive Function in Multiple Sclerosis battery; N.D: no descrito; RMN: resonancia magnética nuclear.
Los diversos estudios han permitido clasificar a pacientes en diversas categorías: EMRR; esclerosis múltiple benigna; síndrome clínico aislado (CIS); síndrome radiológico aislado (RIS); EM pediátrica de inicio temprano o tardío; o por características como la carga lesional o el comienzo en la enfermedad; o incluso atendiendo al grado de discapacidad cognitiva. En cuanto a las variables empleadas para la clasificación también encontramos una gran variedad de opciones. Desde diversos parámetros de la RMN relacionados con la sustancia gris y blanca, pasando por medidas clínicas (EDSS o duración de la enfermedad), hasta medidas no necesariamente cuantitativas como por ejemplo notas clínicas del paciente.
El rango de precisión alcanzado entre los diferentes estudios está comprendido desde el 70,6 hasta el 96%. Estos valores varían mucho entre los diferentes estudios sugiriendo que los procedimientos algorítmicos aplicados, las variables empleadas para la clasificación y los subtipos de la enfermedad que se pretenden identificar son determinantes para el nivel de precisión. Se puede concluir que los algoritmos pueden alcanzar niveles aceptables en la clasificación de los subtipos en pacientes y pueden permitir mejorar la estratificación de los mismos para posteriores estudios de respuesta a intervenciones terapéuticas o de progresión de la enfermedad.
DiagnósticoEn el caso de la aplicación de los algoritmos de ML con el objetivo principal del diagnóstico, hallamos un estudio del 2011 y es necesario esperar hasta el 2016 para encontrar nuevos estudios. Pero sin duda, algo que llama la atención es que en el año 2019 se acumularon una gran cantidad de trabajos en esta categoría. Curiosamente, y de modo distinto a lo que ocurrirá en el apartado de «Predicción», en el año 2020 solo se ha publicado un estudio en los primeros 4 meses (tabla 2)34–50. Otro aspecto destacable en esta categoría es que los trabajos se han orientado principalmente para el diagnóstico de la EM frente a controles sanos (13 estudios). En un número menor (5 estudios), la aplicación del algoritmo persiguió la diferenciación entre diferentes entidades patológicas.
Las principales variables que se han empleado para mejorar nuestra capacidad de diagnóstico proceden de la técnica de RMN en diferentes parámetros de la misma (mapas lesionales, conectividad funcional, etc.). Sin embargo, existen otros marcadores que ofrecen interesantes niveles de precisión como los marcadores serológicos o genéticos, o pruebas como la tomografía de coherencia óptica (TCO) o la electroencefalografía (EEG).
El rango de precisión se presenta desde niveles muy buenos (98,2%) hasta niveles moderados (59%). Curiosamente, ambos resultados se obtuvieron empleando las características de la microbiota intestinal entre diferentes entidades patológicas. En el primer caso, se trataba de distinguir la EM entre otras enfermedades como artritis idiopática o síndrome de fatiga crónica47. En el segundo caso, ese bajo nivel de precisión se produjo a la hora de categorizar entre la EM y la artritis reumatoide40.
PredicciónEs conveniente destacar que en este apartado se han incluido trabajos que en el fondo realizan en algunos casos un proceso de clasificación en subtipos dentro de la enfermedad. Es el caso de estudios en los que se trata de discriminar a buenos de malos respondedores en tratamientos farmacológicos. La decisión de ubicar estos estudios en esta categoría radica en que la finalidad última de la aplicación del algoritmo de ML no es solo la clasificación sino la predicción.
Si se revisa la tabla 351–67 se puede observar que se registró un primer estudio en el año 2008 y tuvieron que pasar varios años (hasta el 2014) para encontrar nuevos estudios con el objetivo de la predicción. Sin embargo, este objetivo es el que hoy por hoy más crecimiento ha experimentado en los dos últimos años con 6 artículos tan solo en los primeros 4 meses del año 2020. Las diversas líneas de actuación en este campo comprenden dos objetivos principales. Por un lado, predecir la posible evolución del paciente (p. ej. riesgo de convertirse en otra forma más evolucionada de la enfermedad), y por otro, anticipar la posible respuesta del paciente a una terapia farmacológica para ayudar al profesional en la elección del tratamiento más adecuado. Es sin duda, una aproximación para un tipo de medicina más personalizada que contempla la posible heterogeneidad de los condicionantes específicos de cada paciente. Respecto a cuánto de precisos son los algoritmos de ML en estos dos casos, en la predicción en la progresión de la enfermedad, los porcentajes de precisión estuvieron en un rango entre 61,8 y 92,9%. En el caso de la precisión en clasificar a los pacientes en función de la respuesta que mostrarán a terapias farmacológicas el rango de precisión es similar con una horquilla entre el 68 y el 89,3%.
Otros enfoquesEn este último apartado hemos querido incluir otras líneas de actuación que se están produciendo en la aplicación del ML en la EM. Una de las líneas más activas en este sentido es la que trata de desarrollar nuevos indicadores (scores) que integran diferentes variables y consigan mejorar la evaluación de los pacientes. Un primer ejemplo de esta aplicación fue desarrollado por el equipo de Kosa et al.68(2016). Este grupo planteó la necesidad de obtener un indicador más sensible para la evaluación de cambios en la progresión de la enfermedad y así poder anticipar lo antes posible la respuesta a tratamientos farmacológicos en pacientes con la forma progresiva. El nuevo evaluador denominado «CombiWISE» integra valores de varias escalas: «Expanded disability status scale» (EDSS), «Scripps neurological rating scale», «25 foot walk test» y el «9 hole peg test». Los resultados mostraron en el seguimiento de 98 pacientes con la forma progresiva que el conjunto de estos indicadores era mejor que las medidas de atrofia en RMN o que la escala EDSS por sí solas. De hecho un punto en la EDSS correspondía con 7,5 puntos en el nuevo indicador aumentando por tanto la resolución en la evaluación de los pacientes. En otro estudio69 se desarrolló un indicador denominado «Brain Age» para poder determinar si vale como marcador de curso clínico y severidad de la enfermedad. Los resultados mostraron que este indicador mostraba, en promedio, un valor superior en pacientes con EM respecto de sujetos sanos y que correlacionaba con la atrofia cerebral y la carga lesional en sustancia blanca. Desafortunadamente no se encontraron buenas correlaciones entre este indicador y diversas variables clínicas de los pacientes, lo cual hace dudar de su potencial uso clínico. En otros casos, los algoritmos de ML se han centrado en optimizar las capacidades de evaluación ya existentes. Una línea de actuación muy activa se ha concentrado en la determinación de la discapacidad motora y su relación con diversos parámetros evaluados del paciente. En estos estudios principalmente se suele analizar la marcha del paciente mediante sensores colocados en la piel70–73. Otra de las líneas que acumula más estudios es la que analiza las posibles causas de la enfermedad. En este sentido, se han desarrollado estudios con dos objetivos: describir posibles factores de riesgo74,75 e identificar potenciales marcadores de la EM para desarrollar posibles dianas terapéuticas útiles en futuros tratamientos76–79. Por último, quisiéramos destacar una línea de actuación que trata de valorar con diversos enfoques si la aplicación del ML resulta beneficiosa para el profesional sanitario en términos eminentemente prácticos. En este sentido, se ha tratado de valorar si la colaboración entre los profesionales sanitarios y la aplicación de los algoritmos de ML dan mejores resultados en la evaluación de los pacientes que cuando se realiza esta por separado. Para ello se realizó un estudio en el que se trataba de predecir el curso de la enfermedad en función de los historiales de los pacientes con EM80. El estudio concluyó que la cooperación entre profesionales humanos y el ML rendía mejores valores que cuando los pronósticos se realizaban por separado.
Además, los algoritmos de ML pueden ayudar al profesional sanitario en reducir la contribución de factores que pueden estar confundiendo para realizar una correcta clasificación. Por ejemplo, un estudio ha mostrado cómo puede reducirse, sin necesidad de la intervención por parte del profesional sanitario, la contribución de factores como la edad que pueden deshacer la homogeneidad de los grupos (clústeres) descritos por el algoritmo de ML81. En otros casos, la aplicación de ML proporciona una mejora en la realización de la prueba reduciendo el tiempo de operación como en el caso de la TCO, donde se consigue una exploración de las superficies de la retina y del edema macular con una precisión mejor que la habitualmente lograda con métodos más costosos en términos de tiempo, y realizada en tan solo 10 segundos82. Otra aplicación en la línea de apoyar al profesional sanitario, en este caso al neuropsicólogo, es la de automatizar parcialmente la evaluación cognitiva en los pacientes83. Durante la progresión de la enfermedad, el paciente puede ir experimentando deterioro cognitivo y uno de los test aplicados para su evaluación es el «Brief Visuospatial Memory Test-Revised» (BVMT-R), en el que el paciente tiene que reconocer figuras geométricas y dibujarlas. Tras la digitalización del test y un entrenamiento del algoritmo, los resultados mostraron una precisión del 80% en la evaluación del test considerando la evaluación del profesional como gold-standard. Los autores concluyeron que la primera evaluación sigue siendo una tarea para el profesional sanitario pero futuras mejoras en el aprendizaje de los algoritmos puedan permitir que las reevaluaciones en estudios longitudinales sean desarrolladas de manera fiable por los algoritmos de ML.
DiscusiónLas aplicaciones más intuitivas de los algoritmos de ML en la EM son sin duda el diagnóstico (o clasificación si hablamos de subtipos dentro de la enfermedad) y la predicción de la evolución o respuesta a las posibles estrategias terapéuticas. Sin embargo, las aplicaciones posibles de los algoritmos de ML son enormes y recientes estudios muestran nuevas vías de investigación que se abren cara al futuro. En el caso particular de la EM, el análisis de todas las ramas «ómicas» con ingentes cantidades de datos pueden ser abordadas con gran potencia de análisis, algo que hasta hace unos años resultaba muy complicado realizar con las técnicas convencionales de análisis estadístico. Particularmente interesante es la aplicación de estos algoritmos en el estudio de posibles biomarcadores identificativos de la enfermedad que nos permitan comprender la etiología precisa de la misma y trazar posibles vías terapéuticas en el futuro.
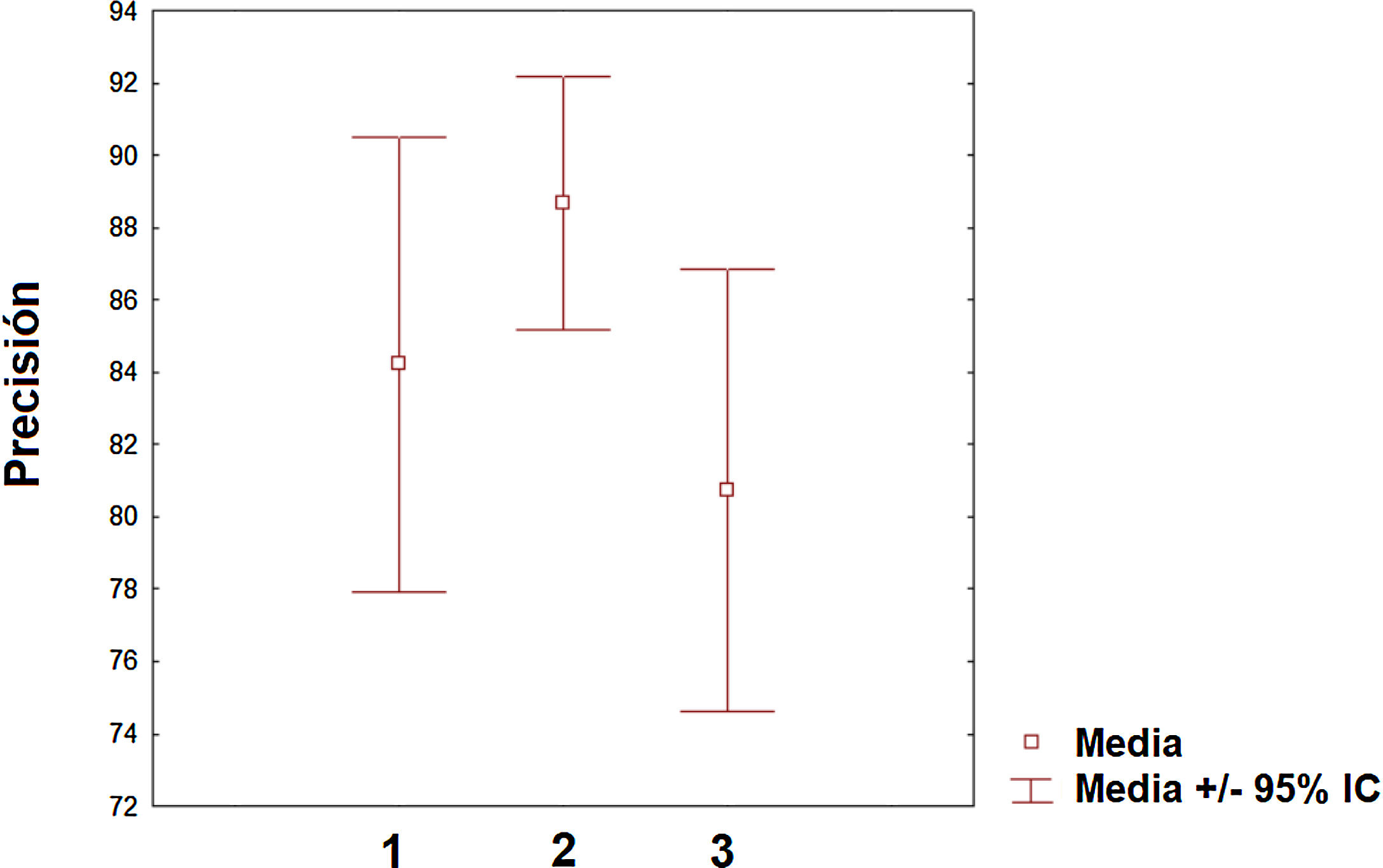
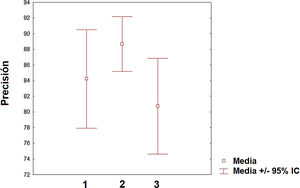
Pero sin duda, el campo con una mayor proyección para su aplicación clínica es el de la predicción de la respuesta ante los diversos tratamientos farmacológicos que existen y se generarán en los próximos años. De hecho, la predicción es la aplicación que ha aumentado más en el número de publicaciones en los dos últimos años. Sin embargo, todavía existe un desequilibrio de los trabajos aplicados para predecir la progresión de la enfermedad (línea mayoritaria) de aquellos que se orientan hacia la predicción de la respuesta terapéutica (línea minoritaria). Sin duda, en los próximos años, esta última línea será líder en la aplicación del ML en la EM así como en otras patologías neurológicas. Respecto de la precisión obtenida entre las diferentes categorías (clasificación, diagnóstico y predicción), se puede decir que el valor medio es similar aunque el intervalo de confianza sea algo distinto entre ellas (fig. 3). Es necesario indicar que la comparación entre las diversas categorías debe considerarse con precaución dado que las medidas de precisión, el número de variables incluidas y los procedimientos de ML empleados difieren de unos estudios a otros. Sin embargo, es posible concluir que existen niveles relevantes de precisión en los primeros estudios realizados en este campo. Dado el carácter de entrenamiento que requieren estos algoritmos de ML, es necesario que las instituciones sanitarias hagan un esfuerzo por cooperar en repositorios de datos para poder incrementar la capacidad de los diversos algoritmos en la descripción de los posibles patrones dentro de la enfermedad. En el caso del contenido genético, este enfoque es particularmente interesante dado que esto permitirá una caracterización más precisa del paciente y personalizar aún más si cabe en el futuro los tratamientos. Sin duda, nos encontramos en el camino hacia una medicina personalizada sugerida desde múltiples estudios84–86. Es esperable que, en los próximos años, aquellos sistemas sanitarios que hagan un esfuerzo en la implementación de este tipo de herramientas de la inteligencia artificial liderarán parte de los avances más relevantes en la práctica clínica. Entre las limitaciones de este estudio debemos destacar algunas consideraciones. El motor de búsqueda PubMed puede cometer errores en su rastreo de los artículos publicados hasta la fecha. Sin embargo, el análisis de los apartados de bibliografía de los diferentes artículos analizados minimiza la posibilidad de que un número elevado de artículos relacionados con el ML y la EM no hayan sido incluidos en la revisión. Otro aspecto que puede tenerse en cuenta es que existen otros conceptos como «deep learning» que pueden ser de interés en su aplicación en la EM y están relacionados con el ML. En futuras revisiones sobre el ML y su aplicación sobre la EM u otras patologías, deberá contemplarse la posibilidad de ampliar las palabras clave a otras posibles acepciones como «deep learning» o «network representation learning».
ConclusiónEl presente trabajo de revisión ha permitido observar que la aplicación de los algoritmos de ML en la EM está siendo un campo muy activo mostrando un crecimiento exponencial en los dos últimos años. Las principales áreas de aplicación son la clasificación de diferentes subtipos dentro de la enfermedad, el diagnóstico frente a sujetos sanos y otras enfermedades y la predicción de la evolución del paciente o la respuesta a un tratamiento terapéutico. Sin embargo, es posible encontrar también estudios que tratan de optimizar técnicas o buscar nuevos indicadores para mejorar la evaluación clínica en términos de tiempo, coste y bienestar para el paciente. En general, lo observado en los diferentes estudios es que la aplicación de este tipo de algoritmos ML obtiene valores de precisión en algunos casos excelentes (>90%), y puede ser un importante apoyo para el profesional sanitario en el diagnóstico y pronóstico dentro de la EM.
FinanciaciónEste trabajo ha sido subvencionado por el proyecto PSI2016-78133-P del Ministerio de Ciencia, Innovación y Universidades (España).
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.